鸡白痢沙门菌spiC基因克隆与生物信息学分析
2016-03-30安树敏郭荣显潘志明耿士忠焦新安
安树敏,郭荣显,薛 颖,潘志明,耿士忠,焦新安
(扬州大学江苏省人兽共患病学重点实验室/江苏省动物重要疫病与人兽共患病防控协同创新中心,江苏扬州 225009)
鸡白痢沙门菌spiC基因克隆与生物信息学分析
安树敏△,郭荣显△,薛颖,潘志明,耿士忠*,焦新安*
(扬州大学江苏省人兽共患病学重点实验室/江苏省动物重要疫病与人兽共患病防控协同创新中心,江苏扬州 225009)
摘要:为了研究鸡白痢沙门菌SpiC蛋白的功能,以鸡白痢沙门菌S06004基因组DNA为模板PCR扩增spiC基因,进行克隆测序和生物信息学分析,并调查该基因在255株不同年代鸡白痢沙门菌分离株中的分布与进化情况。结果显示,鸡白痢沙门菌spiC基因全长384 bp,编码127个氨基酸,在不同年代分离株中稳定存在。推导SpiC蛋白无跨膜区、信号肽和卷曲螺旋结构,有1处酪蛋白激酶Ⅱ磷酸化位点,2处N-豆蔻酰化位点,预测线性抗原表位主要分布在15-20、29-35、52-53、70-72、83-85、89-94 aa。利用SWISS MODEL在线程序建模,得到SpiC三维结构模型。同源性及进化分析显示spiC基因仅存在于沙门菌中且比较保守,鸡白痢沙门菌SpiC蛋白与沙门菌肠道亚种亲缘关系最近。
关键词:鸡白痢沙门菌;SpiC蛋白;生物信息学分析
沙门菌是一类兼性胞内寄生病原菌,能引起人和动物多种不同症状表现的沙门菌病,在医学、兽医学和公共卫生学领域备受重视[1-2]。鸡白痢沙门菌作为一种宿主专嗜性血清型,主要侵害20日龄以内的雏鸡,表现出高发病率和病死率,对我国家禽业的发展危害极大。沙门菌的致病性涉及多种毒力因子,其中毒力岛2 (Salmonellapathogenicity island 2, SPI-2)编码的Ⅲ型分泌系统 (type three secretion system, TTSS)能够将细菌的效应蛋白分泌或转运至宿主细胞内,导致细菌与宿主的一系列相互作用,与沙门菌在胞内存活和发挥毒力密切相关[3-4]。
SpiC蛋白由沙门菌SPI-2编码后通过Spi/Ssa TTSS 转运至宿主细胞内,与宿主膜泡运输相关蛋白TassC 和Hook3 相互作用,在避免吞噬小泡 (Salmonellacontaining vacuoles, SCV)与溶酶体融合过程中起重要作用[5]。体外试验发现SpiC对于SseB、SseC、SseD的分泌是必须的[6]。一些研究表明,SpiC可以通过影响沙门菌鞭毛蛋白FliC、FlhD的表达,参与调控SPI-2依赖的巨噬细胞MAPK (mitogen-activated protein kinase,MAPK)信号通路的活化[7-8]。此外,沙门菌spiC基因缺失株在宿主细胞和组织中的定殖和存活能力明显减弱,致病力显著降低[9-11]。尽管如此,SpiC蛋白作为一个SPI-2编码的效应蛋白颇受争议。本研究对编码鸡白痢沙门菌SpiC蛋白的基因进行克隆测序,并对其序列特点、理化性质及结构特征等进行了分析,以期为下一步的功能研究、减毒活疫苗候选株的基因发掘奠定基础。
1材料与方法
1.1材料
1.1.1质粒和菌株鸡白痢沙门菌S06004株、1962年-2012年间的分离株由江苏省人兽共患病学重点实验室保存;pMD20-T克隆载体、大肠埃希菌Dpα为宝生物工程(大连)有限公司产品。
1.1.2主要试剂TaqDNA聚合酶、DNA Marker、限制性内切酶、细菌基因组DNA提取试剂盒、DNA胶回收试剂盒等为宝生物工程(大连)有限公司产品;其余试剂均为进口或国产分析纯;PCR引物合成和序列测定由南京金斯瑞生物有限公司完成。
1.2方法
1.2.1spiC基因的克隆根据GenBank中鸡白痢沙门菌基因组序列(gbCP006575.1),设计1对特异性引物。上游引物spiC-F:5′-TACTCGAGTTATACCCCACCCGAATAAAGTTTATG-3′,下游引物spiC-R:5′- TAGGATCCATGCTGGCAGTTTTAAAAGGCATT-3′。以提取的鸡白痢沙门菌S06004基因组DNA为模板扩增spiC基因,反应条件为:94 ℃ 5 min;94 ℃ 45 s,58℃ 45 s,72 ℃ 60 s,共30个循环;72 ℃ 10 min。PCR扩增产物经10 g/L琼脂糖凝胶电泳初步验证后,使用DNA胶回收试剂盒回收目的片段,克隆至pMD20-T,转化Dpα感受态细胞,通过氨苄青霉素抗性和蓝白斑筛选等获得阳性克隆,送南京金斯瑞生物有限公司测序。
1.2.2不同年份鸡白痢沙门菌spiC基因的扩增分析对实验室保存的255株鸡白痢沙门菌进行spiC基因的PCR扩增,并按不同年代随机选择10株进行测序分析。
1.2.3SpiC生物信息学分析
1.2.3.1基本理化性质分析应用ProtParam (http://web.expasy.org/protparam/)计算蛋白质的氨基酸组成、理论分子质量等电点等。采用ProtScale (http://web.expasy.org/protscale/),选择Kyte & Doolittle参数,分析蛋白质的疏水性与亲水性。
1.2.3.2结构域分析应用SMART (http://smart.embl-heidelberg.de/)进行结构域预测。采用TMHMM Server v. 2.0 (http://www.cbs.dtu.dk/services/TMHMM/)预测蛋白质的跨膜结构域。通过Motif Scan (http://hits.isb-sib.ch/cgi-bin/PFSCAN)预测蛋白质的磷酸化、糖基化等修饰位点和基序。通过在线分析软件SignalP (http://www.cbs.dtu.dk/services/SignalP/)预测蛋白质的信号肽序列。
1.2.3.3亚细胞定位预测亚细胞定位预测使用PSORT Prediction (http://psort.hgc.jp/form.html)在线软件。
1.2.3.4 B细胞表位分析应用IEDB Analysis Resource (http://tools.immuneepitope.org/main/html/bcell_tools.html/)预测蛋白质可能的线性B细胞抗原表位。
1.2.3.53D结构分析利用SWISS MODEL (http://swissmodel.expasy.org/)程序进行建模,分析蛋白质的3D结构。
1.2.3.6同源性分析利用在线Protein BLAST (http://blast.ncbi.nlm.nihgov/Blast.cgi?PROGRA
M=blastp&PAGE-TYPE=BlastSearch&LINK-LOC=blasthome)软件进行氨基酸序列同源性比对和相似性分析。
1.2.3.7系统进化分析运用MEGA 5.0 软件邻位相连法对选择的序列构建系统进化树。
2结果
2.1spiC基因的克隆
PCR扩增得到大小约380 bp的特异性条带(图1),阳性克隆测序表明,鸡白痢沙门菌spiC基因含有一个384 bp 的开放阅读框 ,编码127个氨基酸(图2)。已将该基因提交至GenBank,登录号为JN673268.1。
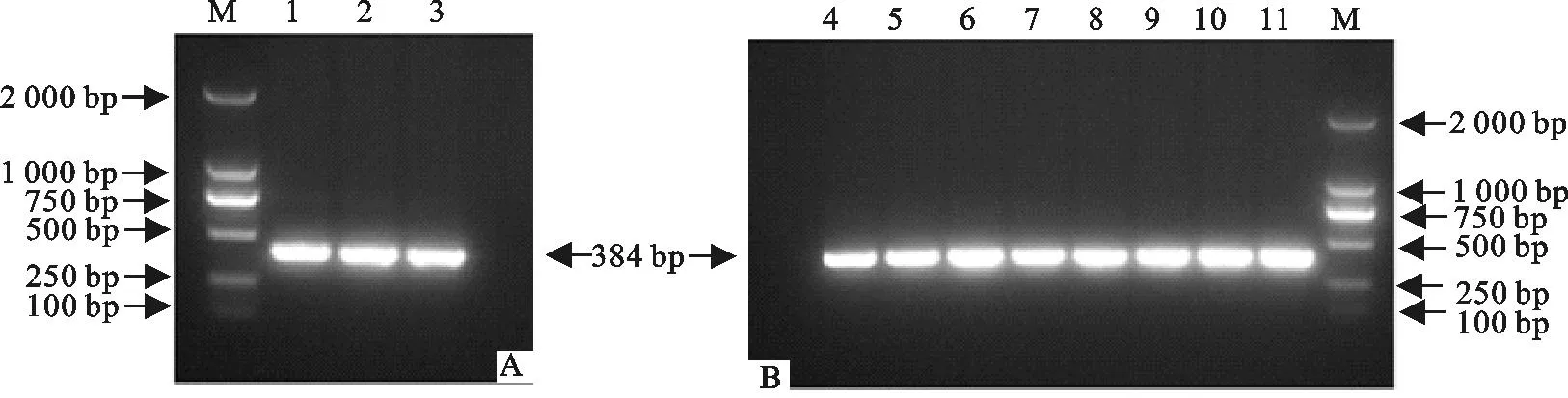
M. DNA标准DL 2 000; 1~11. spiC PCR产物
M. DNAMarker DL 2 000; 1-11. spiC PCR products
图1spiC基因的克隆(A)与菌落PCR扩增(B)
Fig.1Cloning and PCR amplification of spiC gene

图2 spiC基因核苷酸序列及其编码的氨基酸序列
2.2 不同年份spiC基因的检测结果
255株鸡白痢沙门菌均能扩增出约380 bp的spiC基因片段(部分菌株扩增结果见图1B),所选10株不同年代的分离株测序结果显示,与S06004株基因序列完全一致,同源性达100%(表1)。
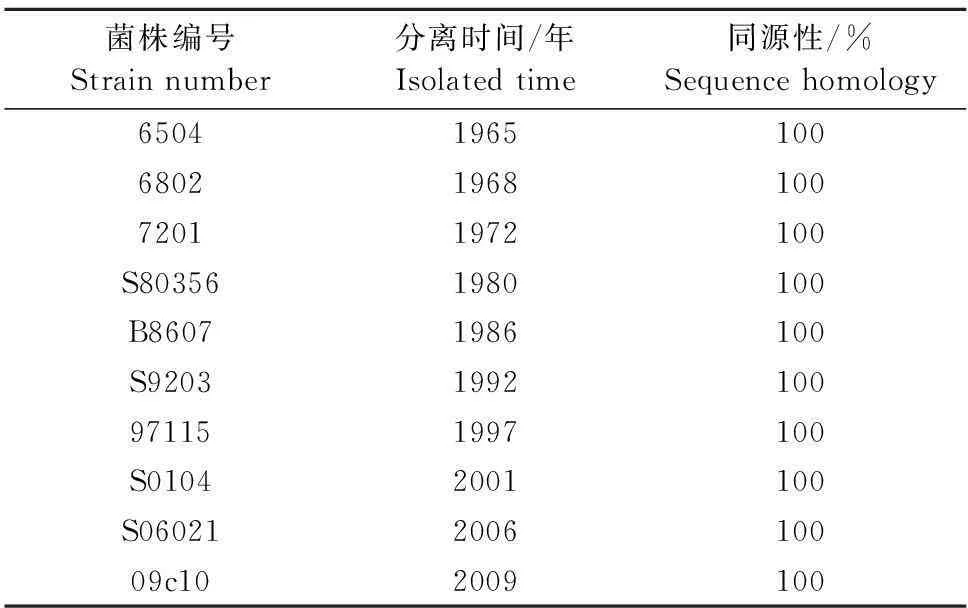
表1 不同年份分离株spiC基因的检测结果
2.3鸡白痢沙门菌SpiC蛋白的生物信息学分析
2.3.1理化性质通过ExPASy在线软件分析,SpiC蛋白由127个氨基酸残基组成,理论分子质量为13.95 ku,理论等电点为5.79,原子总数为1 980,分子式为C633H997N163O183S4。该蛋白不含Pyl和Sec,在其余20种氨基酸中,Leu (L)含量最丰富,约占14.2%,带负电荷的氨基酸残基 (Asp + Glu) 占10.2%,带正电荷残基 (Arg + Lys) 占7.1%。不稳定系数37.94,小于阈值40,表明该蛋白比较稳定。脂肪系数和疏水性平均值表明其可能是一种亲水蛋白(图3)。
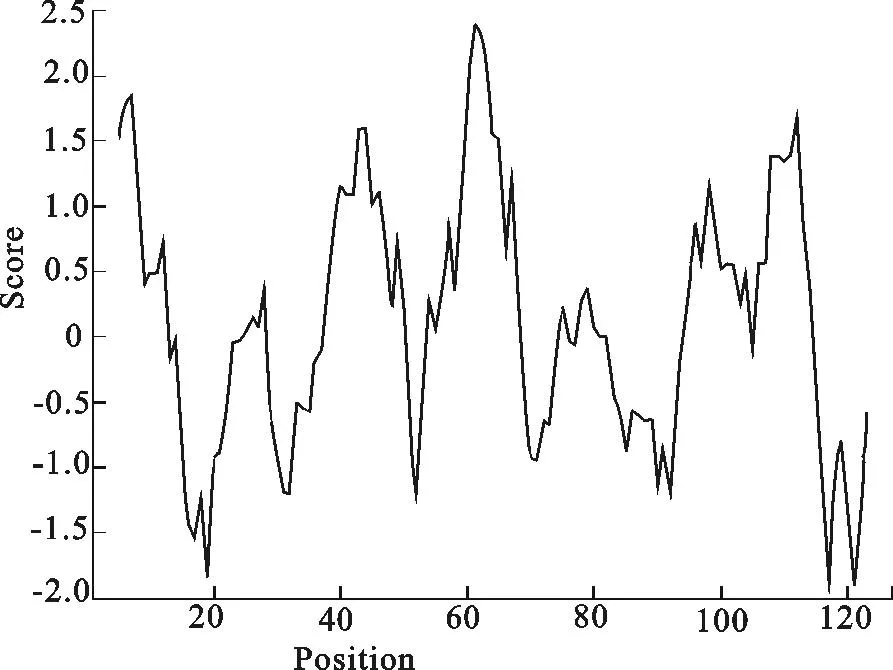
图3 亲水性分析
2.3.2结构分析应用SignalP分析显示,SpiC蛋白无明显的信号肽切割位点,即不存在信号肽。TMHMM Server v. 2.0 预测该蛋白无跨膜区。Motif Scan 预测结果发现,SpiC蛋白可能存在以下基序:1处酪蛋白激酶Ⅱ磷酸化位点 (93SSSE);2处N-豆蔻酰化位点 (18GNSRSW、82GGWLNG)。该蛋白含有一个低复杂度区域 (low complexity regions, LCRs),分布在92-103 aa (SSSSELIAIEIE)。蛋白质亚细胞定位分析结果表明,SpiC极有可能位于细胞质中。IEDB预测SpiC线性抗原表位主要分布在15-20、29-35、52-53、70-72、83-85、89-94 aa(图4)。利用SWISS MODEL在线程序建模,得到SpiC三维结构模型(图5)。
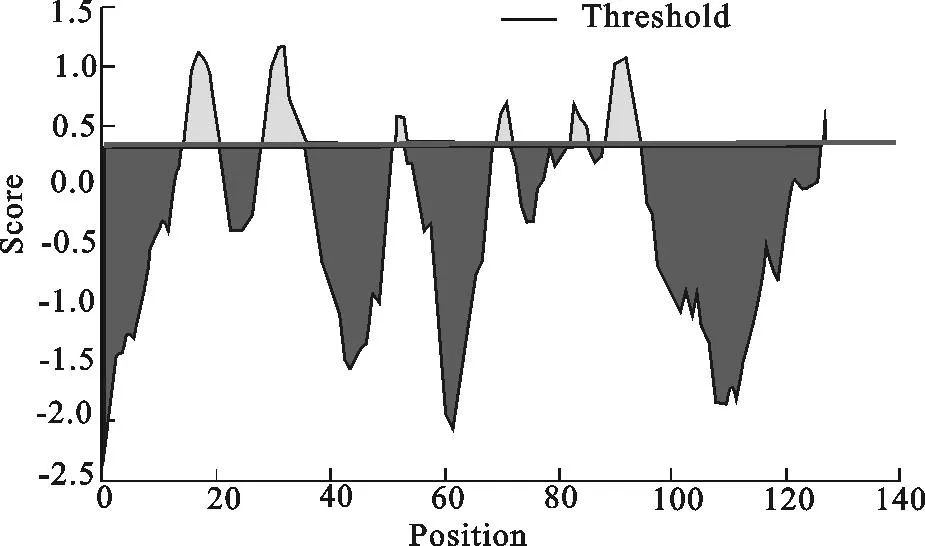
图4 SpiC 蛋白线性表位预测
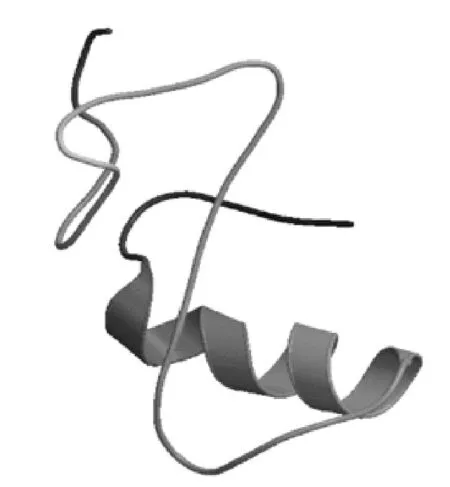
图5 3D结构分析
2.3.3同源性比较及进化分析将spiC基因序列在BlastN中进行比对,结果发现该核苷酸序列仅在沙门菌中存在,菌株之间保守性极高。氨基酸序列在沙门菌中一致性高达98%以上,与其他一些微生物如奈氏西地西菌 (Cedeceaneteri)、摩氏摩根菌 (Morganellamorganii)、雷金斯堡约克菌 (Yokenellaregensburgei)、耶尔森菌 (Yersinia)等同源序列一致性在40%左右。应用MAGE 5.0软件基于Neighbor-joining方法,将SpiC氨基酸序列与其他微生物构建系统进化树(图6),进化树主要分为3个大分支,SpiC属于第1大分支,与沙门菌肠道亚种 (Salmonellaenterica)的蛋白亲缘关系最近,这与鸡白痢沙门菌的细菌学分类结果一致。

图6 SpiC 的系统进化分析
3讨论
本研究以鸡白痢沙门菌基因组为模板,克隆得到spiC基因,推导出其编码的氨基酸序列,并对其进行了生物信息学分析。该基因全长384 bp,编码127个氨基酸,在鸡白痢沙门菌中稳定存在。沙门菌spiC基因在NCBI数据库中无高度同源序列,而且相对保守。Elemfareji O I等[12]对分离自不同地区、不同年代的87株伤寒沙门菌和94株肠炎沙门菌毒力相关基因进行PCR检测得出,spiC基因检出率为100%,充分说明该基因广泛存在于沙门菌中。本研究对近50年跨度的255个分离株的检测结果显示,该基因在鸡白痢沙门菌中稳定存在。此外,鉴于SpiC蛋白在其他致病微生物中不存在或同源性低的特点,我们推测其可作为沙门菌属的一种共同抗原用于该菌的特异性检测。在SPI-2编码的TTSS多种效应蛋白中,存在一段保守的可作为分泌转运信号的结构域,然而SpiC蛋白缺少该N-端序列[13]。跨膜蛋白在细胞与外界之间进行物质和信息交换过程中发挥着重要作用,而且跨膜部分多含有卷曲螺旋结构。通过软件分析发现SpiC无跨膜区或卷曲螺旋结构,其他一些已确定的效应蛋白如SseC、SseF、SseJ、SseG及SsaD等则均具有跨膜区或卷曲螺旋。
沙门菌SPI-2编码的SpiC蛋白最初被鉴定为一种效应蛋白,可转运至巨噬细胞的胞质中,参与调控SCV与吞噬溶酶体的融合以逃避宿主细胞的杀伤作用,但随着研究的不断深入,一些学者对其功能提出质疑,SpiC在TTSS中究竟扮演着何种角色仍值得推敲。尽管已证明高尔基体相关蛋白Hook3的功能涉及囊泡的运输[14],但NIPSNAP作为一个线粒体蛋白,其同系物TassC蛋白是如何结合SpiC以抑制吞噬溶酶体的融合不得而知。SpiC并未在细菌表面或宿主细胞内被检测到,其作为易位子或易位子组件的功能也未得到验证,暗示其很有可能是一类胞质蛋白,在TTSS效应蛋白SseB、SseC的易位过程中发挥分子伴侣或调解作用[15]。沙门菌spiC突变株与另一SPI-2编码的蛋白SsaM的基因突变株具有相似的表型,均丧失了分泌转运组分蛋白及形成沙门菌诱导丝的能力,免疫共沉淀试验也证实了两者有特异性的相互作用。进一步的研究揭示,TTSS的装配受“pH感应器”SsaM/SpiC/SsaL复合物的调节,当沙门菌SCV酸化时,诱导复合体降解,效应蛋白得以分泌[16]。而且,spiC突变株的毒力表型与单个效应蛋白的基因突变有很大差异,表现出类似于SPI-2整个突变的毒力致弱效应。基于此,就有一些研究者认为SpiC蛋白不是SPI-2的效应蛋白,而是作为分子伴侣在TTSS中发挥重要作用。至于这一猜测能否得到公认,需进一步探索,笔者所在实验室也将对此项研究继续做出努力。
参考文献:
[1]郭荣显,耿士忠,焦红梅,等. 噬菌体裂解E蛋白介导鸡白痢沙门菌跨膜孔道的电镜观察 [J]. 电子显微学报,2014, 33(1): 80-83.
[2]熊丹,宋丽,胡茂志,等. 沙门菌免疫逃逸机制及其应用研究进展 [J]. 动物医学进展,2015, 36(4): 96-100.
[3]Figueira R, Holden D W. Functions of theSalmonellapathogenicity island 2(SPI-2) type Ⅲ secretion system effectors [J]. Microbiology, 2012, 158 (5): 1147-1161.
[4]李静怡,崔可琦,冯赛祥,等. 鸡白痢沙门菌的分离鉴定、耐药性及分子分型研究 [J]. 动物医学进展,2014, 35(2): 29-33.
[5]Vonaesch P, Sellin M E, Cardini S, et al. TheSalmonellatyphimuriumeffector protein SopE transiently localizes to the early SCV and contributes to intracellular replication [J]. Cell Microbiol, 2014, 16(12): 1723-1735.
[6]Yu X J, Ruiz-Albert J, Unbworth K E, et al. SpiC is required for secretion ofSalmonellapathogenicity island 2 type Ⅲ secretion system proteins [J]. Cell Microbiol, 2002, 4(8): 531-540.
[7]耿士忠,刘欢,焦新安,等. 鸡白痢沙门菌SpiC 蛋白的原核表达及其应用 [J]. 细胞与分子免疫学杂志,2015, 31(2):149-152,158.
[8]Buckner M M, Croxen M A, Arena E T, et al. A comprehensive study of the contribution ofSalmonellaentericaserovartyphimuriumSPI2 effectors to bacterial colonization, survival, and replication in typhoid fever, macrophage, and epithelial cell infection models [J]. Virulence, 2011, 2(3): 208-216.
[9]Braukmann M, Methner U, Berndt A. Immune reaction and survivability ofSalmonellatyphimuriumandSalmonellainfantisafter infection of primary avian macrophages [J]. PLoS One, 2015, 10 (3): e0122540.doi:10.1371/journal.pone.0122540.
[10]Geng S Z, Jiao X A, Barrow P, et al. Virulence determinants ofSalmonellagallinarumbiovarpullorumidentified by PCR signature-tagged mutagenesis and the spiC mutant as a candidate live attenuated vaccine [J]. Vet Microbiol, 2014, 168(2-4): 388-394.
[11]耿士忠,刘男男,焦新安,等. 鸡白痢沙门菌S06004ΔspiC突变株的构建与鉴定 [J]. 中国兽医科学,2014, 44(4): 379-386.
[12]Elemfareji O I, Thong K L. Comparative virulotyping ofSalmonellatyphiandSalmonellaenteritidis[J]. Indian J Microbiol, 2013, 53(4): 410-417.
[13]Xu L, Sowa M E, Chen J, et al. An FTS/Hook/p107 (FHIP) complex interacts with and promotes endosomal clustering by the homotypic vacuolar protein sorting complex [J]. Mol Biol Cell, 2008, 19(12): 5059-5071.
[14]Leung A W, Halestrap A P. Recent progress in elucidating the molecular mechanism of the mitochondrial permeability transition pore [J]. Biochim Biophys Acta, 2008, 1777(7-8): 946-952.
[15]Tsai C L, Burkinshaw B J, Strynadka N C, et al. TheSalmonellatype Ⅲ sacretion system virulence effector forms a new hexameric chaperone assembly for export of effector/chaperone complexes [J]. J Bacteriol, 2015, 197(4): 672-675.
[16]Yu X J, McGourty K, Liu M, et al. pH sensing by intracellularSalmonellainduces effector translocation [J]. Science,2010, 328(5981): 1040-1043.
Cloning and Bioinformatics Analysis of spiC Gene fromSalmonellapullorum
AN Shu-min,GUO Rong-xian,XUE Ying,PAN Zhi-ming,GENG Shi-zhong,JIAO Xin-an
(KeyLaboratoryofZoonosesofJiangsuProvince,Co-innovationCenterforPreventionandControlofImportantAnimalInfectiousDiseasesandZoonosesofJiangsuProvince,YangzhouUniversity,Yangzhou,Jiangsu,225009,China)
Abstract:To research the function of SpiC protein, Salmonella pullorum spiC gene was amplified by PCR using specific primers which were designed according to the whole genome sequence of S06004 published in GenBank. Bioinformatics analysis was performed with software, and the evolution and presence in 255 Salmonella pullorum strains isolated from different years were investigated. The result showed that spiC gene was stable in all tested isolates. Sequence analysis revealed that spiC gene is 384 bp and encodes a putative protein of 127 amino acids. SpiC without transmembrane domain, signal peptide and coil structure, however, contains a potential casein kinase II phosphorylation site, two potential N-nutmeg acylation sites. The linear epitopes of SpiC are localized in the regions of 15-20, 29-35, 52-53, 70-72, 83-85, 89-94 aa. The three-dimensional structure of SpiC was determined by SWISS-MODEL work space. The homology and phylogenetic analysis showed that SpiC gene is conservative, and only occurs in Salmonella spp. The SpiC protein of Salmonella pullorum and Salmonella pullorum Subsp.enterica has the closest relationship.
Key words:Salmonella pullorum;SpiC protein; bioinformatics analysis
文章编号:1007-5038(2016)02-0006-05
中图分类号:S852.612;Q785
文献标识码:A
作者简介:安树敏 ( 1991-),女,内蒙古巴彦淖尔人,硕士,主要从事畜禽传染病研究。△同等贡献作者 *通讯作者
基金项目:863项目(2011AA10A212);江苏省自然科学基金 (BK20151306);中国博士后基金(2014M551670);国家自然科学基金项目(31230070)
收稿日期:2015-07-07
