濒危药用植物新疆阿魏cDNA文库构建与微卫星标记*
2016-03-13王果平樊丛照罗红梅季爱加李晓瑾
王果平,樊丛照,罗红梅,季爱加,宋 骏,李晓瑾**
(1.新疆维吾尔自治区中药民族药研究所 乌鲁木齐 830002;2.中国医学科学院 中国协和医科大学 药用植物研究所 北京 100193)
濒危药用植物新疆阿魏cDNA文库构建与微卫星标记*
王果平1,樊丛照1,罗红梅2,季爱加2,宋 骏1,李晓瑾1**
(1.新疆维吾尔自治区中药民族药研究所 乌鲁木齐 830002;2.中国医学科学院 中国协和医科大学 药用植物研究所 北京 100193)
目的:测试和分析新疆阿魏转录组,挖掘基因表达谱多态性标记(EST-SSRs),为新疆阿魏属植物遗传多样性、生物学特性、功能基因鉴定和濒危机制的研究奠定基础。方法:提取新疆阿魏茎叶总RNA,反转录合成cDNA,应用454 GS FLX Titanium进行测序,并对其序列进行拼接,获得一致性序列(Unique Sequences);使用Simple Sequence Repeat Identification Tool(Perl Script)对一致性序列进行分析,检索SSRs模体,采用Primer 5及SSR Hunter软件设计引物并进行PCR扩增。结果:高通量测序获得新疆阿魏ESTs序列550 200条,经拼接得到60 248条Unique Sequences,从中发现1 916个EST-SSRs,平均每100 kb出现3.18个SSR,其中二核苷酸最多,GA重复最常见,随机挑选5条序列扩增及测序结果表明,80%的SSR位点与Sanger测序结果一致。结论:通过转录组测序首次获得新疆阿魏EST-SSRs,为新疆阿魏生物学特性及濒危机制研究奠定了基础。
新疆阿魏 EST-SSRs 高通量测序 分子标记
药材阿魏为伞形科阿魏属植物花茎中具有特殊气味的油胶树脂,欧亚多国皆有阿魏的药用历史。在中国,阿魏最早收载于《唐本草》(公元659年),其后历代本草均有记载,具有解痉、祛风、止泻、除肠虫、治疗哮喘、癫痫及抗流感等功效[1-12]。《中华人民共和国药典》收载新疆阿魏Ferula sinkiangensis为阿魏药材基原植物之一,仅分布于中国新疆伊犁地区。因其为多年生一次结果早春草本植物,阿魏药材产自其花茎,过度采收将严重影响植物的繁衍,加之当地过度垦荒及气候变化等原因,阿魏的野生植物资源极度萎缩,现已被列为国家三级濒危保护植物[13]。因此,探索研究其濒危机制对新疆阿魏的资源保护及可持续利用具有重要的现实意义。
微卫星序列(Simple Sequence Repeat,SSR)标记是近年来发展起来的一种以特异引物PCR为基础的分子标记技术。SSR是一类由几个核苷酸(一般为1-6个)为重复单位组成的长达几十个核苷酸的串联重复序列,存在于真核生物的基因组中,因其数量丰富、多态性高、共显性等特点常被作为遗传标记广泛应用于动植物的遗传和育种研究实践中[14]。EST-SSRs是基于表达序列标签建立的分子标记,它在遗传作图、资源多样性、功能基因的发现与定位、物种起源与进化和比较基因组学研究等方面有着重要的利用价值。近年来EST-SSRs作为分子标记在农作物大麦、甜瓜及马铃薯等领域有着广泛的应用,亦有用于红豆杉、金银花等药用植物[15-19]。本研究拟采用高通量测序技术测试其转录组,挖掘基因表达谱多态性标记(EST-SSRs),为新疆阿魏属植物遗传多样性、生物学特性、功能基因鉴定和濒危机制的研究奠定基础。
1 材料与方法
1.1 植物材料
新疆阿魏F. sinkiangensis植物样本采自新疆伊宁县拜石墩新疆中药民族药研究所新疆阿魏野生抚育基地,随机选取植株幼嫩茎叶,采集后即刻用清水清洗5-8次,吸水纸吸干叶表面水分,迅速保存于液氮中备用。
1.2 RNA 提取与cDNA合成
随机选取3份样本,合并后采用总RNA提取试剂盒(北京百泰克生物技术有限公司,批号:RP 2801)提取新疆阿魏茎叶总RNA,Oligotex®mRNA kit(德国Qiagen公司,批号:70022)分离纯化mRNA。以2 μg mRNA为模板,利用SMARTTMPCR cDNA Synthesis kit(美国Clontech公司,批号:634902)反转录合成cDNA。采用PCR Advantage II polymerase(美国Clontech公司,批号:639201)对cDNA 进行扩增,扩增条件为95℃,1 min;94℃,15 s;65℃,30 s;68℃,6 min,13个循环。采用PureLink TM PCR Purification kit(美国Invitrogen公司,批号:K310001)去除体系中小于300 bp的片段。
1.3 454文库构建和测序[21,22]
将5 μg双链cDNA 打断为300-800 bp的片段,两端添加特异性衔接子A和B,变性为单链连接到磁珠上,经emPCR富集,置于PicoTiter Plate板,应用高通量测序平台454 GS FLX Titanium测序。
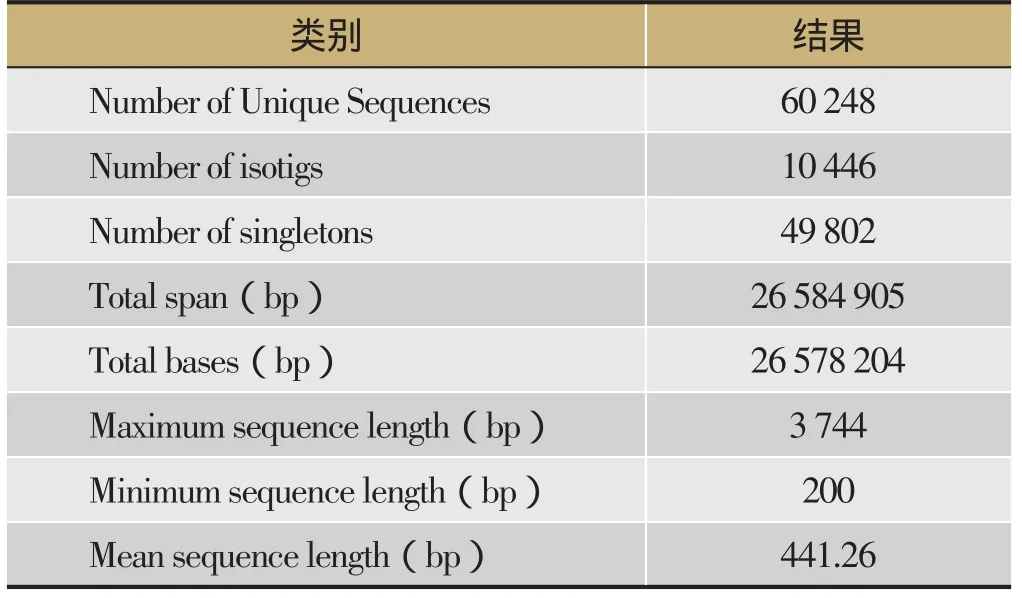
表1 阿魏转录组测序与组装结果
1.4 序列拼接
采用GS-FLX Software去除衔接子区域和低质量序列,屏蔽SMART PCR 引物。将测序序列利用GS De Novo Assembler软件进行拼接,所有分析使用默认参数。
1.5 微卫星标记检索
采用Simple Sequence Repeat Identification Tool检索SSR位点,设置参数如下:序列长度最短为14 bp;二核苷酸、三核苷酸、四核苷酸、五核苷酸、六核苷酸最少重复次数分别为7、5、4、3、3次。
1.6 SSR位点验证1.6.1 引物设计
随机选取5条含有SSR位点的Unique Sequences,采用SSRHunter软件对序列进行分析,采用Primer5软件在SSRs侧翼序列设计PCR引物,引物设计参数采用默认设置。
1.6.2 PCR扩增体系及反应条件
SSRs引物扩增体系为25 μL,包括12.5 μL 2×Taq PCR Mastermix(天根生化科技有限公司,批号:KT201),基因组DNA模板2 μL,上下游引物各1 μL,H2O 8.5 μL。PCR扩增程序为:95℃预变性1 min;94℃变性30 s,55℃退火30 s,72℃延伸1 min,35个循环;72℃延伸5 min。
1.6.3 扩增结果检测
PCR产物1%琼脂糖凝胶电泳检测后由美吉生物测序公司双向测序,测序峰图利用CodonCode Aligner V 4.0.4(美国CodonCode公司)校对拼接,去除引物区,并采用SSRHunter软件检验SSR位点。
2 结果与分析
2.1 测序和序列拼接
以新疆阿魏茎叶为材料,总RNA的提取结果较好,条带清晰。构建的新疆阿魏cDNA文库中,1/2测序反应获得数据总量为167.39 Mb,EST序列550 200条,所有ESTs通过GS FLX ASSEMBLER软件拼接,去除200 bp以下的序列,获得60 248 条Unique Sequences,包括10 446个序列重叠群(isotig)和49 802条单一序列(singleton),碱基总数为26 578 204 bp,最长序列为3 744 bp,最短200 bp,平均长度为441.26 bp。
2.2 新疆阿魏EST-SSRs的特征
在新疆阿魏茎叶60 248个unigene中搜索到1 916个SSR 位点,占unigene序列的3.18%, 平均每100 kb出现3.18个SSR。SSR种类丰富,二至六核苷酸重复类型均存在,重复次数也存有差异(表2)。在不同核苷酸重复类型中,最常见的是二核苷酸,占总ESR-SSRs的31.99%,其次是三核苷酸和六核苷酸(22.7%),出现频率最低的为五核苷酸(6.41%)。
在检出SSR中,共发现445种基序(motif)类型(图1),出现频率最高的10类基序为:GA(135个,7.17%)、AG(108个,5.73%)、TC(103个,5.46%)、TACG(92个,4.88%)、CT/TA(61个,3.24%)、ACGT(54个,2.87%)、GTAC(48个2.55%)、AC(42个,2.23%)、AT(38个,2.02%)、TG(35个,1.86%)。10类基序占所有SSRs出现频率的38.01%,其中六核苷酸基序类型有291种,其总量最丰富,占总类型的58.75%。
2.3 微卫星标记的验证
采用SSRHunter及Primer5软件,在SSRs序列附近设计引物,其中有1 028条EST-SSRs可成功设计正反向引物,包括625条singletons和403条contigs。为了验证EST-SSRs位点的真实性和可靠性,本实验随机选取5条引物进行PCR扩增,5条引物均获得了测序结果,其中编号为isotig00998、isotig03124、isotig09447、isotig04206的 4条 序列测序结果中SSR位点与contigs中一致,编号为isotig06720的序列Sanger测序结果重复序列为(TG)5,而在454测序中的重复序列为(AT)10。其余4条序列Sanger测序结果中的重复序列与SSRs基序序列一致。
3 讨论
近年转录组测序等高通量测序技术在众多植物中得到广泛应用,EST-SSR亦随之得到开发与利用。454 GS FLX Titanium高通量测序技术是一种基于焦磷酸测序原理而建立起来的第二代系统,其测序效率远高于传统的sanger测序法,但对cDNA文库的质量有较高的要求,本研究应用了SMART技术制备GS FLX cDNA文库的方法流程[20,21],构建了新疆阿魏cDNA文库,为新疆阿魏物遗传多样性、生物学特性、功能基因鉴定和濒危机制的研究提供新的切入点。

表2 新疆阿魏Unique Sequences的SSR位点预测
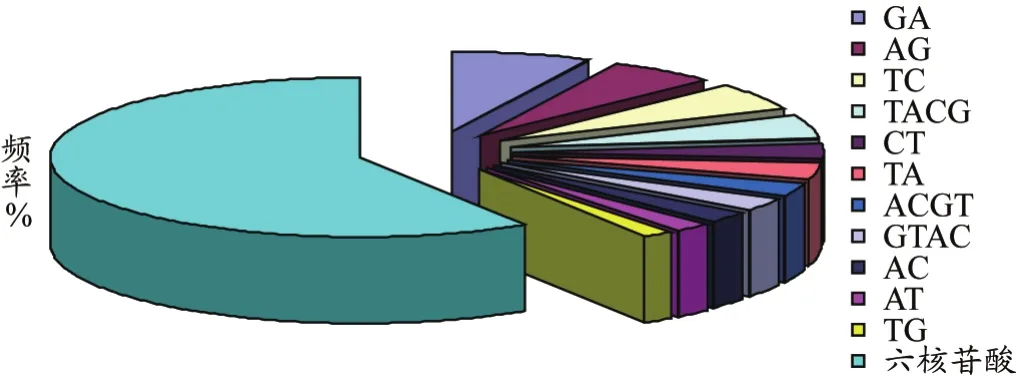
图1 基于模体类型的新疆阿魏EST-SSRs频率分布
新疆阿魏的ESTs中,四核苷酸为分布最少的模体,可能与四核苷酸SSRs主要位于非编码区有关,而双子叶中最具有代表性的重复序列AG在新疆阿魏中占比例较大[22]。在植物中,一般二核苷酸和三核苷酸为最丰富表达模体类型[23],但在新疆阿魏中最丰富表达模体类型为六核苷酸,与东北红豆杉研究结果一致[18]。SSR位点中存在较高的六核苷酸比例,即使六核苷酸位点发生变异,但由于突变压力和某些特定氨基酸的延伸,六核苷酸在基因编码的移码突变区存在很强的阳性选择,使遗传性状相对稳定,有利于物种生存[24]。新疆阿魏是否也与东北红豆杉一样是古老的物种,还有待于进一步深入研究。基于转录组测序的EST-SSRs标记对新疆阿魏及同属植物的生物学特性、遗传多样性、种质资源评价及良种培育等方面具有重要的应用价值。
参考文献
1 Srinivasan K. Spices as influencers of body metabolism: an overview of three decades of research. Food Research International, 2005, 38(1)∶77-86.
2 Zargari A. Medicinal Plants, Sixth ed. Tehran :Tehran University Publications, 1996∶ 312.
3 Ross I A. Medicinal plant of the World:Chemical Constituents, Traditional and Modern Medicinal Uses. Totowa:Humana Press, 2005∶223-224.
4 Iranshahy M, Iranshahi M. Traditional uses, phytochemistry and pharmacology of asafoetida (Ferula assa-foetida oleo-gum-resin)-A review. J Ethnopharmacol, 2011, 134(1)∶ 1-10.
5 Mahendra P, Bisht S. Ferula asafoetida: Traditional uses and pharmacological activity. Pharmacogn Rev, 2012, 6(12)∶ 141-146.
6 Ben Salem S, Jabrane A, Harzallah-Skhiri F. New bioactive dihydrofuranocoumarins from the roots of the Tunisian Ferula lut ea (Poir.) Maire. Bioorg Med Chem Lett, 2013, 23(14)∶ 4248-4252.
7 Nazari Z E, Iranshahi M. Biologically active sesquiterpene coumarins from Ferula species. Phytother Res, 20 11, 25(3)∶ 315-323.
8 Ka voosi G, Tafsiry A, Ebdam A A, et al. Evaluation of antioxidant and antimicrobial activities of essential oils from Carum copticum Seed and Ferula assafoetida Latex. J Food Sci, 2013, 78(2)∶ T356-T361.
9 Iranshahi M, Sahebkar A, Hosseini S T, et al. Cancer chemopreventive activity of diversin from Ferula diversivittata in vitro and in vivo. Phytomedicine, 2010, 17(3-4)∶ 269-273.
10 Abu-Zaiton A S. Anti-diabetic activity of Ferula assafoetida extract in normal and alloxan- induced diabetic rats. Pak J Biol Sci, 2010, 13(2)∶97-100.
11 Fatehi M, Farifteh F, Fatehi-Hassanabad Z. Antispasmodic and hypotensive effects of Ferula asafoetida gum extract. J Ethnopharmacol, 2004, 91(2-3)∶ 321-324.
12 Kumar S, Singh D K, Singh V K. Tertiary combination of freeze-dried urine of Indian breeds of cow with plant products against snail Lymnaea acuminata. Pak J Biol Sci, 2012, 15(20)∶ 992-996.
13 傅立国. 中国植物红皮书(1). 北京:科学出版社, 1992∶ 502.
14 Powell W, Machray G C, Provan J. Polymorphism revealed by simple sequence repeats. Trends in plant Science, 1996, 1(7)∶ 215-222.
15 赖勇,王鹏喜,范贵强,等.大麦SSR标记遗传多样性及其与农艺性状关联分析.中国农业科学, 2013, 46(2)∶ 233-242.
16 宋海斌,崔喜波,马鸿艳,等.基于SSR标记的甜瓜品种(系)DNA指纹图谱库的构建.中国农业科学, 2012, 45(13)∶ 2676-2689.
17 段艳凤,刘杰,卞春松,等.中国88个马铃薯审定品种SSR指纹图谱构建与遗传多样性分析. 作物学报, 2009, 35(8)∶ 1451-1457.
18 吴琼,段小群,肖培根,等.基于高通量测序的红豆杉EST-SSRs标记研究.中国中药杂志, 2012, 37(24)∶ 3728-3733.
19 何柳,徐晓兰,王振中,等.金银花莽草酸/奎宁酸香豆酰转移酶(LjHCT)基因克隆与序列分析. 世界科学技术-中医药现代化, 2014, 16(2)∶ 263-268.
20 Poinar H N, Schwarz C, Qi J, et al. Metagenomics to paleogenomics: large-scale sequencing of mammoth DNA. Science, 2006, 311(5759)∶392-394.
21 Margulies M, Egholm M, Altman W E, et al. Genome sequencing in microfabricated high- density picolitre reactors. Nature, 2005, 437(7057)∶ 376-380.
22 Temnykh S, DeClerck G, Lukashova A, et al. Computat ional and experimental analysis of microsatellites in rice(Oryza satixa L.): frequency, length variation, transposon associations, and genetic marker potential. Genome Res, 2001, 11(8)∶ 1441-1452.
23 伍宝朵,郝朝运,范睿,等.胡椒EST-SSR标记的开发和应用.热带作物学报, 2015, 36(2)∶ 237-243.
24 Chagne D, Chaumeil P, Ramboer A, et al. Cross-species transferability and mapping of genomic and cDNA SSRs in pines. Theor Appl Genet, 2004, 109(6)∶ 1204-1214.
cDNA Library Construction and Microsatellite Marker Analysis of the Endangered Medicinal Plant Ferula sinkiangensis
Wang Guoping1,Fan Congzhao1,Luo Hongmei2,Ji Aijia2,Song Jun1,Li Xiaojin
(1.Xinjiang Institute of Chinese Materia Medica and Ethnical Materia, Urumqi 830002, China; 2.Institute of Medicinal Plant Development, Chinese Academy of Medical Sciences and Peking Union Medical College, Beijing 100193, China)
This study aimed to detect and analyze of the transcriptome data of F. sinkiangensis, as well as identify its genetic polymorphism of simple sequence repeats (SSRs) of expressed sequence tags (EST-SSRs), which laid foundations for studies on genus genetic diversity, biological characteristics, identification of functional gene and the endangered mechanism of F. sinkiangensis. Total RNA from the leaves of F. sinkiangensiswas extracted and reversed to cDNA, in order to construct the 454-EST library. Then 454-EST library was sequenced using 454 GS FLX Titanium. After sequences assembly, the unique sequences of F. sinkiangensis were obtained. The SSR identification tool that named Sequence Repeat Identification Tool Simple (Script Perl) was applied to analyze the SSR motif from the obtained unique sequences. Primers were designed by Primer 5 and SSR Hunter software and amplified by PCR. As a result, 550 200 EST sequences of F. sinkiangensis were obtained by high-throughput sequencing, which were assembled into 60 248 unique sequences. Among these unique sequences, 1 916 EST-SSRs sites were discovered with the average of 3.18 EST-SSRs sites in every 100 kb sequences. Among the EST-SSRs sites, dinucleotide SSRs was the richest with G and A nucleotide repeats mostly. In conclusion, EST-SSRs data of F. sinkiangensis was established firstly in this study, which provided the basis for further researches for biological characteristics and endangered mechanisms of F. sinkiangensis through transcriptome sequencing.
Ferula sinkiangensis, EST-SSRs, high-throughput sequencing, molecular marker
10.11842/wst.2016.02.011
R282.2
A
(责任编辑:马雅静 张志华,责任译审:朱黎婷 王 晶)
2015-11-29
修回日期:2015-12-07
* 新疆维吾尔自治区科技厅优秀青年科技创新人才培养项目(2013721042):基于高通量测序技术的濒危药用植物新疆阿魏开花调控基因的挖掘,负责人:王果平。
** 通讯作者:李晓瑾,研究员,主要研究方向:中药资源。
