基于线粒体COⅠ序列比较长江口中华绒螯蟹放流与野生群体的遗传多样性
2016-03-04彭欣悦朱美贵
彭欣悦,赵 峰,张 涛,耿 智,3,朱美贵,庄 平
(1.中国水产科学研究院东海水产研究所,上海 200090;2.上海海洋大学水产与生命学院,上海 201306;3.华东师范大学生命科学学院,上海 200062)
基于线粒体COⅠ序列比较长江口中华绒螯蟹放流与野生群体的遗传多样性
彭欣悦1,2,赵 峰1,张 涛1,耿 智1,3,朱美贵1,2,庄 平1
(1.中国水产科学研究院东海水产研究所,上海 200090;2.上海海洋大学水产与生命学院,上海 201306;3.华东师范大学生命科学学院,上海 200062)
为探讨长江口中华绒螯蟹(Eriocheir sinensis)放流亲蟹与野生群体的遗传多样性和遗传结构的差异,对77个放流和野生亲蟹个体进行了线粒体细胞色素氧化酶亚基Ⅰ(COⅠ)基因序列的比较分析,结果表明:所检测的样本共计77个COⅠ基因序列(630 bp)中,变异位点(V)41个,简约信息位点(P)37个,A+T(61.7%)的含量明显高于C+G(38.3%),表现出较为明显的碱基组成偏倚性;两个群体共检测出15种单倍型,其中单倍型Hap-1出现频率最大,为两个群体所共享,放流和野生群体各具有5种独有单倍型;两个群体的单倍型多样性指数(H)为0.825,核苷酸多样性指数(π)为0.004 66,平均核苷酸变异数(K)为2.910,其中野生群体的单倍型多样性指数(0.833)和核苷酸多样性指数(0.007 70)均高于放流群体(0.810和0.002 11)。AMOVA分子方差分析表明,长江口中华绒螯蟹放流与野生群体总的遗传变异主要来自群体内,其中96.53%遗传变异来自各群体内部,3.47%遗传变异来自群体间。群体内遗传分化指数(FST)野生群体(0.034 75)高于放流群体(0.034 57),两个群体间遗传分化指数(FST)为0.034 66(P>0.05),两个群体遗传分化不显著。两个群体间的基因流(Nm)为13.93(Nm>1),表明放流群体和野生群体的基因交流较为频繁。
中华绒螯蟹;遗传多样性;COⅠ基因;长江口;增殖放流
中华绒螯蟹(Eriocheir sinensis)俗称河蟹,隶属节肢动物门(Arthropoda),甲壳纲(Crustacea),十足目(Decapoda),方蟹科(Grapsidae),绒螯蟹属,自然分布十分广泛,在我国北起辽宁、南至福建沿海各省份的通海河流中几乎均有其踪迹,但以长江水系中华绒螯蟹群体种质最为优良[1]。长江口由于生境独特、河口浅滩广阔、生物多样性高,是中华绒螯蟹得天独厚的产卵场[2],加之长江源远流长,长江中下游水体中水草茂盛,饵料丰富,对中华绒螯蟹的生长育肥十分有利,由此形成了极具特色的长江水系中华绒螯蟹品系,因其个体肥大、肉质细嫩、味道鲜美而深受广大消费者的喜爱[3]。近几十年,由于过度捕捞、围垦造田、水域环境污染以及长江中下游大型水利工程的建设等原因,中华绒螯蟹生存及洄游的生态环境受到严重破坏,自然资源急剧衰退[4]。自上世纪60年代末至本世纪初长江口的蟹苗产量极速下降,曾一度枯竭,几乎没有产量[5]。为了恢复中华绒螯蟹天然资源,近年来长江中下游各省市均采取了人工增殖放流,特别是长江口水域的亲蟹增殖放流活动取得了良好的效果。
目前,对渔业增殖放流效果评估的主要方法是放流个体标记与回捕率分析,其中实物标记是传统的标记方法,应用也最为广泛[6]。近年来,分子标记技术研究迅猛发展,在虾蟹类的增殖放流效果评估中也得到了一定的应用[7-9]。对长江口中华绒螯蟹增殖放流的研究工作大多集中在标志方法、放流数量、放流区域、放流时间及苗种大小等方面[6],增殖放流的效果究竟如何、放流群体和野生群体间的遗传关系如何、放流群体是否会改变自然水域野生群体的遗传多样性等成为目前人们关注的问题。本实验利用线粒体DNA分子标记技术对长江口中华绒螯蟹增殖放流活动中的养殖亲蟹和长江口自然水域的野生亲蟹两个群体的线粒体细胞色素氧化酶亚基Ⅰ(COⅠ)基因序列进行了初步研究,比较并分析了其群体遗传多样性和遗传结构,旨在探究长江口水域中华绒螯蟹亲蟹的遗传学背景,为系统研究长江口中华绒螯蟹种质资源状况、增殖放流效果评价等提供一定的理论依据。
1 材料与方法
1.1 样本采集
2014年12月采集长江口中华绒螯蟹增殖放流活动的放流亲蟹40 ind,记为放流群体;同时,从2015年3月至2015年6月长江口中华绒螯蟹产卵场定点底拖网调查捕获的野生中华绒螯蟹亲蟹中随机抽取37 ind,记为野生群体。将所有样本活体带回实验室后,测定每个样本的体重、体宽、体高等表型性状,因野外调查捕获的中华绒螯蟹中可能混有放流亲蟹,故需根据曹侦等[10]研究的形态差异分析法进行判别,分别取其螯足或步足,置于-20℃保存备用。
1.2 基因组DNA提取
将置于-20℃保存的样本取出置于碎冰上,取其螯足部分肌肉,为避免肌肉样品反复冻融,每个样本取出的肌肉分装1~3管并编号,以备再次提取基因组DNA。基因组DNA提取方法严格按照海洋动物组织基因组DNA提取试剂盒(TIANGEN)中的说明书操作步骤进行,提取的基因组DNA采用紫外吸收法测定其含量及纯度。
1.3 线粒体COⅠ基因扩增及序列测定
采用FOLMER等[11]的中华绒螯蟹线粒体COⅠ基因引物进行序列扩增和测定,引物序列为COⅠ-F:5′-GGTCAACAATCATAAGATATTGG-3′、COⅠ-R:5′-TAAACTTCAGGGTG ACCAAAAAATCA-3′,引物由上海桑尼生物科技有限公司合成。反应体系总体积为50μL,包括Premix溶液25μL(1.25U/25μL TaKaRa Taq、0.4 mM dNTP、0.4 mM Mixture Taq Buffer等)、模板DNA 5μL(60~100 ng·μL-1)、引物(F/R)各1μL(20μM)、灭菌蒸馏水18μL。在PCR仪(Eppendorf 5331)上进行扩增,共设30个循环。循环前94℃预变性5 min,每个循环包括94℃40 s,55℃退火45 s,72℃延伸60 s;循环结束后于72℃延伸10 min。
PCR扩增产物用1%的琼脂糖凝胶电泳检测,并在凝胶成像系统观察并拍照,结合Marker判断并选取目的条带明亮、清晰、无拖尾、特异性较好的样品,编号并密封包装,送上海桑尼生物科技公司的ABIPRISMTM测序系统进行双向测序,并进行人工序列核对、矫正和拼接,测序后利用GeneDoc软件进行对位排序。
1.4 数据处理
1.4.1 测序结果比对及遗传多样性参数分析
将每个样本测序得到的结果在GenBank数据库中进行BLAST同源性搜索,然后使用Clustal X、MEGA 5.1软件中进行序列比对和重排,同时辅以人工校对;使用MEGA 5.1计算其碱基含量、变异位点数(V)、简约信息位点数(P);使用DNASP 5.1计算其单倍型数目(H)、单倍型多样性指数(Hd)、核苷酸多样性指数(π)、平均核苷酸差异数(K)等。
1.4.2 群体遗传结构分化分析
使用软件MEGA 5.1,并运用Kimura双参数替代模型(Kimura-2-Parameter,K-2-P)计算群体间的遗传距离;使用Arlequin 3.5软件进行分子变异方差分析(AMOVA),计算群体内及群体间的遗传分化指数(FST)及遗传变异在群体内及群体间的分布,并检测群体间FST的显著性(重复次数为1 000次)。通过设定两种AMOVA分析来检验中华绒螯蟹的群体遗传结构,首先根据样本来源,将所有样本划分为放流群体和野生群体两个组群,以验证群体内是否存在显著的群体遗传结构差异;其次将所有样本划分为一个组群,以验证群体间是否存在显著的群体遗传分化,群体间的基因流Nm计算公式为:Nm=(1/FST-1)/2。
2 结果与分析
2.1 中华绒螯蟹线粒体COⅠ基因序列变异
用于研究的77个的中华绒螯蟹个体的COⅠ基因均可被稳定扩增,PCR扩增产物经1%琼脂糖凝胶电泳检测,均能显示明亮清晰的条带,PCR扩增产物经试剂盒纯化回收后送至上海桑尼生物科技有限公司测序。测序得到的所有COⅠ基因序列经比对和校正,在用于群体遗传多样性数据分析的77个630 bpCOⅠ基因序列中,其变异位点数(V)41个,占总位点数的6.51%;简约信息位点数(P)37个,单变异位点数4个(图1);在两个群体所有的COⅠ基因序列的碱基组成中A、T、C、G平均碱基含量分别为27.1%、34.6%、21.0%、17.3%,A+T(61.7%)的含量明显高于C+G(38.3%),表现出较为明显的碱基组成偏倚性(表1)。

图1 中华绒螯蟹线粒体COⅠ基因序列变异位点Fig.1 Variable sites ofm itochondrial COⅠgene for E.sinensis

表1 中华绒螯蟹放流群体与野生群体线粒体COⅠ基因片段的序列组成Tab.1 Nucleotide composition ofm itochondrial COⅠgene in released and w ild E.sinensis populations
2.2 种群遗传结构特征
在2个中华绒螯蟹群体中共检测出15种单倍型,各群体的单倍型分布见表2,其中单倍型Hap-1出现频率最大并为两个群体所共享,单倍型Hap-3、Hap-5、Hap-6、Hap-13、Hap-14为放流群体所独有,单倍型Hap-9、Hap-10、Hap-11、Hap-12、Hap-15为野生群体所独有。
两个中华绒螯蟹群体遗传多样性参数如表3所示。由表3可知,放流群体序列变异程度远低于野生放流群体,在放流群体所有COⅠ基因序列中发现变异位点8个,其中简约信息位点数7个;野生群体所有COⅠ基因序列中发现变异位点41个,其中简约信息位点数35个。放流群体单倍型数、单倍型多样性、核苷酸多样性及平均核苷酸差异数分别为9、0.801 1、0.002 11和1.319,均分别低于野生群体的单倍型数、单倍型多样性、核苷酸多样性及平均核苷酸差异数,其依次为12、0.833 3、0.007 70和4.982。

表2 中华绒螯蟹COⅠ基因单倍型在放流群体和野生群体中的分布Tab.2 Distribution of COⅠgene hap lotypes in released and w ild E.sinensis populations

表3 中华绒螯蟹放流群体与野生群体的遗传多样性参数Tab.3 Parameters of genetic diversity in released and w ild E.sinensis populations
通过Kimura双参数法计算放流群体和野生群体内的遗传距离分别为0.002和0.007,两个群体间的遗传距离为0.005。两个群体间的遗传分化指数(FST)和基因流(Nm)分别为0.034 66、13.93(表4)。两个群体间的FST和Nm显示放流群体和野生群体的基因交流较为频繁(Nm=13.93>1)。
AMOVA分子方差分析表明,中华绒螯蟹放流群体和野生群体两个群体总的遗传变异主要来自群体内,其中96.53%遗传变异来自各群体内部,3.47%遗传变异来自各群体间。群体内遗传分化指数(FST)野生群体(0.034 75)高于放流群体(0.034 57),两个群体间遗传分化指数(FST)为0.034 66(P>0.05),表明两个群体遗传分化不显著(表5)。

表4 中华绒螯蟹种群间的FST(左下角)和Nm(右上角)Tab.4 FST(left bottom)and Nm(right bottom)value of E.sinensis populations
3 讨论
3.1 中华绒螯蟹群体遗传结构特征
线粒体DNA的研究是近20年来分子生物学研究的重要内容,线粒体DNA是核外遗传物质,与核基因存在平行进化关系,为母系遗传并且具有较高的突变率,突变固定后形成的DNA多态性位点可反映出群体遗传特征、种群分化和种属关系[12]。细胞色素氧化酶I亚基(cytochrome oxidase subunit I,COⅠ)是线粒体氧化呼吸链的重要成员,是线粒体基因组中变异性较大、进化速度较快的区域,故适合于种群遗传水平差异的检测,该基因近年来在昆虫、虾类、蟹类、贝类以及鱼类的群体遗传结构和系统进化方面有较多的研究[13-18]。
本研究所得的COⅠ基因序列中,A+T的含量为61.7%,明显高于C+G的含量,表现出较为明显的碱基组成偏倚性。A、T、C和G 4种核苷酸在线粒体基因组中呈不均一性分布,这是动物线粒体基因组的一个共性[19]。有研究者基于线粒体COⅠ基因序列的三疣梭子蟹(Portunus trituberculatus)东海区群体遗传多样性分析研究也得出三疣梭子蟹A+T(64.2%)的含量明显高于C+G(35.9%)[20],这与本研究结果十分接近。三疣梭子蟹和中华绒螯蟹同属十足目蟹类,两者的线粒体基因COⅠ基因核苷酸组成非常接近,均符合节肢动物A、T含量高的特点[21]。在本研究中的放流与野生群体的所有样本的线粒体COⅠ基因中,共测定出变异位点数(V)41个,占总位点数的6.51%,其中简约信息位点数(P)37个,远大于单变异位点数(S)。在群体总的变异位点中,大多位点存在多种变异类型,群体变异位点的变异种类越多,其群体遗传多样性水平就越高。其中,在两个种群线粒体COⅠ基因序列第34~364 bp之间集中27个变异位点,是变异频率较高的区段,可以考虑作为中华绒螯蟹种群鉴别的线粒体DNA分子标记。在对两个群体所有个体的线粒体COⅠ基因序列单独进行分析时发现,在野生群体中检测出变异位点数(V)41个、简约信息位点数(P)35个、单变异位点数(S)6个,而在放流群体中检测出变异位点数(V)8个、简约信息位点数(P)7个、单变异位点数(S)1个。由此可以得知,对两个群体整体和单独分析结果均表明,野生群体的遗传变异程度均远大于放流群体,这意味着目前长江口中华绒螯蟹野生群体遗传信息量丰富,利于物种种质资源的优化。
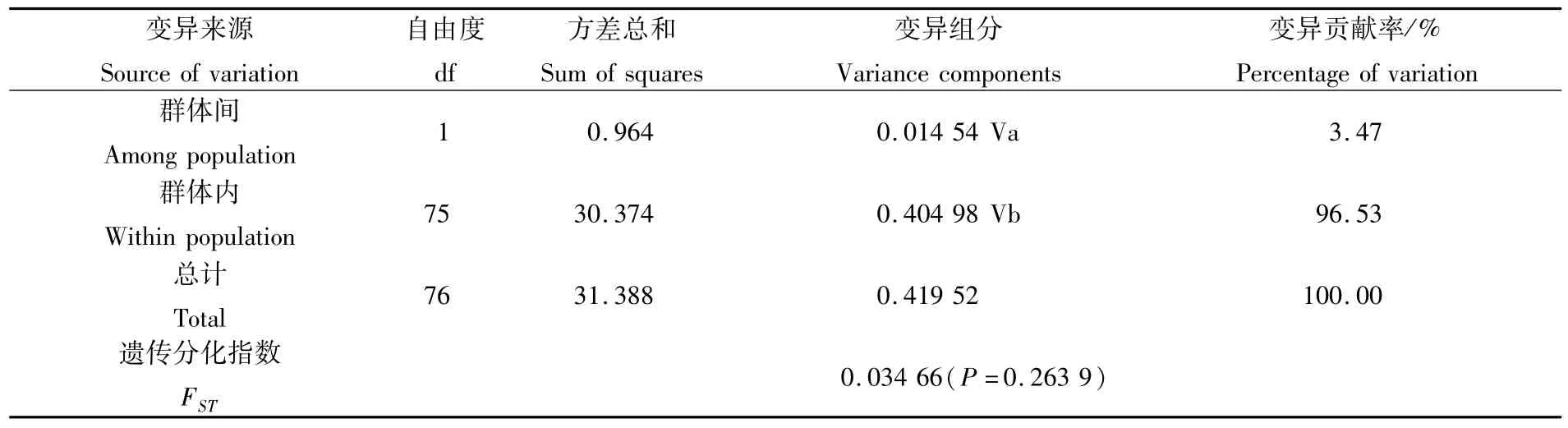
表5 中华绒螯蟹放流群体与野生群体的AMOVA分析Tab.5 AMOVA analysis of released and w ild E.sinensis popu lations
3.2 中华绒螯蟹放流和野生群体的群体遗传多样性
遗传多样性是生物多样性的重要组成部分,同时也是物种进化的本质与保证。群体的遗传变异程度决定着物种进化的趋势,往往一个种群对环境适应能力的强弱恰恰是该种群遗传多样性丰富程度的直观反映,因此对群体遗传多样性的研究极具理论和实践意义。一个物种的群体遗传多样性的水平受到诸多因素的影响,如人工养殖规模大小、群体内的近交及随机遗传漂变、物种自身的生活史特点等[20]。近年来有学者研究表明十足目虾蟹类的遗传多样性水平较低,且变 异 基 本 全 部 来 自 于 群 体 内[22-25]。HEDGECOCK等[26]通过总结65种虾蟹类的平均杂合度,认为甲壳类动物遗传变异较低的原因是其较短的生活史以及缺乏随机遗传漂变。单倍型多样性与群体大小及环境有关,群体大且环境差异大则群体的单倍型多样性就会高[27]。庞大的种群数量、环境的不均一性和适于种群快速增长的生活习性是维持自然种群内较高单倍型多样性的基础[28]。核苷酸多样性(π)是衡量群体遗传多样性和群体遗传分化的重要指标之一,π值越大表示群体遗传多样性越高[29]。本研究结果表明,长江口放流群体和野生群体均具有较高的单倍型多样性指数,分别为0.801 1和0.833 3,而核苷酸多样性指数相对较低,分别为0.002 11和0.007 70,由此推测长江口中华绒螯蟹群体数量相对较大,但群体内各单倍型之间序列差异较小。本研究结果还显示,中华绒螯蟹放流群体的单倍型多样性、核苷酸多样性及平均核苷酸差异数均低于野生群体的单倍型多样性、核苷酸多样性及平均核苷酸差异数,这说明长江口中华绒螯蟹放流群体的遗传多样性低于野生群体,这可能是因为本研究中所用放流群体的亲蟹采购于养殖场,其通过捕捞野生亲本,采用人工育苗技术培育成亲蟹,野生亲本数量的有限性可能是导致其群体遗传多样性较低的原因之一;其次可能是由于养殖环境的单一性所致。有学者采用线粒体COⅠ序列对厚壳贻贝(Mytilus coruscus)和银鲳(Pampus argenteus)的养殖与野生群体遗传多样性比较分析的研究结果均表明,养殖群体的单倍型数量和核酸多样性指数都要比野生群体的单倍型数量和核酸多样性指数低[30-31],这一结果与本研究所得结果一致。FST常用来表示两个群体之间的分化程度,在0~1的范围内,FST值越大表示两个群体的分化程度越高。本研究中,两个群体总的遗传分化指数FST为0.034 66(P>0.05),表明两个群体之间的遗传差异较小,遗传分化不显著。AMOVA分子方差分析表明,两个群体总的遗传变异基本上都是存在各群体内(表5),这与FST的分析结果是一致的。
总之,目前长江口中华绒螯蟹遗传多样性还较为丰富,而且放流与野生群体间存在一定的遗传结构差异,两群体之间并未出现显著遗传分化,这可能与中华绒螯蟹自身的洄游特性、人工增殖放流的规模较小、时间较短有关,但仍需要继续监测两者的遗传状况变化,以防止野生群体基因库受到放流群体的干扰和污染,从而降低野生群体的遗传多样性。同时,建议增殖放流亲本要选择经过遗传检测与评价的亲蟹群体进行增殖放流,以避免其对野生中华绒螯蟹种质资源造成不良影响。
[1] 李晨虹,李思发.中国大陆沿海六水系绒螯蟹(中华绒螯蟹和日本绒螯蟹)群体亲缘关系:形态判别分析[J].水产学报,1999,23(4):337-342.
LI C H,LI S F.Phylogenesis of populations of mitten crabs(Eriocheir sinensisandEriocheirjaponicus)in six river systems of mainland China:morphology discriminant analysis[J].Journal of Fisheries of China,1999,23(4):337-342.
[2] 庄 平,王幼槐,李圣法,等.长江口鱼类[M].上海:科学技术出版社,2006:1-11.
ZHUANG P,WANG Y H,LISF,etal.Fishery of the Yangtze Estuary[M].Shanghai:Shanghai Science and Technology Press,2006:1-11.
[3] 吕永春.长江水系和其他水系中华绒螯的区别[J].中国水产,1998(1):23.
LV Y C.The difference ofEriocheir sinensisbetween the Yangtze River and other river[J].China Fisheries,1998(1):23.
[4] 刘 凯,段金荣,徐东坡,等.长江口中华绒螯蟹亲体捕捞量现状及波动原因[J].湖泊科学,2007,19(2):212-217.
LIU K,DUAN J R,XU D P,et al.Studies on current resource and causes of catch fluctuation of brooders of mittencrab in estuary of the Changjiang River[J].Journal of Lake Sciences,2007,19(2):212-217.
[5] 张航利,王海华,冯广鹏.长江口中华绒螯蟹和中华鲟的增殖放流及其效果评估[J].江西水产科技,2012(3):45-48.
ZHANG H L,WANG H H,FENG G P.Effect evaluation on stock enhancement ofEriocheir sinensisandAcipenser sinensisfrom the Yangtze Estuary[J].Fishery Sciences,2012(3):45-48.
[6] 刘 璐,林 琳,李纯厚,等.海洋渔业生物增殖放流效果评估研究进展[J].广东农业科学,2014(2):133-137.
LIU L,LIN L,LIC H,et al.Effect assessment of marine fishery stock enhancement:A review of the literature[J].Guangdong Agricultural Sciences,2014(2):133-137.
[7] 李伟亚,王伟继,孔 杰,等.中国对虾微卫星四重PCR技术的建立及其在模拟放流效果评估方面的应用[J].海洋学报(中文版),2012,34(5):213-220.
LIW Y,WANG W J,KONG J,et al.Establishment of themicrosatellite quadruple PCR technology forFenneropenaeus Chinensisand its application in the evaluation on the effect of releasing simulation[J].Acta Oceanologica Sinica,2012,34(5):213-220.
[8] 陈睿毅.微卫星标记在牙鲆增殖放流中的应用研究[D].上海:上海海洋大学,2013.
CHEN R Y.Application ofmicrosatellitemarkers to evaluate the effect of Paralichthys olivaceus stock enhancement[D].Shanghai:Shanghai Ocean University,2013.
[9] 杨 爽.基于线粒体DNA控制区标记的三疣梭子蟹和中国对虾增殖放流效果评价研究[D].青岛:中国海洋大学,2013.
YANG S.Effect evaluation on stock enhancement ofPortunus trituberculatusandFenneropenaeus chinensisbased on mtDNA control region[D].Qingdao:Ocean University of China,2013.
[10] 曹 侦,冯广朋,庄 平.长江中华绒螯蟹洄游群体与养殖群体形态差异分析[J].淡水渔业,2013,6(43):3-7.
CAO Z,FENG G P,ZHUANG P.Morphological difference analysis between migratorypopulation and cultured population of Eriocheir sinensis in the Yangtze River[J].Freshwater Fisheries,2013,6(43):3-7.
[11] FOLMER O,BLACK M,HOEH W,et al.DNA primers for amplification ofmitochondrial cytochrome coxidase subunit I from diverse metazoan invertebrates[J].Molecular Marine Biology and Biotechnology,1994,3(5):294-299.
[12] 王 伟,何舜平,陈宜瑜.线粒体DNA d-loop序列变异与鳅鮀亚科鱼类系统发育[J].自然科学进展,2002(1):35-38.
WANGW,HE SP,CHEN Y Y.Sequence variation of mtDNA control region andGobiobotinaefish phylogeny[J].Progress in Natural Science,2002(1):35-38.
[13] VANDEWOESTIJNES,BAGUETTEM,BRAKEFIELD P M,et al.Phylogeography ofAglais urticae(Lepidoptera)based on DNA sequences of the mitochondrial COⅠgene and control region[J].Molecular Phylogenetics and Evolution,2004,31(2):630-646.
[14] HUALKASINW,SIRIMONTAPORN P,CHOTIGEAT W,et al.Molecular phylogenetic analysis of white prawns species and the existence of two clades inPenaeus merguiensis[J].Journal of Experimental Marine Biology and Ecology,2003,296(4):1-11.
[15] MAURO A,ARCULEO M,PARRINELLO N.Morphological and molecular tools in identifying the Mediterranean limpetsPatella caerulea,Patella asperaandPatella rustica[J].Journal of Experimental Marine Biology and Ecology,2003,295(2):131-143.
[16] BURRICP,SMOLENSKIA J.Molecular phylogeny of theCheilodactylidaeandLatridaewith notes on taxonomy and biogeography[J].Molecular Phylogenetics and Evolution,2004,30(2):118-127.
[17] TANG B P,ZHOU K Y,SONG D X,et al.Molecular phylogeny of the Latridae with notes on taxonomy and biogeography[J].Molecular Phylogenetics and Evolution,2004,30(3):118-127.
[18] TANG B P,ZHOU K Y,SONG D X,et al.Molecular systematics of the Asian mitten crabs,genus Eriocheir(Crustacea:Brachyura)[J].Molecular Phylogenetics and Evolution,2003,29(2):309-316.
[19] BROWNW M.The Mitochondrial Genome of Animals[M]//MACLNTYRE R J,ed.Molecular evolutionary genetics.New York:Plenum Press,1985:95-130.
[20] 王 景,张凤英,蒋科技,等.基于线粒体COⅠ基因序列的三疣梭子蟹东海区群体遗传多样性分析[J].海洋渔业,2015,37(2):114-121.
WANG J,ZHANG F Y,JIANG K J,et al.Genetic diversity of Portunus trituberculatus based on the mitochondrial cytochrome oxidase subunit I from the East China Sea[J].Marine Fisheries,2015,37(2):114-121.
[21] 胡 婧,刘 念,黄 原.节肢动物线粒体基因组研究进展与基因顺序分析[J].昆虫分类学报,2006,2(28):153-160.
HU J,LIU N,HUANG Y.Advances on research of arthropoda mitochondrial genomes and analysis of gene order[J].Enotomotaxonomia,2006,2(28):153-160.
[22] 孟宪红,马春艳,刘 萍,等.黄渤海中国对虾6个地理种群的遗传结构极其遗传分化[J].高技术通讯,2004,14(4):97-102.
MENG X H,MA C Y,LIU P,et al.Study on seven geographic populations ofFenneropenaeus chinensisbased on microsatellite DNA[J].Marine Fisheries Esearch,2004,14(4):97-102.
[23] 高志千,周开亚.中华绒螯蟹遗传变异的RAPD分析[J].生物多样性,1998,6(3):186-190.
GAO Z Q,ZHOU K Y.Genetic variation of the Chinese mitten-handed crab populations detected by RAPD analysis[J].Chinese Biodiversity,1998,6(3):186-190.
[24] 邱高峰,常林瑞.我国近海中国对虾种群遗传差异的RAPD分析[J].上海水产大学学报,2001,10(1):1-5.
QIU G F,CHANG L R.Population genetic variation of Chinese shrimp Penaeus Chinensis along the coast of China assessed by randomamplified polymorphic DNA(RAPD)[J].Journal of Shanghai Fisheries University,2001,10(1):1-5.
[25] MULLEY J,LATTER B.Genetic variation and evolutionary relationships within a group of thirteen species of penaeid prawns[J].Evolution,1980(34):904-916.
[26] HEDGECOCK D,TRACCY M,NELSON K.The biology of Crustacea[C].New York:Academic Press,1982:284-430.
[27] REISER F.Molecular Evolutionary Genetics[J].Library Journal,1987,112(4):84.
[28] NEIM.Molecular evolutionary genetics[M].New York:Columbia University Press,1987.
[29] BUONNACORSIV P,MCDOWELL JE.Reconciling pattems of inter ocean molecular variance from for classes of molecular markers in blue marlin(Makaira nigricans)[J].Molecular Ecology,2001,10(5):1179-1196.
[30] 管成林,李继姬,郭宝英.基于线粒体COⅠ基因比较分析野生与养殖厚壳贻贝种群的遗传多样性[J].安徽农业科学,2012,40(13):7660-7662.
GUAN C L,LI J J,GUO B Y.Genetic diversity evaluation by the comparative analysis on mitochondrial COⅠgene between wild and cultured populations ofMytilus couruscus.[J].Journal of Anhui Agricultural Sciences,2012,40(13):7660-7662.
[31] 彭士明,施兆鸿,侯俊利.基于线粒体D-loop区与COⅠ基因序列比较分析养殖与野生银鲳群体遗传多样性[J].水产学报,2010,1(34):19-25.
PENG S M,SHI Z H,HOU J L.Comparative analysis on the genetic of cultured and wild silver pomfret populations based on mtD-loop andCOⅠgene[J].Journal of Fisheries of China,2010,1(34):19-25.
Genetic diversity of Eriocheir sinensis in released and w ild populations based on M itochondrial Cytochrome Oxidase SubunitⅠsequence from the Yangtze Estuary
PENG Xin-yue1,2,ZHAO Feng1,ZHANG Tao1,GENG Zhi1,3,ZHU Mei-gui1,2,ZHUANG Ping1
(1.East China Sea Fisheries Research Institute,Chinese Academy of Fishery Sciences,Shanghai 200090,China;2.College of Fishery and Life Science,ShanghaiOcean University,Shanghai201306,China;3.College of Fishery and Life Science,East China Normal University,Shanghai200062,China)
Chinese Mitten Crab,Eriocheir sinensis,is an important economic crab in China.The crab population has decreased greatly in recent years due to over-exploitation and large-scale water conservation construction in the Yangtze River.To restore the natural resources of Chinese mitten crab in the Yangtze River,measures have been taken by provinces and cities along the Yangtze River,such as taking artificial stockingmeasures.In this study,Mitochondrial Cytochrome Oxidase SubunitⅠ(COⅠ)fragments of released and wildEriocheir sinensisfrom the Yangtze Estuary were amplified via PCR,then PCR productswere purified and sequenced.The population genetic diversity and structure by sequencing themitochondrialCOⅠgene of 77 individuals were analyzed.Results showed that there were 41 variable sites in total 630 sites and 15 haplotypeswere found among 77 individuals of released and wildEriocheir sinensis.Among the total 15 haplotypes,Hap-1 appeared most frequently in released and wildEriocheir sinensisand was shared with two populations.Hap-3,Hap-5,Hap-6,Hap-13 and Hap-14 were unique for the released population while Hap-10,Hap-11,Hap-12,and Hap-15 were unique for thewild.The total genetic diversity indexswere high.Haplotype diversity index(H)was 0.825,nucleotide diversity index(π)was0.004 66,and the average number of nucleotide variation index was2.910.Analyzing separately the genetic diversity of two populations,we found that haplotype diversity and nucleotide diversity index of the wild population were higher than that of the released population.The haplotype diversity index of two populations was 0.833 and 0.810.The nucleotide diversity index of two populations was 0.007 70 and 0.002 11.Analysis ofmolecular variance(AMOVA)showed that the genetic differentiation index(FST)of two populations was 0.034 66(P>0.05),indicating obvious genetic differences among the released and wild individuals,and the genetic variation mainly existed within populations.Index of genetic differentiation(FST)and gene flow(Nm)between released population and wild population were 0.034 66 and 13.93,indicating that there weremore frequent gene flows between two populations(Nm=75.05>1).
Eriocheir sinensis;genetic diversity;Cytochrome Oxidase Subunit I(COⅠ);Yangtze Estuary;stock enhancement
Q 754
A
1004-2490(2016)03-0254-08
2015-09-28
公益性行业(农业)科研专项经费资助(201203065);上海市长江口青草沙水库邻近水域生态修复专项
彭欣悦(1988-),女,硕士研究生。E-mail:xypeng2015@163.com
庄 平,研究员。E-mail:Pzhuang@hotmail.com
