6个不同海域文蛤地理群体的亲缘关系分析
2016-03-04陈爱华吴杨平姚国兴蔡永祥
王 超,陈爱华,曹 奕,吴杨平,张 雨,姚国兴,蔡永祥
(1.上海海洋大学水产与生命学院,上海 201306;2.江苏省海洋水产研究所,南通 226007)
6个不同海域文蛤地理群体的亲缘关系分析
王 超1,2,陈爱华2,曹 奕2,吴杨平2,张 雨2,姚国兴2,蔡永祥2
(1.上海海洋大学水产与生命学院,上海 201306;2.江苏省海洋水产研究所,南通 226007)
为探究中国辽东半岛海域(辽宁群体)、长江口及其两翼海域(江苏群体)、台湾海峡西部海域(福建群体)、珠江口及其两翼海域(广东群体)、北部湾海域(广西群体)以及日本伊势湾海域(三重群体)6个不同海域文蛤(Meretrix meretrix)地理群体的亲缘关系,采用18S rRNA基因、线粒体细胞色素氧化酶c亚基Ⅰ(COⅠ)及16S rRNA基因3种分子标记进行序列测定与分析。测序结果显示,3种基因序列长度分别在1 854、658、596 bp左右;18S rRNA基因碱基组成无偏异,序列较为保守;COⅠ与16S rRNA基因A+T平均含量明显大于G+C含量,符合线粒体基因组成特征。通过MegAlign软件比对6个地理群体文蛤,序列相似百分比分别为99.7%~100.0%(18S)、91.7%~99.8%(COⅠ)、90.2%~99.8%(16S),其中三重文蛤序列差异最大;以文蛤属的帘文蛤(Meretrix lyrata)作外群,采用MEGA 5.03软件中相邻连接法(NJ)构建系统发育树显示,我国沿海5个文蛤群体聚为一枝且与日本三重文蛤分开,自展值分别为67%(18S)、99%(COⅠ)、98%(16S);我国沿海5个文蛤群体中,辽宁、江苏、福建文蛤群体首先聚在一起,其次是广西文蛤群体,最后是广东文蛤群体。研究结果表明,辽宁、江苏、福建文蛤群体同源性较高,亲缘关系最近;广西文蛤群体、广东文蛤群体与我国其它文蛤群体间遗传差异较大,地理遗传分化明显;而日本三重文蛤与我国沿海文蛤可定为文蛤的2个地理亚种。
文蛤;18S rRNA基因;COⅠ基因;16S rRNA基因;系统发育
文蛤(Meretrix meretrix Linnaeus)隶属于软体动物门(Mollusca),瓣鳃纲(Lamellibranchia),帘蛤目(Veneroida),帘蛤科(Veneridae),文蛤属,为广温、广盐性滩涂埋栖型双壳贝类,广泛分布于中国、日本、朝鲜、印度等国家。文蛤肉质鲜美,营养丰富,其生物活性成分具有抗肿瘤、抗氧化、降血脂等功效[1],具有较高的经济价值。目前我国文蛤养殖以野生苗种或近亲繁育苗种为主,随着累代养殖,近交衰退、种质下降等现象明显,导致养殖文蛤生长速度减慢、抗逆力下降。加之,不同海域天然苗种异地移养,促使不同遗传背景文蛤群体基因交流增强,对文蛤增养殖业的健康发展十分不利。因此,文蛤种质资源研究与良种选育工作亟需开展,而从分子水平了解不同海域文蛤的遗传背景,可为以上工作的开展提供科学的理论基础。
目前关于我国文蛤群体间遗传多样性的研究,主要集中在局部地区或少数群体之间[2-5]。而我国海岸线漫长且地形复杂,文蛤在南北沿海均有分布,要想全面了解文蛤种质资源状况,必须扩大研究范围,搜集足够多的文蛤群体。冯建彬等[6]曾采用RAPD技术对我国辽宁、山东、江苏、福建、广东、广西文蛤群体进行遗传分析,为文蛤的种质资源利用提供了参考。但近十年来,我国文蛤增养殖业迅速扩张,异地移养、人工育种等可能对不同地区文蛤的遗传背景产生影响,对当前我国文蛤种质资源状况需重新认识。日本文蛤具有色泽艳丽、生长速度快等优良性状,且与我国文蛤有较高的杂交配合力[7],有望成为我国文蛤良种选育的优势材料之一。因此,本文以我国辽宁丹东、江苏南通、福建长乐、广东珠海、广西北海和日本三重6个地理群体文蛤为研究对象,采用细胞核18S rRNA基因及线粒体COⅠ、16S rRNA基因3种分子标记,希望能够较为全面地了解不同海域文蛤的种质资源状况,通过比较日本三重文蛤与我国沿海文蛤的遗传差异,为我国文蛤良种选育提供理论基础。
1 材料与方法
1.1 样品采集
实验用文蛤为2014年10月采自江苏省文蛤良种场保存的不同地理群体文蛤,采集信息见表1。6个地理群体文蛤分别来自于中国辽东半岛海域辽东丹东(辽宁群体)、长江口及其两翼海域江苏南通(江苏群体)、台湾海峡西部海域福建长乐(福建群体)、珠江口及其两翼海域广东珠海(广东群体)、北部湾海域广西北海(广西群体)及日本伊势湾海域三重(三重群体),具体采集地见图1。各群体均取20 ind个体,壳长约4 cm。采活体至实验室解剖,取闭壳肌组织,保存于95%的酒精中。
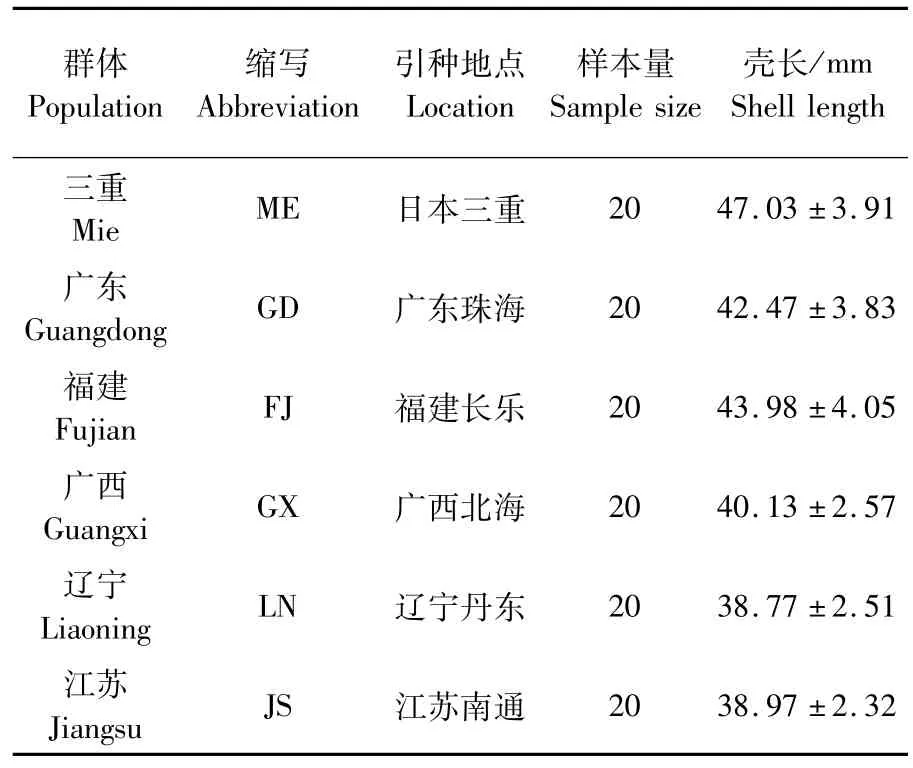
表1 不同地理群体文蛤样品采集信息Tab.1 Information of different populations of M.meretrix

图1 样本采集地理分布图(▲代表采样地点)Fig.1 Geographic distribution of sam pling sites(▲shows a samp ling site)
1.2 DNA提取
取闭壳肌组织,采用传统的酚/氯仿法提取总DNA,-20℃保存备用。
1.3 PCR扩增与产物测序
18S rRNA基因参考程汉良等[8]的方法设计3对引物分别扩增18S rRNA基因全序列的3个基因片段,再拼接为全序列;COⅠ基因采用无脊椎动物通用引物;16S rRNA基因引物参考ANDERSON[9]的序列。引物序列见表2,由生工生物工程(上海)股份有限公司合成。
PCR反应在Eppendorf PCR仪上进行,体系设置25μL。体系组成:模板DNA(50 ng· μL-1)1μL;上下游引物各1μL;10×PCR buffer 2.5μL;dNTP(2.5 mM)2μL;MgCl2(25 mM)2 μL;Taq DNA聚合酶(5 U·μL-1)0.4μL;ddH2O补足25μL。PCR反应条件设定:94℃预变性4 min;94℃变性40 s,58℃(18S)、52℃(COⅠ)、46℃(16S)退火30 s,72℃延伸1 min,35个循环;最后72℃延伸7 min。PCR产物经1%琼脂糖凝胶电泳,EB染色,UV照射下割胶,Sanprep柱式DNA胶回收试剂盒回收纯化。挑选效果理想的PCR纯化产物送铂尚生物技术(上海)有限公司使用扩增引物进行双向测序。
1.4 序列分析
正反向序列用DNAStar Package(version 7.1.0)中的Seqman软件组装,结合测序峰图进行人工校正。18S rRNA基因3个基因片段根据其重叠部分拼接为全序列。最终每个群体均得到18S rRNA、COⅠ、16S rRNA基因3条序列,用Editseq软件编辑和分析,并除去两端的引物部分。通过NCBI的Blast比对,验证所得序列均为目的基因。MegAlign软件比对序列,计算序列相似百分比与差异百分比。MEGA(version 5.03)分析碱基组成,计算变异位点数、简约信息位点数,转换(si)、颠换(sv)位点数及其比率(si/sv),并根据Kimura双参数模型计算遗传距离。以文蛤属(Meretrix)的帘文蛤(Meretrix lyrata)相应序列作外群,GenBank序列号:JN996715.1(18S),JN898944.1(COⅠ),JN969948.1(16S),采用NJ法构建分子系统发育树。
2 结果与分析
2.1 序列比对
对6个群体文蛤基因序列分析显示,18S rRNA基因碱基平均含量分别是T 24.0%、C 24.2%、A 23.7%、G 28.1%、A+T 47.7%(表3)。日本三重文蛤的序列长度为1 849 bp,而我国沿海5个地理群体文蛤的序列长度均为1 854 bp,两者相差5 bp是因为在位点231~235处存在5个碱基的插入/缺失(图2)。由于18S rRNA基因具有较高的保守性,三重文蛤与我国文蛤仅有1个位点发生颠换(G/T),我国文蛤各群体间无变异发生。
COⅠ基因碱基平均含量为T 45.4%、C 13.6%、A 20.6%、G 20.4%、A+T 66.0%(表3)。序列不存在碱基的插入/缺失,长度均为658 bp(图3),共有73个变异位点,其中简约信息位点30个,平均转换位点29个,平均颠换位点2个,比值R=si/sv=13.12。变异位点大多发生于密码子的第三位,由于密码子的简并性,COⅠ基因编码的219个氨基酸中只有3个位点发生变异。
16S rRNA基因碱基平均含量T 37.0%、C 12.0%、A 28.9%、G 22.2%、A+T 65.9%(表3)。序列共出现15个碱基的插入/缺失,分布于250~360之间的7个位点,三重文蛤、广东文蛤、福建文蛤、广西文蛤、辽宁文蛤、江苏文蛤的序列长度依次为597、593、595、596、596、596 bp(图4),共有变异位点71个,简约信息位点11个,平均转换位点20个,平均颠换位点7个,比值R=si/sv=2.94。

表2 引物序列Tab.2 Primer sequences

表3 不同地理群体文蛤18S rRNA、16S rRNA和COⅠ基因片段碱基组成百分比Tab.3 The base com position(%)of 18S rRNA,16S rRNA and COⅠgene fragments in different populations of M.meretrix (%)

图2 不同地理群体文蛤18S rRNA基因序列比对Fig.2 Sequence alignment of partial 18S rRNA gene fragment in different populations of M.meretrix
用MegAlign软件采用Jotun Hein方法对6个文蛤群体的18S rRNA基因、COⅠ基因及16S rRNA基因序列分别进行比对,序列相似百分比与差异百分比见表4。18S rRNA基因序列三重文蛤与我国沿海文蛤差异百分比为0.3%,我国各群体间相似百分比均达100.0%。COⅠ基因序列6个文蛤群体相似百分比91.7%~99.8%,三重文蛤与我国文蛤差异均达8.0%及以上,我国辽宁、江苏、福建文蛤群体之间序列相似度较高,与广西、广东文蛤群体差异相对较大。16S rRNA基因序列6个群体相似百分比90.2%~99.8%,三重文蛤与我国文蛤差异度水平均达10.0%以上,序列相似度也是辽宁、江苏、福建文蛤群体较高,其次是广西、广东文蛤群体。3种基因序列分析结果较为一致,我国辽宁、江苏、福建文蛤群体同源性较高,与广西、广东文蛤之间差异明显,三重文蛤与我国文蛤间基因序列差异最大。

图3 不同地理群体文蛤COⅠ基因序列比对Fig.3 Sequence alignment of partial COⅠgene fragment in different populations of M.meretrix

表4 基于Jotun Hein法计算6个地理群体文蛤18S rRNA基因、COⅠ基因、16S rRNA基因序列相似百分比与差异百分比Tab.4 Percentage of sequence identity and divergence of 18S rRNA,16S rRNA and COⅠgene in different populations of M.meretrix by Jotun Hein method (%)
用MegAlign软件采用Jotun Hein方法对6个文蛤群体的18S rRNA基因、COⅠ基因及16S rRNA基因序列分别进行比对,序列相似百分比与差异百分比见表4。18S rRNA基因序列三重文蛤与我国沿海文蛤差异百分比为0.3%,我国各群体间相似百分比均达100.0%。COⅠ基因序列6个文蛤群体相似百分比91.7%~99.8%,三重文蛤与我国文蛤差异均达8.0%及以上,我国辽宁、江苏、福建文蛤群体之间序列相似度较高,与广西、广东文蛤群体差异相对较大。16S rRNA基因序列6个群体相似百分比90.2%~99.8%,三重文蛤与我国文蛤差异度水平均达10.0%以上,序列相似度也是辽宁、江苏、福建文蛤群体较高,其次是广西、广东文蛤群体。3种基因序列分析结果较为一致,我国辽宁、江苏、福建文蛤群体同源性较高,与广西、广东文蛤群体之间差异明显,三重文蛤与我国文蛤间基因序列差异最大。
2.2 遗传距离与系统发育
用MEGA软件根据Kimura 2-parametermodel计算包括外群在内的遗传距离(D),用Bootstrap方法(1 000 replications)计算标准误差。由18S rRNA基因计算得出的遗传距离中(表5),三重文蛤与我国文蛤的遗传距离D=0.001,与帘文蛤之间D=0.007;我国文蛤群体间遗传距离为0,与帘文蛤之间D=0.008。
有研究表明,帘蛤科贝类DNA条形码间隙区为0.040~0.100[10]。本研究根据COⅠ基因得出的遗传距离中(表6),三重文蛤与我国江苏(0.084)、广西(0.091)、辽宁(0.080)、福建(0.082)、广东(0.087)文蛤群体遗传距离处于间隙区内且靠近0.100水平,已接近属内种间差异;我国江苏、广西、福建、辽宁文蛤群体之间遗传距离0.006~0.028,皆小于条形码间隙0.040水平,即属于种内差异;广东文蛤群体与江苏、辽宁、福建文蛤群体遗传距离(0.066)处于间隙区且靠近0.040,与广西文蛤群体距离0.036,可视为种内差异。
16S rRNA基因中(表7),三重文蛤与我国文蛤遗传距离为0.092~0.098;我国辽宁、江苏、福建、广西文蛤群体遗传距离较小(0.003~0.016),与广东文蛤群体遗传距离为0.039~0.054。THORP[11]提出群体间16S rRNA基因的遗传距离是0.030~0.200,可见6个文蛤群体遗传差异并没有达到种间水平,辽宁、江苏、福建文蛤群体甚至低于群体间水平。

图4 不同地理群体文蛤16S rRNA基因序列比对Fig.4 Sequence alignment of partial 16S rRNA gene fragment in different populations of M.meretrix

表5 6个地理群体文蛤及外群18S rRNA基因序列之间的遗传距离和标准误差Tab.5 Genetic distances(D)and corresponding standard errors of 18S rRNA gene among six populations of M.meretrix and outgroup

表6 6个地理群体文蛤及外群COⅠ基因序列之间的遗传距离和标准误差Tab.6 Genetic distances(D)and corresponding standard errors of COⅠgene among six populations of M.meretrix and outgroup

表7 6个地理群体文蛤及外群16S rRNA基因序列之间的遗传距离和标准误差Tab.7 Genetic distances(D)and corresponding standard errors of 16S rRNA gene am ong six populations of M.meretrix and outgroup
以帘文蛤作外群,采用相邻连接法(NJ法)分别构建不同海域文蛤18S rRNA基因、COⅠ基因、16S rRNA基因序列的系统发育树,节点数值表示Bootstrap 1 000次检验自展值。18S rRNA基因序列NJ树拓扑结构显示(图5),6个地理群体文蛤首先聚为一枝与外群分开;文蛤种群中,我国沿海文蛤聚为一枝与三重文蛤分开,得到67%的支持率。COⅠ基因与16S rRNA基因序列的NJ树拓扑结构一致(图6-7),首先是6个文蛤种群聚为一枝与外群分开;其次我国沿海文蛤聚为一枝与三重文蛤分开,自展值分别高达99%(COⅠ)与98%(16S);我国不同地区文蛤中,辽宁、江苏、福建文蛤群体亲缘关系较近聚在一起,自展值分别是65%(COⅠ)与97%(16S),其次是广西文蛤群体,最后是广东文蛤群体。

图5 基于18S rRNA基因序列的不同地理群体文蛤NJ树(K imura 2-parameter model,1 000 replications)Fig.5 NJ phylogenetic tree based on 18S rRNA gene of different populations of M.meretrix

图6 基于COⅠ基因序列的不同地理群体文蛤NJ树(K imura 2-parameter model,1 000 replications)Fig.6 NJ phylogenetic tree based on COⅠgene of different populations of M.meretrix
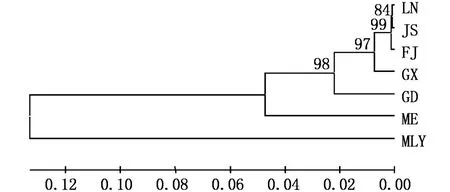
图7 基于16S rRNA基因序列的不同地理群体文蛤NJ树(K imura 2-parameter model,1 000 replications)Fig.7 NJ phylogenetic tree based on 16S rRNA gene of different populations of M.meretrix
2.3 不同文蛤群体的亲缘关系
由以上分析可知,我国沿海辽宁、江苏、福建文蛤群体遗传分化低于地理群体间变异水平,亲缘关系最近;广西文蛤群体、广东文蛤群体与我国其它文蛤群体间遗传差异较大,地理遗传分化明显;日本三重文蛤与我国文蛤之间3种基因序列均有较大差异,按照帘蛤科贝类0.040~0.100条形码间隙以及16S rRNA基因群体间0.030~0.200的遗传距离,三重文蛤与我国文蛤之间超出种内分化但未达到种间水平,可认为二者达到地理亚种分化,3种基因序列拓扑结构均把我国文蛤群体聚为一枝与三重文蛤分开,且可信度较高,也支持把三重文蛤与我国文蛤定为文蛤的2个地理亚种。
3 讨论
3.1 3种分子标记相结合应用于文蛤种群的亲缘关系分析
18S rRNA基因在生物体内含量较大且在进化过程中具有保守性,适合不同层次系统发育的研究;COⅠ基因是线粒体氧化呼吸链的重要成员,基因变异较大;16S rRNA基因是非编码蛋白质基因,不受密码子编码的选择压力影响,进化速度适中。本研究结合18S rRNA基因、COⅠ与16S rRNA基因分析不同地理群体文蛤的遗传差异,利用3种基因序列不同的保守性,在聚类分析中得到了一致的拓扑结构,能够有效反映不同地理群体文蛤的亲缘关系。根据本文研究,18S rRNA基因碱基平均含量分别是T 24.0%、C 24.2%、A 23.7%、G 28.1%,4种碱基基本无偏异,与程汉良等[8]发表的帘蛤科贝类碱基组成一致;COⅠ与16S rRNA基因碱基A+T含量明显高于G+C含量,符合线粒体基因组成特征。COⅠ基因共有73个变异位点,其中简约信息位点30个,16S rRNA基因有71个变异位点,简约信息位点11个,表明16S rRNA基因比COⅠ基因序列保守,与头足纲(Cephalopoda)[12]、鲍科(Haliotidae)[13]等多个研究结果一致。
3.2 日本三重文蛤与我国沿海文蛤之间遗传分化已达到亚种水平
DNA条形码是HEBERT等[14]提出的一种基于COⅠ基因进行物种鉴定的分子技术。随着研究的深入,HEBERT等[15]、BURNS等[16]提出以条形码间隙(种内基因序列的最大遗传距离与种间基因序列的最小遗传距离之间的空隙)界定物种。陈军[10]在帘蛤科贝类的DNA条形码研究中,把0.040~0.100定义为帘蛤科的条形码间隙,据此将315个样品中的289个成功鉴定到种,并在BOLD数据库中验证了此间隙区的存在。因此,本研究以0.040~0.100的条形码间隙探讨不同群体文蛤的亲缘关系是合理的。
在以往的研究中,DNA序列差异通常也被用来鉴定物种。有研究表明,马蹄螺科(Trochidae)属内种间COⅠ基因序列差异为5.7%~12.1%[17];珍珠蚌科(Margaritiferidae)属内种间COⅠ基因序列差异11.57%~12.33%[18]。帘蛤科贝类的研究中,波纹巴非蛤(P.undulata)与织锦巴非蛤(P.textile)16S rRNA基因序列差异7.8%~8.0%,薄片镜蛤(D.corrugate)与薄壳镜文蛤(D.angulosa)序列差异为10.7%,两者都被认为是相互独立的种,防城港文蛤与其它地理群体文蛤COⅠ基因序列差异达到6.6%,被研究者认为具有两个地理亚种的可能性[19]。本研究中日本三重文蛤与我国沿海文蛤COⅠ基因序列平均差异8.4%,16S rRNA基因序列平均差异10.4%,已接近种间分化水平。在聚类分析中,COⅠ基因与16S rRNA基因具有高度一致性,我国5个地理群体文蛤聚在一起,可信度达到99%(COⅠ)、98%(16S),三重文蛤则单独形成一个分枝,即使在保守性较高的18S rRNA基因聚类中,三重文蛤也单独形成一枝与我国沿海文蛤分开。
综上,作者认为可将三重文蛤与我国沿海文蛤定为文蛤的2个地理亚种。吴杨平等[20]基于形态分析研究认为,日本三重文蛤与丽文蛤(Meretrix cusoria)相近,应为丽文蛤,并与福建长乐丽文蛤达到亚种水平。而潘宝平等[21]、CHEN等[22]、程汉良等[19]的研究则支持将丽文蛤订为文蛤的同物异名或地理亚种的观点。因此,本文将日本三重文蛤与我国沿海文蛤定为文蛤的2个地理亚种的结论与上述研究有一定的相似之处。
3.3 我国文蛤种质资源的现状与保护
冯建彬等[6]的研究结果显示,辽宁、山东、江苏文蛤群体亲缘关系较近,其次是福建、广西、广东群体。而在本研究中,福建文蛤群体与辽宁、江苏文蛤群体聚在一起,其次是广西、广东群体,可见福建文蛤群体与辽宁、江苏文蛤群体间亲缘关系增近,我国其它文蛤群体间亲缘关系并未发生较大改变。福建与辽宁、江苏文蛤群体可能是由于近些年异地移养、苗种引入等造成了种质混杂,其它文蛤群体种质保护较好。日本三重县(伊势湾)与我国存在地理隔离,两地气候及海水理化因子差异较大,长期的自然选择造成三重文蛤与我国文蛤达到亚种分化。为保证我国文蛤种质资源的可持续利用及增养殖业的健康发展,必须加强实施保护与改良措施,如:文蛤的增养殖中应避免近亲繁殖,减少累代养殖;合理引种,文蛤异地移养要严格监管,避免对当地自然种群的基因污染;开展文蛤种质资源调查工作,对未受污染的种群建立种质资源保护区,对野生种已经大量减少的海域进行保种及人工增殖。
[1] 杜正彩,侯小涛,黄 庆,等.文蛤化学成分与药理作用研究进展[J].安徽农业科学,2014,42(2):439-441.
DU Z C,HOU X T,HUANG Q,et al.Research progress of chemical composition and pharmacological activity ofMeretrix meretrix[J].Journal of Anhui Agricultural Sciences,2014,42(2):439-441.
[2] 杜晓东,邓岳文,叶富良,等.广东和广西地区野生文蛤的遗传多样性[J].中国水产科学,2004,11(1):41-47.
DU X D,DENG Y W,YE F L,et al.Genetic diversity of seven wild populations ofMeretrix meretrix[J].Journal of Fishery Sciences of China,2004,11(1):41-47.
[3] 刘 馨,孙祥山,高悦勉.文蛤北方种群生化遗传结构与变异的研究[J].水产科学,2006,25(4):179-183.
LIU X,SUN X S,GAO Y M.Genetic Structure and Variation inMeretrix meretrixfrom Northern China[J].Fisheries Science,2006,25(4):179-183.
[4] 林志华,董迎辉,李 宁,等.基于形态参数和AFLP标记的文蛤(Meretrix meretrix)不同地理群体遗传变异分析[J].海洋与湖沼,2008,39(3):245-251.
LIN Z H,DONG Y H,LIN,et al.The genetic structure and diversity analysis of different geographical populations ofMeretrix meretrixusing morphological parameters and AFLP markers[J].Oceanologia Et Limnologia Sinica,2008,39(3):245-251.
[5] 董迎辉,姚定余,林志华,等.浙江和广西两种文蛤的分子鉴定及形态特征分析[J].水产学报,2011,35(10):1505-1502.
DONG Y H,YAO D Y,LIN Z H,et al.Molecular classification and morphological traits of two species ofMeretrix(Mollusca,Bivalvia)[J].Journal of Fisheries of China,2011,35(10):1505-1502.
[6] 冯建彬,李家乐,王美珍,等.我国沿海不同群体文蛤遗传差异RAPD分析[J].海洋渔业,2005,27(4):281-285.
FENG J B,LI J L,WANG M Z,et al.RAPD analysis of genetic variations among the seven populations ofMeretrix meretrixin China’s coastal zone[J].Marine Fisheries,2005,27(4):281-285.
[7] 吴杨平,陈爱华,姚国兴,等.3个不同地理群体红壳色文蛤杂交的配合力分析[J].海洋渔业,2014,36(4):314-319.
WU Y P,CHEN A H,YAO G X,et al.Combining ability analysis on diallel cross from three different redMeretrix meretrixpopulations[J].Marine Fisheries,2014,36(4):314-319.
[8] 程汉良,彭永新,王 芳,等.6种帘蛤科贝类18S rRNA基因全序列比较分析[J].中国水产科学,2008,15(4):559-567.
CHENG H L,PENG Y X,WANG F,et al.Sequence analysis of 18S rRNA gene of six Veneridae clams(Mollusca:Bivalvia)[J].Journal of Fishery Sciences of China,2008,15(4):559-567.
[9] ANDERSON FE.Phylogeny and historical biogeography of the Loliginid squids(Mollusca:Cephyalopoda)based on mitochondrial DNA sequence data[J].Molecular Phylogenetics and Evolution,2000,15(2):191-214.
[10] 陈 军.帘蛤科贝类分子系统学研究[D].青岛:中国海洋大学博士学位论文,2012.
CHEN J.Molecular Systematics of Veneridae(Bivalvia,Mollusca)[D].Qingdao:Doctoral Dissertation of Ocean University of China,2012.
[11] THORP J P.The molecular dock hypothesis:Biochemical evaluation,genetic differentiation and systematics[J].Annual Review of Ecology and Systematics,1982,13(1):139-168.
[12] LIN X Z,ZHENG X D,XIAO S,et al.Phylogeny of the cuttlefishes(Mollusca:Cephalopoda)based on mitochondrialCOⅠand 16S rRNA gene sequence data[J].Acta Oceanologica Sinica,2004,23(4):699-707.
[13] AN H S,JEE Y J,MIN K S,et al.Phylogenetic analysis of six species of Pacific abalone(Haliotidae)based on DNA sequences of16S rRNA and cytochrome oxidase subunit I mitochondrial genes[J].Marine Biotechnology,2005,7(4):373 -380.
[14] HEBERT PD N,CYWINSKA A,BALL SL,etal.Biological identifications through DNA barcodes[J].Proceedings of the Royal Society of London B,2003(270):313-322.
[15] HEBERT PD N,RATNASINGHAM S,DEWAARD J R.Barcoding animallife:cytochrome c oxidase subunit I divergences among closely related species[J].Proceedings of the Royal Society of London B,2004(270):S96-S99.
[16] BURNS JM,JANZEN D H,HAJIBABAEIM,et al.DNA barcodes and cryptic species of skipper butterflies in the genus Perichares in Area de Conservacion Guanacaste,Costa Rica[J].Proceedings of the National Academy of Sciences of the United States of America,2008,105(17):6350-6355.
[17] DONALD K M,KENNEDY M,SPENCER H G.The phylogeny and taxonomy of australmonodontine topshells(Mollusca:Gastropoda:Trochidae),inferred from DNA sequences[J].Molecular Phylogenetics and Evolution,2005,37(2):474-483.
[18] MACHORDOM A,ARAUJO R,ERPENBECK D,et al.Phylogeography and conservation genetics of endangered European Margaritiferidae(Bivalvia:Unionoidea)[J].Biological Journal of the Linnean Society,2003,78(2):235-252.
[19] 程汉良,周旻纯,陈冬勤,等.基于16S rDNA序列的帘蛤科贝类分子系统发育研究[J].水产科学,2012,31(11):657-662.
CHENG H L,ZHOU M C,CHEN D Q,et al.Phylogenetic Analysis of Veneridae(Mollusca:Bivalvia)based on Mitochondrial 16S rDNA[J].Fisheries Science,2012,31(11):657-662.
[20] 吴杨平,姚国兴,陈爱华,等.文蛤属2种贝类多变量形态分析及日本文蛤的物种有效性[J].水产学报,2011,35(9):1410-1417.
WU Y P,YAOG X,CHEN A H,etal.Multivariate morphometric analysis of two species ofMeretrix,withnotes on JapaneseMeretrix lusoria’s validity[J].Journal of Fisheries of China,2011,35(9):1410-1417.
[21] 潘宝平,吴琪,张素萍,等.文蛤属(Meretrix)16S rRNA基因及ITS1序列的系统学分析[J].海洋与湖沼,2006,37(4):342-347.
PAN B P,WU Q,ZHANG S P,et al.Molecular phylogeny ofMeretrix(mollusca,bivalvia)based on 16s rRNA genes and ITS1 sequences[J].Oceanologia Et Limnologia Sinica,2006,37(4):342-347.
[22] CHEN A H,LI Z X,FENG G N.Phylogcnctic relationships of the genusMeretrix(Mollusca:Veneridae)based on mitochondrialCOⅠgene sequences[J].Zoological Research,2009,30(3):233-239.
Genetic relationship analysis of six Meretrix meretrix populations from different sea areas
WANG Chao1,2,CHEN Ai-hua2,CAO Yi2,WU Yang-ping2,ZHANG Yu2,YAO Guo-xing2,CAIYong-xiang2
(1.College of Fisheries and Life Science,Shanghai Ocean University,Shanghai201306;2.Jiangsu Marine Fisheries Research Institute,Nantong226007)
In this paper,18S ribosomal RNA gene,mitochondrial cytochrome oxidase c subunit I(COⅠ)gene and 16S rRNA gene sequences of six differentMeretrixmeretrixpopulations from Liaodong Peninsula sea area(Liaoning population),the Yangtze Estuary and its twowing sea areas(Jiangsu population),Western sea area of Taiwan Strait(Fujian population),Pearl River Estuary and its two wing sea areas(Guangdong population),Beibu Bay sea area(Guangxi population)and Ise Bay sea area,Japan(Mie population)were sequenced and analyzed to study their genetic relationship.The genetic background of differentMeretrix meretrixpopulations was analyzed in molecular level,to provide theoretical basis for germplasm resources research and breeding.Results of sequence analysis showed that:the length of three genetic sequences was about 1 854 bp,658 bp and 596 bp respectively;base composition of18S rRNA genewas unbiased,and its sequence was conservative;COⅠgene had 73 variable sites,among which 30 were parsim-informative sites,the average content of A+T was 66.0%;16S rRNA gene had 71 variable sites,among which 11 were parsim-informative sites,the average contentof A+Twas65.9%.A+T contentwas obviously higher than G+C content in bothCOⅠand 16S rRNA gene,which was in accordance with features of mitochondrial genes.Gene sequences of sixMeretrix meretrixpopulations were aligned by MegAlign,the percentage of sequence identity was 99.7%-100.0%(18S),91.7%-99.8%(COⅠ),90.2%-99.8%(16S)respectively.Meretrix meretrixfrom Mie had the largest difference.Phylogenetic trees were constructed by MEGA5.03 using adjacent connection method(NJ)with Meretrix lyrata as outgroup.Five Meretrix meretrix populations from China coast areas were clustered into one clade and separated fromMeretrix meretrixfrom Mie,Japan,bootstrapping value was 67%(18S),99%(COⅠ)and 98%(16S)respectively;among the five populations from China coast areas,Meretrix meretrixfrom Liaoning,Jiangsu,Fujian clustered together first,and then the population from Guangxi,the last was the population from Guangdong.Results indicated that:Meretrixmeretrixfrom Liaoning,Jiangsu,Fujian had a higher homology and their genetic relationship was the closest;a larger genetic difference was found amongMeretrixmeretrixfrom Guangxi,Guangdong and other populations from China,and their geographic genetic differentiation was obvious;Meretrixmeretrixfrom Mie,Japan and China coast areas could be described as two geographic subspecies.
Meretrixmeretrix;18S rRNA gene;COⅠgene;16S rRNA gene;phylogeny
Q 951
A
1004-2490(2016)03-0262-11
2015-09-12
江苏省科技厅重点研发项目(BE2015324);江苏省水产三新工程重大项目(D2014-16);江苏省属公益类科研院所能力提升项目(BM2015017);江苏省水产良种保种和亲本更新项目(BZ2014,2015);南通市农业科技创新项目(HL2014007)
王 超(1990-),男,河南信阳人,硕士研究生,主要从事贝类增养殖学研究。
E-mail:wangchao7198@163.com
陈爱华,研究员。Tel:0513-85228272,E-mail:chenah540540@aliyun.com
