利用CRISPR/Cas9技术敲除斑马鱼ifi30基因
2016-02-07曹顶臣佟广香吕伟华孙志鹏匡友谊郑先虎孙效文
曹顶臣,佟广香,吕伟华,孙志鹏,匡友谊,郑先虎,孙效文
(中国水产科学研究院黑龙江水产研究所,黑龙江 哈尔滨 150070)
利用CRISPR/Cas9技术敲除斑马鱼ifi30基因
曹顶臣,佟广香,吕伟华,孙志鹏,匡友谊,郑先虎,孙效文
(中国水产科学研究院黑龙江水产研究所,黑龙江 哈尔滨 150070)
利用CRISPR/Cas9技术,在斑马鱼Danio rerio干扰素γ诱导蛋白30(ifi30)基因的第一个外显子处选取靶位点,用PCR法构建gRNA,并进行体外转录。将体外转录的sgRNA和Cas9mRNA显微注射到斑马鱼1细胞期的受精卵中,实现对靶基因ifi30的沉默。注射后24h检测发现,基因突变率在79%~95%之间,平均突变率为87.2%。为了获得稳定遗传的基因突变纯合系,将F0代与野生型斑马鱼进行配组,获得了3种突变类型的F1代,其突变率为30%。三种突变类型中突变1和突变2为移码突变,突变3删除了6个碱基,并未造成移码。突变1和突变2的F1代杂合个体生长发育未见异常。将杂合F1代自交,理论上将获得突变纯合个体,但仅在受精2h以内的胚胎中检测到了突变纯合个体,受精24h后的胚胎中,只有野生型和杂合型,未见突变纯合个体。推测ifi30基因的移码突变造成了干扰素γ诱导蛋白的缺失,导致胚胎死亡。
斑马鱼;ifi30;CRISPR/Cas9技术;敲除
斑马鱼Danio rerio个体小、发育快、易于饲养、性成熟周期短、繁殖力强、体外受精、胚胎在母体外发育,且受精卵透明、易于操作,已成为常用的动物模型[1],越来越多地被用于免疫相关研究中。干扰素γ诱导蛋白30(ifi30)作为一种可溶性的蛋白质,能够在催化二硫键的还原,参与抗原呈递和加工以及对机体病毒免疫、肿瘤免疫、寄生虫抗原免疫应答产生影响,ifi30的缺失可能会对机体病毒免疫[2,3]、肿瘤免疫[4,5]、寄生虫的抗原免疫应答造成影响,还可能引起自身免疫病的发生和发展[6]。因此,研究ifi30基因突变对斑马鱼的影响,具有重要的意义。
CRISPR/Cas9技术[7-9]简单、易行,作为一种备受瞩目的基因组定向编辑技术,在人类细胞、小鼠和斑马鱼等物种中成功实现了基因组的定点修饰,并且具有较高的突变率[10-13]。本研究利用CRISPR/ Cas9技术编辑斑马鱼ifi30基因,定点敲除特定位点的序列,以获得稳定遗传的ifi30基因缺失的纯合个体,为研究该基因在免疫中的功能提供材料。
1 材料与方法
1.1 材料
试验用AB品系斑马鱼,来自中国水产科学研究院黑龙江水产研究所自动循环水系统中。光暗周期比率为14h∶10h(14 Light∶10 Dark)。按雌雄3∶2的比例将性成熟、健康的斑马鱼放入产卵盒内,用挡板将雌雄鱼分开。次日注射前将挡板取出,使其完成产卵与受精。
1.2 基因靶位点设计
本试验采用特定的限制性内切酶检测突变,利用ChopChop设计ifi30的靶位点、PCR检测引物和内切酶。ifi30基因具有7个外显子,在第一个外显子上选择GGACATAGCAGCTGAATGT为靶位点。采用PCR扩增质粒的方法获得gRNA序列,在靶位点前增加上T7启动子序列,后增加上gRNA序列,获得的gRNA引物和检测PCR引物见表1。Chop-Chop设计的ifi30检测内切酶为Pvu II(NEB公司),该酶在相应PCR扩增片段中有一个酶切位点。酶切后,ifi30野生型(+/+)有2条带,分别为294bp和147bp;纯合突变型(-/-)仅1条带,为441bp;杂合型(+/+)有3条带,包括了野生型条带和突变型条带,分别是294bp、47bp和441bp。
1.3 PCR扩增获得gRNA
用引物ifi30和PT7reverse组合扩增,加入2ng的pT7-gRNA质粒(Addgeng网站购买)为模板。扩增程序为:95℃,3min;95℃,30s;58℃,30s;72℃,30s;35个循环,72℃,5min。用2%琼脂糖凝胶检测PCR产物,产物约120bp。检测后用PCR产物纯化试剂盒(Axygen)纯化回收PCR产物,测定浓度,作为gRNA待用。
1.4 gRNA和Cas9体外转录
gRNA用MEGAshortscriptTMT7 High Yield Transcription Kit体外转录;Cas9表达质粒pCS2-nCas9n(Addgeng网站购买)用内切酶Not I(NEB公司)线性化,回收后用mMESSAGE mMACHINE SP6体外转录,转录结束后,用DNase去除DNA残留,试剂盒回收RNA,检测RNA的浓度和纯度,-80℃冰箱保存待用。
1.5 显微注射及检测
体外转录的sgRNA和Cas9 mRNA混合后加入25%的酚红后,显微注射到单细胞期的斑马鱼胚胎中。sgRNA和 Cas9 mRNA的注射量分别为200pg/μL和300pg/μL,对照组为等量的25%的酚红。注射后24~48h之间检测突变率。在发育良好的胚胎中加入40mL裂解液[10mMTris(1mol/L)、pH8.0,50mM KCl(3mol/L)、0.3%的 Tween20和 NP40、0.5mg/mL的蛋白酶K],55℃裂解2h,98℃10min。取出后震荡混匀,离心,取上清液为模板。用PCR检测引物扩增,20μL PCR扩增体系,程序同gRNA的PCR扩增。酶切鉴定PCR产物。将获得的突变进行TA克隆,检测变异克隆,测序,获得突变序列。
1.6 变异检测
注射的斑马鱼与野生型斑马鱼交配获得F1代,筛选出F1突变个体,突变个体自交获得F2。F2代中理论上包含纯合突变个体、杂合个体和野生型。F2代突变纯合个体群交,获得稳定遗传的突变系(图1)。
2 结果与分析
2.1 gRNA靶位点的选择及载体的构建

表1 引物序列Tab.1 The sequences of primer used in the experiment

图1 交配策略Fig.1 Mateing tactics in various types of zebrafish
通过ChopChop系统搜索和确定gRNA的靶位点。输入ifi30基因外显子序列获得一系列关于sgRNA识别靶位点,选择ifi30基因第一个外显子上的GGACATAGCAGCTGAATGT为靶位点。合成ifi30和PT7 reverse引物(引物序列见表1),以pT7-gRNA质粒为模板进行PCR扩增,获得l 120bp左右的产物片段(图2)。

图2 gRNA的电泳图谱(M:DL2000)Fig.2 Electrophoresis patterns of gRNA(Marker:DL2000)
2.2 突变效率计算
显微注射24h后,选择发育良好的胚胎进行裂解提取DNA,每组检测50枚胚胎,共计5组,分别用引物ifi30F和ifi30R进行PCR扩增,酶切检测突变率。结果表明:1~5组的突变效率分别为89%、83%、95%、90%和79%,平均突变率为87.2%。
2.3 F1代突变个体获得
将注射的斑马鱼F0代饲养至性成熟,与野生型(AB系)斑马鱼进行1∶1配组,产生F1子代,共配10组。受精24h后,提取受精卵DNA进行PCR后酶切,检测突变情况。10组F1代斑马鱼中,有3组检测到突变,突变率为30%。图3为其中一组突变检测结果,图中1号和3号样本为三条带,属于杂合型突变,说明DNA序列发生了变异,改变了酶的识别位点。将突变个体DNA进行PCR扩增,回收产物进行TA克隆测序。图4为突变个体1的测序结果,与参照序列对比,在箭头位置缺失了5个碱基CTGAA。图5显示了3个突变体的突变位点缺失情况。由图5可知,F1代ifi30基因序列产生了不同程度的缺失。突变个体1删除了5个碱基CTGAA,突变个体2删除了4个碱基TGAA,突变个体3删除了6个碱基TGAATG。
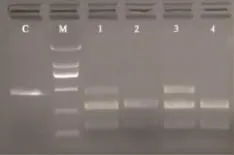
图3 F1代突变电泳图Fig.3 Electrophoresis patterns of F1mutations
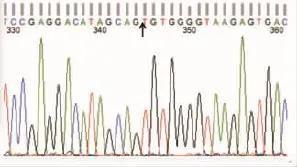
图4 突变1的测序图(箭头代表缺失位点的位置)Fig.4 The sequence diagram of mutation 1(the arrow showing the missing position)
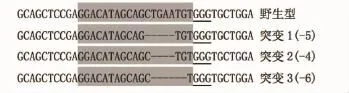
图5 F1代ifi30基因位点的突变Fig.5 Mutations of F1at ifi30 gene target sites
突变1和突变2变异的碱基数不是3的整倍数,理论上能够在蛋白翻译过程中造成移码,生成新的蛋白。将突变1和突变2饲养至性成熟,分别自交,获得2组自交F2代个体,F2代个体中理论上包含纯合突变个体(-/-),杂合个体(+/-)和野生型(+/+)。取受精后2h和24h的成活卵检测2组F2代受精卵的突变,每次每组检测50个样本。检测发现,受精2h的样本包含10个纯合突变个体(-/-)、28个杂合突变个体(+/-)和12个野生型个体(+/+),基本符合孟德尔遗传定律;受精24h的样本包括0个纯合突变个体(-/-)、33个杂合突变个体(+/-)和17个野生型个体(+/+)(图6和图7)。受精2h检测,样本4、7、8和12仅含1条带,与对照带基本一致,为纯合突变个体(-/-),而在受精24h没有纯合突变个体(-/-),说明实验成功地沉默了ifi30基因;ifi30是胚胎发育中的一个关键基因,ifi30基因突变,使胚胎死亡。
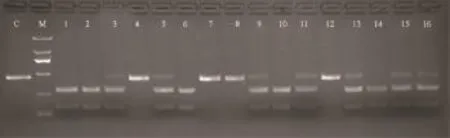
图6 受精2h样本突变的检测结果Fig.6 The mutations in 2 h post-fertilization

图7 受精24h样本突变的检测结果Fig.7 The mutations in 24 h post-fertilization
3 讨论
基因组靶向编辑技术能使生物基因组DNA中的特定位点发生改变[13-15],是研究基因功能的重要工具,用人工核酸酶可以提高基因编辑效率。目前常见的基因敲除系统有3种:ZFNs、TALENs和CRISPR/Cas9。ZFNs和TALENs对每个新的DNA靶点必须要经过一个再加工过程,费时费力[16,17]。与ZFNs和TALENs相比,CRISPR/Cas9更易于操作,效率更高,更容易得到纯合子突变体,可以在不同位点同时引入多个突变,仅需对gRNA进行编辑,是目前应用最广泛的基因编辑技术,已成功应用于微生物、动植物等诸多物种的基因组改造[8,9]。CRISPR/Cas9系统介导的基因突变具有较高的突变率,本试验检测了5组注射后的样本,最高突变率的为95%,最低为79%,平均为87.2%,表现出较高的突变效率。性成熟F0代与野生型AB配组的突变率为30%。注射后,检测发现,F0代样本突变率高于F0与野生型配组产生的F1代的突变率,这是由于注射后,在CRISPR/Cas9系统的作用下,基因组发生了编辑。注射一般在1细胞期,但已经有部分细胞已经发育,每个胚胎的注入位置也不同,在细胞内能发生基因编辑的位置也不确定,因此只能得到F0代嵌合体。注射24h采用的是整个胚胎,包含了所有组织的变异,具有较高的突变率。而为了获得纯合系,要选择生殖细胞发生突变,且能够稳定遗传的突变体。本研究发现,突变个体也是仅有部分卵细胞发生突变,因此F0代与野生型AB配对的突变率要远低于注射后检测的突变率。
本研究用CRISPR/Cas9系统编辑ifi30基因。ifi30基因编码干扰素γ诱导蛋白30,位于斑马鱼2号染色体上,具有7个外显子,全长1 057bp。靶位点起始于mRNA的第211个碱基,位于第一外显子区域。突变1删除的5bp,序列在222~226bp,突变2删除的4bp,序列是223~226bp,均不是3的整数倍。移码突变的结果将引起该段肽链的改变,而肽链的改变将引起蛋白质性质的改变,最终引起性状的变异,严重时会导致个体的死亡。突变1和突变2均能够引起移码突变,且位于第一外显子能更大程度地影响基因的转录和翻译,达到沉默基因的效果。对ifi30基因突变的杂合子观察未发现明显的异常,但ifi30基因突变的纯合子在胚胎发育过程中死亡。这是由于杂合子还存在完整的ifi30基因,能够正常翻译具有功能的干扰素γ诱导蛋白30,因此未见异常。在突变纯合子中,ifi30基因不能被翻译成具有功能的干扰素γ诱导蛋白30,因此胚胎死亡,可见ifi30蛋白是胚胎发育过程中必不可少的一种蛋白,能够参与抗原呈递和加工,影响对机体病毒免疫、肿瘤免疫、寄生虫的抗原免疫应答[4-6]。推测ifi30基因突变的纯合体,造成免疫缺陷,因而在胚胎期发生死亡。为了进一步了解ifi30基因在免疫过程中发挥的作用,今后应通过条件性敲除,研究不同发育阶段和不同组织中ifi30基因的功能。
[1]贾顺姬,孟安明.中国斑马鱼研究发展历程及现状[J].遗传,2012,34(9):1082-1088.
[2]SinnathambyG,Maric M,Cresswell P,et al.Differential requirements for endosomal reduction in the presentation of two H2-Ed-restricted epitopes from influenza hemagglutinin[J].The Journal ofImmunology,2004,172(11):6607-6614.
[3]Sealy R,Chaka W,Surman S,et al.Target peptide sequence within infectious human immunodeficiency virus type 1 does not ensure envelope-specific T-helper cell reactivation:influences of cysteine protease and gamma interferon-induced thiolreductase activities[J].Clinical and Vaccine Immunology,2008,15(4):713-719.
[4]Haque MA,Li P,Jackson S K,et al.Absence of γ-Interferon-inducible lysosomal thiol reductase in melanomas disrupts T cell recognition of select immunodominant epitopes[J].The Journal of experimental medicine,2002,195(10):1267-1277.
[5]Li P,Haque MA and Blum J S.Role of disulfide bonds in regulating antigen processing and epitope selection[J]. Journal ofImmunology,2002,169(5):2444-2450.
[6]Maric M,Barjaktarevic I,Bogunovic B,et al.Cutting edge: developmental up-regulation of IFN-gamma-inducible lysosomalthiol reductase expression leads to reduced T cell sensitivity and less severe autoimmunity[J].Journal of Immunology,2009,182(2):746-750.
[7]Doudna J A and Charpentier E.Genome editing.The new frontier of genome engineering with CRISPR-Cas9[J]. Science,2014,346(6213):1258096.
[8]Gaj T,Gersbach C A and Barbas C F.ZFN,TALEN and CRISPR/Cas-based methods for genome engineering[J]. Trends in Biotechnology,2013,31(7):397-405.
[9]Barrangou R.Cas9 targeting and CRISPR revolution[J]. Science,2014,344(6185):707-708.
[10]Cong L,Ran F A,Cox D,et al.Multiplex genome engineering using CRISPER/Cassystem[J].Science,2013, 339(6121):819-823.
[11]Mali P,Yang L H,Esvwlt K M,et al.RNA-guided human genomeengineeringviaCas9[J].Science,2013,339(6121): 823-826.
[12]Hwang W Y,Fu Y F,Reyon D,et al.Efficient genome editing in zebrafish using a CRISPER-Cassyetem[J]. Nature Biotechnology,2013,31(3):227-229.
[13]Carroll D.Genome engineering with zinc-finger nucleases[J].Genetics,2011,188(4):773-782.
[14]Perez-Pinera P,Ousterout D G and Gersbach C A.Advances in targeted genome editing[J].Current opinion in chemical biology,2012,16(3):268-277.
[15]Urnov F D,Rebar E J,Holmes MC,et al.Genome editing with engineered zinc finger nucleases[J].Nature Reviews Genetics,2010,11(9):636-646.
[16]Geurts A M,Cost G J,Freyvert Y,et al.Knockout rats via embryo microinjection of zinc-finger nucleases[J].Science,2009,325(5939):433-433.
[17]Cermak T,Doyle E L,Christian M,et al.Efficient design and assembly of custom TALEN and other TAL effector-based constructs for DNA targeting[J].Nucleic Acids Research,2011,39(12):e82.
Knock Out of Interferon-gamma-inducible Gene 30 in Zebrafish by CRISPR/Cas9
CAO Ding-chen,TONG Guang-xiang,LV Wei-hua,SUN Zhi-peng,KUANG You-yi,ZHENG Xian-hu,SUN Xiao-wen
(Heilongjiang Fisheries Research Institute,Chinese Academy of Fishery Sciences,Harbin 150070,China)
The interferon-gamma-inducible protein 30(ifi30)gene,as central regulators of immunity in vertebrates,was knocked out in zebrafish using CRISPR/Cas9 to investigate regulation of the immunity by interferons.The small guide RNAs were designed using CHOPCHOP software to target the first exon of ifi30 gene,and synthesized with PCR.After transcribed in vitro,the mixed mRNAs of guide RNAs and Cas9 were micro-injected into fertilized eggs.The restricted enzyme Pvu II was used for screening genetic mutation in 24hpf embryos,and the mutation rate was found to be ranged from 79%to 95%,with mean value of 87.2%.In order to obtain stable genetic homozygous mutant lines,the F0individuals were crossed with wild type zebrafish,and 3 mutant alleles were identified in F1,in which two alleles(named Mut1 and Mut2)caused frameshifts in ifi30 gene,while the other allele did not cause the stop-gained mutant with 6 bp deletion.The heterozygous individuals with Mut1 and Mut2 alleles were normal in growth and development,while the homozygous embryos with Mut1 and Mut2 allele in F2generation did not develop into larva,the homozygous mutant genotypes only found in survival embryos after 24hpf.The findings suggested that the functional deficiency of ifi30 may result from frame-shift caused death of the embryos during development.
zebrafish;ifi30;CRISPR/Cas9;knock-out
S917
A
1005-3832(2016)06-0026-05
2016-10-11
中国水产科学研究院基本科研业务费(2016HY-ZD0301);农业部948项目(2016-X15).
曹顶臣(1973-),男,副研究员,从事鱼类分子育种研究.E-mail:caodingchen@hrfri.ac.cn
孙效文,研究员.E-mail:sunxw2002@163.com
