HDAC9基因SNPs与大动脉粥样硬化缺血性卒中的相关性研究
2016-01-12柳维林林云娇陶静彭军李光谱陈立典
柳维林,林云娇,陶静,彭军,李光谱,4,陈立典
HDAC9基因位于7号染色体q21.1,全长935.47kb,可变剪切产生29种转录产物。HDAC9基因是组蛋白Ⅱ类HDACs家族成员之一,调控染色体结构和基因转录[1]。已知HDAC9基因在肌肉、心脏和脑组织中高表达[2]。近年研究表明,该基因与大动脉粥样硬化性(large vessel atherosclerosis,LVD)缺血性卒中相关联。2012年对欧洲人群缺血性卒中患者进行全基因组关联研究(genome-wide association study,GWAS)发现,HDAC9基因单核苷酸多态性(single nucleotide polymorphisms,SNPs)rs11984041位点是大动脉粥样硬化性缺血性卒中的首个遗传危险因子(OR=1.42,95%CI1.28~1.57)[3]。随后METASTROKE大规模meta分析发现HDAC9基因rs2107595位点增加39%患病风险(OR=1.39,95%CI1.27~1.53)[4]。2015年Su等[5]通过1632病例-对照检测rs2107595位点发现该等位基因与中国南方汉族人群缺血性卒中患者无相关。2013年对汉族人群缺血性卒中患者GWAS分析,发现HDAC9基因rs2389995(OR=1.33,95%CI1.01~1.75)和rs2240419位点(OR=1.29,95%CI1.02~1.63)与大动脉粥样硬化性缺血性卒中密切相关[6]。前面欧洲人群GWAS研究通过10个独立队列中心大样本量的验证,而汉族人群仅1个研究团队对237例大动脉粥样硬化性缺血性卒中患者的SNPs进行GWAS初筛研究。因此,HDAC9基因不同SNPs对汉族人群大动脉粥样硬化性缺血性卒中的遗传易感性仍有待进一步研究证实,且HDAC9基因SNPs在大动脉粥样硬化性缺血性脑卒中的作用机制目前知之甚少。本研究利用国际人类基因组单体型图计划(the international HapMap project,HapMap)公布的北京汉族人群(Han Chinese in Beijing,China,CHB)信息,对HDAC9基因的最小等位基因频率(minor allele frequency,MAF)、连锁不平衡(linkage disequilibrium,LD)、单体域(haplotype block)、单体型频率和标签SNPs(haplotype tagging SNPs,htSNPs)以及HDAC9潜在功能预测和风险程度进行分析,旨在为进一步关联研究中SNPs的确定及机制研究提供分析基础。
1 对象与方法
1.1 研究对象 本文研究对象为千人基因组计划所确定的北京汉族样本45例,信息为该计划公布的HDAC9基因SNPs基因型测定数据。
1.2 方法
1.2.1 SNPs确定标准 依据GWAS发现HDAC9在大动脉粥样硬化性缺血性卒中的热点区域和阳性突变位点集中在末端(3'端)区域[3]。欧洲和汉族大动脉粥样硬化性缺血性卒中人群中已报道的4个SNPs均出现在HDAC9(Chr7:18086949-19002416,ENSG00000048052.17)的末端区域,如rs11984041(Chr7:189923121)、rs2107595(Chr7:19009765),rs2389995(Chr7:18933395)和rs2240419(Chr7:18935566)。因此,本研究选择中国汉族人群(CHB,release#27,Chr7)HDAC9(Chr:18930kb-19020kb),长度为80kb的基因区域,利用Haploview软件对该区域进行信息分析。确定SNPs的标准:基因型测定率大于80%、Hardy-Weinberg平衡(P>0.05)、MAF>0.05。
1.2.2 MAF和LD分析 对任一给定SNPs位点,MAF的计算公式为MAF=(2n1+n2)/2N。其中N代表样本个体数,n1表示基因型为最小等位基因纯合子的个体数,n2表示杂合子的个体数[7]。
对MAF>5%且满足Hardy-Weinberg平衡(P>0.05)的SNPs,进行LD plot分析。连锁不平衡程度采用LD系统D’和SNPs间的关联强度r2检验。D’的计算公式为D’=D/Dmax,其中参数D=Pij-PiPj,D的最大可能值Dmax=min[Pi(1-Pj),(1-Pi)Pj],i和j分别代表相邻的两个基因位点,Pi和Pj分别为相应基因位点的等位基因频率。对任意2位点i和j间r2估计的计算公式为r2=2(PAB-PAPB)/PAPaPBPb,其中PA、Pa分别为位点i等位基因和最小等位基因频率,PB、Pb分别为位点j等位基因和最小等位基因频率,PAB代表i~j单体型为AB时的单体型频率[8]。
1.2.3 单体域构建和单体型频率分析 根据SNPs的连锁不平衡程度采用confidence intervals(Gabriel等)(CI)算法[9]、four Gamete rule(FGR)算法[10]和solid spine of LD(SSOL)算法[8]构建单体域和单体型频率的计算。
1.2.4 htSNPs的选择标准 取r2≥0.8,两点最大似然值[11]LOD≥3.0和aggressive-tagging(two- and three- maker)选择htSNPs。LOD(θ)=LOD10[L()/L(0.5)]。θ为重组分数,为重组分数估计值。
1.2.5 htSNPs潜在功能和风险程度 通过SNP function prediction、miRWalk软件预测htSNPs潜在功能和HDAC9基因3'端htSNPs位点与miRNA结合关系。
2 结果
2.1 基本情况 HDAC9末端区域(Chr7:18930kb-19010kb),共包括75个SNPs。不符合条件的SNPs共24个,其中2个不符合Hardy-Weinberg平衡(P<0.05),16个属于纯合子基因型(21.3%),6个SNPs最小等位基因频率存在性别差异(P<0.05)。符合条件的SNPs共51个(68.0%)。
2.2 单体域及单体型频率
2.2.1 基于confidence intervals算法的单体域和单体型频率 基于confidence intervals计算HDAC9末端区域(Chr7:18930kb-19010kb)构建了11个单体域(图1)。分别包含8,5,5,3,2,5,4,3,3,3,9个单体型,第4,5,9个单体域两个相邻SNPs发生完全连锁(D’=1);其他单体域以1~4个单体型为主,累计频率88.6%,95.2%,89.9%,66.7%,68.5%,70.8%,84.0%,82.8%(表1)。

图1 基于confidence intervals计算HDAC9末端单体域

表1 基于confidence intervals计算HDAC9的单体型及频率

图2 基于four Gamete rule计算HDAC9末端单体域

表2 基于four Gamete rule计算HDAC9的单体型及频率
2.2.2 基于four Gamete rule计算单体域和单体型频率 基于four Gamete rule计算HDAC9末端区域(Chr7:18930kb-19010kb)构建了14个单体域(图2)。分别包含6,3,4,5,2,3,3,3,4,5,3,3,3,4个单体型,第2,3,4,6,7,8,9,12,13个单体域相邻SNPs发生完全连锁(D’=1);其他单体域以2~3个单体型为主,累计频率80.9%,73.8%,70.9%,84.0%,97.6%(表2)。
2.2.3 基于solid spine of LD计算单体域和单体型频率 基于solid spine of LD计算HDAC9末端区域(Chr7:18930kb-19010kb)构建了8个单体域(图3)。分别包含8,4,6,3,9,4,7,10个单体型,第2,4个单体域相邻SNPs发生完全连锁(D’=1);其他单体域以3~4个单体型为主,累计频率78.5%,88.1%,85.1%,94.7%,71.4%,83.4%(表3)。
2.3 HDAC9重组热点 HDAC9基因Chr7:18930kb-19010kb区域间发现一个重组热点,rs2717356位点低LD,不在单体域内。该位点相对背景强度增加约40倍(图4)。
2.4 htSNPs及其特征 符合研究条件的51个SNPs中,经过3种不同连锁不平衡计算方法,结合htSNs选择的标准进行筛选,30个SNPs确定为htSNPs,占58.8%(表4)。标签SNPs中,MAF>0.10组段占100%;MAF>0.20组段占90%;MAF>0.30组段占50%;MAF>0.40组段占20%。
2.5 HDAC9 htSNPs位点与micoRNAs结合情况以及相关功能 对30个htSNPs通过SNP function prediction预测其功能,发现rs2526630位点位于HDAC9的3’UTR区域,miRand、miRWalk和Targetscan软件分析,发现rs2256630[C/T]结合has-miR-1184,hsamiR-1273,hsa-miR-190,hsa-miR-545,hsamiR-576-3p,hsa-miR-576-5p,hsa-miR-616,hsa-miR-891a和hsa-miR-922,与神经系统病理生理密切相关,包括神经干细胞分化、血管新生和炎症、动脉硬化等(表5)。

图3 基于solid spine of LD计算HDAC9末端单体域

图4 HDAC9末端重组热点区域

表3 基于solid spine of LD计算HDAC9的单体型及频率
3 讨论
HDAC9主要位于细胞质,但能够穿梭于细胞核和细胞质之间,调控组蛋白及非组蛋白的乙酰化修饰[25]。作为组蛋白乙酰化修饰调控基因转录的重要调节因子,使基因启动子不易接近转录调控元件,从而抑制转录。非组蛋白的乙酰化和去乙酰化平衡的改变影响细胞多方面的功能。近1/4的卒中患者属于由动脉粥样硬化导致的大血管卒中。大血管卒中与冠状动脉疾病(coronary artery disease,CAD)间有一些共同的风险因素,并且两者均具有很强的遗传成分。至今为止,染色体7p21.1处HDAC9基因区域发现是与大血管卒中关系最密切的风险位点[26]。通过外周血单核细胞基因表达的重复验证研究显示,大规模meta分析报道的动脉粥样硬化缺血性卒中rs2107595风险等位基因(与颈动脉斑块的存在相关)携带者中,HDAC9 mRNA水平增加,但相邻基因(TWIST1/FERD3L)没有改变。与HDAC9+/+、ApoE-/-小鼠相比,HDAC9-/-、ApoE-/-小鼠主动脉粥样硬化病变体积显著降低,并且晚期病变降低。此外,HDAC9-/-、ApoE-/-小鼠动脉斑块内,Mac-3阳性巨噬细胞的比例更高,这可能归因于晚期病变比例的降低。人类动脉粥样硬化斑块的分析显示,此前rs2107595与特异的斑块特征无关[27]。
HapMap计划是人类基因组计划的延伸,由日本、英国、加拿大、中国、尼日利亚和美国的科学家们共同合作完成,它向公众免费开放全部数据,为基因及病因学研究提供了基本数据。SNPs是病因学研究中重要的遗传标记,无论对疾病危险因子的筛选还是重复验证性的遗传关联研究,SNPs的筛选均为研究的一个重要环节,对提高研究发现的把握度和研究效率有至关重要的意义[28]。
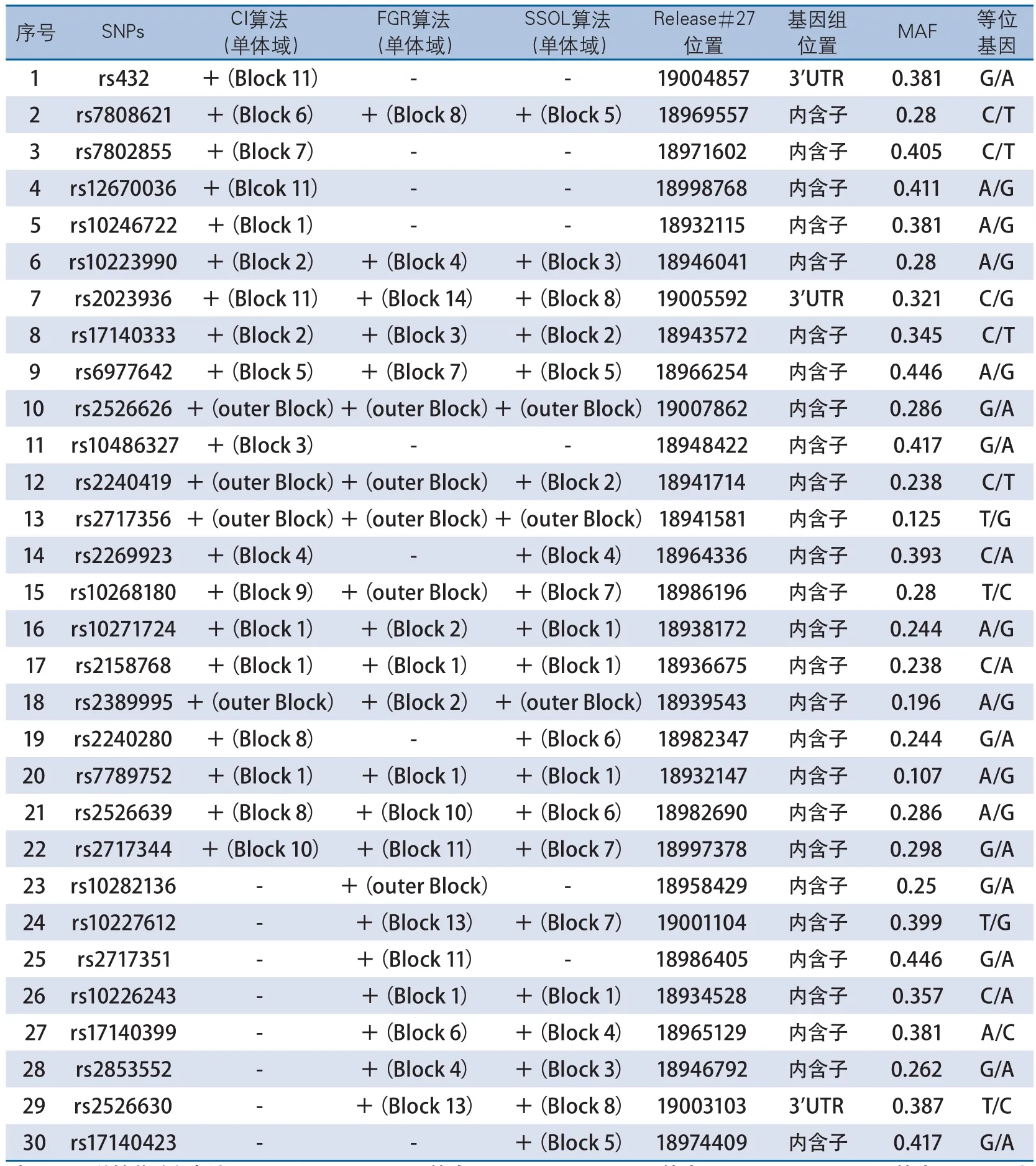
表4 北京汉族人群HDAC9基因18930kb-19010kb区域的标签SNPs及其MAF值
HapMap计划将MAF>0.05的SNPs作为首要研究目标,MAF广泛应用于复杂疾病的全基因组关联研究。在关联研究中,有研究表明,MAF>0.01所构建的图谱可捕获最多的遗传信息,同时成本也最高,MAF阈值一般取大于0.05或0.10[29]。本研究显示HDAC9基因Chr7:18930kb-19020kb区域共测定75个SNPs,其中Hardy-Weinberg平衡P<0.05,且最小等位基因频率P>0.05的SNPs共51个。对SNPs进行连锁不平衡及单体域分析有研究推荐采用confidence intervals、four Gamete rule和solid spine of LD3种计算方法[30]。本研究利用3种计算方法分别构建了11、14和8个单体域,每个单体域包括了1~4个主要单体型。此外,还发现rs2717356位点低LD,不在单体域内,成为HDAC9基因的重组热点区域。
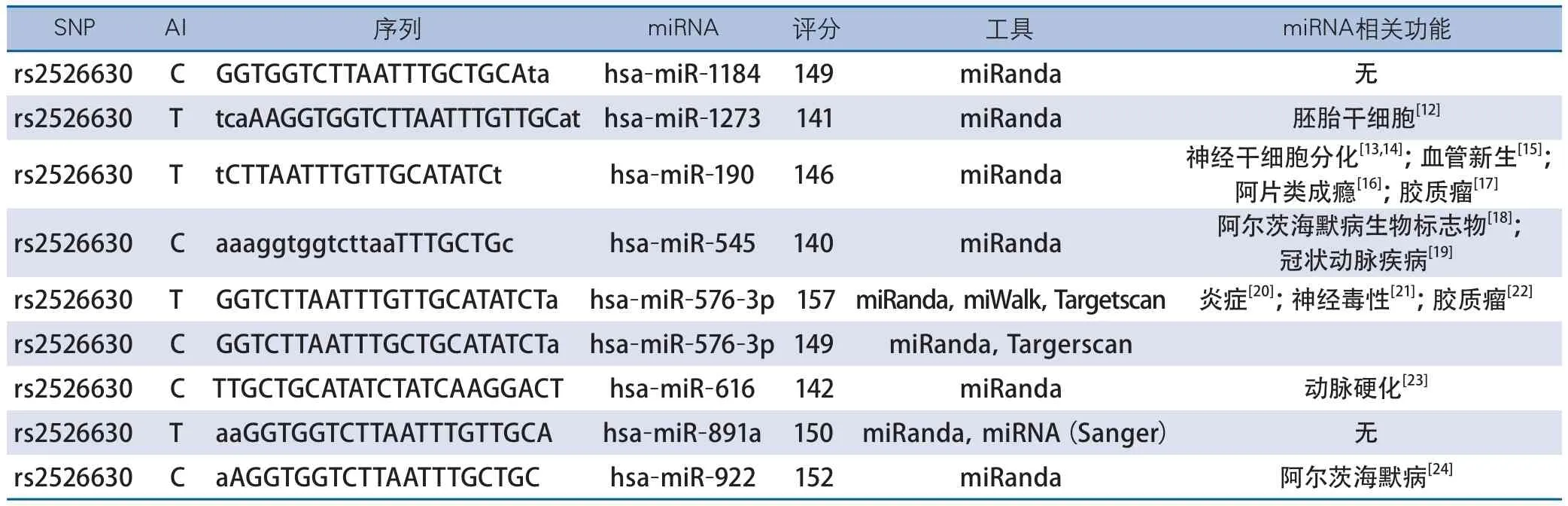
表5 rs2526630与miRNA结合情况
利用标签htSNPs进行大样本量的筛查疾病易感基因SNPs位点或重复验证,不仅可以大大减少用于基因型与疾病关联分析中SNPs的数量,还能提高研究效能、降低成本。由于强关联SNPs之间可相互随机替代[31]。本研究利用Haploview软件取r2≥0.8,且LOD≥3.0确定30个SNPs为htSNPs,且MAF均大于0.10。为进一步研究不同人群HDAC9基因SNPs易感位点的筛选或重复验证提供依据。因此,今后需要对汉族其他人群进行大动脉粥样硬化卒中患者HDAC9基因易感位点筛选,就可以采用本人所筛选的30个标签SNPs位点。
此外,对htSNPs功能分析中,发现rs2526630位点[C/T]位于HDAC9的3’UTR区域,与has-miR-1184,hsa-miR-1273,hsamiR-190,hsa-miR-545,hsa-miR-576-3p,hsa-miR-576-5p,hsa-miR-616,hsa-miR-891a和hsa-miR-922,其中hsa-miR-545,hasmiR-576,hsa-miR-616与神经系统病理生理密切相关,包括神经干细胞分化、血管新生和炎症,动脉硬化等息息相关。因此,本研究推测hsa-miR-545,has-miR-576,hsa-miR-616作为大动脉粥样硬化缺血性脑卒病理生理机制研究具有一定的价值。
1 Haberland M,Montgomery RL,Olson EN.The many roles of histone deacetylases in development and physiology:implications for disease and therapy[J].Nat Rev Genet,2009,10:32-42.
2 Chang S,McKinsey TA,Zhang CL,et al.Histone deacetylases 5 and 9 govern responsiveness of the heart to a subset of stress signals and play redundant roles in heart development[J].Mol Cell Biol,2004,24:8467-8476.
3 International Stroke Genetics Consortium (ISGC),Wellcome Trust Case Control Consortium 2(WTCCC2),Bellenguez C,et al.Genome-wide association study identifies a variant in HDAC9 associated with large vessel ischemic stroke[J].Nat Genet,2012,44:328-333.
4 Traylor M,Farrall M,Holliday EG,et al.Genetic risk factors for ischaemic stroke and its subtypes (the METASTROKE collaboration):a meta-analysis of genome-wide association studies[J].Lancet Neurol,2012,11:951-962.
5 Su L,Shen T,Liang B,et al.Association of GWAS-supported loci rs2107595 in HDAC9 gene with ischemic stroke in southern Han Chinese[J].Gene,2015,570:282-287.
6 Han Y,Sun W,Wang L,et al.HDAC9 gene is associated with stroke risk in a Chinese population[J].Exp Biol Med (Maywood),2013,238:842-847.
7 Panagiotou OA,Evangelou E,Ioannidis JP.Genomewide significant associations for variants with minor allele frequency of 5% or less--an overview:A HuGE review[J].Am J Epidemiol,2010,172:869-889.
8 Barrett JC,Fry B,Maller J,et al.Haploview:analysis and visualization of LD and haplotype maps[J].Bioinformatics,2004,21:263-265.
9 Gabriel SB,Schaffner SF,Nguyen H,et al.The structure of haplotype blocks in the human genome[J].Science,2002,296:2225-2229.
10 Hudson R,Kaplan N.Statisticial properties of the number of recombination events in the history of a sample of DNA sequences[J].Genetics,1985,111:147-164.
11 Qin ZS,Niu T,Liu JS.Partition-ligation-expectationmaximization algorithm for haplotype inference with single-nucleotide polymorphisms[J].Am J Hum Genet,2002,71:1242-1247.
12 Morin RD,O'Connor MD,Griffith M,et al.Application of massively parallel sequencing to microRNA profiling and discovery in human embryonic stem cells[J].Genome Res,2008,18:610-621.
13 Zheng H,Zeng Y,Zhang X,et al.mu-Opioid receptor agonists differentially regulate the expression of miR-190 and NeuroD[J].Mol Pharmacol,2010,77:102-109.
14 Li W,He S,Zhou Y,et al.Neurod1 modulates opioid antinociceptive tolerance via two distinct mechanisms[J].Biol Psychiatry,2014,76:775-784.
15 Hao Y,Yang J,Yin S,et al.The synergistic regulation of VEGF-mediated angiogenesis through miR-190 and target genes[J].RNA,2014,20:1328-1336.
16 Zheng H,Law PY,Loh HH.Non-coding RNAs regulating morphine function:with emphasis on the in vivo and in vitro functions of miR-190[J].Front Genet,2012,3:113.
17 Slaby O.MiR-190 leads to aggressive phenotype of neuroblastoma through indirect activation of TrkB pathway[J].Med Hypotheses,2013,80:325-326.
18 Kumar P,Dezso Z,MacKenzie C,et al.Circulating miRNA biomarkers for Alzheimer's disease[J].PLoS One,2013,8:e69807.
19 Chen F,Zhao X,Peng J,et al.Integrated microRNA-mRNA analysis of coronary artery disease[J].Mol Biol Rep,2014,41:5505-5511.
20 Yarbrough ML,Zhang K,Sakthivel R,et al.Primatespecific miR-576-3p sets host defense signalling threshold[J].Nat Commun,2014,5:4963.
21 Rodriguez-Lebron E,Paulson HL.Allele-specific RNA interference for neurological disease[J].Gene Ther,2006,13:576-581.
22 Dong L,Li Y,Han C,et al.miRNA microarray reveals specific expression in the peripheral blood of glioblastoma patients[J].Int J Oncol,2014,45:746-756.
23 Raitoharju E.MicroRNA-616 and atherosclerosis--a commentary on the paper by Liu et al[J].Atherosclerosis,2013,228:42-43.
24 Zhao ZB,Wu L,Xiong R,et al.MicroRNA-922 promotes tau phosphorylation by downregulating ubiquitin carboxy-terminal hydrolase L1 (UCHL1)expression in the pathogenesis of Alzheimer's disease[J].Neuroscience,2014,275:232-237.
25 Lahm A,Paolini C,Pallaoro M,et al.Unraveling the hidden catalytic activity of vertebrate class IIa histone deacetylases[J].Proc Natl Acad Sci USA,2007,104:17335-17340.
26 Markus HS,Mäkelä KM,Bevan S,et al.Evidence HDAC9 genetic variant associated with ischemic stroke increases risk via promoting carotid atherosclerosis[J].Stroke,2013,44:1220-1205.
27 Azghandi S,Prell C,van der Laan SW,et al.Deficiency of the stroke relevant HDAC9 gene attenuates atherosclerosis in accord with allele-specific effects at 7p21.1[J].Stroke,2015,46:197-202.
28 Chen Q,Wang H,Hetmanski JB,et al.BMP4 was associated with NSCL/P in an Asian population[J].PLoS One,2012,7:e35347.
29 RaoS Q,Huang WJ,Hu B,et al.Effects of cutoff thresholds for minor allele frequencies on HapMap resolution:A real dataset-based evaluation of the Chinese Han and Tibetan populations[J].Chinese Science Bulletin,2009,54:2069-2075.
30 Nicodemus KK,Liu W,Chase GA,et al.Comparison of type I error for multiple test corrections in large single-nucleotide polymorphism studies using principal components versus haplotype blocking algorithms[J].BMC Genet,2005,6 Suppl 1:S78.
31 Carlson CS,Eberle MA,Rieder MJ,et al.Selecting a maximally informative set of single nucleotide polymorphisms for association analyses using linkage disequilibrium[J].Am J Hum Genet,2004,74:106-120.
