利用无标记分子信标及核酸染料TOTO-3检测转基因食品
2015-12-10向东山
向东山
(生物资源保护与利用湖北省重点实验室,湖北民族学院化学与环境工程学院,湖北 恩施 445000)
利用无标记分子信标及核酸染料TOTO-3检测转基因食品
向东山
(生物资源保护与利用湖北省重点实验室,湖北民族学院化学与环境工程学院,湖北 恩施 445000)
利用无标记的分子信标,结合核酸染料TOTO-3,采用同步荧光分析法,建立一种高灵敏、高选择性的花椰菜花叶病毒CaMV 35S启动子核酸片段(目标DNA)的定量检测方法。在优化条件下,目标DNA浓度为2×10—10~200×10—10mol/L之间时,TOTO-3的荧光强度(∆I)与目标DNA浓度(C)之间具有良好的线性关系,其拟合的回归方程为Δ I = 2.022 2 C+18.411 1。方法检出限(3σ)为1×10—10mol/L。该方法重复性好、灵敏度高、选择性好、检出限低。利用该方法,结合不对称聚合酶链式反应技术,实现了对转基因食品的检测。
无标记分子信标;TOTO-3;荧光;转基因食品;检测
随着生物科技的飞速发展,转基因技术在农业各领域已得到广泛应用。转基因技术的使用,可显著增加农作物的产量,提高农作物抗病、抗虫、抗冻等抗逆能力,改进农作物的营养品质等[1-5]。将各类转基因作物经过加工而得到的食品被称为转基因食品。
转基因食品是一把双刃剑,在带给人类巨大实惠的同时也带来了潜在的安全隐患,由于人们对基因工程掌握的不确定性及不可预测性,使得转基因食品在安全性、伦理学以及环境污染等方面存在潜在的威胁[6-9],这是人们不得不面对的一个重要问题。因此对转基因食品的检测具有十分重要的现实意义。
目前,转基因食品的检测方法主要有两类:一类是对外源基因的表达产物即产生的特异蛋白质进行检测,另一类是直接对引入的外源基因序列进行检测[10-12]。前一类检测方法特异性高、操作简单、成本低、稳定性好,但在食品中被检测蛋白浓度较低时会出现假阴性。此外,食品的加工程度对这类检测方法影响较大,若食品在加工过程中使目标蛋白变性也会性导致假阴性的产生。相对于第一种检测方法而言,后一类检测方法绝大数结合了不对称聚合酶链式反应(polymerase chain reaction,PCR)技术,其检测的灵敏度大大提高,且这类检测方法较少受食品加工程度的影响。因此,直接对引入的外源基因序列进行检测的方法在转基因食品的检测中具有广泛的应用前景。
据报道,绝大多数的转基因植物使用花椰菜花叶病毒CaMV 35S启动子[13-14],因此,可以通过检测样品中是否含有CaMV 35S启动子来判断样品是否为转基因植物。
分子信标是一种呈发夹结构的茎-环双标记的寡核苷酸探针,具有合成容易、操作简单、选择性好以及不必与未反应的探针分离即可实时检测等特点,在特定序列单链核酸的检测中扮演着越来越重要的角色[15-18]。近年来,有关应用分子信标对特定序列单链核酸检测的报道越来越多[19-22]。因此分子信标在核酸的定性及定量分析中具有十分广阔的应用前景。
TOTO-3(quinolinium, 1,1′- [1,3-propanediylbis [(dimethyliminio)-3,1-propanedi-yl]]bis[4-[3-(3-methyl-2(3H)-benzothiazolylidene)-1-propen-1-yl]]-, iodide)是一种不对称的菁酸类染料,它与双链核酸具有高度的亲和性。TOTO-3在水溶液中荧光很弱,与单链DNA也几乎不发生作用,但它与双链核酸结合后其荧光强度会增加1 000 倍以上[23]。
本实验利用无标记的分子信标(无有机荧光基团和荧灭基团)及核酸染料TOTO-3,采用同步荧光分析法,建立了一种高灵敏、高选择性的CaMV 35S启动子基因序列检测方法,并结合不对称PCR技术,实现转基因食品的检测。
1 材料与方法
1.1 材料与试剂
实验用的大豆油为具有转基因标示的金龙鱼转基因大豆油,购于湖北省恩施市市场。
TOTO-3 美国Molecular Probes公司;其他所有化学试剂均为分析纯,购于美国Sigma化学有限公司和国药集团化学试剂有限公司。所有的核酸均由上海生工生物工程技术服务有限公司(中国)合成并用高效液相色谱法进行纯化,其核苷酸序列如下:
上游引物:5’-CGA CAG TGG TCC CAA AGA -3’;下游引物:5’-AAG ACG TGG TTG GAA CGT CTT C-3’;无标记分子信标:5’-GCG CGC TGG ACC CCC ACC CAC GAG GAG CAT CGC GCG C-3’;CaMV 35S启动子寡核苷酸片段序列(目标DNA,T):5’-GAT GCT CCT CGT GGG TGG GGG TCC A-3’。
目标核酸的错配序列:一个碱基错配序列(MT1):5’-GAT GCT CCT CGT AGG TGG GGG TCC A -3’;两个碱基错配序列(MT2):5’-GAT GCT CAT CGT GGG TAG GGG TCC A-3’;3个碱基错配序列(MT3):5’-GAT GCG CCT CGC GGG TGA GGG TCC A-3’。
1.2 仪器与设备
RF-5301PC型荧光光谱仪 日本Shimadzu公司;PB-21型pH计 德国Sartorius公司;System 2700 PCR仪美国Applied Biosystems公司。
1.3 方法
1.3.1 检测原理
核酸染料TOTO-3在水溶液中的荧光很弱,与单链DNA的作用也很微弱,但它与双链DNA具有很强的亲和性,且与双链DNA结合后,其荧光信号会显著增强。在没有目标DNA时,分子信标处于茎-环结构状态,分子信标的茎较短(只有6 个碱基对),与TOTO-3作用后,荧光信号较弱;在有目标DNA时,目标DNA与分子信标发生杂交反应,形成较长的双链,与TOTO-3作用后荧光强度显著增强。根据分子信标与目标DNA杂交后所形成的双链DNA与TOTO-3结合后,荧光信号增加的程度,实现对特定单链DNA的检测。
1.3.2 样品制备
杂交反应由分子信标与目标DNA 在一定条件下完成。首先,将目标DNA溶解在缓冲溶液中,配制成浓度为1×10—6mol/L的储备液。取一定量的目标DNA溶液加入到适当浓度的分子信标溶液中并混合均匀,加入缓冲溶液至总体积达到450 μL,在一定的温度条件下孵育一定的时间。在本实验中,孵育的目的是为了破坏分子信标中碱基互补部分的双链,从而加速分子信标与目标DNA的反应速度。然后,将孵育后的溶液冷却至室温,在冷却的过程中使分子信标与目标DNA进行杂交反应,待杂交完成后,加入一定浓度的核酸染料TOTO-3 50 μL,使杂交后的双链DNA与核酸染料TOTO-3结合。在条件优化的反应中,目标DNA及分子信标的浓度均为2×10—8mol/L,孵育温度为60 ℃,孵育时间为6 min,TOTO-3的浓度为1×10—7mol/L,双链NDA与核酸染料TOTO-3结合的时间为20 min。
1.3.3 目标DNA的检测
目标DNA的检测通过对TOTO-3荧光强度的测定而实现,TOTO-3的荧光强度通过同步荧光扫描获得。TOTO-3最大发射波长与最大激发波长的波长间隔20 nm,因此本实验中同步扫描的波长间隔(Δλ)设置为20 nm。荧光分光光度计的激发及发射狭缝宽度均设置为10 nm。
1.3.4 碱基错配分析
50 μL浓度为2×10—7mol/L的目标DNA序列和相同浓度的碱基错配序列均按照上述样品制备过程进行准备,然后分别检测相应的荧光信号,最后进行比较和分析。
1.3.5 实际样品检测
针对真实样品的分析,先通过不对称PCR获得含目标DNA的单链核酸,然后利用无标记的分子信标及核酸染料TOTO-3对其进行检测。不对称PCR扩增可得到大量含目标DNA的单链核酸,分子信标在与其反应时不需要相互竞争,可显著地提高检测的灵敏度。不对称PCR扩增的条件为:PCR反应液总体积为50 μL(上游引物与下游引物的比例为40∶1,引物序列见1.1节)。4 μL模板(来自于转基因大豆油),5 mmol/L的 MgCl2溶液8 μL,PCR 缓冲液5 μL,2.5 mmol/L 的dNTPs 4 μL,20 μmol/L的上游引物2 μL,1 μmol/L的下游引物1 μL,5 U Taq聚合酶,加蒸馏水至总体积为50 μL。反应程序为:94 ℃预变性3 min后,按94 ℃变性30 s,55 ℃复性45 s,72 ℃延伸50 s进行PCR循环反应,35 次循环。PCR反应结束后用缓冲溶液将反应体系进行稀释,然后加入50 μL 2×10—7mol/L的分子信标,用蒸馏水稀释至450 μL,在60 ℃孵育6 min。冷却至室温,再将50 μL 1×10—6mol/L TOTO-3溶液加入至上述溶液中摇匀,室温放置20 min后,用同步分析法对其荧光进行检测。
2 结果与分析
2.1 不同缓冲溶液对测定结果的影响
不同缓冲的溶液所含的阴、阳离子各不相同,离子强度也各不一样,它们对DNA的杂交及荧光染料都有影响。实验测试了伯瑞坦-罗宾森缓冲溶液(brittonrobinson buffer,BR)、磷酸缓冲溶液(phosphate buffer,PB)、三(羟甲基)氨基甲烷-盐酸缓冲溶液(Tris (hydroxymethyl) aminomethane-HCl buffer,Tris)及硼酸-硼砂缓冲溶液(boric acid-borax buffer,B)对测定结果的影响,结果(图1)表明,缓冲溶液对测定结果影响不大,Tris缓冲溶液效果略好,在后续实验中选择Tris缓冲溶液。

图 1 不同缓冲溶液对荧光强度的影响Fig.1 Effect of buffer solutions on the fl uorescence intensity
2.2 pH值对测定结果的影响
缓冲溶液的pH值可以影响单链DNA的杂交反应,另外核酸染料TOTO-3的荧光强度随也pH值的变化而变化。实验测试了6 个不同pH值的Tris缓冲溶液对测定结果的影响。结果(图2)表明,在pH 7.0~8.2的范围内,核酸染料TOTO-3的荧光强度随pH值的增加而增加,当pH值大于8.2时,荧光强度逐渐下降。因此选择缓冲溶液的pH值为8.2。

图 2 pH值对荧光强度的影响Fig.2 Effect of pH on the fl uorescence intensity
2.3 孵育温度对测定结果的影响
常温时,分子信标与目标DNA之间的杂交反应较慢,为了加快反应速度,通过加热的方式打开分子信标末端互补的碱基对。为了找到合适的孵育温度,实验考察不同孵育温度对测定结果的影响。结果(图3)表明,孵育温度在30~60 ℃之间时,核酸染料TOTO-3的荧光强度随着温度的升高而增加,当孵育温度在大于60 ℃时,其荧光强度基本不再发生变化。因此最佳孵育温度为60 ℃。
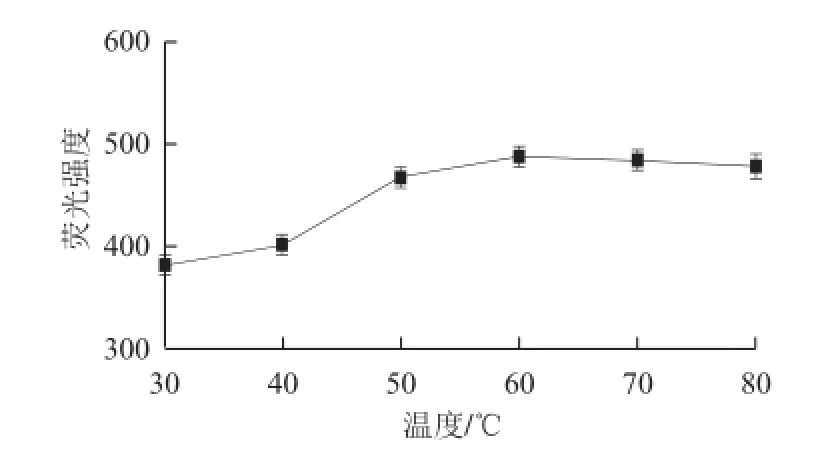
图 3 孵育温度对荧光强度的影响Fig.3 Effect of incubation temperature on the fl uorescence intensity
2.4 孵育时间对测定结果的影响

图 4 孵育时间对荧光强度的影响Fig.4 Effect of incubation time on the fl uorescence intensity
本实验中,孵育的主要目的是为了将分子信标的茎打开,因此孵育时间是影响分子信标与目标DNA杂交反应速率的重要因素。实验对不同的孵育时间进行测试。结果(图4)表明,反应时间在2~6 min内,核酸染料TOTO-3的荧光强度随时间的延长而增加,当孵育时间超过6 min时,荧光强度趋于稳定,说明在6 min内即可将分子信标的茎完全打开。因此,本实验选择的孵育时间为6 min。
2.5 离子强度对测定结果的影响
离子具有稳定双链DNA的作用。实验通过加入不同浓度的NaCl考察离子强度对荧光强度的影响。实验设计了6 个不同NaCl浓度,并测试了它们对测定结果的影响。结果(图5)表明,NaCl的浓度在0~80 mmol/L之间时,TOTO-3的荧光强度随着NaCl浓度的增加而增加。当NaCl浓度达到80 mmol/L后,其荧光强度趋于稳定。本实验选择NaCl浓度为80 mmol/L。

图 5 离子强度对荧光强度的影响Fig.5 Effect of ionic strength on the fl uorescence intensity
2.6 双链DNA与TOTO-3结合时间对测定结果的影响
核酸染料TOTO-3只有与双链DNA结合之后才会发出较强的荧光,因此它与双链DNA的结合时间是影响其荧光强度的一个重要因素。实验对TOTO-3与双链DNA的作用时间进行了考察。结果(图6)表明,TOTO-3与双链DNA的作用在20 min内即可完成,当结合时间超过20 min后,其荧光强度趋于稳定。因此本实验选择TOTO-3与双链DNA的最佳结合时间为20 min。

图 6 双链DNA与TOTO-3结合时间对荧光强度的影响Fig.6 Effect of binding time between ds-DNA and TOTO-3 on the fl uorescence intensity
2.7 工作曲线及检出限
在最优实验条件下,对目标DNA的浓度与相应TOTO-3荧光强度之间的关系进行测试,并找出目标DNA浓度与染料TOTO-3荧光强度之间的线性范围。结果(图7)表明,在目标DNA浓度为2×10—10~200×10—10mol/L之间时,TOTO-3的荧光强度(∆I)与目标DNA浓度(C)之间具有良好的线性关系。其拟合的回归方程为∆I=2.022 2 C+18.411 1(R2= 0.995 9)。对7个浓度为2×10—9mol/L的平行样品分别进行检测以评价方法的精密度,7个检测结果的相对标准偏差(relative standard deviation,RSD)为3.33%,说明方法具有较好的精密度。用空白试剂标准偏差(n = 11)的3 倍除以回归方程的斜率得方法检出限为1×10—10mol/L。

图 7 TOTO-3的荧光强度与目标DNA浓度之间的线性关系Fig. Linear relationship between the fl uorescence intensity of TOTO-3 and the concentration of target DNA
2.8 碱基错配实验
按照碱基错配的实验方法,通过对不同碱基错配序列的分析,测试了方法的特异性。图8表明,在目标DNA与错配DNA浓度相同的情况下,目标DNA所对应的荧光强度(已扣除荧光背景)远大于一个碱基错配的DNA序列,这表明该方法具有很高的选择性。
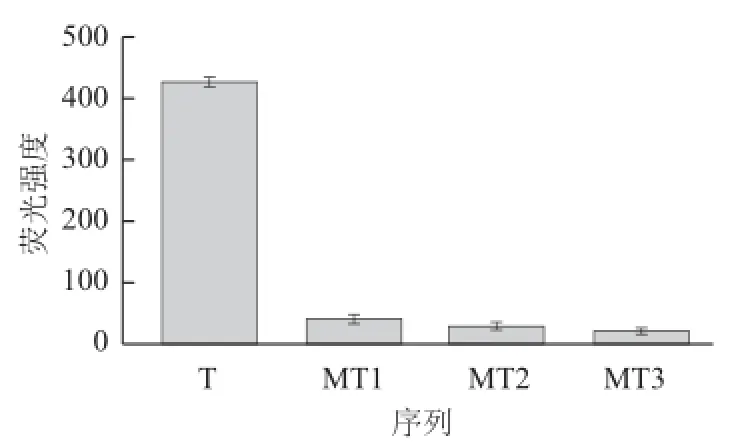
图 8 方法特异性测试Fig.8 Specifi city of the method
2.9 实际样品的分析
利用上述分析方法,结合不对称PCR技术对实际样品进行分析。根据实际样品分析步骤,实验比较了有模板DNA和没有模板DNA存在的分析结果。结果表明,单独的TOTO-3溶液荧光信号很弱(图9a),没有模板DNA时,核酸染料TOTO-3 的荧光信号也较弱(图9b),这表明在扩增产物中没有含目标DNA的核酸片段。当有模板DNA时,核酸染料TOTO-3 的荧光信号显著增强(图9c),这表明在扩增产物中有较多的含目标DNA的核酸片段。这充分说明,该方法能用于转基因食品的分析。

图 9 没有模板DNA存在(a、b)和有模板DNA存在(c)的不对称PPCCRR比较Fig.9 Comparison of asymmetric PCR without (a, b) and with (c) template DNA
3 结 论
本实验利用无标记的分子信标及核酸染料TOTO-3,建立了一种特定序列核酸检测的新方法。利用该方法,并结合不称PCR技术,实现了对转基因食品的检测。该方法操作简单、检测速度快、灵敏度高、重复性好、检出限低。
[1] MUMM R H. A look at product development with genetically modifi ed crops: examples from maize[J]. Journal of Agricultural and Food Chemistry, 2013, 61(35): 8254-8259.
[2] SCHWEMBER A R. An update on genetically modified crops[J]. Ciencia E Investigacion Agraria, 2008, 35(3): 231-250.
[3] QAIM M, ZILBERMAN D. Yield effects of genetically modified crops in developing countries[J]. Science, 2003, 299: 900-902.
[4] RYFFEL G U. Dismay with GM maize a science-based solution to public resistance against genetically modified crops that could be compatible with organic farming[J]. EMBO Reports, 2011, 12(10): 996-999.
[5] QIAO F B, WILEN J, ROZELLE S. Dynamically optimal strategies for managing resistance to genetically modified crops[J]. Journal of Economic Entomology, 2008, 101(3): 915-926.
[6] HILBECK A, WEISS G, OEHEN B, et al. Ranking matrices as operational tools for the environmental risk assessment of genetically modified crops on non-target organisms[J]. Ecological Indicators, 2014, 36: 367-381.
[7] OH J P, CHUNG K H. Analysis of agricultural characteristics to establish the evaluation protocol and environmental risk assessment for genetically modifi ed hot pepper crops[J]. Horticulture Environment and Biotechnology, 2012, 53(5): 349-356.
[8] YANG Jun, WANG Zhirui, YANG Deli, et al. Ecological risk assessment of genetically modifi ed crops based on cellular automata modeling[J]. Biotechnology Advances, 2009, 27(6): 1132-1136.
[9] MAGANA-GOMEZ J A, de la BARCA A M C. Risk assessment of genetically modified crops for nutrition and health[J]. Nutrition Reviews, 2009, 67(1): 1-16.
[10] KIM J H, KIM E H, YEA M C, et al. Validation of a multiplex PCR detection kit for screening of herbicide-tolerant genes in genetically modified crops[J]. Journal of the Korean Society for Applied Biological Chemistry, 2013, 56(2): 251-254.
[11] KUMAR R, SINGH C K, KAMLE S, et al. Development of nanocolloidal gold based immunochromatographic assay for rapid detection of transgenic vegetative insecticidal protein in genetically modifi ed crops[J]. Food Chemistry, 2010, 122(4): 1298-1303.
[12] MANO J, OGUCHI T, AKIYAMA H, et al. Simultaneous detection of recombinant DNA segments introduced into genetically modifi ed crops with multiplex ligase chain reaction coupled with multiplex polymerase chain reaction[J]. Journal of Agricultural and Food Chemistry, 2009, 57(7): 2640-2646.
[13] TAKABATAKE R, TAKASHIMA K, KURASHIMA T, et al. Interlaboratory study of qualitative PCR methods for genetically modified maize events MON810, Bt11, GA21, and CaMV P35S[J]. Journal of Aoac Internationa, 2013, 96(2): 346-352.
[14] HOHNE M, SANTISI C R, MEYER R. Real-time multiplex PCR: An accurate method for the detection and quantification of 35S-CaMV promoter in genetically modifi ed maize-containing food[J]. European Food Research and Technology, 2002, 215(1): 59-64.
[15] ZHANG Kai, WANG Ke, XIE Minhao, et al. DNA-templated silver nanoclusters based label-free fluorescent molecular beacon for the detection of adenosine deaminase[J]. Biosensors Bioelectronics, 2014, 52: 124-128.
[16] XIANG Dongshan, ZHAI Kun, WANG Lianzhi. Multiplexed DNA detection with a composite molecular beacon based on guaninequenching[J]. Analyst, 2013, 138(18): 5318-5324.
[17] WEI Xinpan, SU Shao, GUO Yuanyuan, et al. A molecular beaconbased signal-off surface-enhanced raman scattering strategy for highly sensitive, reproducible, and multiplexed DNA detection[J]. Small, 2013, 9(15): 2493-2499.
[18] DENG Shengyuan, CHENG Lingxiao, LEI Jianping, et al. Label-free electrochemiluminescent detection of DNA by hybridization with a molecular beacon to form hemin/G-quadruplex architecture for signal inhibition[J]. Nanoscale, 2013, 5(12): 5435-5441.
[19] ZHANG Min, GUO Sumiao, LI Yingru, et al. A label-free fl uorescent molecular beacon based on DNA-templated silver nanoclusters for detection of adenosine and adenosine deaminase[J]. Chemical Communication, 2012, 48(44): 5488-5490.
[20] XUAN F, LUO X T, HSING I M. Ultrasensitive solution-phase electrochemical molecular beacon-based DNA detection with signal amplifi cation by exonuclease Ⅲ-assisted target recycling[J]. Analytical Chemistry, 2012, 84(12): 5216-5220.
[21] RAI V, NYINE Y T, HAPUARACHCHI H C, et al. Electrochemically amplified molecular beacon biosensor for ultrasensitive DNA sequence-specific detection of Legionella sp.[J]. Biosensors Bioelectronics, 2012, 32(1): 133-140.
[22] WANG J, ONOSHIM A D, AKI M, et al. Label-free detection of dnabinding proteins based on microfluidic solid-state molecular beacon sen sor[J]. Analytical Chemistry, 2011, 83(9): 3528-3532.
[23] CHIANG C K, HUANG C C, LIU C W, et al. Oligonucleotide-based fl uore scence probe for sensitive and selective detection of mercury (Ⅱ) in aqueous solution[J]. Analytical Chemistry, 2008, 80(10): 3716-3721.
Detection of Genetically Modifi ed Food with a Non-Labeled Molecular Beacon and a Nucleic Acid Dye TOTO-3
XIANG Dongshan
(Key Laboratory of Biologic Resources Protection and Utilization of Hubei Province, School of Chemical and Environmental Engineering, Hubei Minzu University, Enshi 445000, China)
A highly sensitive and selective detection method for DNA of caulifl ower mosaic virus (CaMV) 35S promoter (target DNA) was developed using a non-labeled molecular beacon (MB) and a nucleic acid dye TOTO-3. Under the optimum conditions, the fl uorescence intensity of TOTO-3 exhibited good linear dependence on the quantity of the target DNA in the range of 2×10-10–200×10-10mol/L. The fi tted regression equation was ∆ I = 2.022 2 C + 18.411 1, and the limit of detection was 1 ×10-10mol/L (3σ). The proposed method had a good precision, low detection limit, good selection and high sensitivity. The detection of genetically modifi ed food could be achieved by this proposed method based on asymmetric polymerase chain reaction (PCR).
S132
A
1002-6630(2015)02-0174-05
10.7506/spkx1002-6630-201502033
2014-04-08
国家自然科学基金地区科学基金项目(21465010);湖北省教育厅重点项目(D20131905;D20131906);
生物资源保护与利用湖北省重点实验室开放基金项目(PKLHB1316);湖北民族学院团队项目(MY2013T003);
湖北省林学一级学科资助项目
向东山(1974—),男,副教授,博士,研究方向为分析化学。E-mail:zk3100@sohu.com
Kay words: non-labeled molecular beacons; TOTO-3; fl uorescence; genetically modifi ed food; detection
