基于RNA-seq的崇左金花茶EST-SSR标记开发
2015-11-19黄连冬高继银李昕骥张文驹
邵 阳,范 文,黄连冬,高继银,李昕骥,张文驹
(1.复旦大学 生命科学学院 生物多样性科学研究所 生物多样性与生态工程教育部重点实验室,上海 200438;2.广西南宁市金花茶公园,南宁 530022;3.广东棕榈风景园林科学研究院,中山 528416;4.中国林业科学研究院亚热带林业研究所,富阳 311400)
崇左金花茶(Camellia chuongtsoensis S.Y.Liang et L.D.Huang)为山茶科(Theaceae)山茶属(Camellia)金花茶组(Sect.Chrysantha H.T.Chang)常绿灌木,主产广西崇左等地区,是近年来新发现的四季开花的珍稀山茶类群[1].崇左金花茶花朵分布均匀,花色鲜黄并呈蜡质光泽,具有极高的观赏价值,是一种优良的育种材料[2],在家庭盆栽、园林美化等方面具有广阔的应用前景[3].此外,其叶片和花果中含有丰富的蛋白质、人体必需的氨基酸及微量元素,又具有重要的保健价值[4].作为新发现的物种,目前有关崇左金花茶的研究主要集中于栽培、组织培养、化学成分分析等方面[4-6],其遗传背景尚不明确,亟待开发稳定、高效的分子标记,为该种种质的鉴定、居群遗传结构分析以及系统分类等研究工作提供依据.
现有的分子标记中,简单重复序列(Simple Sequence Repeats,SSR),又名微卫星(Microsatellites),具有高丰度、高变异、共显性等特点[7],已被广泛应用于植物的保护遗传学研究中[8].常用的SSR 标记分为基因组SSR(G-SSR)和转录组SSR(EST-SSR)两类[9].之前有关SSR 开发的研究,往往基于Sanger测序,不仅需要投入大量人力物力和时间,得到的序列也有限.二代测序的发展,特别是RNA-seq的应用,使得快速获得某一特定物种的转录组信息成为可能,极大地丰富了物种序列背景数据库.由RNA-Seq测序拼装得到的Unigene含有丰富的SSR 位点,得到的EST-SSR 能够直接反映基因的表达信息,在亲缘植物间具有良好的通用性[10].因此,相关方法已成为SSR 引物开发的新途径.本研究基于崇左金花茶花转录组获得的Unigene进行微卫星位点搜索,设计引物并对引物种内扩增的多态性进行检验.开发的ESTSSR 分子标记可用于崇左金花茶遗传多样性与资源保护等多方面的研究.
1 材料与方法
1.1 材料及数据获取
崇左金花茶转录组测序样本采自上海山茶园,样品处于花苞现蕾期.采摘下的花芽立即保存于液氮中.采用改良的CTAB-LiCl法[11]直接提取RNA,交由上海晶能生物技术有限公司完成建库及测序,实验包括以mRNA 为模板,用CreatorTMSmartTMcDNA 文库构建(Clontech)试剂盒反转录合成cDNA.采用QIAquick PCR 纯化试剂盒(Qiagen,Valencia,CA)对cDNA 双末端修复并加A,进行PCR 扩增.构建好的文库用Agilent 2100 Bioanalyzer 和ABI StepOnePlus Real-Time PCR System 质检合格后,使用Illumina HiSeqTM2500 测序.测序得到的原始RNA-seq 数据,交由华大基因公司进行拼装,获得Unigene.SSR 引物验证材料采自广西省崇左市的一个崇左金花茶居群,共24个个体,每一个体采一新鲜叶片,用硅胶干燥迅速干燥并保存.采用改良的CTAB法[12]提取基因组总DNA,实验流程详见文献[13].最后将DNA 稀释至约30ng/μL,-20℃保存备用.
1.2 EST-SSR位点检索
基于崇左金花茶花苞RNA-Seq数据组装获得的Unigene,利用MISA(MIcroSAtellite identification tool,http:∥pgrc.ipk-gatersleben.de/misa/)扫描SSR 位点,搜索参数设置为:二核苷酸重复次数≥6,三核苷酸重复次数≥5,四、五、六、七核苷酸重复次数≥4.单核苷酸重复单元的SSR 位点由于在实际研究中应用较少,故不进行筛选.
1.3 EST-SSR引物设计及验证
调用Primer 3.0批量引物设计程序[14]对含有SSR 位点的序列设计引物,引物设计原则:引物长度18~22bp;退火温度55~65℃;上、下游引物的Tm值相差不大于5℃;PCR 产物长度80~500bp;GC含量在40%~60%之间.每个位点程序默认产生至少5 对候选引物.传统的SSR 开发流程中,会利用Primer Premier 5.0基于每对引物的性质和其他副产物等对每对引物给出排名,基于排名可找到相应序列最理想的引物[15-16].为优化引物设计,我们利用引物所对应的Unigene在Primer Premier 5.0中对引物进行进一步检验,尽量避免发夹结构,错配,引物二聚体的出现.之后,随机选择10对引物对崇左金花茶居群进行多态性验证.随机挑选的10 对引物交由桑尼公司合成.采用10μL 的PCR 体系,包括:10×PCR Buffer(Mg2+Free),0.8μL;25mmol/L MgCl2,0.8μL;10mmol/L dNTP,0.6μL;10μmol/L正向和反向引物各0.5μL;5U/μL Taq,0.1μL;基因组DNA,0.4μL;用3dH2O 补齐至10μL.PCR 反应条件为:94℃预变性5min;94℃变性30s,退火温度30s,72℃延伸30s,循环30次;72℃延伸7min.每对引物的最适退火温度通过梯度PCR 实验确定,PCR 产物经6%聚丙烯酰胺凝胶电泳,用银染法检验条带.对于存在种内多态性的位点,用软件POPGENE 32[17]计算相关的群体遗传学数据.
2 结果与分析
2.1 崇左金花茶EST-SSR频率与分布
按照搜索标准,利用MISA 在崇左金花茶转录组数据组装获得的35 410 个Unigene中共鉴定出7 754个2~7核苷酸重复类型的SSR 位点,分布于6 394条序列中,其中1 121条序列具有两个以上的位点.在获得的Unigene中SSR 位点出现频率为21.90%,分布密度为1/3.60kb.
崇左金花茶EST-SSR重复单元丰富,包括二碱基重复到七碱基重复.重复单元的频率不同,出现最多的为二碱基重复单元,占SSR 位点总数的61.30%,其次是三碱基重复单元,占27.02%.其余类型的重复单元出现频率较低:四碱基重复单元占5.35%,五碱基重复单元占3.26%,六碱基重复单元占2.99%,七碱基重复单元占0.08%(表1).
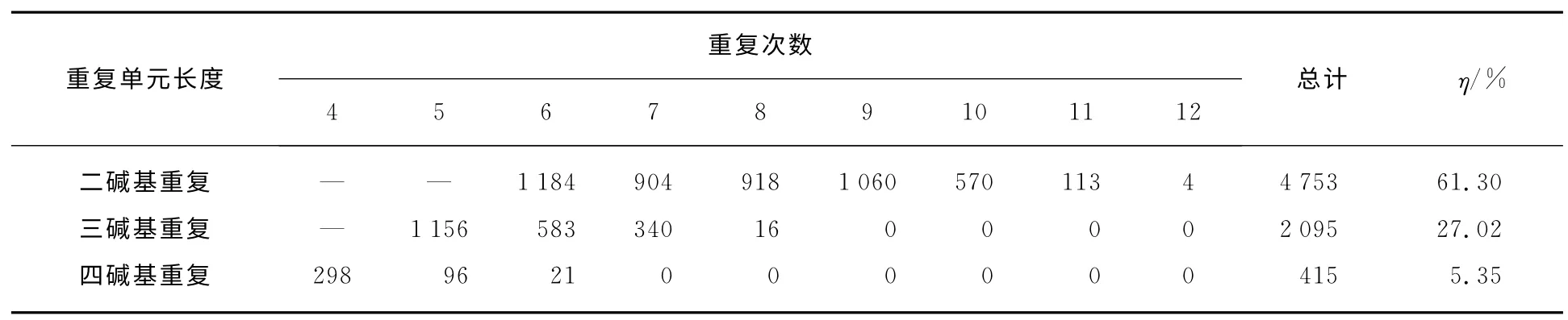
表1 崇左金花茶转录组中不同EST-SSR的数量、类型以及频率Tab.1 The number,type and frequency of EST-SSR in C.chuongtsoensis
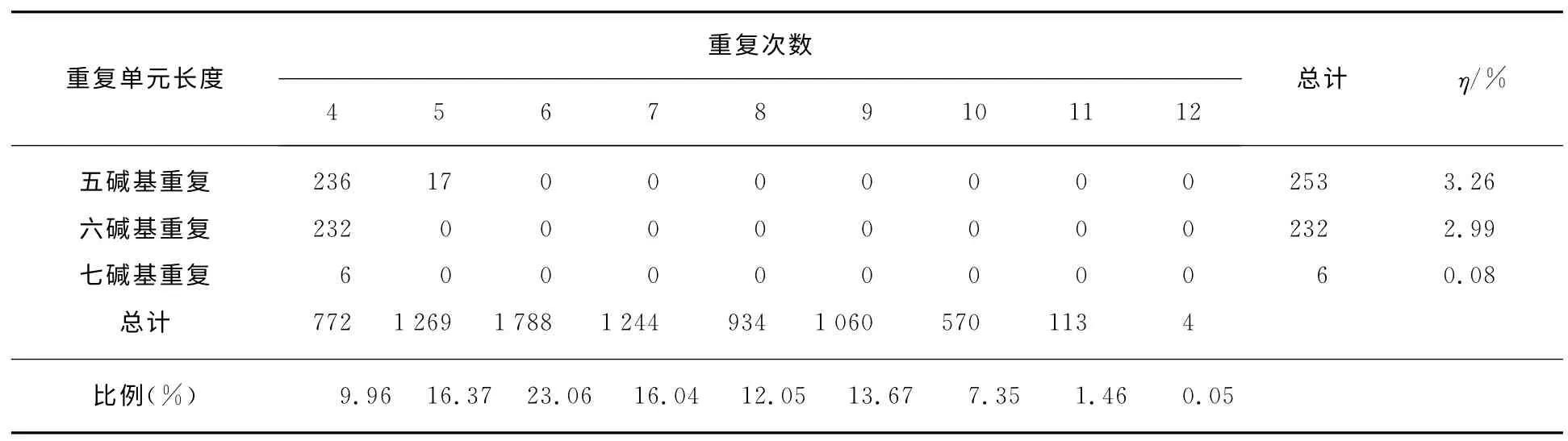
(续表)
2.2 崇左金花茶EST-SSR的特性
在参考了序列完整性的基础上,我们对出现重复单元的类型进行了进一步分类与合并.在崇左金花茶的EST-SSR中,共发现235种重复单元,包括4种二碱基重复单元,10种三碱基重复单元,26种四碱基重复单元,50种五碱基重复单元,138种六碱基重复单元,7 种七碱基重复单元.二碱基重复单元中,AG/CT 含量最多,占二碱基重复单元的80.60%,其次是AT/TA,占12.96%.三碱基重复单元中,AAG/CTT 含量最高,占三碱基重复单元的25.49%,其余重复单元类型及发生频率见表2.
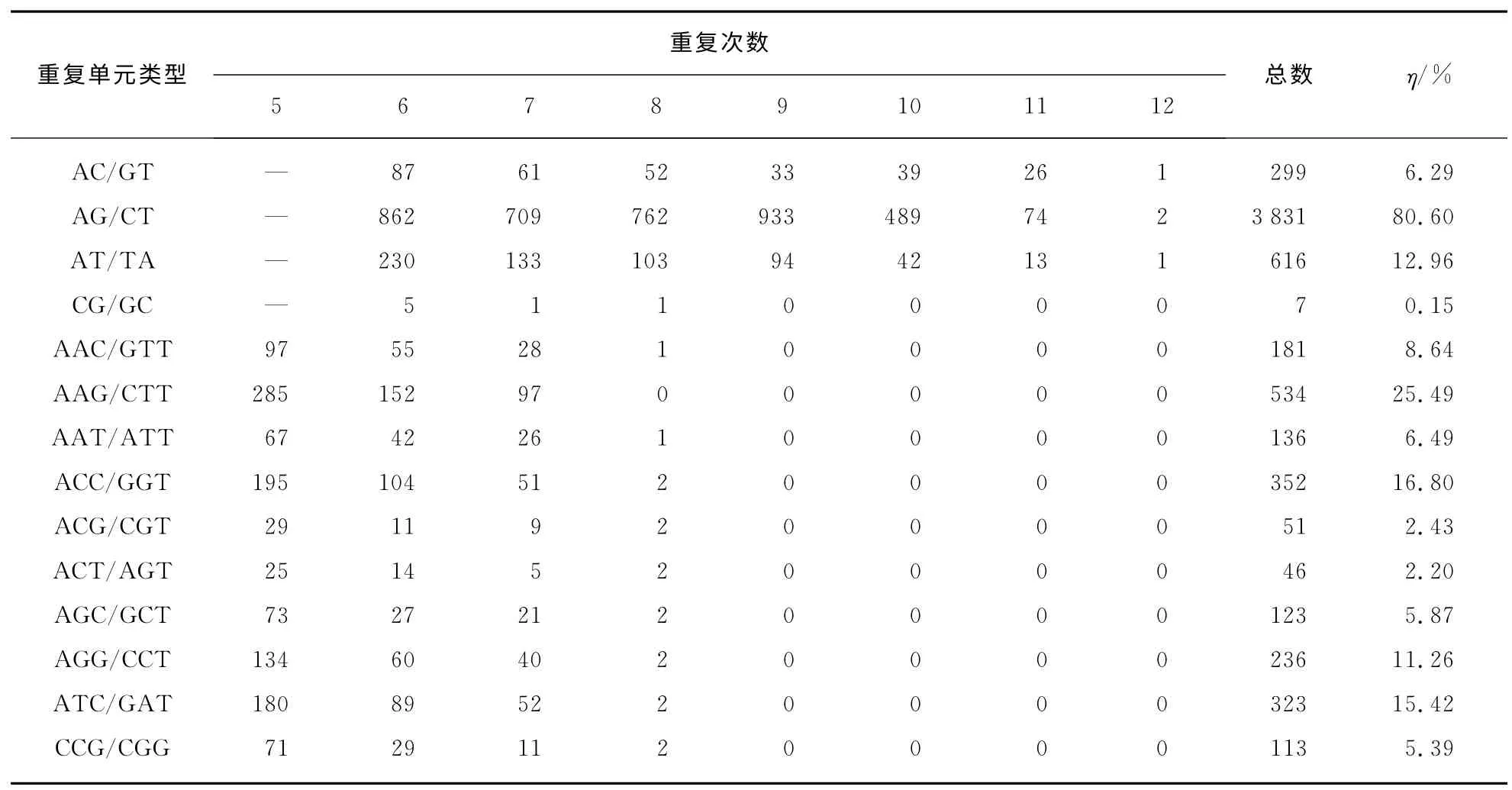
表2 崇左金花茶EST-SSR中二、三碱基重复单元的类型及频率Tab.2 The type and frequency of di and tri-nucleotide repeats in the EST-SSR data of C.chuongtsoensis
2.3 崇左金花茶EST-SSR引物验证以及多态性评价
在崇左金花茶花转录组中,共计3 303条包含EST-SSR 位点的Unigene可成功设计出引物.随机抽取10对引物,部分引物信息见表3(第764页).以崇左金花茶广西崇左居群DNA 为模板进行PCR 扩增.结果表明,10对引物中的8对成功扩增出与预期大小相同的条带,其中4对引物扩增的DNA 条带在居群内具有多态性(图1),EST-SSR5引物中,有8个个体扩增出可读条带,存在零等位基因.表4显示这四个位点的观测杂合度(HO)为0.00到1.00不等,期望杂合度(HE)为0.38到0.85不等,且显著地偏离了Hardy-Weinberg平衡(P<0.001).其余两对引物一对无法扩增出条带,另一对则扩增出大量非特异性条带.本研究的引物有效扩增率为80%,其中多态性占40%.
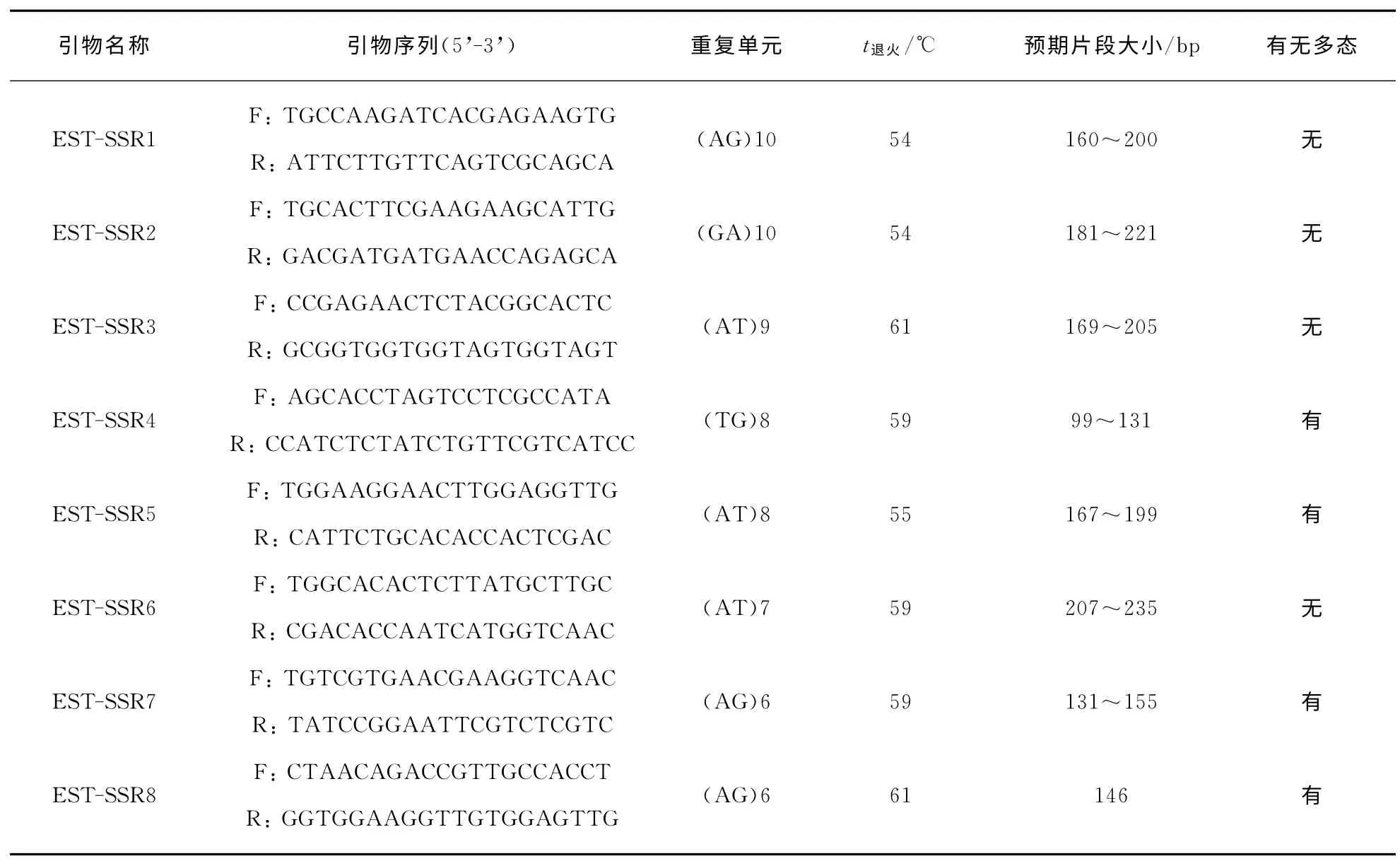
表3 崇左金花茶中8对EST-SSR引物的特点Tab.3 The character of 8pairs of primers in C.chuongtsoensis
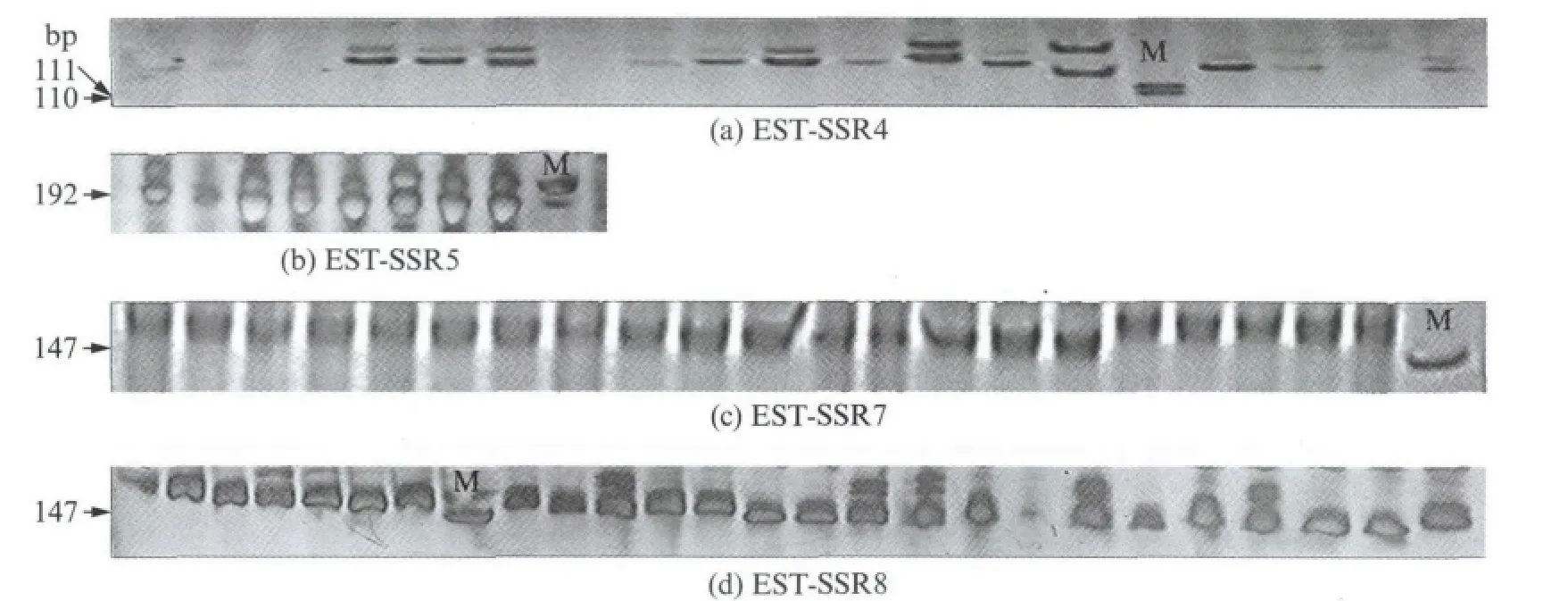
图1 部分SSR 引物扩增崇左金花茶花个体的聚丙烯酰胺凝胶电泳照片(M 为分子质量标记)Fig.1 SDS-PAGE of C.chuongtsoensisindividuals amplified by the SSR primers(M is DNA marker)
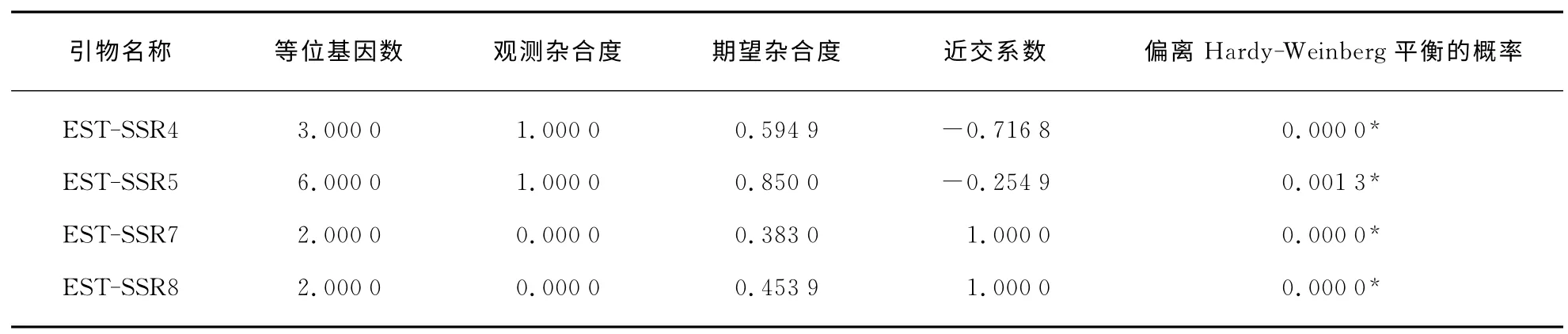
表4 崇左金花茶采样居群的遗传学分析Tab.4 The population genetic analysis in one population of C.chuongtsoensis
3 讨论
3.1 SSR 引物开发策略比较
传统的SSR 引物开发方法分为四大类.第一类是经典方法,包括构建与筛选大(小)插入片段基因组文库方法,这种方法的主要缺点是阳性克隆得率较低;第二类是富集法,包括引物法富集以及磁珠富集法,这类方法存在的问题在于其获得有用序列的概率较低;第三类是省略筛库法,包括STMP以及SAM,这种方法得到的阳性克隆较多,但过程较为繁琐[18-19];第四种是数据库筛选法,这种方法的限制性在于研究的目的物种不一定在数据库中有足够的数据量.在应用微卫星标记进行遗传研究时,选用何种方法获得SSR 引物,往往取决于研究目的和实验条件[20].诸如崇左金花茶此类没有参考基因组的物种,若想快速高效地得到大量EST-SSR引物,利用转录组测序,de novo 组装后检测SSR 位点并开发引物是一种可行且高效的途径.近年来转录组测序发展势头迅猛,已成为了开发SSR 分子标记的重要方法[21,22].然而,通过不同的软件,设定不同的参数,搜索得到的SSR 频率以及数量都有所差异.本研究从转录组组装的Unigene中搜索得到7 754个SSR 位点,分布于6 394条序列中.SSR 位点出现频率为21.9%,高于茶树花(16.66%)、野三七(16.86%)、刺梨(20.37%)、半夏(16.24%),低于石山蜘蛛抱蛋(27.8%)等用转录组数据检测出SSR 位点的频率[21,23-26].研究结果的差异一方面归因于筛选标准的不同,另一方面也和不同来源的EST(如:不同物种,组织或时期)相关[27].以往研究用MISA 进行SSR 位点搜索均检测单碱基重复单元,这样会提高SSR 位点的检出率.然而在本研究中,不检测单核苷酸重复单元,崇左金花茶的EST-SSR 检出率在被子植物中依旧较高,这充分说明了崇左金花茶转录组中存在丰富的SSR 位点,使用转录组数据开发SSR 引物合理而高效.
3.2 导致偏离Hardy-Weinberg平衡的可能因素
经过检验,本研究四对具有多态性的引物均偏离了Hardy-Weinberg平衡.本研究所选用的考察种群个体数(n=24),相比其他研究并不在少数,然而,由于我们采样的崇左金花茶野外居群尺度小,采样个体之间距离近,可能是导致偏离Hardy-Weinberg平衡的主要原因.纯合子过量对种群的遗传结构所造成的影响也会造成偏离Hardy-Weinberg平衡.另外,基于转录组开发的微卫星标记来源于表达序列,本文通过Blast检测了8对成功扩增的SSR 引物的侧翼区,发现7对引物所在序列可能具有功能(表5),因而会受到自然选择的影响,相关序列不再进行中性进化而偏离Hardy-Weinberg平衡.但换个角度而言,有“功能”的引物也提供了检验居群适应性进化的标记,从而使我们能估计环境因子的作用.
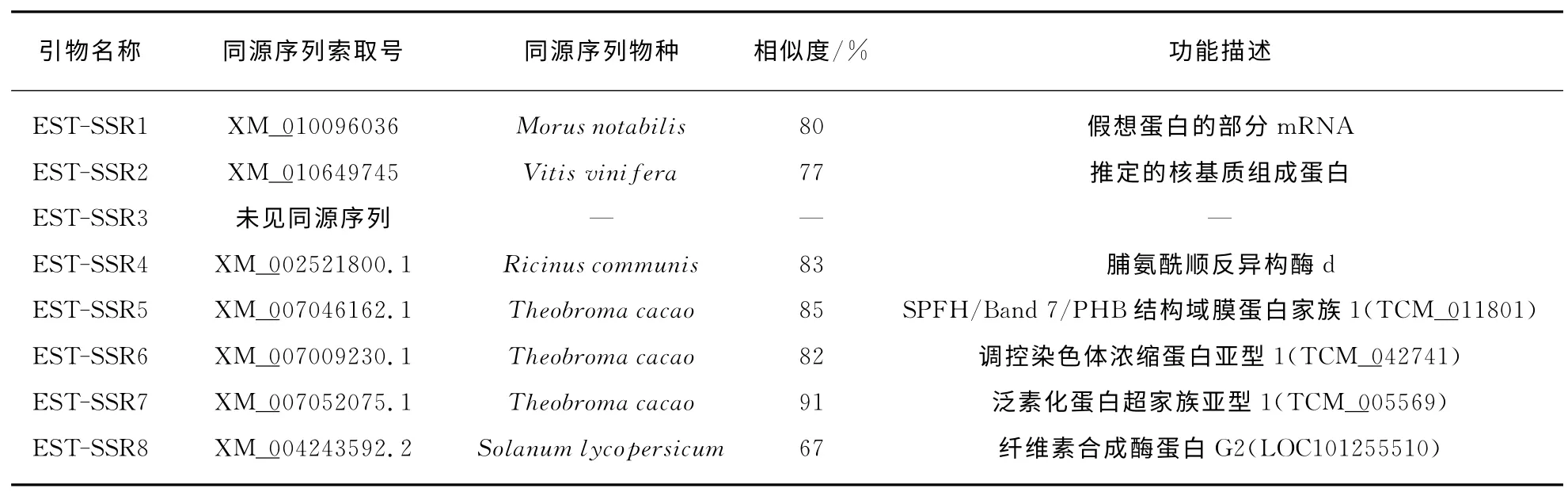
表5 崇左金花茶部分引物的功能预测Tab.5 The functional prediction of 8pairs of primers in C.chuongtsoensis
3.3 小结
SSR 一直以来都被认为是很好的分子标记,是构建遗传连锁图谱、研究群体遗传学、进行分子辅助育种、品种指纹图谱绘制的理想工具[28-30].在本研究中我们发现崇左金花茶转录组中存在丰富的SSR 位点,这说明相当一部分SSR 是转录的.RNA-seq促进了这种引物的挖掘,同时这些有“功能”的引物提供了检验居群适应性进化的标记,从而使我们能估计相关基因在群体遗传变异上的作用.
[1]黄连冬,梁盛业,叶创兴.四季花金花茶——金花茶一新种[J].广东园林,2014,36(1):69-70.
[2]游慕贤,游鸣飞.崇左金花茶——黄色茶花育种的希望[J].中国花卉园艺,2012(18):32-33.
[3]高继银,刘信凯,黄连冬,等.四季开花的茶花又一重大发现——金花茶家族中的佼佼者“崇左金花茶”[J].中国花卉盆景,2010(1):2-4.
[4]莫昭展.崇左金花茶的氨基酸成分研究[J].时珍国医国药,2013,24(6):1385-1386.
[5]杨舒婷,林 茂,王华新,等.崇左金花茶的组培诱导愈伤研究[J].安徽农业科学,2013(1):17-18.
[6]胡文质.崇左金花茶的栽培[J].花卉,2010(8):26.
[7]POWELL W,MACHRAY G C,PROVAN J.Polymorphism revealed by simple sequence repeats[J].Trends in Plant Science,1996,1(7):215-222.
[8]KANTETY R V,LA ROTA M,MATTHEWS D E,et al.Data mining for simple sequence repeats in expressed sequence tags from barley,maize,rice,sorghum and wheat[J].Plant Molecular Biology,2002,48(5):501-510.
[9]WEN M,WANG H,XIA Z,et al.Developmenrt of EST-SSR and genomic-SSR markers to assess genetic diversity in Jatropha Curcas L.[J].BMC Research Notes,2010,3(1):42.
[10]WEI W,QI X,WANG L,et al.Characterization of the sesame(Sesamum indicum L.)global transcriptome using Illumina paired-end sequencing and development of EST-SSR markers[J].BMC Genomics,2011,12(451).
[11]CHANG S,PURYEAR J,CAIRNEY J.A simple and efficient method for isolating RNA from pine trees[J].Plant Molecular Biology Reporter,1993,11(2):113-116.
[12]POREBSKI S,BAILEY L G,BAUM B R.Modification of a CTAB DNA extraction protocol for plants containing high polysaccharide and polyphenol components[J].Plant Molecular Biology Reporter,1997,15(1):8-15.
[13]徐 颖.山茶属植物个体内rDNA 多态性及杜鹃红山茶的空间遗传结构[D].上海:复旦大学生命科学学院,2012.
[14]KORESSAAR T,REMM M.Enhancements and modifications of primer design program Primer3[J].Bioinformatics,2007,23(10):1289-1291.
[15]LI X,ZHOU Y,WANG Y,et al.Isolation and characterization of twelve novel microsatellite markers for the sea-bulrush(Scirpus mariqueter Tang et Wang,Cyperaceae)[J].Conservation Genetics Resources,2014,6(3):677-678.
[16]许 汀,许 璐,王 昊,等.基于5’锚定PCR 法和磁珠富集法的西藏沙棘多态性SSR 引物开发[J].复旦学报(自然科学版),2014,48(4):543-549.
[17]YEH F C,YANG R,BOYLE T J,et al.PopGene32,Microsoft Windows-based freeware for population genetic analysis,version 1.32[M].Edmonton,Alberta,Canada:Molecular Biology and Biotechnology Centre,University of Alberta,2000.
[18]BROWN S M,HOPKINS M S,MITCHELL S E,et al.Multiple methods for the identification of polymorphic simple sequence repeats(SSRs)in sorghum[Sorghum bicolor(L.)Moench][J].Theoretical and Applied Genetics,1996,93(1-2):190-198.
[19]李明芳,郑学勤.开发SSR 引物方法之研究动态[J].遗传,2005,26(5):769-776.
[20]陈怀琼,隋 春,魏建和.植物SSR 引物开发策略简述[J].分子植物育种,2009,7(4):845-851.
[21]HUANG D,ZHANG Y,JIN M,et al.Characterization and high cross-species transferability of microsatellite markers from the floral transcriptome of Aspidistra saxicola(Asparagaceae)[J].Molecular Ecology Resource,2014,14(3):569-577.
[22]ZALAPA J E,CUEVAS H,ZHU H,et al.Using next-generation sequencing approaches to isolate simple sequence repeat(SSR)loci in the plant sciences[J].American Journal of Botany,2012,99(2):193-208.
[23]王 森,张 震,姜倪皓,等,半夏转录组中的SSR 位点信息分析[J].中药材,2014,37(9):1567-1570.
[24]王丽鸳,韦 康,张成才,等.茶树花转录组微卫星分布特征[J].作物学报,2014,40(1):80-85.
[25]鄢秀芹,鲁 敏,安华明.刺梨转录组SSR 信息分析及其分子标记开发[J].园艺学报,2015,42(2):341-349.
[26]李翠婷,张广辉,马春花,等.野三七转录组中SSR 位点信息分析及其多态性研究[J].中草药,2014,45(10):1468-1472.
[27]邓科君,张 勇,熊丙全,等.药用植物丹 参EST-SSR 标 记的鉴定[J].药学学报,2009,44(10):1165-1172.
[28]POWELL W,MORGANTE M,ANDRE C,et al.The comparison of RFLP,RAPD,AFLP and SSR(microsatellite)markers for germplasm analysis[J].Molecular Breeding,1996,2(3):225-238.
[29]ZIETKIEWICZ E,RAFALSKI A,LABUDA D.Genome fingerprinting by simple sequence repeat(SSR)-anchored polymerase chain reaction amplification[J].Genomics.1994,20(2):176-183.
[30]RONGWEN J,AKKAYA M S,BHAGWAT A A,et al.The use of microsatellite DNA markers for soybean genotype identification[J].Theoretical and Applied Genetics.1995,90(1):43-48.
