棉花Trihelix转录因子GhGT29基因的克隆及功能分析
2015-10-29李月刘晓东董永梅谢宗铭陈受宜
李月,刘晓东,董永梅,谢宗铭,陈受宜
棉花Trihelix转录因子基因的克隆及功能分析
李月1,刘晓东1,董永梅2,谢宗铭2,陈受宜3
1. 新疆农业大学农学院,农业生物技术重点实验室,乌鲁木齐830052;2. 新疆农垦科学院生物技术研究所,作物种质创新与基因资源利用兵团重点实验室,石河子 832000;3. 中国科学院遗传与发育生物学研究所,国家植物基因组重点实验室,北京100101
Trihelix转录因子在植物抵御各种逆境胁迫中扮演重要作用,克隆棉花Trihelix转录因子基因并分析其表达特性和功能,为最终利用转基因手段改良棉花抗逆性奠定基础。本文依据生物信息学分析,采用RT-PCR方法从陆地棉中克隆了一个Trihelix转录因子基因,命名为(GenBank登录号:JQ013097)。该基因最大开放阅读框(ORF)为1092 bp,编码363个氨基酸,预测分子量为40.9 kDa,等电点为5.45。SMART蛋白结构预测发现,该蛋白含有1个Trihelix家族典型的SANT结构域。系统进化树分析表明,属于Trihelix转录因子SH4亚家族,与拟南芥、2亲缘关系最近。实时荧光定量PCR结果表明,受高盐、干旱、低温胁迫和ABA诱导表达;在陆地棉的根、茎、叶、花、开花后当天胚珠以及开花后12 d(12 DPA)纤维中均有表达,其中在花中表达量最高,在茎中表达量最低。利用拟南芥原生质体系统进行分析,结果显示GhGT29主要定位于细胞核中,并且具有转录激活活性。以上结果表明基因可能参与棉花逆境信号通路中对抗逆功能基因表达的调控。
棉花;Trihelix转录因子;非生物胁迫;亚细胞定位;转录激活
高盐、低温和干旱等非生物逆境胁迫是影响植物生长和发育的重要限制因子。近年关于植物抗性机制的研究表明,转录因子在植物逆境胁迫信号传导中起重要作用[1]。转录因子是一类调控蛋白,通过特异序列组成的DNA结合功能域与下游调控基因的启动子结合,从而诱导或抑制下游抗逆基因的表达,参与生命体不同生理生化途径的调控。转录因子根据其DNA结合功能域的初级结构或三维结构的相似性和多聚化结构域的结构特点,可以分为不同的家族。目前,研究表明在植物中存在至少64个转录因子家族[2]。其中,NAC、MYB、bZIP和WRKY转录因子家族是研究最多而且广泛参与非生物胁迫的几个大家族,在拟南芥中都超过100多个成员[3]。
Trihelix转录因子是一类植物特有的转录因子小家族,大约有60多个亚家族成员[4],其中拟南芥中有30个,水稻中31个[5]。Trihelix转录因子根据其高度保守的三螺旋结构域而命名,该结构域能特异性地结合GT元件,所以该家族又被称为GT因子[6]。Trihelix转录因子的DNA结合功能域富含碱性、酸性氨基酸以及脯氨酸/谷氨酸,含3个α-螺旋,能形成螺旋-环-螺旋-环-螺旋构象,一般以1或2个形式存在于Trihelix蛋白的N端或C端[7],功能域中每个串联重复的内部疏水区都有一个色氨酸残基,即W-Xn-W-Xn-W,但是第3个保守的色氨酸残基容易发生变化,多被苯丙氨酸(F)、异亮氨酸(I)所取代。Kaplan-Levy等[4]基于GT蛋白结构域的保守氨基酸序列,将Trihelix家族分为GT-1、GT-2、GTγ、SH4和SIP1 5个亚家族,其中GT-2有2个DNA结构域,其他亚家族只有1个,这与Xie等[8]根据DNA结合功能域的数目将Trihelix转录因子分为GT-1、GT-2和GT-3型(GT-2型转录因子有2个DNA结构域)的分类相一致。虽然GT-1、GTγ、SH4和SIP1 4个亚家族只有1个DNA结合功能域,但这些结合功能域每个串联重复的内部疏水区的保守氨基酸残基有变化,GT-1、SH4和GT-2亚家族C端的第3个保守的氨基酸是色氨酸残基(W-Xn-W-Xn-),GTγ亚家族和GT-2的N端是苯丙氨酸(W-Xn-W-Xn-),在SIP1亚家族中是异亮氨酸(W-Xn-W-Xn-)[4]。GT元件作为光应答调控元件在豌豆(L.)叶片基因的启动子中首先被鉴定出来,目前已在拟南芥()、水稻(L.)、玉米(L.)、菠菜(L.)和大豆(L.)等多种植物中的启动子区克隆出多种功能的GT元件[9]。GT因子与相应的GT元件特异结合,调控基因的表达,发挥不同的生物学功能。Trihelix转录因子不仅调控光应答基因的表达[10~12],而且参与植物生长发育的各个过程,如花器官形态建成、胚囊、种子离层、表皮毛和气孔形成以及植物晚期胚胎发育[13~19]。另外研究还发现,细菌等病原物侵染植物后,一些Trihelix转录因子基因受到强烈诱导表达,如从水稻中鉴定出一个响应稻瘟病的RML1蛋白[20],以及在病原菌处理后快速诱导表达的基因[21]。结果显示Trihelix转录因子也参与植物对生物胁迫的应答反应。最近,越来越多的研究表明,Trihelix转录因子GT-2亚家族作为干旱、低温及盐害等非生物胁迫下的主要响应因子,在植物抗逆能力综合改良中也具有重要作用。例如:转基因的过表达植株增强了对冷害和盐胁迫的耐受性[22];杨树()基因和拟南芥基因通过控制叶片气孔密度来调节植物的水分利用率,从而提高转基因植株的耐旱能力[23, 24];在大豆中,过量表达和可以显著提高拟南芥对盐害,干旱和冷害的抗性[8]。
棉花(L.)作为重要的纤维和油料作物,在全球范围内广泛种植。与水稻、小麦、玉米等主要作物相比,棉花具有较高的抗旱、耐盐性。尽管如此,由于全球气候变化和环境污染,非生物胁迫已成为影响棉花正常生长和产量的主要限制因子[25]。因此,揭示棉花耐逆的分子机理、克隆与逆境胁迫相关的重要功能基因对于棉花耐逆性和广适应性的基因工程改良,扩大棉花生产具有重要的意义。本实验室在前期研究中对棉花Trihelix转录因子进行了功能分析,发现其对低温、高盐、干旱胁迫和外源ABA处理均有不同程度的胁迫应答[26]。本文采用表达序列标签(EST)的电子拼接结合RT-PCR技术从陆地棉中克隆出1个新的Trihelix转录因子基因,对于是否响应非生物胁迫应答以及在非生物胁迫调控中具有何种功能进行了分析,并对其序列特征、亚细胞定位及转录激活活性进行研究,为进一步了解Trihelix转录因子基因家族功能及其分子机制提供理论依据,以期为棉花抗逆性基因工程改良奠定基础。
1 材料和方法
1.1 植物材料
陆地棉品种新陆早26号由新疆农垦科学院生物技术研究所种质资源室提供。
1.2 方法
1.2.1 播种、生长条件和胁迫处理
将新陆早26号种子均匀播种于装满蛭石的营养钵中,在植物培养间培养幼苗,培养温度为23℃、湿度为65%、光周期为16 h/8 h。选取生长一致的15 d苗龄(一对真叶期)的棉花,参照文献[26]已报道的棉花胁迫处理方法,分别进行干旱、高盐和4℃低温和外源ABA(100 μmol/L)处理,以正常生长的幼苗为对照。同时将另一部分正常生长的幼苗移至土壤进一步生长,于花期分别采集花、开花后(Days post anthesis, DPA)当天(0 DPA)胚珠以及开花后12 d (12 DPA)纤维,提取总RNA,用于组织表达分析。上述各种胁迫处理后,分别于0、1、3、6和12 h采集叶片,同时采集对照组的根、茎、叶,将以上采集的所有材料迅速置于液氮,用于总RNA提取。
1.2.2 总RNA的提取和cDNA的制备
采用改良的CTAB法,参照文献[27]的方法,提取棉花不同胁迫处理时间点叶片和不同组织的总RNA,用RNase-Free DNase I(TaKaRa公司)去除基因组DNA污染,并在Thermo Scientific(NanoDrop 1000)分光光度计上进行浓度和质量检测,1.2%琼脂糖凝胶电泳检测RNA的完整性。按照M-MLV反转录试剂盒(Promega公司)操作说明合成单链cDNA,于-20℃保存,用于基因克隆、基因胁迫响应和组织表达分析。
1.2.3 陆地棉基因的克隆
根据Plant Transcription Factor Database[28]中棉花Trihelix转录因子EST序列,将Ghi010304的EST序列所代表的基因命名为,以此EST序列在已公布的棉花基因组数据库中比对,获得了cDNA序列。根据最大开放阅读框(ORF)设计引物:GT29HⅠRⅠF:5′-GCCGGATCCGAATTCATGGCC TCGGAGCAGTTAAGC-3′;GT29ⅠR: 5′-GCCGTC- GACTAACTTGTCGGCGATCCTCC-3′。
在引物上引入HⅠ、RⅠ和Ⅰ酶切位点,以新陆早26号叶片 cDNA为模板,以高保真聚合酶TransStar KD Plus(TransGen)进行RT-PCR扩增。扩增产物连接构建到Peasy-Blunt Zero克隆载体(TransGen公司)上,构建Peasy T-GhGT29载体,并测序验证。高保真TransStar KD Plus(TransGen)扩增体系为:5×TransStar KD Plus Buffer 10 μL,2 mmol/L dNTP 5 μL,TransStar KD Plus DNA Polymerase 1 μL,模板 1 μL,正反向引物各1 μL,补充水到50 μL。扩增条件:94℃ 2 min;94℃ 10 s,60℃ 30 s,68℃ 2 min,30个循环;最后再68℃延伸10 min。
1.2.4 陆地棉GhGT29蛋白序列分析及系统进化树的构建
利用DNAStar软件将基因ORF翻译成蛋白序列,并用EXPASy的ProtParam tool在线程序(http://web.expasy.org/cgi-bin/protparam/protparam)预测蛋白的分子量和等电点。用Psort(http: //www.psort.org/)在线工具完成蛋白的亚细胞定位预测。ScanProsit和SMART(http://smart.embl-heidelberg. de/)程序对基因进行蛋白家族预测,以确定该基因所属的蛋白家族类别。利用NCBI中BlastP和DNAMAN软件对GhGT29蛋白序列进行氨基酸序列相似性分析和多重序列比对。
Kaplan-Levy等[4]根据GT蛋白结构域的保守氨基酸序列,将Trihelix家族分为GT-1、GT-2、GTγ、SH4和SIP1 5个亚家族。本文以此为基础,分别从GenBank中下载拟南芥()、水稻(L.)、白杨()、大豆(L.)等Trihelix转录因子亚家族蛋白序列,并与GhGT29蛋白一起,利用ClustalX和MEGA5.0软件构建系统进化树。
1.2.5 陆地棉基因的表达分析
采用CTAB法提取经高盐、干旱、低温和ABA处理的不同时间点的叶片和不同生育时期各组织的总RNA,用反转录试剂盒合成cDNA序列。根据基因的EST序列设计qPCR引物:
GhGT29F:5′-AACCAATGGAGACGAAGAACC-3′;
GhGT29R:5′- AGCTATCGGCGTGGTCTTTACG-3′。
以棉花基因[29](GenBank登录号:DQ116441)引物U7F(5′-AGAGGTCGAGTCTTCGG- ACA-3′)和U7R(5′-GCTTGATCTTCTTG GGCTTG-3′)为qPCR内参引物。根据TOYOBO的SYBR qPCR Mix Kit说明操作,反应体系:一链cDNA 2 μL, 10 μmol/L正反向引物各0.5 μL,SYBR qPCR Mix 10 μL,用灭菌超纯水补齐到20 μL。利用Roche LightCycler 480II实时定量PCR仪进行PCR扩增。反应条件:95℃ 30 s;95℃ 5 s,58℃ 30 s,72℃ 30 s,40个循环;72℃ 30 s。数据采用2-△△CT法分析数据,确定基因的相对表达量。利用DPS(7.05)软件对定量数据进行单因素方差分析。
1.2.6 GhGT29蛋白在拟南芥原生质体中的亚细胞定位分析
利用pBI221作为瞬时表达载体,Ⅰ和HⅠ限制性内切酶酶切Peasy T-GhGT29质粒和pBI221-GFP载体,凝胶回收酶切片段,将基因的全长序列与基因融合,构建由35S启动子驱动的融合表达蛋白GhGT29-GFP,由空载体pBI221-GFP做为正对照。参照http://genetics. mgh.harvard.edu/sheenweb/protocols/方法,将GhGT29- GFP和pBI221-GFP载体质粒转化拟南芥原生质体,用Leica TCS SP5显微镜观察分析。
1.2.7 GhGT29蛋白原生质体转录激活活性分析
利用Ⅰ和HⅠ限制性内切酶将扩增的基因全长序列进行双酶切,酶切片段融合到GAL4效应基因载体上,构建GAL4DBD-GhGT29融合蛋白。参照Hao等[30]的方法分析GhGT29蛋白的转录激活活性。该方法的双荧光素酶报告基因测试系统(Dual-luciferase reporter assay system)包含报告基因(Reporter)和效应基因(Effecter)两个载体系统,其中报告基因载体系统5×GAL4-LUC具有5个串联的GAL4转录因子结合位点(5×GAL4-binding site),并驱动萤火虫荧光素酶报告基因的表达。效应基因载体系统是在pRT107载体的基础上加上GAL4转录因子的DNA结合功能域(GAL4DBD),该系统由35S启动子驱动(图1)。如果待研究的蛋白具有转录激活域,它与GAL4DBD的融合蛋白就能与GAL4结合位点结合,激活报告基因的表达,产生的荧光素酶发出的荧光可以被荧光仪测定。pTRL载体是内参对照,含有组成型表达海洋腔肠(Renilla)荧光素酶报告基因,作为内参控制转化效率。以两个报告基因表达后发生的荧光量比值来测试蛋白的转录调控能力。将全长基因构建到效应基因载体上。以空GAL4DBD为阴性对照,以转化的GAL4的DNA结合/激活结构域载体质粒VP16作为阳性对照。将上述效应基因载体、报告基因载体和内参(pTRL)载体按照6:6:2(μg)的比例共转化拟南芥叶片原生质体细胞,检测出的相对荧光强度即代表转录激活活性的强度。

图1 拟南芥原生质体瞬时检测中的报告基因载体系统和效应基因载体系统
2 结果与分析
2.1 陆地棉基因的克隆与序列分析
根据Plant Transcription Factor Database中棉花Trihelix转录因子EST序列,以蛋白ID号为Ghi010304的EST序列在已公布的棉花基因组数据库(http://www.phytozome.net/)中比对,获得了一个最大开放阅读框为1092 bp的cDNA序列,在ORF上下游设计引物进行RT-PCR扩增,其产物经克隆测序验证,并将该基因命名为(GenBank登录号:JQ013097)。
根据测序结果,利用在线程序EXPASy的ProtParam tool(http://web.expasy.org/cgi-bin/protparam/ protparam)和SMART(http://smart.embl-heidelberg.de/)对GhGT29蛋白进行生物信息学分析。基因编码一个363氨基酸的蛋白,预测蛋白分子量为40.9 kDa,pI(等电点)为5.45,带正电荷的氨基酸(Arg+Lys)占13.7%,带负电荷的氨基酸(Asp+Glu)占15.4%,蛋白不稳定系数(Instability index)为48.32,说明该蛋白是一种不稳定蛋白。经SMART预测,结果表明该蛋白含有1个Trihelix SANT结构域,由47位亮氨酸(L)至118位的谷氨酰胺(Q)组成。
2.2 GT蛋白多序列比对及系统进化树分析
利用DNAMAN软件,对陆地棉(GhGT29)、拟南芥(AtASIL1、AtASIL2、AtGT-3a和AtGT-1)、水稻(OsGTγ-1、OsGTγ-2、OsGTγ-3和OsGT-2)和大豆(GmGT-2A和GmGT-2B)GT蛋白DNA结合功能域进行多重序列比对。结果表明(图2),GhGT29蛋白具有1个功能结构域,含有3个α-螺旋,该功能域符合Trihelix转录因子的DNA结合功能域的特征,富含碱性、酸性氨基酸以及脯氨酸/谷氨酸,每个串联重复的内部疏水区都有一个色氨酸残基(-Xn--Xn-),并且具有GT-1、SH4亚家族的结构特点,因此推测GhGT29可能属于GT-1、SH4亚家族成员。

图2 GhGT29蛋白的氨基酸序列比对
*表示高度保守的氨基酸残基用;:表示部分保守的氨基酸残基;虚线表示Trihelix结构域中的螺旋结构;黑线表示可能的核定位信号;At:;Os:;Gm:;Gh;。
采用MEGA5邻近相连法对陆地棉GhGT29转录因子进行序列比对并构建系统进化树。结果表明,GhGT29与拟南芥AtSH4-like1、AtSH4-like2亲缘关系比较近,聚类为一类(图3),AtSH4-like1、AtSH4-like2基因属于SH4亚家族,因此将GhGT29归为SH4亚家族,这与利用SMART蛋白结构预测和氨基酸序列比对推断GhGT29属于SH4亚家族的结果一致。

图3 GhGT29蛋白与其他植物Trihelix转录因子蛋白的进化树分析
GT-1亚家族包括AtGT-1(At1g13450)、AtGT-4(At3g25990)和OsRML1(AL627350);GT-2亚家族包括AtGT-2(At1g76890)、AtGTL1(At1g33240)、AtGT2L(At5g28300)、AtPTL(At5g03680)、GmGT-2A(EF221753)、GmGT-2B(EF221754)和PtaGTL1(JN113092);SH4亚家族包括AtSH4-like1(At2g35640)和AtSH4-like2 (At1g31310);GTγ亚家族包括OsGTγ-1(Os02g33770)、OsGTγ-2 (Os11g06410)和OsGTγ-3(Os12g06640);SIP亚家族包括AtASIL1(At1g54060)、AtASIL2(At3g14180)和AtFIP2(At4G17060)。
2.3基因的诱导表达和组织表达分析
利用实时荧光定量PCR对陆地棉基因在各种非生物胁迫下和不同组织中的表达进行了分析。结果表明(图4),基因对高盐、干旱、低温和ABA处理都有不同程度的应答。在200 mmol /LNaCl处理后,基因在0 h、1 h和6 h的表达量都比较低,3 h和12 h的表达量达到最大,是其他时间段的6倍。干旱处理下,基因在1 h的表达量最大,在3 h、6 h 和12 h的表达量基本一致,无显著性差异,略高于0 h的表达。基因对冷胁迫有微弱应答,总体上呈降低的趋势,在3 h表达量达到最大,之后随着胁迫处理时间的延长,表达量逐渐下降,在12 h降到最低。ABA处理后,基因在1 h的表达量最高,在3 h表达量急剧下降,达到最低,随后随着胁迫处理时间的延长,基因表达又逐渐升高,在12 h恢复到和0 h一样。

图4 GhGT29基因在4种不同胁迫处理下的表达模式分析
以未处理(0 h)的表达量做为参照,不同胁迫处理时间相同小写字母表示差异不显著(>0.05),不同小写字母之间表示差异显著(<0.05)。
同时,利用实时荧光定量PCR分析了基因的时空表达模式。结果表明(图5),基因在根、茎、叶、花、0 DPA胚珠及12 DPA纤维中都有表达,但在根、茎、叶中的表达量处于较低水平,在花中的表达量最高,其次是纤维和胚珠,推测基因除参与棉花非生物胁迫应答外,还可能参与棉花的纤维发育。
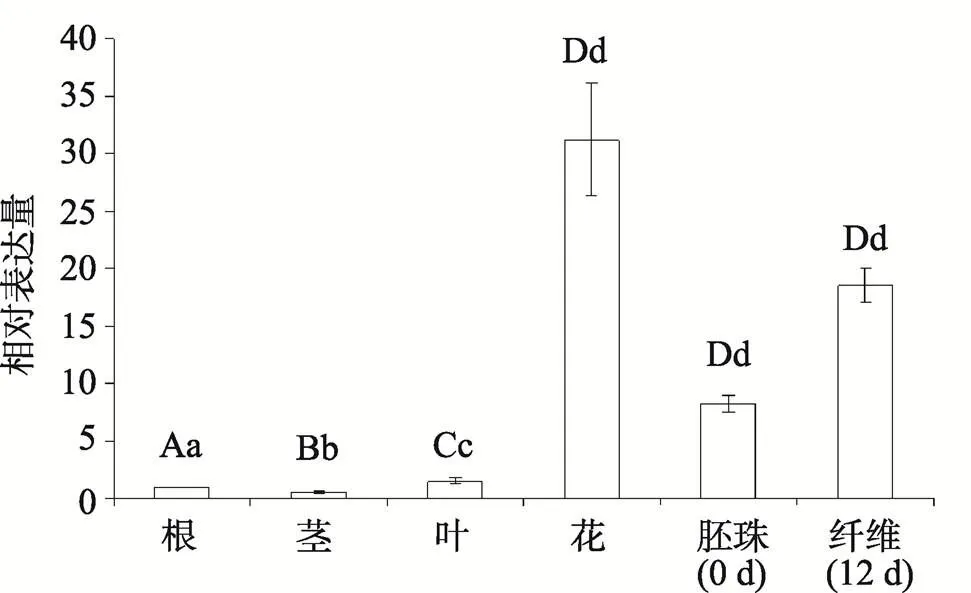
图5 GhGT29基因在不同组织表达量的比较
以根的表达量做为参照,不同组织相同小写字母表示差异不显著(>0.05),不同大写字母之间表示差异极显著(<0.01),不同小写字母之间表示差异显著(<0.05)。
2.4基因编码蛋白的亚细胞定位
经https://www.predictprotein.org/和http:// www.bioinfo.tsinghua.edu.an/SubLoc/网站预测GhGT29蛋白具有核定位信号。因此本研究利用GFP蛋白融合方法对GhGT29蛋白的细胞定位进行了实验验证。结果表明,对照35S:GFP的绿色荧光蛋白分布在整个细胞,而GhGT29蛋白仅在细胞核处检测到荧光,表明GhGT29蛋白定位在细胞核(图6),这与GhGT29蛋白定位预测的结果相一致。

图6 GhGT29蛋白在拟南芥原生质体中的亚细胞定位分析
图像是由激光共聚焦显微镜扫面GFP和叶绿体两种波长荧光拍照而成,其中GFP荧光的激发波长488 nm,发射波509 nm;叶绿体自发荧光的激发波长448 nm,发射波647 nm,标尺:200 μm。
2.5 GhGT29转录因子的转录激活活性分析
利用双荧光素酶报告基因测试系统,在拟南芥原生质体中检测陆地棉GhGT29蛋白的转录激活活性。结果表明(图7),阳性对照VP16蛋白调控的报告基因表达量与阴性对照相比差异极显著,GAL4DBD-GhGT29的报告基因表达量也明显高于阴性对照,且呈现极显著差异,说明GhGT29蛋白具有转录激活活性。

图7 GhGT29蛋白转录激活活性分析
阴性对照:35S:GAL4DBD;阳性对照:35S:GAL4DBD-VP16;误差线为标准差(sd);**表示<0.01。
3 讨论
由于全球耕地面积的减少,粮棉争地矛盾加剧,积极开发旱地、盐碱地植棉潜力,将成为棉花生产保持相对稳定的重要应对策略,因此培育多抗棉花品种已是当前棉花育种的重要目标。一些转录因子通过调控下游一系列耐逆基因的表达,进而增强植物的抗逆性,因此在植物耐逆改良中具有重要作用。近年来,已从棉花自身分离了多个耐逆关键转录因子基因,如CCCH-type的锌指结构基因、WRKY转录因子基因,这些基因超表达均能增强植物的抗逆性[31,32]。Trihelix转录因子作为植物特有的一类转录因子小家族,已经在多种植物中被鉴定出响应冷害、干旱和高盐等非生物逆境胁迫以及ABA、SA等各种激素的处理。如大豆63个基因中有11个基因参与生物或非生物胁迫应答反应[33]。其中将大豆和[8],拟南芥[22]、基因[23]、[5],水稻GTγ亚家族的、、基因[34]以及杨树的[24]基因转入拟南芥后,都能增强过表达转基因植株对非生物胁迫的耐受性,表明Trihelix转录因子在植物抵御非生物胁迫中起重要的调控作用。本文通过EST序列的电子拼接结合RT-PCR方法从陆地棉新陆早26号中克隆了一个Trihelix转录因子基因,命名为。SMART分析表明,该蛋白只有1个SANT结构域。同时系统进化树分析GhGT29与拟南芥TrihelixSH4亚家族的AtSH4-like1和AtSH4-like2共同组成一个分支,表明GhGT29属于SH4亚家族。
目前研究表明,SH4亚家族主要参与调控种子离层发育和植物早期胚胎发育,如水稻()基因[35]。本研究通过实时荧光定量PCR分析表明,Trihelix转录因子SH4亚家族的基因对干旱、高盐、低温和ABA处理均有应答,推测基因作为棉花中一个新的SH4亚家族基因,在非生物胁迫信号传递过程中,可能是一个信号连接点,通过依赖于ABA途径参与不同逆境信号传导。组织特异性表达分析显示,基因在陆地棉的花器官和纤维中的表达量比较高,推测基因除可能参与棉花非生物胁迫应答外,还可能参与棉纤维的发育。
转录因子在细胞质中合成,由核定位信号引导进入细胞核后才能发挥调控其他基因表达的作用。通过在线软件预测,GhGT29定位在细胞核中,亚细胞定位实验结果表明,GhGT29蛋白确实定位在细胞核内,与转录因子是核定位特征相一致。转录激活域和转录抑制域是转录因子调节下游靶基因表达的区域,对转录因子功能的执行至关重要。转录因子通过转录激活域提高下游基因的表达量,通过转录抑制域降低下游基因的表达,因此明确Trihelix转录因子基因在信号传递中的调控作用,对于解析其生物学功能有重要意义。Hao等[30]利用双荧光素酶基因报告测试系统在拟南芥中研究大豆NAC转录因子和基因的转录活性,发现前者具有转录激活活性,后者有转录抑制活性,其中NRAD结构域是NAC型转录因子的转录活性区域。目前关于Trihelix类蛋白的转录激活活性已在多种植物中进行了研究。水稻OsGT-2蛋白在体内具有转录激活活性[36],大豆GmGT2B具有转录激活活性,其N端的1-153残基是其激活中心[8],拟南芥AtGT2L的 N端结构域(1-383)是其激活中心[22]。拟南芥AtGT-4具有转录激活活性,C端的114-372残基是其激活中心[5]。本研究表明,GhGT29蛋白具有转录激活的活性,但是关于其具体的激活中心,还有待于进一步研究。
本研究在新陆早26中克隆了一个Trihelix转录因子基因,该基因编码蛋白含有1个SANT结构域,属于Trihelix转录因子家族的SH4亚家族;主要定位于细胞核中;通过其转录激活活性调控下游基因的表达;响应高盐、干旱、低温和ABA处理;在棉纤维和花中的表达量较高。目前已经将基因转化野生型拟南芥,并获得了转基因纯系植株,对其具体的抗逆生物学功能还在进一步研究中。
参考文献
[1] Ma LF, Zhang JM, Huang GQ, Li Y, Li XB, Zheng Y. Molecular characterization of cotton C-repeat/ dehydration-responsive element binding factor genes that are involved in response to cold stress., 2014, 41(7): 4369–4379.
[2] Pérez-Rodríguez P, Riaño-Pachón DM, Corrêa LGG, Rensing SA, Kersten B, Mueller-Roeber B. PlnTFDB: updated content and new features of the plant transcription factor database., 2010, 38(Database issue): D822–D827.
[3] Llorca CM, Potschin M, Zentgraf U. bZIPs and WRKYs: two large transcription factor families executing two different functional strategies., 2014, 5: 169.
[4] Kaplan-Levy RN, Brewer PB, Quon T, Smyth DR. The Trihelix family of transcription factors-light, stress and development., 2012, 17(3): 163–171.
[5] Wang XH, Li QT, Chen HW, Zhang WK, Ma B, Chen SY, Zhang JS. Trihelix transcription factor GT-4 mediates salt tolerance via interaction with TEM2 in., 2014, 14: 339–353.
[6] 罗军玲, 赵娜, 卢长明. 植物Trihelix转录因子家族研究进展. 遗传, 2012, 34(12): 1551–1560.
[7] Qin Y, Ma X, Yu GH, Wang Q, Wang L, Kong LR, Kim W, Wang HW. Evolutionary history of Trihelix family and their functional diversification., 2014, 21(5): 499–510.
[8] Xie ZM, Zou HF, Lei G, Wei W, Zhou QY, Niu CF, Liao Y, Tian AG, Ma B, Zhang WK, Zhang JS, Chen SY. Soybean Trihelix transcription factors GmGT-2A and GmGT-2B improve plant tolerance to abiotic stresses in transgenic., 2009, 4(9): e6898.
[9] 关秋玲, 陈焕新, 张毅, 李秋莉. 植物GT元件和GT因子的研究进展. 遗传, 2009, 31(2): 123–130.
[10] Nagata T, Niyada E, Fujimoto N, Nagasaki Y, Noto K, Miyanoiri Y, Murata J, Hiratsuka K, Katahira M. Solution structures of the Trihelix DNA-binding domains of the wild-type and a phosphomimetic mutant ofGT-1: mechanism for an increase in DNA-binding affinity through phosphorylation., 2010, 78(14): 3033–3047.
[11] Dehesh K, Bruce WB, Quail PH. A trans-acting factor that binds to a GT-motif in a phytochrome gene promoter., 1990, 250(4986): 1397–1399.
[12] Gilmartin PM, Memelink J, Hiratsuka K, Kay SA, Chua NH. Characterization of a gene encoding a DNA binding protein with specificity for a light-responsive element., 1992, 4(7): 839–849.
[13] O'Brien M, Kaplan-Levy RN, Quon T, Sappl PG, Smyth DR. PETAL LOSS, a Trihelix transcription factor that represses growth inthaliana, binds the energy-sensing SnRK1 kinase AKIN10., 2015, 66(9): 2475–2485.
[14] Weselake RJ, Taylor DC, Rahman MH, Shah S, Laroche A, McVetty PBE, Harwood JL. Increasing the flow of carbon into seed oil., 2009, 27(6): 866–878.
[15] Gao MJ, Lydiate DJ, Li X, Lui HL, Gjetvaj B, Hegedus DD, Rozwadowski K. Repression of seed maturation genes by a Trihelix transcriptional repressor in Arabidopsis seedlings., 2009, 21(1): 54–71.
[16] Gao MJ, Li X, Lui HL, Gropp GM, Lydiate DD, Wei S, Hegedus DD. ASIL1 is required for proper timing of seed filling in., 2011, 6(12): 1886–1888.
[17] Barr MS, Willmann MR, Jenik PD. Is there a role for Trihelix transcription factors in embryo maturation?, 2012, 7(2): 205–209.
[18] Lampugnani ER, Kilinc A, Smyth DR. PETAL LOSS is a boundary gene that inhibits growth between developing sepals inthaliana., 2012, 71(5): 724–735.
[19] Zhou Y, Lu DF, Li CY, Luo JH, Zhu BF, Zhu JJ, Shangguan YY, Wang ZX, Sang T, Zhou B, Han B. Genetic control of seed shattering in rice by the APETALA2 transcription factor SHATTERING ABORTION., 2012, 24(3): 1034–1048.
[20] Wang R, Hong GF, Han B. Transcript abundance of rml1, encoding a putative GT1-like factor in rice, is up-regulated by Magnaporthe grisea and down-regulated by light., 2004, 324: 105–115.
[21] Park HC, Kim ML, Kang YH, Jeon JM, Yoo JH, Kim MC, Park CY, Jeong JC, Moon BC, Lee JH, Yoon HW, Lee SH, Chung WS, Lim CO, Lee SY, Hong JC, Cho MJ. Pathogen- and NaCl-induced expression of the SCaM-4 promoter is mediated in part by a GT-1 box that interacts with a GT-1-like transcription factor., 2004, 135(4): 2150−2161.
[22] Xi J, Qiu YJ, Du LQ, Poovaiah BW. Plant-specific Trihelix transcription factor AtGT2L interacts with calcium/calmodulin and responds to cold and salt stresses., 2012, 185–186: 274–280.
[23] Yoo CY, Pence HE, Jin JB, Miura K, Gosney MJ, Hasegawa PM, Mickelbart MV. The Arabidopsis GTL1 transcription factor regulates water use efficiency and drought tolerance by modulating stomatal density via transrepression of SDD1., 2010, 22(12): 4128–4141.
[24] Weng H, Yoo CY, Gosney MJ, Hasegawa PM, Mickelbart MV. Poplar GTL1 is a Ca2+/calmodulin-binding transcription factor that functions in plant water use efficiency and drought tolerance., 2012, 7(3): e32925.
[25] Ahuja I, de Vos RCH, Bones AM, Hall RD. Plant molecular stress responses face climate change., 2010, 15(12): 664–674.
[26] 李月, 孙杰, 陈受宜, 谢宗铭. 棉花转录因子GhGT30基因的克隆及转录功能分析. 作物学报, 2013, 39(5): 806–815.
[27] 胡根海, 喻树迅. 利用改良的CTAB法提取棉花叶片总RNA. 棉花学报, 2007, 19(1): 69–70.
[28] Zhang H, Jin JP, Tang L, Zhao Y, Gu XC, Gao G, Luo JC. PlantTFDB 2.0: update and improvement of the comprehensive plant transcription factor database., 2011, 39(Suppl. 1): D1114–D1117.
[29] Shi YH, Zhu SW, Mao XZ, Feng JX, Qin YM, Zhang L, Cheng J, Wei LP, Wang ZY, Zhu YX. Transcriptome profiling, molecular biological, and physiological studies reveal a major role for ethylene in cotton fiber cell elongation., 2006, 18(3): 651–664.
[30] Hao YJ, Song QX, Chen HW, Zou HF, Wei W, Kang XS, Ma B, Zhang WK, Zhang JS, Chen SY. Plant NAC-type transcription factor proteins contain a NARD domain for repression of transcriptional activation., 2010, 232(5): 1033–1043.
[31] Zhou T, Yang XY, Wang LC, Xu J, Zhang XL. GhTZF1 regulates drought stress responses and delays leaf senescence by inhibiting reactive oxygen species accumulation in transgenic., 2014, 85(1–2): 163–177.
[32] Shi WN, Hao LL, Li J, Liu DD, Guo XQ, Li H. The Gossypium hirsutum WRKY gene GhWRKY39-1 promotes pathogen infection defense responses and mediates salt stress tolerance in transgenic., 2014, 33(3): 483–498.
[33] Osorio MB, Bücker-Neto L, Castilhos G, Turchetto-Zolet AC, Wiebke-Strohm B, Bodanese-Zanettini MH, Margis-Pinheiro M. Identification andcharacterization of soybean Trihelix-GT and bHLH transcription factors involved in stress responses., 2012, 35(Suppl. 1): 233–246.
[34] Fang YJ, Xie KB, Hou X, Hu HH, Xiong LZ. Systematic analysis of GT factor family of rice reveals a novel subfamily involved in stress responses., 2010, 283(2): 157–169.
[35] Li CB, Zhou AL, Sang T. Rice domestication by reducing shattering., 2006, 311(5769): 1936–1939.
[36] Dehesh K, Hung H, Tepperman JM, Quail PH. GT-2: A transcription factor with twin autonomous DNA-binding domains of closely related but different target sequence specificity., 1992, 11(11): 4131–4144.
Cloning and functional analysis of the cotton Trihelix transcription factor
Yue Li1, Xiaodong Liu1, Yongmei Dong2, Zongming Xie2, Shouyi Chen3
Trihelix transcription factors are important proteins involved in response to abiotic stresses in plants. Understanding the molecular mechanisms of Trihelixin cottons will lay the foundation to improve stress tolerance by gene engineering. In this study, a gene encoding Trihelix transcription factor was isolated in upland cottons using reverse transcription PCR according to bioinformatic analysis. The gene was named as(GenBank accession No. JQ013097), which was 1 092 bp, contained a 1 089 bp open reading frame and encoded a protein of 363 amino acids with a predicted molecular weight of 40.9 kDa and a isoelectric point of 5.45. SMART analysis showed GhGT29 contained one typical SANT motif. Phylogenetic analysis showed thatbelonged to the SH4 subfamily of the Trihelix family and was most closely related toand. Quantitative real-time PCR (qRT-PCR) analysis revealed thatwas induced by high salt, drought, cold and abscisic acid. The expression profile also revealed thatwas constitutively expressed in all tested tissues, such as roots, stems, leaves, flowers, ovules (0 DPA) and fibers (12 DPA). The expression level ofwas the highestin flowers and the lowest in stems. Using theprotoplasts assay system, we found that the GhGT29 protein was located in cell nuclei and had trans-activation activity. These results revealed thatmight be involved in the regulation of stress resistance-related genes in stress signaling pathways in upland cottons.
cotton; Trihelix transcription factor; abiotic stress; subcellular localization; trans-activation activity
2015-07-15;
2015-09-07
国家转基因生物新品种培育科技重大专项(编号:2009ZX08009-090B),国家自然科学基金项目(编号:31470289)和新疆农业大学校前期课题(编号:XJAU201311)资助
李月,博士,讲师,研究方向:植物分子生物学。E-mail:liyue6905@126.com
谢宗铭,研究员,博士,研究方向:棉花分子育种。E-mail:xiezmchy@163.com陈受宜,研究员,博士,研究方向:植物对逆境应答的分子机制和大豆基因组学。E-mail:sychen@genetics.ac.cn
10.16288/j.yczz.15-320
网络出版时间: 2015-9-29 8:49:53
URL: http://www.cnki.net/kcms/detail/11.1913.R.20150929.0849.004.html
(责任编委: 李付广)
