遗传风险评分在复杂疾病遗传学研究中的应用
2015-10-29牛大彦严卫丽
牛大彦,严卫丽
遗传风险评分在复杂疾病遗传学研究中的应用
牛大彦,严卫丽
复旦大学附属儿科医院临床流行病学教研室,上海 201102
心血管疾病、2型糖尿病、原发性高血压、哮喘、肥胖、肿瘤等复杂疾病在全球范围内流行,并成为人类死亡的主要原因。越来越多的人开始关注遗传易感性在复杂疾病发病机制中的作用。至今,与复杂疾病相关的易感基因和基因序列变异仍未完全清楚。人们希望通过遗传关联研究来阐明复杂疾病的遗传基础。近年来,全基因组关联研究和候选基因研究发现了大量与复杂疾病有关的基因序列变异。这些与复杂疾病有因果和(或)关联关系的基因序列变异的发现促进了复杂疾病预测和防治方法的产生和发展。遗传风险评分(Genetic risk score,GRS)作为探索单核苷酸多态(Single nucleotide polymorphisms,SNPs)与复杂疾病临床表型之间关系的新兴方法,综合了若干SNPs的微弱效应,使基因多态对疾病的预测性大幅度提升。该方法在许多复杂疾病遗传学研究中得到成功应用。本文重点介绍了GRS的计算方法和评价标准,简要列举了运用GRS取得的系列成果,并对运用过程中所存在的局限性进行了探讨,最后对遗传风险评分的未来发展方向进行了展望。
遗传风险评分;复杂疾病;单核苷酸多态
复杂疾病,如心血管疾病、2型糖尿病、原发性高血压、肥胖以及哮喘等疾病的遗传不遵循孟德尔遗传模式,其发生受多个微效基因及环境因素的影响,并存在基因–基因、基因–环境间相互作用[1]。复杂疾病在全球范围内广泛流行,严重危害着人类的健康,人们迫切希望从根本上找到这些疾病的发病机理,从而为疾病的诊断、治疗以及预防提供基础和保障。
随着“人类基因组计划”和“人类基因组单体型图计划”的相继完成,遗传因素在疾病中扮演的角色逐渐被发掘出来。为了更大限度、更有效地探索遗传因素在复杂疾病中的作用,人们构建出了一系列分析方法如单体型分析、全基因组关联研究(Genome-wide association study, GWAS)、遗传风险评分(Genetic risk score,GRS)等分析遗传序列变异与复杂疾病表型之间的关系。大量的模拟研究[2]和实验研究[3]证明,在研究多个处于连锁不平衡的位点与复杂疾病的关联时, 基于单体型的关联性研究要比单个位点的分析更加高效。利用分子实验推断每个个体的单体型态较为昂贵,且费时费力,虽然根据一些算法可以推断个体单体型态的分布,但是这造成了信息浪费、低效以及偏倚,也不能通过统计的方法找出真正有功能的多态位点。随着高通量基因检测技术的快速发展,GWAS开始进入研究人员的视野,它有着极高的效费比,能够从整体上对全基因组进行全面而综合地评估和考察, 并且事先不需要基因的背景知识, 即不需要在研究前建立任何假设,广泛被大家接受。鉴于这些优势,GWAS被广泛应用于复杂疾病的遗传学研究[4]并取得了一系列成果,如基因与体重指数(肥胖)关系的发现[5]、肺结核易感位点的发现[6]等等。但是,GWAS也面临着一些挑战,比如复杂疾病相关基因位点数目众多,每一个位点只起到很小的作用,这给判断基因序列变异和复杂疾病之间的关系带来了很大困难。
GRS能整合多个单核苷酸多态(Single nucleotide polymorphisms,SNPs)的综合信息来评价基因序列变异和疾病之间的联系,且重复性较好,是解决GWAS上述问题的合适方法[7]。2005年,Horne等[8]尝试着运用GRS来推测SNPs和冠心病之间的关联,并指出GRS在复杂疾病遗传学研究中具有很大潜力,值得在包括冠心病在内的复杂疾病遗传学研究中推广。近几年,随着GWAS的开展,GRS才在复杂疾病遗传学研究中得到广泛的应用。本文将重点介绍GRS的计算方法和评价标准,简要列举运用GRS取得的系列成果并对运用过程中所存在的局限性进行探讨。
1 GRS的算法
首先,选择合适的SNPs。这些SNPs可以从以往的GWAS研究中筛选得到,也可以根据发病机制从病理生理学通路中得到,或者将通过以上两种途径得到的SNPs整合在一起计算GRS。例如,在前列腺癌GRS的研究中,研究者从以往发表的GWAS研究中筛选了29个SNPs[9]用于GRS的计算;Sorosina等[10]利用以往文献中与免疫相关的106个SNPs计算多发性硬化症的GRS,并分析它和不同类型多发性硬化症之间的关系。关于纳入的SNPs的数目应根据研究疾病的不同而不同,并不是越多越好。Futema等[11]通过比较3个不同数目SNPs(6个SNPs、12个SNPs、33个SNPs)构建的GRS,发现6个SNPs和12个SNPs构成的GRS对高胆固醇血症的预测能力一样,将SNPs增加到33个并不增加GRS辨别有无家族型高胆固醇血症的能力。
然后,对纳入的SNPs进行运算。GRS 的构建基于多基因模型,假定疾病的遗传效应等于各个位点的效应之和,算法分两种:简单的GRS和加权的GRS。(1)简单的GRS:GRS=ΣS(为相应SNPs的个数)。该算法认为每个风险等位基因的作用相等,只根据相关风险等位基因的个数来计算。Borghini等[12]在计算乳腺癌放射治疗后皮肤急性反应的GRS时规定高风险等位基因的纯合子(有两个高风险等位基因)记为2分,杂合子记为1分,低风险等位基因的纯合子记为0分。另一项关于肥胖GRS的研究也是基于此算法[13]。(2)加权GRS:GRS=SS(为第个SNPs的权重,S为第个SNPs)。该算法认为每个风险等位基因对疾病的影响不同,通过给每个风险等位基因赋予一个相应的权重来显示不同SNPs对疾病的影响程度不同。这个权重通常为该SNPs的优势比的自然对数,常通过GWAS研究中的优势比取对数或相关回归模型中回归系数得到。相对而言,加权GRS广泛被运用。León-Mimila等[14]在计算非酒精性脂肪肝的GRS时将每个SNPs风险等位基因的个数和它的估测效应(系数)相乘然后求和。除此之外,包括Maehlen等[15]关于类风湿性关节炎自身抗体GRS的计算、Abdullah等[16]关于2型糖尿病GRS的计算等一系列研究[17~22]都是基于此种算法。关于哪一种GRS更有优势,Che和Motsinger-Reif[23]在临床表型相关基因存在交互作用和连锁不平衡情况下,对两种方法在检验效能、Ⅰ类错误、受试者操作曲线下面积(Area under the receiver operating characteristic curve,AUC)、模型拟合情况这几个方面进行比较,得出加权GRS整体效果比简单GRS更好一些的结论。GRS构建方法的框架见图1。
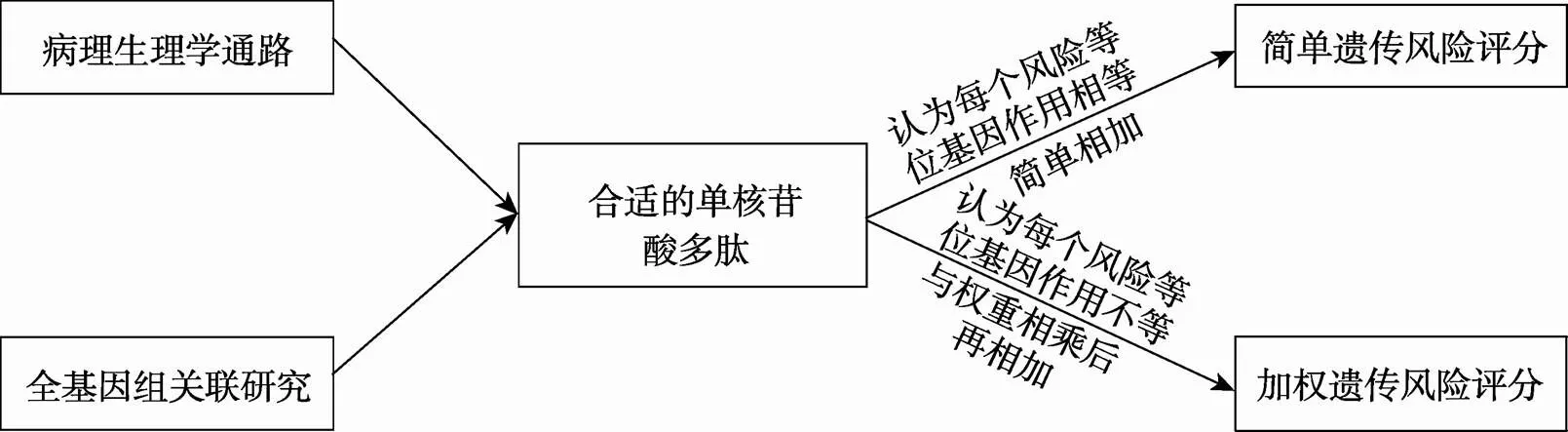
图1 遗传风险评分(GRS)的构建方法框架图
2 GRS的评价
构建的GRS与临床表型(疾病)之间的关联可以通过许多指标进行评价。比较简单的评价指标是通过比较GRS和一些连续变量(BMI、胆固醇浓度等)之间的相关系数。Belsky等[7]分析白人肥胖GRS和BMI之间关系时得出,矫正年龄、性别、地理差异后不超重的GRS得分和BMI的相关系数=0.12,超重的=0.13。其他评价指标包括:(1)优势比(Odds ratio,OR),表示GRS得分高的人群出现相应临床表型的风险是得分低的人群的倍数,可以通过Logistic回归分析获得。León-Mimila等[14]发现非酒精性脂肪肝GRS≥6的研究对象患非酒精性脂肪肝的风险是GRS≤5的研究对象的2.55倍(=2.55,=0.045)。一项关于肥胖GRS对胰岛素抵抗风险预测的研究得出肥胖GRS和胰岛素抵抗之间的关联具有统计学意义(=1.08, 95% CI 1.04~1.12,=1.18×10–4)[24]。(2)风险函数比(Hazard ratio,HR),可以通过构建Cox比例风险模型来实现。与OR相似,HR是通过比较出现结局的风险大小来进行评价,如乳腺癌放射治疗后急性皮肤毒性GRS得分高的一组出现急性皮肤毒性反应的风险是得分低一组的5倍(=5.1, 95% CI 1.2~22.8,=0.03)[12],其他几项研究也运用此评价指标进行评价[19,25]。
GRS对风险模型预测能力的提高可以通过以下4个指标进行评价:(1)方程决定系数(R),利用GRS可解释变异占的比重的变化来评价,可通过构建多因素回归方程得到。如Fava等[26]研究了高血压GRS与缺血性脑中风之间的关系,将GRS加入到传统风险因素组成的回归模型后,模型可解释变异从0.167增加到0.170;(2)AUC,AUC越接近1越好,当AUC值为0.5时,提示GRS的预测能力和机遇的预测能力没有不同,不具备预测价值。AUC的具体分析方法可参阅Janes的研究[27]。Morrison等[28]发现在非洲裔美国人中,将冠心病GRS加入到年龄、高血压、总胆固醇、高密度脂蛋白、糖尿病、吸烟等传统风险因素组成的基线模型后AUC从0.758增加到0.769,Goni等[29]关于肥胖GRS的研究及其他许多研究[17,21,30~33]均是采用这个评价指标进行评价的。AUC的大小也可以用C统计量来表示,最近一项关于高血压GRS的研究显示,由4个SNPs构成的加权GRS并不能提高模型对高血压的预测能力(加入GRS前和加入GRS后的C统计量分别为0.810和0.811,=0.1057)[34];(3)重分类改善指标(Net reclassification improvement,NRI),表示加入GRS后研究对象重新分类导致模型的改善程度。NRI=(up,events–down,events)–(up,nonevents–down,nonevents),即NRI=(出现结局的个体中经重新分类后风险向上移动的比例–出现结局的个体经重新分类后风险向下移动的比例)–(没有出现结局的个体中经重新分类后风险向上移动的比例–没有出现结局的个体重经重新分类后风险向下移动的比例)[35]。若所得到的NRI的值大于0则为正向改善,小于0则为负向改善,等于0则代表无改善,具体可参考文献[36]。Tam等[31]根据临床特征将研究对象分为(<5%, 5%~10%, 10%~15%, 15%~20%, ≥20%)五类,发现在基线模型中加入简单GRS和加权GRS后NRI分别改善11.0%和11.4%;(4)综合辨别指数(Integrated discrimination index, IDI),可以采用构建probit回归模型对灵敏性和特异性进行整体评价,通过比较含有风险标记的基线模型和加入新风险标记的测试模型的预测能力对GRS进行评价。当IDI为0时,说明两个模型预测能力一样。以肥胖为例,IDI=(test,obese–test,non-obese)– (baseline,obese–baseline,non-obese),指某组特定模型的平均预测能力。Belsky等[7]利用IDI对肥胖GRS进行评价得出该肥胖GRS对肥胖的预测能力强于人口地理信息对肥胖的预测,另外还有一些研究也是运用此指标对GRS进行评价[19,21]。在对GRS进行评价时,这些指标常常结合起来使用。相对来说,AUC这一指标应用的更加广泛。
对于构建的同一GRS来说,当它应用于不同人群时,预测能力也是不同的。朱瑶等[37]发现运用24个SNPs构建的GRS 对中国60~70岁的男性前列腺癌的预测能力较好,但对小于60岁或大于70岁的男性预测能力降低。
3 GRS的具体应用
GRS应用广泛,本文以Thanassoulis等[19]关于心血管疾病的GRS为例进行具体介绍。该研究以弗雷明汉心脏队列研究人群为研究对象,以风险等位基因纯合子(有两个高风险等位基因)记2分,杂合子记1分,没有风险等位基因记0分,基于GWAS筛选出与冠心病高度相关的13个SNPs(<5×10–8,且至少在两个以上人群得到验证)和冠心病主要危险因素相关的89个SNPs(危险因素包括低密度脂蛋白、高密度脂蛋白、甘油三酯、糖尿病、高血压、C反应蛋白)分别计算得到2个GRS,其中一个只含前13个SNPs(GRS总分0~26),另一个包含所有SNPs (102个SNPs,GRS总分0~204)。作者又以原始GWAS研究中β系数作为权重,分别计算了相应的加权GRS。以10年随访中出现心血管事件为结局,建立了3个cox比例风险模型,模型1校正性别和年龄,模型2校正年龄、性别和心血管危险因素(包括:高血脂、糖尿病、高血压),模型3校正年龄、性别、心血管危险因素和父母心血管病史,评价GRS和心血管事件的关联,得到13个SNPs的GRS和冠心病之间存在着显著的关联(=1.07, 95% CI 1.00~1.15,=0.04),而102个SNPs的GRS和心血管事件之间不存在关联(=1.01,95% CI 0.99~1.03,=0.48),使用加权GRS进行分析并没有对结果造成显著影响。该研究再以冠状动脉钙化为结局进行 Logistic回归分析,发现13个SNPs的GRS与结局之间存在显著关联(每个风险等位基因的=1.18, 95% CI 1.11~1.26,=3.4×10–7),将GRS得分从高到低排序,发现GRS得分在前1/3的结局的风险是后1/3组的2倍(=2.04,95% CI 1.48–2.83)。为了进一步评价GRS是否提高模型预测能力,作者又以0~6%、6%~20%、>20%为界值将研究对象分为3类,发现在包含年龄、性别、心血管危险因素的基线模型中加入13个SNPs的GRS后,NRI提升了0.29。
此研究中用于构建GRS的SNPs均来自于前期的GWAS研究,可以看出GWAS侧重于筛选出有意义的位点,而GRS则更注重评价这些SNPs的综合效应。
4 GRS的应用及局限
GRS整合了多个SNPs的信息,将每个SNPs的微弱效应进行叠加,大大提高了对疾病风险的预测,因此在不同领域取得了一系列成果。例如:利用GRS发现冠状动脉硬化和血压GRS之间的关系[38],说明血压相关基因变异可以影响冠状动脉硬化的形成,更清晰地阐明了血压对冠状动脉硬化的影响;肾免疫球蛋白GRS和肾脏疾病在不同人群之间关系的研究揭示了肾免疫球蛋白A引起的肾衰竭在北欧更流行的趋势[39];心脑血管疾病GRS的研究显示,心血管遗传标记物对心血管疾病预测方面还存在局限性[40];甲状腺过氧化物酶抗体的GRS和甲状腺疾病之间关系的研究发现二者之间存在很强的联系,GRS得分高的比得分低的亚临床甲状腺低下风险高1.80倍,甲状腺功能明显减退风险高1.89倍[41]。关于基因多样性对心脏移植患者术后生存率的影响的研究显示,在GRS得分高的患者中非洲裔美国人的生存率低于白种人[42]。另外,GRS在对2型QT间期延长综合征[43]、阿尔茨海默病[44]、骨折风险[45]、肥胖遗传风险不同人群体育锻炼获益情况[46]、结肠(直肠)癌[47]、孕妇先兆子痫风险[48]的研究中也取得了丰富成果。
GRS也存在一些局限性,比如需要大量的GWAS作为支撑,受到以前GWAS研究人群的限制,研究对象十分有限,尤其是关于儿童的调查比较少;在一些环节如筛选合适的SNPs、纳入SNPs的个数以及等位基因权重的取值以及GRS如何评价都没有统一的标准;另外,GRS没有考虑基因之间的相互作用和罕见基因突变的影响,可能影响GRS对疾病预测能力。
鉴于GRS应用以来取得的一系列成果,我们有理由相信随着复杂疾病遗传学研究的进一步发展,GRS会被进一步地完善并取得更多、更有影响的成果。
参考文献
[1] Keavney B. Genetic association studies in complex diseases.,2000, 14(6): 361–367.
[2] Morris RW, Kaplan NL. On the advantage of haplotype analysis in the presence of multiple disease susceptibility alleles., 2002, 23(3): 221–233.
[3] Martin ER, Lai EH, Gilbert JR, Rogala AR, Afshari AJ, Riley J, Finch KL, Stevens JF, Livak KJ, Slotterbeck BD, Slifer SH, Warren LL, Conneally PM, Schmechel DE, Purvis I, Pericak-Vance MA, Roses AD, Vance JM. Snping away at complex diseases: Analysis of single-nucleotide polymorphisms aroundin alzheimer disease.,2000, 67(2): 383–394.
[4] 邵谦之, 姜毅, 吴金雨. 全基因组测序及其在遗传性疾病研究及诊断中的应用. 遗传, 2014, 36(11): 1087–1098.
[5] Frayling TM, Timpson NJ, Weedon MN, Zeggini E, Freathy RM, Lindgren CM, Perry JRB, Elliott KS, Lango H, Rayner NW, Shields B, Harries LW, Barrett JC, Ellard S, Groves CJ, Knight B, Patch AM, Ness AR, Ebrahim S, Lawlor DA, Ring SM, Ben-Shlomo Y, Jarvelin MR, Sovio U, Bennett AJ, Melzer D, Ferrucci L, Loos RJF, Barroso I, Wareham NJ, Karpe F, Owen KR, Cardon LR, Walker M, Hitman GA, Palmer CNA, Doney ASF, Morris AD, Smith GD, Hattersley AT, McCarthy MI. A common variant in thegene is associated with body mass index and predisposes to childhood and adult obesity., 2007, 316(5826): 889–894.
[6] 郑伟, 季林丹, 邢文华, 涂巍巍, 徐进. 肺结核全基因组关联研究进展. 遗传, 2013, 35(7): 823–829.
[7] Belsky DW, Moffitt TE, Sugden K, Williams B, Houts R, McCarthy J, Caspi A. Development and evaluation of a genetic risk score for obesity.,2013, 59(1): 85–100.
[8] Horne BD, Anderson JL, Carlquist JF, Muhlestein JB, Renlund DG, Bair TL, Pearson RR, Camp NJ. Generating genetic risk scores from intermediate phenotypes for use in association studies of clinically significant endpoints.,2005, 69(2): 176–186.
[9] Wei HM, Chen HT, Wang P, Wu YS, Na R, Liu F, Sun JS, Jiang DK, Lu DR, Xu JF. Prostate cancer antigen 3 and genetic risk score as markers for the detection of prostate cancer in the chinese population., 2015, 17(1): 168–170.
[10] Sorosina M, Esposito F, Guaschino C, Clarelli F, Barizzone N, Osiceanu AM, Brambilla P, Mascia E, Cavalla P, Gallo P, Martinelli V, Leone M, Comi G, D'Alfonso S, Martinelli BF. Inverse correlation of genetic risk score with age at onset in bout-onset and progressive-onset multiple sclerosis., 2014, doi: 10.1177/1352458514561910.
[11] Futema M, Shah S, Cooper JA, Li K, Whittall RA, Sharifi M, Goldberg O, Drogari E, Mollaki V, Wiegman A, Defesche J, D'Agostino MN, D'Angelo A, Rubba P, Fortunato G, Walu-Miarka M, Hegele RA, Aderayo Bamimore M, Durst R, Leitersdorf E, Mulder MT, Roeters van Lennep JE, Sijbrands EJG, Whittaker JC, Talmud PJ, Humphries SE. Refinement of variant selection for the ldl cholesterol genetic risk score in the diagnosis of the polygenic form of clinical familial hypercholesterolemia and replication in samples from 6 countries., 2014, 61(1): 231–238.
[12] Borghini A, Vecoli C, Mercuri A, Petruzzelli MF, D'Errico MP, Portaluri M, Andreassi MG. Genetic risk score and acute skin toxicity after breast radiation therapy., 2014, 29(7): 267–272.
[13] Lv D, Zhang DD, Wang H, Zhang Y, Liang L, Fu JF, Xiong F, Liu GL, Gong CX, Luo FH, Chen SK, Li ZL, Zhu YM. Genetic variations in,,andwere associated with childhood obesity and interacted with dietary behaviors in chinese school-age population., 2015, 560(2): 149–155.
[14] León-Mimila P, Vega-Badillo J, Gutiérrez-Vidal R, Villamil-Ramírez H, Villareal-Molina T, Larrieta-Carrasco E, López-Contreras BE, Kauffer LRM, Maldonado-Pintado DG, Méndez-Sánchez N, Tovar AR, Hernández-Pando R, Velázquez-Cruz R, Campos-Pérez F, Aguilar-Salinas CA, Canizales-Quinteros S. A genetic risk score is associated with hepatic triglyceride content and non-alcoholic steatohepatitis in mexicans with morbid obesity.,2015, 98(2): 178–183.
[15] Maehlen MT, Olsen IC, Andreassen BK, Viken MK, Jiang X, Alfredsson L, Källberg H, Brynedal B, Kurreeman F, Daha N, Toes R, Zhernakova A, Gutierrez-Achury J, de Bakker PIW, Martin J, Teruel M, Gonzalez-Gay MA, Rodríguez-Rodríguez L, Balsa A, Uhlig T, Kvien TK, Lie BA. Genetic risk scores and number of autoantibodies in patients with rheumatoid arthritis., 2013, 74(4): 762–768.
[16] Abdullah N, Abdul Murad NA, Attia J, Oldmeadow C, Mohd Haniff EA, Syafruddin SE, Abd Jalal N, Ismail N, Ishak M, Jamal R, Scott RJ, Holliday EG. Characterizing the genetic risk for type 2 diabetes in a malaysian multi-ethnic cohort., 2015, 32(10): 1377–1384.
[17] De Jager PL, Chibnik LB, Cui J, Reischl J, Lehr S, Simon KC, Aubin C, Bauer D, Heubach JF, Sandbrink R, Tyblova M, Lelkova P, Havrdova E, Pohl C, Horakova D, Ascherio A, Hafler DA, Karlson EW. Integration of genetic risk factors into a clinical algorithm for multiple sclerosis susceptibility: A weighted genetic risk score., 2009, 8(12): 1111–1119.
[18] Hu PZ, Muise AM, Xing X, Brumell JH, Silverberg MS, Xu W. Association between a multi-locus genetic risk score and inflammatory bowel disease., 2013, 7: 143–152.
[19] Thanassoulis G, Peloso GM, Pencina MJ, Hoffmann U, Fox CS, Cupples LA, Levy D, D'Agostino RB, Hwang SJ, O'Donnell CJ. A genetic risk score is associated with incident cardiovascular disease and coronary artery calcium: The framingham heart study.,2012, 5(1): 113–121.
[20] van Dijk SC, Enneman AW, van Meurs J, Swart KMA, Ham AH, van Wijngaarden JP, Brouwer-Brolsma EM, van der Zwaluw NL, van Schoor NM, Dhonukshe-Rutten RAM, de Groot LCPGM, Lips P, Uitterlinden AG, Blom H, Geleijnse JM, Feskens E, de Jongh RT, Smulders YM, van den Meiracker AH, Mattace-Raso FUS, van der Velde N. B-vitamin levels and genetics of hyperhomocysteinemia are not associated with arterial stiffness., 2014, 24(7): 760–766.
[21] Yarwood A, Han B, Raychaudhuri S, Bowes J, Lunt M, Pappas DA, Kremer J, Greenberg JD, Plenge R, Worthington J, Barton A, Eyre S. A weighted genetic risk score using all known susceptibility variants to estimate rheumatoid arthritis risk., 2015, 74(1): 170–176.
[22] Lee CPL, Irwanto A, Salim A, Yuan JM, Liu JJ, Koh WP, Hartman M. Breast cancer risk assessment using genetic variants and risk factors in a singapore chinese population.,2014, 16(3): R64.
[23] Che RL, Motsinger-Reif AA. Evaluation of genetic risk score models in the presence of interaction and linkage disequilibrium.,2013, 4: 138.
[24] Xi B, Zhao XY, Shen Y, Wu LJ, Hou DQ, Cheng H, Mi J. An obesity genetic risk score predicts risk of insulin resistance among chinese children., 2014, 47(3): 825–832.
[25] Andersson EA, Allin KH, Sandholt CH, Borglykke A, Lau CJ, Ribel-Madsen R, Sparsø T, Justesen JM, Harder MN, Jørgensen ME, Jørgensen T, Hansen T, Pedersen O. Genetic risk score of 46 type 2 diabetes risk variants associates with changes in plasma glucose and estimates of pancreatic β-cell function over 5 years of follow-up., 2013, 62(10): 3610–3617.
[26] Fava C, Sjögren M, Olsson S, Lövkvist H, Jood K, Engström G, Hedblad B, Norrving B, Jern C, Lindgren A, Melander O. A genetic risk score for hypertension associates with the risk of ischemic stroke in a swedish case–control study.,2015, 23(7): 969–974.
[27] Janes H, Pepe MS. Adjusting for covariate effects on classification accuracy using the covariate-adjusted receiver operating characteristic curve.,2009, 96(2): 371–382.
[28] Morrison AC, Bare LA, Chambless LE, Ellis SG, Malloy M, Kane JP, Pankow JS, Devlin JJ, Willerson JT, Boerwinkle E. Prediction of coronary heart disease risk using a genetic risk score: The atherosclerosis risk in communities study., 2007, 166(1): 28–35.
[29] Goni L, Cuervo M, Milagro FI, Martínez JA. A genetic risk tool for obesity predisposition assessment and personalized nutrition implementation based on macronutrient intake.,2015, 10: 445.
[30] Eriksson J, Evans DS, Nielson CM, Shen J, Srikanth P, Hochberg M, McWeeney S, Cawthon PM, Wilmot B, Zmuda J, Tranah G, Mirel DB, Challa S, Mooney M, Crenshaw A, Karlsson M, Mellström D, Vandenput L, Orwoll E, Ohlsson C. Limited clinical utility of a genetic risk score for the prediction of fracture risk in elderly subjects.,2015, 30(1): 184–194.
[31] Tam CHT, Ho JSK, Wang Y, Lam VKL, Lee HM, Jiang GZ, Lau ESH, Kong APS, Fan XD, Woo JLF, Tsui SKW, Ng MCY, So WY, Chan JCN, Ma RCW. Use of net reclassification improvement (NRI) method confirms the utility of combined genetic risk score to predict type 2 diabetes.,2013, 8(12): e83093.
[32] Nobili V, Donati B, Panera N, Vongsakulyanon A, Alisi A, Dallapiccola B, Valenti L. A 4-polymorphism risk score predicts steatohepatitis in children with nonalcoholic fatty liver disease., 2014, 58(5): 632–636.
[33] Ribeiro RJT, Monteiro CPD, Azevedo ASM, Cunha VFM, Ramanakumar AV, Fraga AM, Pina FM, Lopes CMS, Medeiros RM, Franco EL. Performance of an adipokine pathway-based multilocus genetic risk score for prostate cancer risk prediction.,2012, 7(6): e39236.
[34] Lim NK, Lee JY, Lee JY, Park HY, Cho MC. The role of genetic risk score in predicting the risk of hypertension in the korean population: Korean genome and epidemiology study., 2015, 10(6): e0131603.
[35] 陈雅惠. 以重分类方法评估台湾修订版简易营养评估量表预测台湾中老年人死亡风险的改进能力[学位论文]. 中国台湾: 亚洲大学健康产业管理学系, 2012.
[36] Pencina MJ, D'Agostino RB Sr, Steyerberg EW. Extensions of net reclassification improvement calculations to measure usefulness of new biomarkers., 2011, 30(1): 11–21.
[37] Zhu Y, Han CT, Chen HT, Liu F, Zhang GM, Yang WY, Xu JF, Ye DW. Influence of age on predictiveness of genetic risk score for prostate cancer in a Chinese hospital-based biopsy cohort., 2015, 6(26): 22978–22984.
[38] Takeuchi F, Isono M, Yamamoto K, Yokota M, Akiyama K, Katsuya T, Kim HS, Park JE, Jang Y, Lee JY, Lee JY, Kato N. Heterogeneous effects of association between blood pressure loci and coronary artery disease in east asian individuals., 2015, 79(4): 830–838.
[39] Kiryluk K, Novak J, Gharavi AG. Pathogenesis of immunoglobulin a nephropathy: Recent insight from genetic studies.,2013, 64(1): 339–356.
[40] Raffield LM, Cox AJ, Carr JJ, Freedman BI, Hicks PJ, Langefeld CD, Hsu FC, Bowden DW. Analysis of a cardiovascular disease genetic risk score in the diabetes heart study., 2015, 52(4): 743–751.
[41] Schultheiss UT, Teumer A, Medici M, Li Y, Daya N, Chaker L, Homuth G, Uitterlinden AG, Nauck M, Hofman A, Selvin E, Völzke H, Peeters RP, Köttgen A. A genetic risk score for thyroid peroxidase antibodies associates with clinical thyroid disease in community-based populations., 2015, 100(5): jc20144352.
[42] Coleman B, Guo X, Patel J, Reinsmoen N, Jia Y, Kobashigawa JA. Genetic Risk Score (GRS) Predicts Worse Survival in African American Heart Transplant Recipients at 10 Years., 2015,34(4): S114–S115.
[43] Kolder ICRM, Tanck MWT, Postema PG, Barc J, Sinner MF, Zumhagen S, Husemann A, Stallmeyer B, Koopmann TT, Hofman N, Pfeufer A, Lichtner P, Meitinger T, Beckmann BM, Myerburg RJ, Bishopric NH, Roden DM, Kääb S, Wilde AAM, Schott JJ, Schulze-Bahr E, Bezzina CR. Analysis for genetic modifiers of disease severity in patients with long QT syndrome type 2., 2015, 8(3): 447–456.
[44] Chauhan G, Adams HHH, Bis JC, Weinstein G, Yu L, Töglhofer AM, Smith AV, Van der Lee SJ, Gottesman RF, Thomson R, Wang J, Yang Q, Niessen WJ, Lopez OL, Becker JT, Phan TG, Beare RJ, Arfanakis K, Fleischman D, Vernooij MW, Mazoyer B, Schmidt H, Srikanth V, Knopman DS, Jack CR, Jr., Amouyel P, Hofman A, DeCarli C, Tzourio C, van Duijn CM, Bennett DA, Schmidt R, Longstreth WT, Jr., Mosley TH, Fornage M, Launer LJ, Seshadri S, Ikram MA, Debette S. Association of alzheimer's disease GWAS loci with MRI markers of brain aging., 2015, 36(4): 1765.e7–1767.e16.
[45] Warrington NM, Kemp JP, Tilling K, Tobias JH, Evans DM. Genetic variants in adult bone mineral density and fracture risk genes are associated with the rate of bone mineral density acquisition in adolescence., 2015, 24(14): 4158–4166.
[46] Klimentidis YC, Bea JW, Lohman T, Hsieh PS, Going S, Chen Z. High genetic risk individuals benefit less from resistance exercise intervention.,2015, 39(9): 1371–1375.
[47] Thrift AP, Gong J, Peters U, Chang-Claude J, Rudolph A, Slattery ML, Chan AT, Locke AE, Kahali B, Justice AE, Pers TH, Gallinger S, Hayes RB, Baron JA, Caan BJ, Ogino S, Berndt SI, Chanock SJ, Casey G, Haile RW, Du MM, Harrison TA, Thornquist M, Duggan DJ, Le Marchand L, Lindor NM, Seminara D, Song MY, Wu KN, Thibodeau SN, Cotterchio M, Win AK, Jenkins MA, Hopper JL, Ulrich CM, Potter JD, Newcomb PA, Hoffmeister M, Brenner H, White E, Hsu L, Campbell PT. Mendelian randomization study of body mass index and colorectal cancer risk.,2015, 24(7): 1024–1031.
[48] Smith CJ, Saftlas AF, Spracklen CN, Triche EW, Bjonnes A, Keating B, Saxena R, Breheny PJ, Dewan AT, Robinson JG, Hoh J, Ryckman KK. Genetic risk score for essential hypertension and risk of preeclampsia., 2015, doi: 10.1093/ajh/hpv069.
The application of genetic risk score in genetic studies of complex human diseases
Dayan Niu, Weili Yan
Complex diseases such as cardiovascular disease, type 2 diabetes, essential hypertension, asthma, obesity and cancer have spread across the globe and become the predominant cause of death. There are growing concerns over the role of genetic susceptibility in pathogenesis of complex diseases. However, the related susceptibility genes and sequence variations are still unknown. To elucidate the genetic basis of complex diseases, researchers have identified a large number of genetic variants associated with complex diseases through genome-wide association studies (GWAS) and candidate gene studies recently. The identification of these causal and/or associated variants promotes the development of approaches for complex diseases prediction and prevention. Genetic risk score (GRS), an emerging method for exploring correlation between single nucleotide polymorphisms (SNPs) and clinical phenotypes of complex diseases, integrates weak effects of multiple SNPs and dramatically enhances predictability of complex diseases by gene polymorphisms. This method has been applied successfully in genetic studies of many complex diseases. Here we focus on the introduction of the computational methods and evaluationcriteria of GRS, enumerate a series of achievements through GRS application, discuss some limitations during application, and finally prospect the future of GRS.
genetic risk score; complex diseases; single nucleotide polymorphisms
2015-05-26;
2015-09-19
国家自然科学基金项目(编号:81273168)资助
牛大彦,在读硕士研究生,研究方向:复杂疾病遗传流行病学。E-mail: niudayan14@163.com
严卫丽,博士,博士生导师,教授,研究方向:复杂疾病遗传流行病学。E-mail: yanwl@fudan.edu.cn
10.16288/j.yczz.15-228
网络出版时间: 2015-9-29 8:49:53
URL: http://www.cnki.net/kcms/detail/11.1913.R.20150929.0849.002.html
(责任编委: 卢大儒)
