醋酸菌中CRISPR位点的比较基因组学与进化分析
2015-10-29夏凯梁新乐李余动
夏凯,梁新乐,李余动
醋酸菌中CRISPR位点的比较基因组学与进化分析
夏凯,梁新乐,李余动
浙江工商大学食品与生物工程学院,杭州 310018
CRISPR (Clustered regularly interspaced short palindromic repeats)是近几年发现的一种广泛存在于细菌和古菌中,能够应对外源DNA干扰(噬菌体、病毒、质粒等),并提供免疫机制的重复序列结构。CRISPR系统通常由同向重复序列、前导序列、间隔序列和CRISPR相关蛋白组成。本研究以醋酸发酵中常见3个属醋杆菌属()、葡糖醋杆菌属()和葡糖杆菌属()的48个菌株为研究对象,通过其基因组上CRISPR相关基因序列的生物信息学分析,探索CRISPR位点在醋酸菌中的多态性及其进化模式。结果表明48株醋酸菌中有32株存在CRISPR结构,大部分CRISPR-Cas结构属于type I-E和type I-C类型。除了葡糖杆菌属外,葡糖醋杆菌属和醋杆菌属中的部分菌株含有II类的CRISPR-Cas系统结构(CRISPR-Cas9)。来自不同属菌株的CRISPR结构中重复序列具有较强的保守性,而且部分菌株CRISPR结构中的前导序列具有保守的motif (与基因的转录调控有关)及启动子序列。进化树分析表明适合用于醋酸菌株的分类,而不同菌株间基因的进化与重复序列的保守性相关,预示它们可能受相似的功能选择压力。此外,间隔序列的数量与噬菌体数量及插入序列(Insertion sequence, IS)数量有正相关的趋势,说明醋酸菌在进化过程中可能正不断受新的外源DNA入侵。醋酸菌中CRISPR结构位点的分析,为进一步研究不同醋酸菌株对醋酸胁迫耐受性差异及其基因组稳定性的分子机制奠定了基础。
醋酸菌;CRISPR;重复序列;遗传进化
醋酸菌隶属于α变形杆菌纲,红螺菌目,醋酸杆菌科[1]。醋酸菌一般可以从水果、花卉以及发酵食品中分离获得,由于其能在乙醇脱氢酶和乙醛脱氢酶的作用下将乙醇氧化为乙酸,因而在醋的工业化生产过程中扮演着非常重要的角色[2]。截止到目前,醋酸菌主要包括12个属,其中研究最多的是醋酸杆菌()、葡糖醋杆菌()及葡糖杆菌()。醋酸杆菌和葡糖醋杆菌拥有较高的耐酸性因而被广泛地用于醋的发酵[3]。醋酸菌容易受到外源DNA的干扰,特别是噬菌体的侵染。虽然噬菌体在基因水平转移和遗传多样性方面发挥着重要作用,但在实际的发酵生产过程中,噬菌体的浸染将会造成巨大的经济损失[4]。另外,噬菌体的侵染也造成了醋酸菌基因组的不稳定,进而影响到菌种的性能[5]。为了抵御来自外源DNA的干扰,特别是噬菌体、病毒以及质粒,自然界的细菌在进化过程中形成了一系列的防御机制,包括流产性感染、表面排斥以及限制性调节系统等[6]。CRISPR (Clustered regularly interspaced short palindromic repeats)是近几年发现的一种广泛存在于细菌和古菌中,能够应对外源DNA干扰并提供免疫机制的重复序列结构[7]。因此,醋酸菌中CRISPR结构位点的分析对于研究醋酸菌耐酸能力差异的遗传机制具有重要的意义[8]。
CRISPR系统发挥作用主要分为3步:第一步是适应阶段,通过将外源核酸片段整合到CRISPR位点形成新的间隔序列;第二步是合成CRISPR RNA (crRNA);最后一步是干扰,在crRNA的指引下Cas核酸酶可以将特定新侵入的外源核酸同源片段序列切割[9]。根据CRISPRdb[10]数据库的统计信息,目前已报道的细菌和古菌的基因组序列中约45%的细菌基因组和83%的古菌基因组中都含有CRISPR结构。一般微生物中CRISPR结构的数量有1~2个,而多可达10或30个[9]。CRISPR系统主要由同向重复序列 (Direct repeat, DR)、前导序列(Leader sequence)、间隔序列(Spacer sequence)和CRISPR相关(CRISPR- associated, Cas)蛋白组成[11]。一般来说,CRISPR-Cas系统及其各个组分在不同微生物基因组中的数量、基因序列、出现的频率以及大小等方面具有较高的变异[12]。Makarova等[13]根据基因的结构排列顺序,将CRISPR-Cas系统主要分为3个大类型type I (含有和), type II (含有)和type III (含有)以及10个亚类型(I-A、I-B、I-C、I-D、I-E、I-F、II-A、II-B、III-A和III-B)[13]。
重复序列的长度在不同物种中差别较大,而在同一物种中保守性较高[14]。一般重复序列的长度为23~55 bp,重复序列中存在着短的回文序列,可以形成发夹结构。间隔序列长度一般为21~72 bp,间隔序列来自外源核酸DNA,具有很高的多态性,有时在同一种的不同菌株之间也会出现差异[9]。间隔序列的重复数在不同的菌种中差异明显,据统计最高的重复数可以达到588 (),但一般都在50以下,较低的只有2。前导序列在CRISPR结构中发挥着重要的作用,其通常位于CRISPR结构第一个重复序列的上游一段AT富含区域,该段区域在不同物种间的保守性不高[15]。研究发现,新插入的间隔序列总是会加在前导序列和第一个重复序列之间[16]。同时由于前导序列的特殊结构,前导序列通常含有下游重复序列的转录启动子[17]。基因位于CRISPR结构中重复序列的上游或下游区域,据Daniel等的研究,目前共有45个基因家族,并大致可分为8个亚型,每个亚型均由该亚型特定的基因组成[18]。另外,有6个基因()与大多数不同亚型的基因相关联,被认为是核心基因,尤以为CRISPR结构的标志性基因[18]。研究表明,CRISPR系统中基因和重复序列相互关联起作用,存在协同进化现象[19]。
目前有关细菌CRISPR结构的研究主要集中在大肠杆菌()、乳酸菌()、嗜热链球菌()等[20],但有关醋酸菌基因组中CRISPR结构的分析未见有相关报导。现有研究表明[1],醋杆菌基因组序列中存在大量的前噬菌体DNA和转座子,导致醋杆菌基因组的不稳定性。与醋杆菌相比,葡糖杆菌和葡糖醋杆菌的基因组序列中含有较少的前噬菌体DNA序列,推测可能与其基因组中的CRISPR结构及数量有关。本文对醋酸菌中的CRISPR结构进行了比较分析,探索CRISPR位点在醋酸菌不同属中的变异规律,并对醋酸菌基因组中的前噬菌体序列及插入序列(Insertion sequence,IS)进行了预测,揭示噬菌体数量与CRISPR位点之间的关系[8]。
1 材料和方法
1.1 材料
Ab3基因组序列的测序与分析是由浙江工商大学梁新乐实验室完成(GenBank编号:CP012111),其余27株醋杆菌,9株葡糖醋杆菌以及11株葡糖杆菌的基因组序列GenBank编号见附表1。上述基因组序列均可从NCBI网站下载(http://www. ncbi.nlm.nih.gov/genomes/lproks.cgi)。
1.2 方法
1.2.1 醋酸菌中CRISPR位点查找
完整基因组中CRISPR结构的查找主要根据CRISPRdb数据库[10]。Draft基因组序列中CRISPR的查找主要通过CRISPRfinder(http://crispr.u-psud.fr/)以及CRT软件完成[21]。
1.2.2 CRISPR位点的生物信息学分析
同向重复序列的比对通过ClustalW完成,Draft 基因组中的基因的查找通过本地BLAST完成。前导序列的多序列比对分析同过ClustalW 以及BLAST 和WEBLOGO[22]完成。对获得的CRISPR位点中的同向重复序列进行归类分析,共分为8个(CRISPR 1~8)。通过MEGA 6.0对不同种类的同向重复序列进行遗传进化聚类分析[23]。选取CRISPR结构位点中第一个重复序列上游1000 bp序列作为前导序列查找的对象,通过ClustalW反复比对分析,找出保守的motif区。基因序列的聚类分析方法和同向重复序列相同。
1.2.3 基因组中噬菌体区域及IS序列预测
醋酸菌基因组中噬菌体区域的预测通过PHAST[24]以及Prophinder[25]共同完成。插入序列由ISfinder(https://www-is.biotoul.fr/)[26]完成(=1e-10)。原核生物启动子的预测由在线软件(http://www.fruitfly. org/seq_tools/promoter.html) 进行(采用默认参数)。
1.2.4 CRISPR与噬菌体及IS序列的统计分析
通过非参数检验Wilcoxon rank sum test比较存在CRISPR与不存在CRISPR位点的菌株之间的噬菌体及IS数量的差异。由Spearman相关性检验进行CRISPR的间隔序列或重复序列与噬菌体及IS序列数量的相关性分析。所有统计分析由软件Origin 8.5完成。
2 结果与分析
2.1 醋酸菌CRISPR结构位点的基因组分布概况
48株醋酸菌CRISPR位点分析结果表明,32株醋酸菌含有至少一个CRISPR 结构(附表2),其中醋杆菌属19株,葡糖醋杆菌属6株,葡糖杆菌属7株。根据CRISPRdb数据库信息,4株具有完整基因组序列菌株的9个CRISPR结构位点如图1所示。醋杆菌属CRISPR的平均个数是1.8个(Ab3基因组序列中不含有CRISPR结构),低于葡糖醋杆菌属的2.7个以及葡糖杆菌属的3.3个。醋杆菌属中,CRISPR数量最少的是DSM 23921和DMCS_004以及巴氏醋杆菌(),都只有1个CRISPR,特别是巴氏醋杆菌的CRISPR结构均位于质粒上,在其染色体上未发现CRISPR结构的存在;CRISPR结构数最多的是4H-1和NBRC 16470 (各4个)。葡糖醋杆菌属中,CRISPR数最少的是E25,只有1个,其他葡糖醋杆菌则含有2~4个CRISPR结构。葡糖杆菌属中,CRISPR结构数量相对最多,几乎都含有3~4个。
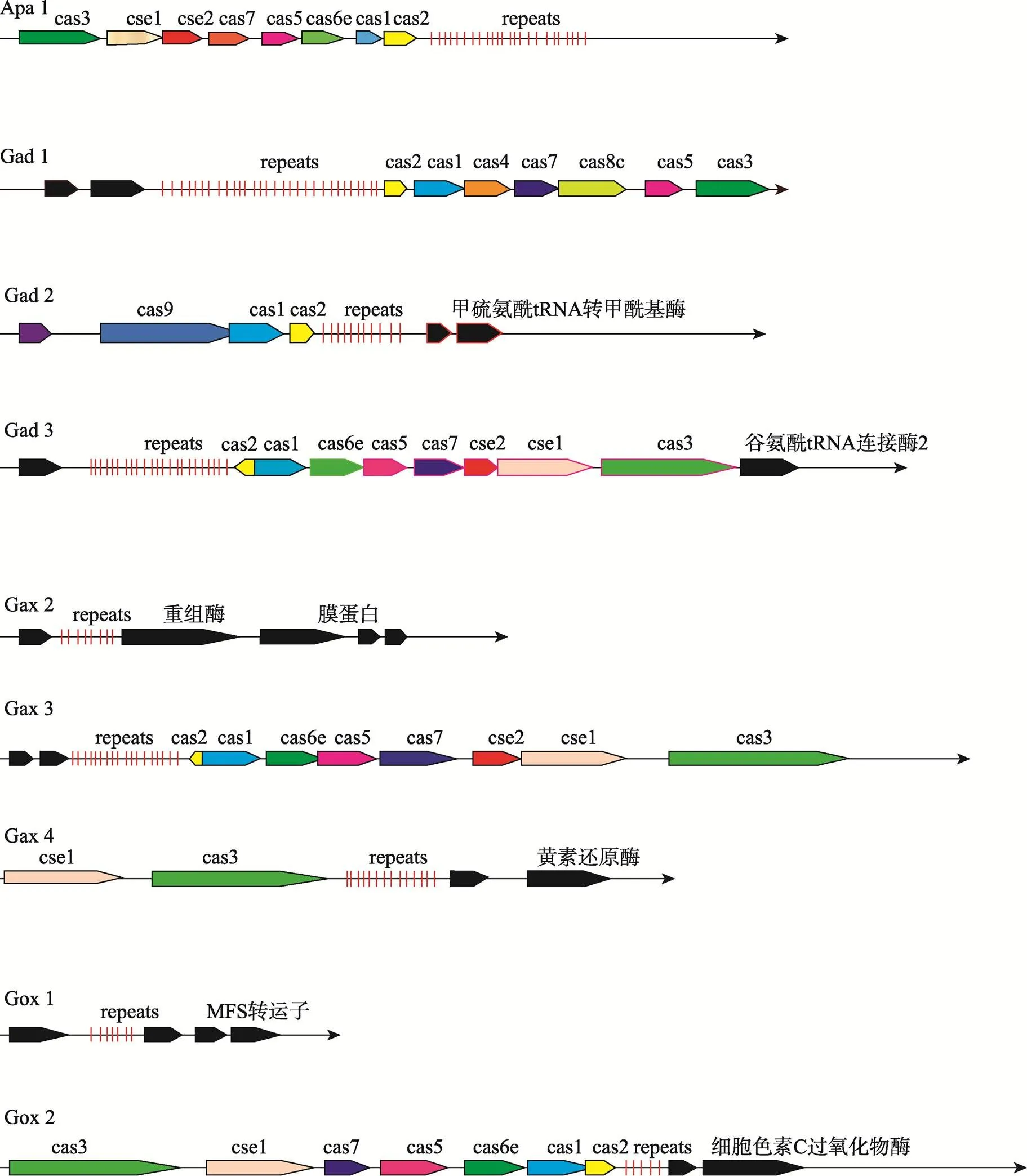
图1 4株醋酸菌中9个CRISPR位点结构示意图
Apa:巴氏醋杆菌IFO 3283-01;Gad:PAI 5;Gax:s NBRC 3288;Gox:H24。不同颜色的箭头代表不同的基因;红色线条代表重复序列(Repeats)。
对32株醋酸菌的CRISPR-Cas系统进行分类,发现醋酸杆菌属中的CRISPR-Cas系统主要属于I-E、I-F、I-C等类型(包含和基因),其中ATCC 23746的基因组序列中存在着II类Cas系统(附表2,附图1)。葡糖醋杆菌属中的CRISPR-Cas系统主要属于I-E和I-D,其中在PAI 5的基因组序列中存在着3种CRISPR-Cas系统:I-E类,I-D类以及II类。而在葡糖杆菌属的7个基因组序列中,CRISPR-Cas系统均属于I-E类。3个属所有菌株的基因组序列中皆不含III类CRISPR-Cas系统,不同菌株基因组中的CRISPR-Cas结构情况如附图1所示。不同醋酸菌属的菌种基因组序列中具有相同类型的CRISPR-Cas系统结构。
本研究所用的48个菌株共含有73个CRISPR, 其中确定位点有51个,可疑的有22个。为了便于分析,根据重复序列的相似情况将CRISPR分为9组,分别为8个类别(CRISPR 1~8)及其他(表1)。其中在CRISPR 1~8中重复序列的结构具有较强的规律性,而且在各个类型中重复序列具有较好的保守性。在其他类型中的重复序列多是可疑位点,保守性较差,没有一定的规律性。CRISPR 1~7在醋杆菌属、葡糖醋杆菌属和葡糖杆菌属中都存在,而CRISPR 8只存在于NBRC 3255和NBRC 3257中。另外,具有相同类型CRISPR-Cas系统的菌株中,CRISPR的数量和结构却存在着较大的差异(附表2)。
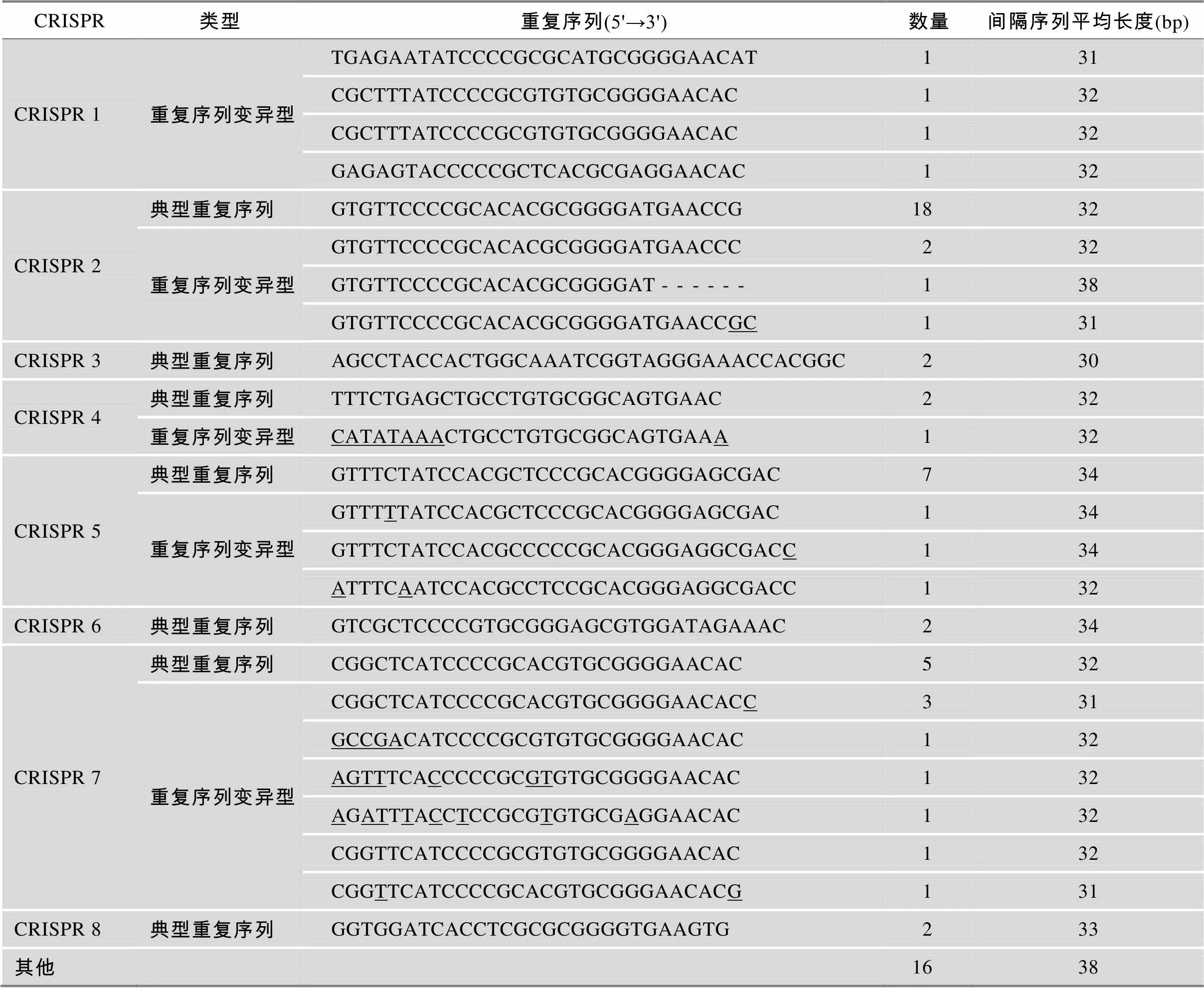
表1 CRISPR重复序列的分类分析
注:下划线代表重复序列变异型中突变的碱基(和典型重复序列相比)。
2.2 重复序列与Cas蛋白的进化分析
根据48株醋酸菌重复序列的比对分析发现,相同的重复序列在不同属的菌株中广泛分布且具有一定的保守性(附表2)。而在同种的不同菌株中,重复序列也存在着差异性,这种差异性体现在部分碱基突变以及CRISPR结构的不同(表1)。以重复序列构建的系统进化树表明醋杆菌属、葡糖醋杆菌属和葡糖杆菌属的菌种互相混杂,不能彼此分开,因此不能利用该序列对不同属的菌种进行分类(图2A)。每一个CRISPR类型中(1~8)都包含了来自不同属的菌种,这说明菌种在遗传进化过程中可能形成了相同的防御体系。聚类分析同时也显示,同一种内的CRISPR具有完全不同的重复序列,如NBRC 3255中的Gth 5和Gth 6(附表2,图2A)。
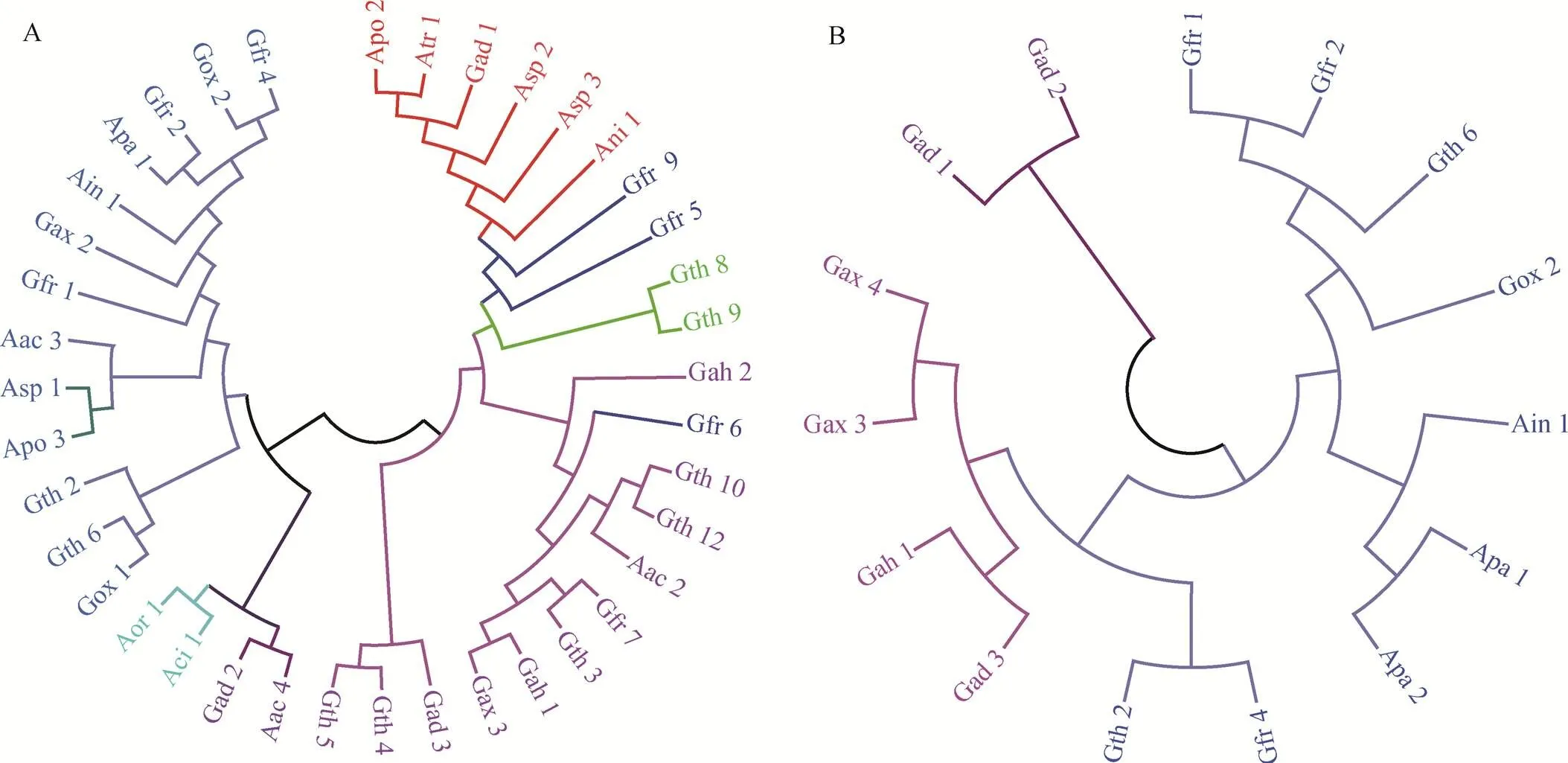
图2 利用醋杆菌属(A)、葡糖醋杆菌属(Ga)和葡糖杆菌属(G)中的重复序列以及cas1基因序列构建的系统发生树拓扑结构图
A:利用重复序列构建的系统发生树;B:利用基因序列构建的系统发生树。不同颜色的分支代表来源于不同种属的菌株。
重复序列的重复数在不同的物种间的差异明显,即使在不同的种或者同一种内不同分离株的CRISPR结构中,重复数的差异依然存在。醋杆菌属中34个CRISPR位点共有443个重复序列,长度在23 bp到38 bp不等(包括确定的和可疑的,确定的CRISPR中重复序列长度在30 bp左右)。在这34个CRISPR中重复数最高可达38个(ATCC 23746),最低只有2个。葡糖醋杆菌属中的16个CRISPR位点中共有131个重复序列,长度在23 bp 到36 bp 不等(同样,确定的CRISPR中重复序列长度在30 bp左右)。在这些CRISPR中重复数最高可达30个(PAI 5),最低也只有2个。葡糖杆菌属中共有23个CRISPR结构,共有136个重复序列, 长度在23 bp 到30 bp不等,大多数在30 bp左右,而CRISPR中重复数最高只有10个,远低于醋杆菌属以及葡糖醋杆菌属中的菌株,最低的重复数只有2个(附表2)。
Cas蛋白的比较基因组分析表明,在绝大多数的CRISPR结构重复序列上游或者下游位置存在着和Cas蛋白编码相关的基因序列。根据CRISPRdb数据库中的数据分析,巴氏醋杆菌() CRISPR结构Apa 1中重复序列的上游存在着、7、、、以及基因(图1)。而葡糖醋杆菌中的Gax 2和葡糖杆菌中的Gox 1 (图1)不存在基因。同时,在Gad 3和Gax 3中基因的排列顺序高度一致,依次是----7---。基因的进化聚类分析发现基因的保守性和重复序列相关,重复序列在不同物种间的保守性也使得基因保守(图2B),与前人的研究结果相吻合[19]。
2.3 间隔序列与前导序列的比较分析
在48株醋酸菌的73个CRISPR位点中共含有637个间隔序列,长度从18~60 bp不等。虽然在同一物种中不同CRISPR中间隔序列长度不一,但分析结果表明在CRISPR1~8分类中,CRISPR 1、CRISPR 2、CRISPR 3、CRISPR 4、CRISPR 6 以及CRISPR 7的间隔序列长度几乎都在32 bp, CRISPR 5的间隔序列长度在34 bp, CRISPR 8中的间隔序列长度在33 bp(表1)。
对48株醋酸菌的51个确定的CRISPR结构中的前导序列进行比对分析,比对时选取各个位点第一个重复上游1000 bp富含AT区域作为研究对象。经过ClustalW以及本地BLAST的反复比对分析,在7个菌种的13个CRISPR位点(Aac 3、Gox 1、Gox 2、Gfr 1、Gfr 2、Gfr 3、Gfr 4、Gth 2、Gth 4、Gth 5、Gth 6、Gth 7、Gth 12)的上游序列中发现一段较为保守的短motif序列(图3A)。该序列靠近第一个重复序列,以“AAATCGG”碱基序列开始,以“GAAGAG”结束。保守结构中存在着众多的连续的“TTTT”结构,并有较明显的碱基突变,说明这些富含AT的区域与基因启动子的转录调控有关。进一步启动子预测分析,表明大部分前导序列中含有原核生物的启动子序列及其典型的转录因子结合位点(图3B)。
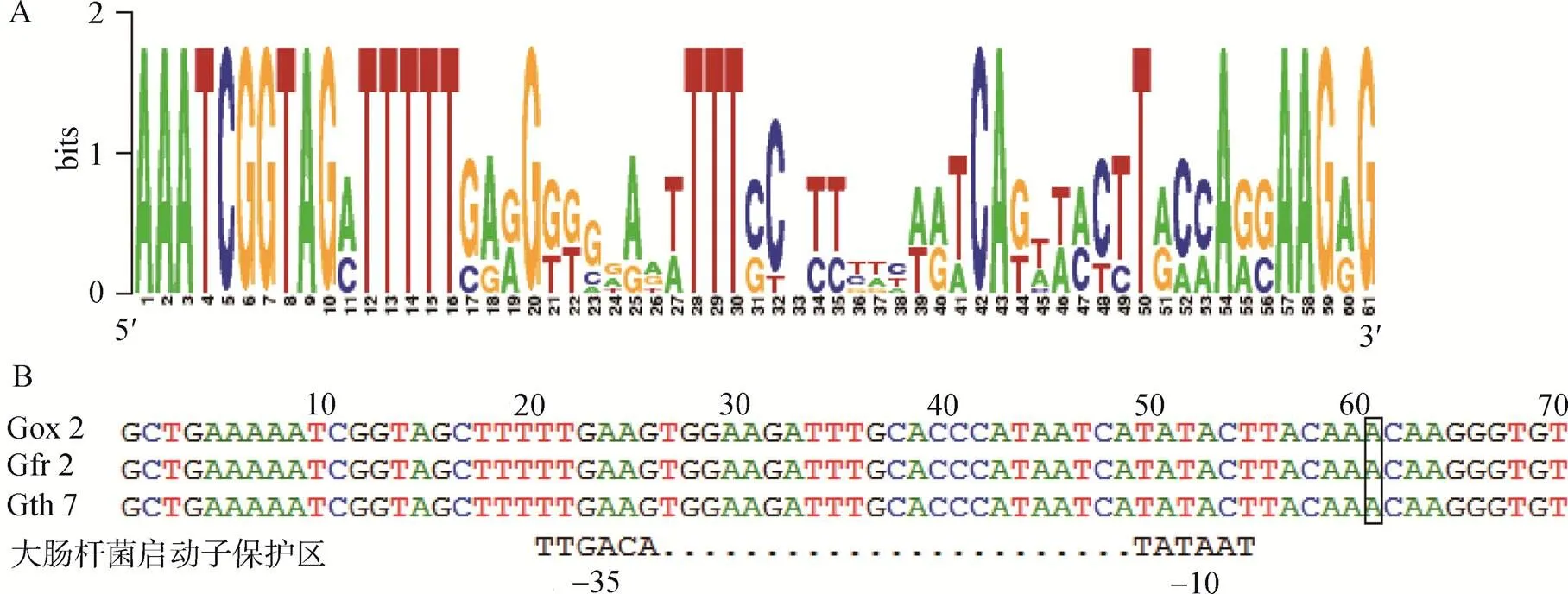
图3 CRISPR前导序列的的保守motif区域与启动子位点比较分析
A:13个CRISPR前导序列具有保守motif;B:3个菌株预测启动子的序列比对,黑框代表可能的转录位点。
2.4 间隔序列数量与前噬菌体数量、IS序列数量的相关性分析
对32株醋酸菌基因组中的前噬菌体序列预测发现,在醋酸菌的基因组序列中皆存在着前噬菌体
(至少1个,最多6个)。醋杆菌属15个基因组序列中的前噬菌体平均数量为2.49个,低于葡糖醋杆菌属(7个基因组序列)中的3.86个,高于葡糖杆菌属(10个基因组序列)中的1.8个(附表3)。在研究前噬菌体数量与CRISPR数量之间的关系发现,在不存在CRISPR结构的基因组序列中的前噬菌体数量并未多于其他含有多个CRISPR结构的基因组序列中的前噬菌体个数,32株醋酸菌基因组序列中的前噬菌体个数与CRISPR未有显著的相关关系(=0.58)。而醋酸菌基因组序列中的前噬菌体个数与间隔序列个数在整体上存在着正相关趋势(菌株174Bp2、LMG 18494和LMG 18890不能在其基因组序列中找到CRISPR- Cas系统的相关编码基因(附表 2),为了减少统计结果产生的误差,因此将这3株菌排除在外)(图4A) (Spearman相关性检验:=0.40,=0.04)。对醋酸菌基因组中IS序列的预测发现,葡糖杆菌属基因组序列中的IS序列个数是最多的(平均53.71),远高于醋杆菌属的22.33个以及葡糖醋杆菌属的24.2个,并与前噬菌体数量呈正相关关系。基因组序列中IS序列个数在PAI 5中最多(84个),在21F-2中最少(4个)。同时分析IS序列与间隔序列个数的关系发现,插入序列和间隔序列间存在着显著的正相关趋势(图4B)(Spearman相关性检验:=0.65,=4.32E-4),与前噬菌体数量和间隔序列个数之间的关系时相一致。另外,葡糖醋杆菌属中的部分菌株拥有较多的插入序列可能与其基因组整体大小较大相关。
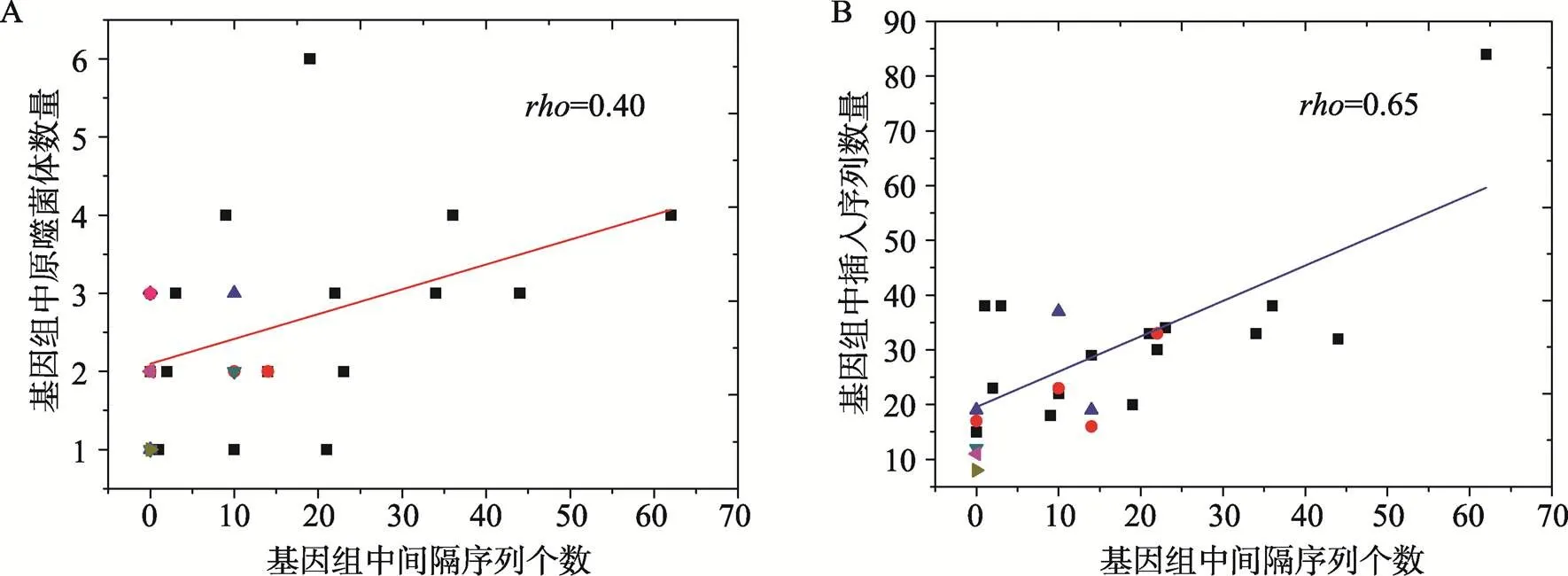
图4 噬菌体数量及IS序列与CRISPR数量关系图
A:噬菌体数量与间隔序列数量的关系;B:IS序列数量与间隔序列数量的关系。
3 讨 论
CRISPR结构作为一种新发现的抵御噬菌体、病毒等外源核酸入侵的细胞防御机制,越来越受到人们的关注。微生物在发酵过程中容易受到噬菌体的污染, CRISPR可以抵抗噬菌体的侵染,这给微生物发酵工业解决噬菌体污染带来全新的解决办法。随着人们对CRISPR功能的深入理解,CRISPR-Cas9基因编辑系统正被广泛应用于微生物的基因组改造[17]。而对醋酸菌CRISPR的研究,将有助于醋酸发酵菌种的进一步改造与控制。传统醋酸发酵过程中产生很高的酸度环境,用于醋酸发酵生产的醋酸菌,特别是醋杆菌属和葡糖醋杆菌属,通常具有较高耐酸性能,而其基因组如何适应高酸度发酵环境的进化机制还有待研究。比较基因组研究发现醋杆菌属和葡糖醋杆菌属的基因组序列中存在大量的噬菌体核酸序列[1],这与醋酸菌种性能的稳定性及醋酸发酵质量都有重要的影响。
本文对醋酸菌3个主要属中的48株菌进行了CRISPR位点分析,发现48株菌中有32株含有CRISPR结构,而CRISPR-Cas系统的分类表明大部分CRISPR-Cas系统属于I类中的I-E 和I-F亚类型,少数菌株的基因组序列中存在着II类的Cas系统,而所有菌株中皆未发现III类Cas系统。从CRISPR的分布情况来看,葡糖杆菌属和葡糖醋杆菌属的CRISPR数量多于醋杆菌属,这与醋杆菌基因组序列中含有较多的噬菌体序列和转座子的现象相对应。相同的CRISPR在不同属的各菌株中均有分布,具有一定的保守性,推测这3个菌属的遗传性质比较接近。而重复序列进化分析表明重复序列不适合用于不同属的菌株之间的分类,说明CRISPR主要是通过水平转移机制在菌株间传递并独立进化。不同CRISPR类别(CRISPR 1~8)中的间隔序列长度几乎相同,预示醋酸菌在发酵过程中受到相同或相似的噬菌体入侵。另外,CRISPR结构在同属而不同种中的差异性可能揭示了不同菌种对外源DNA干扰的抵御能力的差异性,而CRISPR结构在同种不同菌株中的差异性也提示外源DNA入侵的多样性。
通过s基因的研究发现,基因在不同属中存在着保守性,而这一现象刚好和重复序列的保守性相吻合。此结果与文献报道的基因的作用和重复序列相关相一致[19],说明它们可能受相似的功能选择压力。前导序列在不同物种间并不保守[27],对醋酸菌51个已确定的CRISPR位点的前导序列进行分析后发现,在7个种的13个CRISPR位点序列上游有一段短的相对保守的motif。该段序列在不同的物种中碱基突变率较大,没有明显的回文特征,推测可能是由于生长环境不同,导致醋酸菌基因组部分序列在遗传进化过程中发生了较大的变化,进而形成了菌株之间的差异,这也与Yoshinao等[2]全基因组比较分析结果相似。
Takashi等[28]研究发现在中,前噬菌体的数量与CRISPR的数量存在着负相关关系,CRISPR限制了噬菌体的侵入。然而,对醋酸菌基因组序列中前噬菌体区域数量与CRISPR以及间隔序列数量关系的研究发现,醋酸菌基因组中前噬菌体区域数量与间隔序列数量呈现正相关趋势,即前噬菌体的数量并不会因为间隔序列数量增多而减少。出现这个现象的原因可能是醋酸菌在进化过程中,由于生存环境的变化,噬菌体的侵染概率以及侵染噬菌体种类都会有差异,因此当外界噬菌体以及病毒的侵染数量和种类增加时,醋酸菌基因组中的CRISPR位点会相应的增加。另外,这种现象可能也与醋酸菌基因组有较强的不稳定性相关,容易受外源DNA的侵入。因为在IS序列分析时发现,醋酸菌基因组中IS序列与间隔序列数量也同样呈现正相关趋势。
总之,本文首次对醋酸菌3个主要属中的CRISPR结构进行了生物信息学分析,其CRISPR结构的阐明为今后抗噬菌体工程醋酸菌的构建以及醋酸菌CRISPR-Cas9介导的基因敲除研究提供了思路。同时CRISPR位点的研究有助于揭示醋酸菌基因组的不稳定性及其对高酸环境适应性进化的分子遗传机制。
附录:附图和附表见电子版www.chinagene.cn。
参考文献
[1] Illeghems K, Vuyst LD, Weckx S. Complete genome sequence and comparative analysis of386B, a strain well-adapted to the cocoa bean fermentation ecosystem., 2013, 14: 526.
[2] Azuma Y, Hosoyama A, Matsutani M, Furuya N, Horikawa H, Harada T, Hirakawa H, Kuhara S, Matsushita K, Fujita N, Shirai, M. Whole-genome analyses reveal genetic instability of., 2009, 37(17): 5768–5783.
[3] Matsutani M, Hirakawa H, Saichana N, Soemphol W, Yakushi T, Matsushita K. Genome-wide phylogenetic analysis of differences in thermotolerance among closely relatedstrains., 2012, 158(Pt 1): 229–239.
[4] Hambly E, Suttle CA. The viriosphere, diversity, and genetic exchange within phage communities., 2005, 8(4): 444–450.
[5] Matsutani M, Hirakawa H, Yakushi T, Matsushita K. Genome-wide phylogenetic analysis of,, and., 2011, 315(2): 122–128.
[6] Heler R, Marraffini LA, Bikard D. Adapting to new threats: the generation of memory by CRISPR-Cas immune systems., 2014, 93(1): 1–9.
[7] 李君, 张毅, 陈坤玲, 单奇伟, 王延鹏, 梁振, 高彩霞. CRISPR/Cas系统: RNA靶向的基因组定向编辑新技术. 遗传, 2013, 35(11): 1265–1273.
[8] 李铁民, 杜波. CRISPR-Cas系统与细菌和噬菌体的共进化. 遗传, 2011, 33(3): 213–218.
[9] Barrangou R, Marraffini LA. CRISPR-Cas systems: Prokaryotes upgrade to adaptive immunity., 2014, 54(2): 234–244.
[10] Grissa I, Vergnaud G, Pourcel C. The CRISPRdb database and tools to display CRISPRs and to generate dictionaries of spacers and repeats., 2007, 8: 172.
[11] Millen AM, Horvath P, Boyaval P, Romero DA. Mobile CRISPR/Cas-mediated bacteriophage resistance in Lactococcus lactis., 2012, 7(12): e51663.
[12] Terns RM, Terns MP. CRISPR-based technologies: prokaryotic defense weapons repurposed., 2014, 30(3): 111–118.
[13] Makarova KS, Haft DH, Barrangou R, Brouns SJJ, Charpentier E, Horvath P, Moineau S, Mojica FJM, Wolf YI, Yakunin AF, van der Oost J, Koonin EV. Evolution and classification of the CRISPR-Cas systems., 2011, 9(6): 467–477.
[14] Pourcel C, Salvignol G, Vergnaud G. CRISPR elements inacquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies., 2005, 151(Pt 3): 653–663.
[15] Horvath P, Romero DA, Coûté-Monvoisin AC, Richards M, Deveau H, Moineau S, Boyaval P, Fremaux C, Barrangou R.Diversity, activity, and evolution of CRISPR loci in., 2008, 190(4): 1401–1412.
[16] Barrangou R, Fremaux C, Deveau H, Richards M, Boyaval P, Moineau S, Romero DA, Horvath P. CRISPR provides acquired resistance against viruses in prokaryotes., 2007, 315(5819): 1709–1712.
[17] Brouns SJJ, Jore MM, Lundgren M, Westra ER, Slijkhuis RJH, Snijders APL, Dickman MJ, Makarova KS, Koonin EV, van der Oost J. Small CRISPR RNAs guide antiviral defense in prokaryotes., 2008, 321(5891): 960–964.
[18] Haft DH, Selengut J, Mongodin EF, Nelson KE. A guild of 45 CRISPR-associated (Cas) protein families and multiple CRISPR/Cas subtypes exist in prokaryotic genomes., 2005, 1(6): e60.
[19] Horvath P, Coûté-Monvoisin AC, Romero DA, Boyaval P, Fremaux C, Barrangou R. Comparative analysis of CRISPR loci in lactic acid bacteria genomes., 2009, 131(1): 62–70.
[20] Díez-Villaseñor C, Almendros C, García-Martínez J, Mojica FJ. Diversity of CRISPR loci in., 2010, 156(Pt 5): 1351–1361.
[21] Bland C, Ramsey TL, Sabree F, Lowe M, Brown K, Kyrpides NC, Hugenholtz P. CRISPR recognition tool (CRT): a tool for automatic detection of clustered regularly interspaced palindromic repeats., 2007, 8: 209.
[22] Crooks GE, Hon G, Chandonia JM, Brenner SE. WebLogo: a sequence logo generator., 2004, 14(6): 1188–1190.
[23] Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. MEGA6: molecular evolutionary genetics analysis version 6.0., 2013, 30(12): 2725–2729.
[24] Zhou Y, Liang YJ, Lynch KH, Dennis JJ, Wishart DS. PHAST: a fast phage search tool., 2011, 39(Web Server issue): W347–W352.
[25] Lima-Mendez G, Van Helden J, Toussaint A, Leplae R. Prophinder: a computational tool for prophage prediction in prokaryotic genomes., 2008, 24(6): 863–865.
[26] Siguier P, Perochon J, Lestrade L, Mahillon J, Chandler M. ISfinder: the reference centre for bacterial insertion sequences., 2006, 34(Database issue): D32–D36.
[27] Jiang FG, Doudna JA. The structural biology of CRISPR- Cas systems., 2015, 30: 100–111.
[28] Nozawa T, Furukawa N, Aikawa C, Watanabe T, Haobam B, Kurokawa K, Maruyama F, Nakagawa I. CRISPR inhibition of prophage acquisition in., 2011, 6(5): e19543.
Comparative genomics and evolutionary analysis of CRISPR loci in acetic acid bacteria
Kai Xia, Xinle Liang, Yudong Li
The clustered regularly interspaced short palindromic repeat (CRISPR) is a widespread adaptive immunity system that exists in most archaea and many bacteria against foreign DNA, such as phages, viruses and plasmids. In general, CRISPR system consists of direct repeat, leader, spacer and CRISPR-associated sequences. Acetic acid bacteria (AAB) play an important role in industrial fermentation of vinegar and bioelectrochemistry. To investigate the polymorphism and evolution pattern of CRISPR loci in acetic acid bacteria, bioinformatic analyses were performed on 48 species from three main genera (,r and) with whole genome sequences available from the NCBI database. The results showed that the CRISPR system existed in 32 species of the 48 strains studied. Most of the CRISPR-Cas system in AAB belonged to type I CRISPR–Cas system (subtype E and C), but type II CRISPR-Cas system which containgene was only found in the genusand. The repeat sequences of some CRISPR were highly conserved among species from different genera, and the leader sequences of some CRISPR possessed conservative motif, which was associated with regulated promoters. Moreover, phylogenetic analysis ofdemonstrated that they were suitable for classification of species. The conservation ofgenes was associated with that of repeat sequences among different strains, suggesting they were subjected to similar functional constraints. Moreover, the number of spacer was positively correlated with the number of prophages and insertion sequences, indicating the acetic acid bacteria were continually invaded by new foreign DNA. The comparative analysis of CRISR loci in acetic acid bacteria provided the basis for investigating the molecular mechanism of different acetic acid tolerance and genome stability in acetic acid bacteria.
acetic acid bacteria; CRISPR; repeat sequence; genetic evolution
2015-06-01;
2015-09-07
国家自然科学基金(编号:3117175)和浙江省自然科学基金(编号:LY14C060001)资助
夏凯,硕士研究生,专业方向:醋酸菌分子生物学。E-mail: xiakai333@126.com
李余动,博士,副教授,研究方向:微生物基因组学。E-mail: lyd@zjsu.edu.cn
10.16288/j.yczz.15-244
网络出版时间: 2015-10-8 15:57:12
URL: http://www.cnki.net/kcms/detail/11.1913.R.20151008.1557.002.html
(责任编委: 谢建平)
