甲基对硫磷水解酶的酿酒酵母表面展示及在甲基对硫磷降解中的应用
2015-10-26王星星池哲
王星星池哲
(1.甘肃省科学技术情报研究所,兰州 730000;2.中国海洋大学,青岛 266000)
甲基对硫磷水解酶的酿酒酵母表面展示及在甲基对硫磷降解中的应用
王星星1池哲2
(1.甘肃省科学技术情报研究所,兰州 730000;2.中国海洋大学,青岛 266000)
PCR扩增假单胞菌WBC-3的甲基对硫磷水解酶基因,插入表面展示质粒pYD1的多克隆位点,构建pYD1-MPH重组质粒。重组质粒转化酿酒酵母EBY100,2%半乳糖诱导甲基对硫磷水解酶表达,并利用免疫荧光检测甲基对硫磷水解酶在酿酒酵母细胞表面的表达展示。研究了表面展示甲基对硫磷水解酶的酶学性质和酵母工程菌对水体中甲基对硫磷的降解效果。结果表明成功构建具有全细胞甲基对硫磷水解酶催化活性的酵母工程菌,经2%半乳糖诱导48 h,表面展示甲基对硫磷水解酶比酶活力为18.2 U/mg细胞干重。表面展示甲基对硫磷水解酶的最适作用pH为9.5,最适作用温度为30℃,在pH4.0-10.5之间和45℃以下稳定性较好,Mn2+、Co2+、Zn2+、Ca2+、Hg2+、K+、Ni2+对表面展示甲基对硫磷水解酶活性有激活作用,Na+、Fe3+、Ag+对展示酶活力有抑制作用。工程菌在1 h内对淡水中20 mg/L的甲基对硫磷的降解率在80%以上。
甲基对硫磷水解酶;表面展示;酿酒酵母;甲基对硫磷降解
甲基对硫磷是一种高毒的有机磷农药,使用后残留期长,是一种持久性有机污染物[1],在国内水源和食物中时有检出[2,3]。甲基对硫磷作为一种神经毒剂,可通过抑制生物体内乙酰胆碱酯酶的活性而造成乙酰胆碱累积,引起神经功能紊乱,严重时导致个体死亡,对生物体有较大的毒害作用[4]。
甲基对硫磷水解酶(Methyl parathion hydrolase,MPH)是由甲基对硫磷水解酶基因编码的酯酶,可破坏甲基对硫磷中的磷酯键,使甲基对硫磷的毒性下降约120倍[5]。因此,利用基因工程技术生产高效、廉价的甲基对硫磷水解酶制剂是解决环境中甲基对硫磷残留的一种非常具有潜力的方法。目前国内外已分离得到许多甲基对硫磷降解菌,克隆了编码甲基对硫磷水解酶的基因并且进行了异源表达[6-11]。但是分离纯化甲基对硫磷水解酶操作繁琐,成本较高,而且会造成酶活性的损失;而直接利用全细胞进行甲基对硫磷的降解,却因细胞内表达的酶受到细胞膜的阻隔不能与胞外底物充分接触而限制了酶活性的发挥[12]。
微生物表面展示技术是近年来发展较快的一种基因工程技术,其通过将外源蛋白与特定的载体蛋白(锚定蛋白)基因序列融合,使外源蛋白表达锚定在微生物细胞表面[13],在全细胞催化、蛋白分离纯化、环保领域具有良好的应用前景。因此,利用表面展示技术将甲基对硫磷水解酶分泌并锚定在微生物细胞表面,无需酶的纯化,并且突破细胞膜的阻碍使酶和底物充分接触,成为开发高效、简便的全细胞甲基对硫磷水解酶的有效手段。
目前,国内外研究者利用大肠杆菌表面展示系统和假单胞菌表面展示系统进行包括甲基对硫磷水解酶在内的有机磷水解酶的表面展示[14-17],酵母表面展示系统在有机磷水解酶表达展示中的研究较少。酵母表面展示系统是一种常见的展示系统,其锚定蛋白为酵母细胞壁中存在的一类甘露糖蛋白,称为细胞壁蛋白,其中包括酿酒酵母的α和a凝集素。这类蛋白通过共价键与酵母细胞壁的葡聚糖相连接[13]。相较于细菌表面展示系统,酵母表面展示系统具有较完善的蛋白质分泌机制和翻译后修饰,以及安全无毒等优点,利用酵母表面展示系统构建安全、高效的有机磷农药降解工程菌株用于有机磷农药污染治理具有广阔的研究空间。酿酒酵母是酵母表面展示的宿主细胞之一,为食品级微生物,遗传操作方便,安全、容易培养,适合大规模工业生产,适用于开发安全、经济的全细胞甲基对硫磷水解酶。本研究将来源于假单胞菌Pseudomonas sp. WBC-3的甲基对硫磷水解酶表达展示于酿酒酵母细胞表面,检测全细胞酶的活性,并且进行展示酶的酶学性质研究以及全细胞酶对水体中甲基对硫磷的降解研究,为开发新型、高效的全细胞甲基对硫磷水解酶奠定基础,为甲基对硫磷的生物降解提供理论依据和技术支撑。
1 材料与方法
1.1 材料
1.1.1 菌种与质粒 质粒pYD1和其受体菌酿酒酵母EBY100购自美国Invitrogen公司;质粒pET20b带有假单胞菌WBC-3的甲基对硫磷水解酶基因(GenBank accession No.AY251554),由武汉病毒所张先恩研究员提供;E.coli DH5α,购自北京TIANGEN生物公司;pMD19-T simple vector购自宝生物(大连)有限公司(TaKaRa)。
1.1.2 分子生物学酶及试剂 限制性内切酶EcoR I和Xho I、T4 DNA连接酶购自FERMENTAS(MBI)公司;质粒提取试剂盒购自北京TIANGEN生物公司;LA Taq DNA聚合酶、DNA琼脂糖凝胶回收试剂盒、DNA Marker购自宝生物(大连)有限公司(TaKa-Ra);甲基对硫磷纯品(Dr. Ehrenstorfer GmbH,Germany)购自青岛艾科宝生物技术有限公司,纯度为99.5%,配制为10 mg/mL的母液(溶于甲醇);小鼠抗6×His单克隆抗体购于美国Clontech公司;异硫氰酸荧光素(FITC)标记的羊抗鼠IgG抗体购自北京TIANGEN生物公司;其他试剂均为国产分析纯。
1.2 方法
1.2.1 甲基对硫磷水解酶基因的克隆及鉴定 根据假单胞菌WBC-3的甲基对硫磷水解酶基因序列和表面展示质粒pYD1多克隆位点设计引物mphpyd1-se和mphpyd1-an,在上下游引物中分别引入EcoR I和Xho I限制性内切酶位点,引物序列分别 为:mphpyd1-se:GAATTCGCCGCACCGCAGGTG(下划线处为EcoR I酶切位点,mphpyd1-an:CTCGAGCTTGGGGTTGACGACCG(下划线处为Xho I酶切位点)。以质粒pET20b为模板,mphpyd1-se和mphpyd1-an为引物进行扩增。
PCR扩增体系为:2×GC buffer 25.0 μL,2.5 mmol/L dNTP 8.0 μL;20 μmol/L引物(mphpyd1-se、mphpyd1-an)各1.0 μL,质粒pET20b 1.0 μL,LA Taq DNA聚合酶 0.5 μL,ddH2O 13.5 μL。PCR反应参数:94℃预变性3 min;94℃变性30 s,63℃退火30 s,72℃延伸1 min,共30个循环;72℃延伸10 min。胶回收PCR扩增条带,与pMD19-T simple Vector连接,转化E.coli DH5α。挑取白斑克隆培养,提取质粒,用EcoR I、Xho I双酶切验证。选取酶切验证正确的质粒,送博尚生物技术(上海)有限公司测序、比对。将测序正确的质粒命名为pMD19-T simple-MPH。
1.2.2 pYD1-MPH重组质粒的构建及鉴定 将质粒pMD19-T simple-MPH和pYD1进行EcoR I和Xho I双酶切,将酶切回收后的甲基对硫磷水解酶基因片段和质粒pYD1连接、转化E.coli DH5α,再对重组质粒进行EcoR I和Xho I双酶切鉴定,将验证正确的重组质粒命名为pYD1-MPH。
1.2.3 酿酒酵母EBY100转化及MPH的诱导表达 按照Invitrogen公司说明书方法将重组质粒pYD1-MPH转化酿酒酵母EBY100,同时以转入不含MPH基因片段的pYD1作为阳性对照,在添加了亮氨酸的选择培养基平板上筛选阳性转化子。挑取转化子接种于2.0%葡萄糖的YNB-CAA的培养基中30℃振荡培养过夜,菌体用PBS缓冲液洗涤后,重悬于含2.0% D-半乳糖的YNB-CAA液体培养基中25℃振荡培养,诱导目的蛋白表达。每隔12 h取样测定展示表达的酶活,以含pYD1空质粒的转化子作对照。
1.2.4 表面展示甲基对硫磷水解酶活性测定 表面展示甲基对硫磷水解酶活力测定参照Yu等[10]的方法进行。反应体系为1 000.0 μL,其中含6.0 μL 10.0 mg/mL甲基对硫磷母液、929.0 μL 40.0 mmol/L 甘氨酸-氢氧化钠缓冲液(pH9.5)和65.0 μL菌体浓度为1.6×108个细胞/mL的菌悬液。30℃反应10 min,加入200.0 μL 10% HNO3终止反应。离心反应液后取 500.0 μL上清液,加入5.0 mL 0.5 mol/L甘氨酸-氢氧化钠缓冲液(pH10.0)混匀,405 nm下测定吸光值。OD405值反映了甲基对硫磷水解产物对硝基苯酚的生成量。表面展示甲基对硫磷水解酶酶活力单位定义为:在30℃、pH9.5的条件下,每分钟水解1.0 μmol底物即产生1.0 μmol对硝基苯酚所需要的表面展示甲基对硫磷水解酶酶量。比活力为每毫克菌体干重具有的甲基对硫磷水解酶活力,单位为U/mg细胞干重。
1.2.5 细胞免疫荧光检测 取诱导表达48 h相当于含OD600值为2.0的菌液,以转入空载体pYD1的转化子和EBY100分别作为阳性对照和阴性对照,分别以小鼠抗6×His单克隆抗体和异硫氰酸荧光素(FITC)标记的羊抗鼠IgG抗体作为抗体进行免疫荧光检测。具体试验操作方法参照Yue等[18]的方法进行。
1.2.6 表面展示甲基对硫磷水解酶的酶学性质研究 分别用pH值为3.0、3.5、4.0、4.5、5.0、5.5、6.0、6.5、7.0、7.5、8.0、8.5、9.0、9.5、10.0、10.5、11.0的酶反应缓冲液取代酶活测定方法中的缓冲液,按照1.2.4的方法测定酶活,确定酶的最适反应pH。将细胞悬液分别置于上述pH值的缓冲液,4℃保温24 h,测定剩余酶活力,确定酶的pH稳定性。
按照1.2.4的方法,分别在20、25、30、35、40、45、50、55、60、65、70℃水浴反应10 min,测定展示酶活性,确定酶的最适反应温度。将细胞悬液分别在上述温度保温1 h,按照1.2.4的方法,测定剩余酶活,以放置于4℃的酵母细胞的酶活力作为对照(100%),确定酶的温度稳定性。
1.2.7 金属离子对表面展示甲基对硫磷水解酶活性的影响 将表面展示甲基对硫磷水解酶的酵母细胞悬液与不同金属离子混合,金属离子终浓度为5.0 mmol/L。混合液于4.0℃下保温1 h,然后按照1.2.4的方法,测定剩余酶活。对照为加同等体积蒸馏水的细胞酶活,根据剩余酶活判断金属离子对展示酶活性的影响。
1.2.8 水体中甲基对硫磷降解研究 在1 000.0 mL的三角瓶中加入500.0 mL甲基对硫磷浓度为20.0 mg/L的淡水,水体的pH为其自然pH和全细胞酶作用的最适pH,再加入适量的全细胞酶,使菌体终浓度为3×107个细胞/mL。充分混匀,30℃、180 r/min振荡反应1 h,检测对硝基苯酚的生成量,计算甲基对硫磷的降解率。反应结束后离心收集菌体用于下一次降解试验,并测定每轮反应后全细胞酶的剩余活力,以第一次反应前全细胞酶活力作为对照(100%)。
2 结果
2.1 甲基对硫磷水解酶基因片段的扩增和重组质粒pYD1-MPH的构建
PCR扩增后得到903 bp的特异性条带(图1),克隆入pMD19-T-simple Vector,EcoR I、Xho I酶切验证正确后测序分析,结果与假单胞菌WBC-3的甲基对硫磷水解酶基因序列(GenBank accession No.AY251554)相一致。质粒pMD19-T simple-MPH与质粒pYD1双酶切,回收MPH基因片段和pYD1载体片段,连接、转化E.coli DH5α,阳性转化子质粒EcoR I、Xho I双酶切验证结果见图2,质粒pYD1-MPH被切为两个片段,小片段为MPH基因片段,大片段为pYD1片段,表明重组质粒pYD1-MPH构建成功。
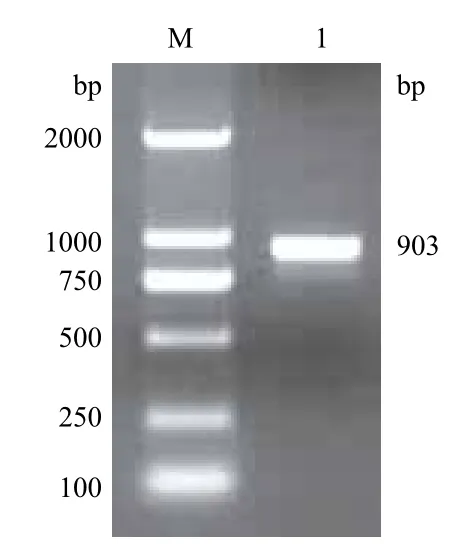
图1 甲基对硫磷水解酶基因PCR扩增产物电泳图

图2 重组质粒pYD1-MPH EcoR I、Xho I双酶切鉴定
2.2 表面展示甲基对硫磷水解酶活力的测定
挑取验证正确的转化子培养测定展示酶活力,以含pYD1空质粒的转化子进行对照。结果表明阳性转化子在诱导培养48 h时酶活最高,为(18.2±1.1)U/mg细胞干重。而转化入空表面展示质粒pYD1的酿酒酵母EBY100没有甲基对硫磷水解酶活力。
2.3 细胞免疫荧光检测
质粒pYD1含有6×His标签,可以利用鼠抗6×His单克隆抗体作为一抗,异硫氰酸荧光素(FITC)标记的羊抗鼠IgG抗体作为二抗,对重组酵母细胞进行免疫荧光检测。从图3中可以看到,转入质粒pYD1-MPH和空载体pYD1的转化子细胞表面可以观察到荧光,而未转化菌株EBY100则检测不到荧光。说明目的蛋白展示到了酿酒酵母EBY100的表面,达到了表面展示的目的。
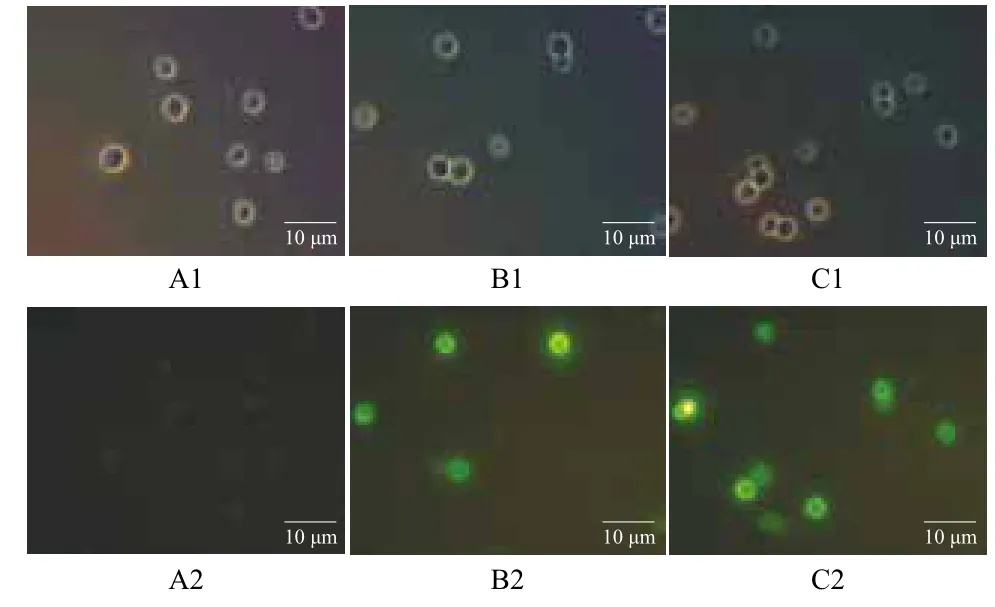
图3 免疫荧光检测表面展示的甲基对硫磷水解酶
2.4 表面展示甲基对硫磷水解酶最适反应pH值和pH值稳定性
从图4可以看出,表面展示甲基对硫磷水解酶的最适作用pH为9.5,在pH4.0-10.5之间酶的稳定性较好,在pH4.0-10.5的缓冲液中4℃下处理24 h后,其仍能保持80%以上的活性。
2.5 表面展示甲基对硫磷水解酶最适反应温度和温度稳定性
图5的结果表明,表面展示甲基对硫磷水解酶的最适作用温度为30℃。在45℃以下稳定性较好,在45℃保温1 h仍能保留80.0%的活性,但在高于45℃时活性下降很快,在55℃保温1 h后表面展示甲基对硫磷水解酶几乎完全失活。
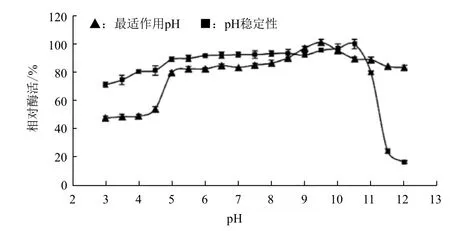
图4 pH对表面展示甲基对硫磷水解酶的活性和稳定性的影响
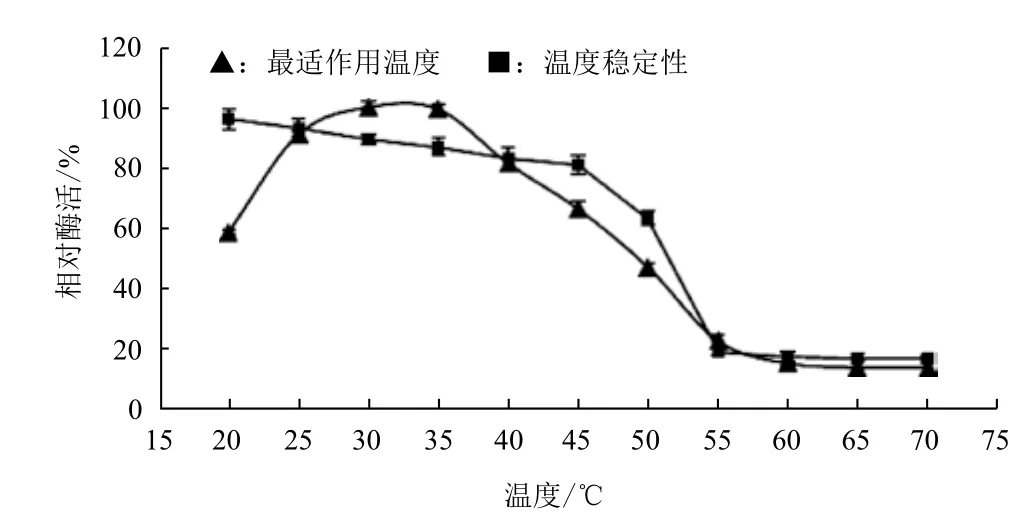
图5 温度对表面展示甲基对硫磷水解酶的活性和稳定性的影响
2.6 金属离子对表面展示甲基对硫磷水解酶活力的影响
如表1所示,5.0 mmol/L的Mn2+对表面展示甲基对硫磷水解酶的激活作用最显著,使展示酶的活力提高将近3倍,Co2+对展示酶也有显著的激活作用,酶活力提高两倍多。Zn2+、Ca2+、Hg2+、K+、Ni2+对表面展示甲基对硫磷水解酶也有激活作用,但是Na+、Fe3+、Ag+对展示酶活力有抑制作用。Mg2+、Cu2+、Fe2+、Ba2+、Li+对展示酶活力没有明显的影响。

表1 金属离子对表面展示甲基对硫磷水解酶活力的影响
2.7 水体中甲基对硫磷降解研究
全细胞酶在1 h内对不同pH淡水中20.0 mg/L甲基对硫磷的降解率均可达到80%以上,受水体pH的影响较小(图6)。全细胞酶重复使用3次后,仍能保持70%以上的活性(表2)。
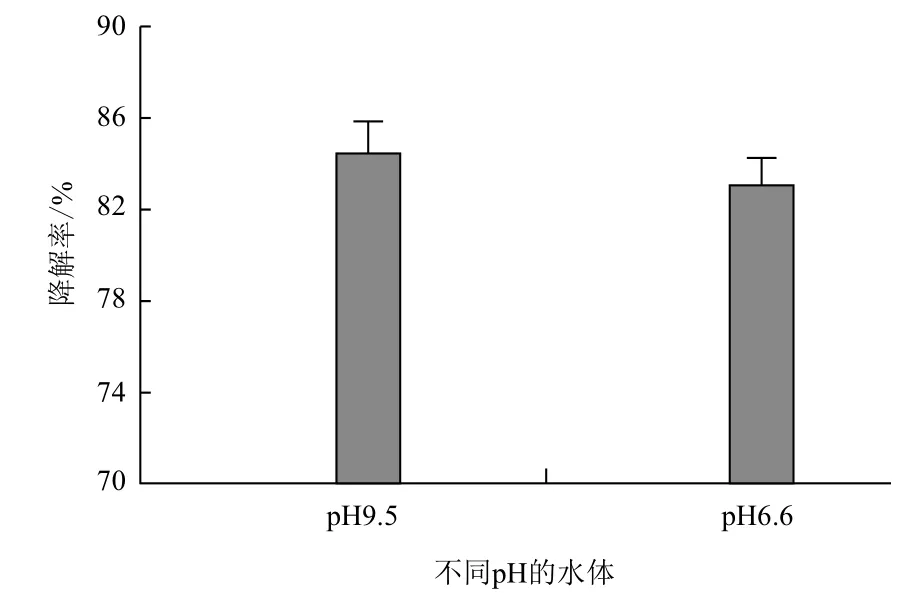
图6 不同pH的淡水中甲基对硫磷的降解率

表2 全细胞酶的重复利用性
3 讨论
由于表面展示技术将外源蛋白锚定在微生物细胞表面,可直接利用全细胞进行催化反应,避免了酶的分离纯化,并且酶与底物充分接触,不受细胞膜的阻隔,因此近年来,表面展示技术在有机磷农药降解的研究应用中发展迅速。目前国内用于包括甲基对硫磷水解酶在内的有机磷水解酶表面展示的宿主细胞多集中于大肠杆菌、假单胞菌[14-17],酵母表面展示系统在有机磷水解酶表达展示中的研究很少,甲基对硫磷水解酶在酿酒酵母中的表面展示尚未有报道。本研究成功将假单胞菌WBC-3的甲基对硫磷水解酶展示在酿酒酵母EBY100细胞表面,构建了表面展示甲基对硫磷水解酶的酵母工程菌株,其甲基对硫磷水解酶比酶活为18.2 U/mg细胞干重。
本研究中的表面展示甲基对硫磷水解酶最适作用pH值为9.5,最适作用温度为30℃,而假单胞菌WBC-3所产甲基对硫磷水解酶的最适作用pH和最适作用温度值为11.0、40℃[19],这可能是由于表面展示重组表达的甲基对硫磷水解酶与锚定蛋白融合表达以及在表达时发生N-糖基化造成的[20]。Yang等[11]在大肠杆菌BL21中重组表达的甲基对硫磷水解酶在25-35℃和pH7.0-9.0时能保持90%以上的活性,本研究中,表面展示甲基对硫磷水解酶在pH4.0-10.5和45℃以下稳定性较好,在25-35℃和pH7.0-9.0能保持约90%的活性。说明本研究中的全细胞酶具有良好的稳定性,在不同温度、pH的环境条件下不易变性失活,而且较非表面展示表达的甲基对硫磷水解酶更经济、方便。
金属离子对本研究中表面展示的甲基对硫磷水解酶活性具有一定影响,其中加入5.0 mmol/L的Mn2+可使展示酶比酶活达到71.9 U/mg细胞干重。Co2+、Zn2+、Ca2+、Hg2+、K+、Ni2+对展示酶均有激活作用,Na+、Fe3+、Ag+对酶活力有抑制作用,Mg2+、Cu2+、Fe2+、Ba2+、Li+对酶活力没有明显影响。楚晓娜等[19]研究发现不同金属离子对假单胞菌WBC-3所产天然甲基对硫磷水解酶的活性也有影响,因此推测该酶为金属酶。通过对该酶三维结构的研究发现,该酶的活性中心由Zn2+和Cd2+组成[21]。据报道,有机磷水解酶是一种金属酶,具有由双Zn2+或Co2+组成的活性中心,活性中心的金属离子可以被Ni2+、Ca2+、Mn2+、Cd2+取代而保持活性[22]。因此推测表面展示的甲基对硫磷水解酶活性中心的Zn2+和Cd2+可能也可以被相应的金属离子取代而使活性发生改变。
本研究的全细胞酶对水体中甲基对硫磷具有良好的降解效果,受水体pH的影响较小,而且操作稳定性较好,可多次重复利用,具有一定的实际应用潜力。但是本研究构建的酵母工程菌其甲基对硫磷水解酶的表达和展示必须要经过2%的半乳糖的诱导,不利于全细胞酶投入生产;质粒pYD1上携带有抗生素抗性基因,酵母工程菌在实际应用过程中存在抗生素抗性基因扩散的潜在危险。因此,本研究构建的酵母工程菌成为具有实用性的全细胞催化剂还需要进一步的探索。通过质粒改造使目的基因的表达不再需要诱导剂,并且移除抗生素抗性基因,同时尝试选用不同的表面展示锚定蛋白,探索不同锚定蛋白对酶表达和活性的影响,进一步提高表面展示酶的活性,将是以后构建安全、高效、适用的表面展示甲基对硫磷水解酶工程菌研究的方向。
4 结论
本研究将假单胞菌WBC-3的甲基对硫磷水解酶在酒精酵母细胞中进行了表达展示,构建了具有全细胞甲基对硫磷水解酶催化活性的酵母工程菌。表面展示甲基对硫磷水解酶的比酶活为18.2 U/mg细胞干重,最适作用pH和最适作用温度为9.5、30℃,在pH4.0-10.5之间和45℃以下稳定性较好。Mn2+、Co2+、Zn2+、Ca2+、Hg2+、K+、Ni2+对展示酶有激活作用,Na+、Fe3+、Ag+对展示酶活力有抑制作用。工程菌在1 h内对淡水中20 mg/L的甲基对硫磷的降解率在80%以上。
[1]Liu FY, Hong MZ, Liu DM, et al. Biodegradation of methyl parathion by Acinetobacter radioresistens USTB-04[J]. J Environ Sci, 2007,19(10):1257-1260.
[2]朱丽芳, 周文军, 王经委. 杭嘉湖地区饮用水源有机农药污染现状调查与分析[J]. 浙江水利水电专科学校学报, 2012, 24(2):51-59.
[3]于志勇, 金芬, 孙景芳, 等. 北京市场常见淡水食用鱼体内农药残留水平调查及健康风险评价[J]. 环境科学, 2013, 34(1):251-256.
[4]Theriot CM, Grunden AM. Hydrolysis of organophosphorus compounds by microbial enzymes[J]. Appl Microbiol Biotechnol,2011, 89(1):35-43.
[5]Cui ZL, Li SP, Fu GP. Isolation of methyl parathion degrading strain M6 and cloning of the methyl parathion hydrolase gene[J]. Appl Environ Microbiol, 2001, 67:4922-4925
[6]Pakala SB, Gorla P, Pinjari AB, et al. Biodegradation of methyl parathion and p-nitrophenol:evidence for the presence of a p-nitrophenol 2-hydroxylase in a gram-negative Serratia sp. strain DS001[J]. Appl Microbiol Biotechnol, 2007, 73:1452-1462.
[7]Fu GP, Cui Z, Huang T, et al. Expression, purification, and characterization of a novel methyl parathion hydrolase[J]. Prot Expres Purif, 2004, 36:170-176.
[8]Liu H, Zhang JJ, Wang SJ, et al. Plasmid-borne catabolism of methyl parathion and p-nitrophenol in Pseudomonas sp. strain WBC-3[J]. Biochem Biophys Res Commun, 2005, 334:1107-1114.
[9]Shen YJ, Lu P, Mei H, et al. Isolation of a methyl parathiondegrading strain Stenotrophomonas sp. SMSP-1 and cloning of the ophc2 gene[J]. Biodegradation, 2010, 21(50):785-792.
[10] Yu H, Yan X, Shen W, et al. Expression of methyl parathion hydrolase in Pichia pastoris[J]. Curr Microbiol, 2009, 59:573-578.
[11] Yang JJ, Yang C, Jiang H, et al. Overexpression of methyl parathion hydrolase and its application in detoxification of organophosphates[J]. Biodegradation, 2008, 19:831-839.
[12] Yang C, Zhao Q, Liu Z, et al. Cell surface display of functional macromolecule fusions on Escherichia coli for development of an autofluorescent whole-cell[J]. Biocatalyst Environ Sci Technol,2008, 42(16):6105-6110.
[13]Ueda M, Tanaka A. Genetic immobilization of proteins on the yeast cell surface[J]. Biotechnol Adv, 2000, 18:121-140.
[14] Li CK, Zhu YR, Benz I, et al Presentation of functional organophosphorus hydrolase fusions on the surface of Escherichia coli by the AIDA-I autotransporter pathway[J]. Biotechnol Bioeng, 2008, 99:485-490.
[15]张红星, 李茜茜, 叶婷, 等. 有机磷水解酶的大肠杆菌细胞表面展示与酶活特性[J]. 华中农业大学学报, 2008, 54(2):227-233.
[16]张红星, 李茜茜, 叶婷, 等. 细胞表面展示有机磷水解酶的恶臭假单胞菌工程菌的构建及全细胞酶活性分析[J]. 华中农业大学学报, 2008, 27(1):65-70.
[17]Yang C, Cai N, Dong M, et al. Surface display of MPH on Pseudomonas putida JS444 using ice nucleation protein and its application in detoxification of organophosphates[J]. Biotechnol Bioeng, 2008, 99(1):30-37.
[18]Yue LX, Chi ZM, Wang L, et al. Construction of a new plasmid for surface display on cells of Yarrowia lipolytica[J]. J Microbiol Method, 2008, 72:116-123.
[19]楚晓娜, 张先恩, 陈亚丽, 等. 假单胞菌WBC-3甲基对硫磷水解酶性质的初步研究[J]. 微生物学报, 2003, 43(4):453-459.
[20]Ryckaert S, Martens V, De Vusser K, et al. Development of a Saccharomyces cerevisiae whole cell biocatalyst for in vitro sialylation of oligosaccharides[J]. J Biotechnol, 2005, 119(4):379-388.
[21]Dong YJ, Bartlam M, Sun L, et al. Crystal structure of methyl parathion hydrolase from Pseudomonas sp. WBC-3[J]. J Mol Biol, 2005, 353(3):655-663.
[22]Efremenko EN, Sergeeva VS. Organophosphate hydrolase-an enzyme catalyzing degradation of phosphorus containing toxin and pesticides[J]. Russ Chem Bull Intern Ed, 2001, 50(10):1826-1832.
(责任编辑 李楠)
Surface Display of Methyl Parathion Hydrolase on Saccharomyces cerevisiae and Its Application in Degradation of Methyl Parathion
Wang Xingxing1Chi Zhe2
(1. Institute of Science and Technology Information of Gansu,Lanzhou 730000;2.Ocean University of China,Qingdao 266000)
The methyl parathion hydrolase(MPH)gene of Pseudomonas sp.WBC-3 was amplified by PCR and cloned into the multiple cloning site of the surface display vector pYD1 to construct a recombinant plasmid pYD1-MPH. Then plasmid pYD1-MPH was transformed into Saccharomyces cerevisiae EBY100. The 2% galactose was used to induce the expression of MPH on the cell surface of EBYl00, and the display of MPH on the cell surface of S.cerevisiae was confirmed by immunofluorescence. The characteristic of the displayed MPH and degradation effect of methyl parathion in water by the engineered yeast were also investigated. The result showed that the engineered yeast strain, which have a whole cell catalytic activity of MPH, was successfully constructed. The activity of MPH displayed on the yeast cells was 18.2 U/mg of cell dry cells by the induction of 2% galactose for 48 h. The displayed MPH had the optimal pH of 9.5 and the optimal temperature of 30℃, respectively and was stable in the pH range of 4.0-10.5 and up to 45℃. The displayed MPH was stimulated by Mn2+,Co2+,Zn2+,Ca2+,Hg2+,K+,Ni2+, and was inhibited by Na+,Fe3+,Ag+. The engineered yeast strain could hydrolyze over 80% of 20.0 mg/L methyl parathion in tap water in 1 h.
methyl parathion hydrolase;surface display;Saccharomyces cerevisiae;methyl parathion degradation
10.13560/j.cnki.biotech.bull.1985.2015.04.026
2014-08-06
甘肃省科技计划资助项目(145RJZA042)
王星星,女,助理研究员,研究方向:污染生态学;E-mail:mmwxx1105@163.com
