氨基甲酸乙酯水解酶的家族生物信息学分析
2022-04-01李若熙杨丽娟
张 献,彭 涛,张 耀,李若熙,杨丽娟
(酿酒生物技术及应用四川省重点实验室(四川轻化工大学),四川 宜宾 644000)
氨基甲酸乙酯(Ethyl carbamate 或Urethane,简称EC),存在于多种发酵食品和酒精饮料中,是一种具有致癌性的物质[1-2]。2007年,EC被国际癌症研究机构IARC(the International Agency for Rescarch on Cancer)归类为2A类致癌物质[3]。日本、加拿大等[4-5]国家对酒精饮料中EC含量有着严格的限量标准。目前消除EC有多种方法,包括对发酵工艺的优化、物理吸附、代谢工程改造酵母以及生物酶法消除等,其中用生物酶法来消除EC被认为是最为理想的一种方法[6]。关于消除EC的生物酶研究中,用于EC消除的酶有两种,一种是酸性脲酶(Acid urease);另外一种就是氨基甲酸乙酯水解酶(Urethane hydrolase或urethanase)。1990年,Kobashi[7]等日本学者从小鼠肠道中的Citrobactersp.中第一次发现EC水解酶,证实其能够降解EC。2006年,Akutsu-ShigenoY[8]等从马红球菌(RhodococcusequistrainTB-60)中得到了EC水解酶,并实现了该酶的异源表达。李京京[9]等从小鼠的胃中筛选出一株具有EC降解能力的赖氨酸芽孢杆菌LysinibacillusfusiformisSC02,并通过对其蛋白质的N段测序,获得了该酶的氨基酸序列。EC水解酶存在于多种微生物中,并且不同微生物的EC水解酶的酶学性质差异较大,普遍存在对乙醇和酸的耐受力差、对底物的亲和力低的问题[8, 10]。2016年,刘晓慧[11]等人通过计算机辅助以及定向改造技术对来自于赖氨酸芽孢杆菌LysinibacillusfusiformisSC02的EC水解酶进行改造,提高了该酶的温度稳定性,但其对乙醇的耐受力并未有所提升。此外,EC水解酶的氨基酸序列未得到有效解析。2019年,Masaki[12]等日本学者在假丝酵母Candidaparapsilosis得到了EC水解酶,并获得了该酶的基因序列。目前,已报道的EC水解酶均属于酰胺酶家族,从EC分子的结构上来看,酯酶家族部分酶也能够对其进行水解,但是还未见有酯酶降解EC的报道[6](见图1)。EC水解酶存在的这些问题限制了EC水解酶的发展与应用。若采用基因工程以及蛋白质工程的手段,对EC水解酶进行分析优化等研究,解决其存在的问题,可进一步推进EC水解酶在食品中的应用。

图1 氨基甲酸乙酯水解机制[6]Fig.1 Hydrolysis mechanism of urethane[6]
本研究采用生物信息学的方法对EC水解酶进行分析,结合ProtParam、ProtScale、SignalP 5.0 Server、NetPhos 2.0 Server等生物信息学软件,对微生物的EC水解酶氨基酸序列分别进行理化性质、序列分子进化、亲水或疏水性、二级结构、功能域、信号肽以及蛋白的磷酸化等方面进行分析和预测,为下一步EC水解酶的酶分子改造、基因工程菌的构建、表达等奠定基础。
1 材料与方法
1.1 数据来源
试验中的EC水解酶(UH)序列均来自美国国家生物信息中心(NCBI)(https://www.ncbi.nlm.nih.gov/)中已收录的完整的氨基酸序列,分别来自于马红球菌、赖氨酸芽孢杆菌以及假丝酵母(见表1)。

表1 氨基酸序列基本信息Table 1 Basic information of amino acid sequence
1.2 方法
采用Expasy网站提供的ProtParam工具对EC水解酶的氨基酸序列进行理化性质分析;亲疏水性特征使用Expasy网站的ProtScale工具进行分析;使用在线工具NPSA server中的SOPMA进行微EC水解酶的氨基酸序列二级结构分析和预测[13];使用CDD和SMART[14]对EC水解酶保守结构的分析;EC水解酶信号肽的分析和预测采用工具SignalP 5.0 server;运用TMHMM 2.0 Server进行EC水解酶的跨膜结构域的分析和预测;NetPhosK 3.1 Server进行EC水解酶的氨基酸序列磷酸化位点分析;使用SWISS-MODEL[15]和I-TASSER对EC水解酶进行3D建模,以及使用SAVES[16]对模型进行评价。利用Clustal Omega[17]进行氨基酸的多序列比对分析,并用ESPript3.0进行序列对比的结果显示。在线分析工具网址(见表2)。

表2 生物学在线分析工具网址Table 2 Websites of online biology analysis tools
2 结果与分析
2.1 EC水解酶蛋白序列理化性质分析
使用ProtParam工具分析3条完整的EC水解酶蛋白序列(见表3)。结果显示,目前在NCBI上所能检索到的EC水解酶氨基酸序列中,氨基酸数量在472-551之间,氨基酸数量最大的是假丝酵母,为551 aa,分子量为61.74 kDa左右。理论等电点(pI)范围在5.03~5.71之间,最大的为Candidaparapsilosis。三个蛋白的负电荷氨基酸残基总数(Asp + Glu)都大于正电荷氨基酸残基总数(Arg + Lys)。同时对三个EC水解酶的氨基酸残基组成也进行了分析(见表4)。发现3个EC水解酶蛋白共有的且含量丰富的主要氨基酸为丙氨酸(Ala)、甘氨酸(Gly)、亮氨酸(Leu)。在蛋白质稳定性方面,RhodococcusequistrainTB-60的EC水解酶不稳定指数为34.70,显示为较稳定蛋白,其余两种蛋白为不稳定蛋白。此外,脂肪系数的结果显示,3个蛋白均表现为亲水性。

表3 EC水解酶的氨基酸序列理化性质Table 3 Physicochemical properties of amino acid sequence of urethane hydrolase

表4 EC水解酶家族成员氨基酸组成成分分析Table 4 Analysis of amino acid composition of members of urethane hydrolase family %
2.2 EC水解酶氨基酸序列的亲水性/疏水性
经过ProtScale对EC水解酶氨基酸序列进行亲水性/疏水性预测。Hphob. /Kyte & Doolittle量表规定疏水性越强的氨基酸标度值越高,当蛋白质的氨基酸标度值大于0时为疏水,小于0时为亲水。
在亲疏性分析中,马红球菌RhodococcusequistrainTB-60的多肽链中465位Trp的值为-2.156,亲水性最强,第283位Cys的值为2.089,疏水性最强(见图2a);赖氨酸芽孢杆菌LysinibacillusfusiformisSC02的多肽链中第463位Asn的值是-3.344,是最低分,亲水性最强,第229位Leu值是1.978,为最高分,疏水性最强(见图2b);假丝酵母Candidaparapsilosis的多肽链中第324位Pro的值为-3.244,亲水性最强,第453位Val的值为2.156,疏水性最强(见图2c);且在亲疏水性分析中三种微生物的EC水解酶都属于亲水性蛋白。

2.3 EC水解酶的信号肽及跨膜区预测
利用SignalP 5.0 server对三株菌的EC水解酶的氨基酸序列进行信号肽分析,都表示EC水解酶的氨基酸序列不含有信号肽(见图3)。
利用在线工具TMHMM 2.0 Server对三株菌的EC水解酶的跨膜区进行分析及预测,3株菌的EC水解酶在膜内的概率为0,在膜外的概率为100%(见图4),说明EC水解酶不存在跨膜结构域。

2.4 EC水解酶的磷酸化位点预测
磷酸化作为蛋白质翻译后的修饰之一,在细胞的信号转导中起着重要的作用[18]。蛋白质的磷酸化主要集中在肽链中具有游离羟基的酪氨酸、丝氨酸、苏氨酸残基上,这些残基本身不带电荷,当磷酸化作用后,便具有了电荷,从而使结构发生变化,进一步引起蛋白质活性的变化。通过在线软件NetPhosK 3.1 Server对EC水解酶的氨基酸序列进行预测。EC水解酶氨基酸序列磷酸化位点(见表5)。RhodococcusequistrainTB-60的EC水解酶中含有15个丝氨酸位点、10个苏氨酸位点、6个酪氨酸位点;LysinibacillusfusiformisSC02的EC水解酶中含有23个丝氨酸位点、11个苏氨酸位点、7个酪氨酸位点;Candidaparapsilosis的EC水解酶中含有30个丝氨酸位点、16个苏氨酸位点、12个酪氨酸位点。

表5 EC水解酶氨基酸序列磷酸化位点统计Table 5 Statistics of phosphorylation sites of amino acid sequence of urethane hydrolase
2.5 二级结构分析
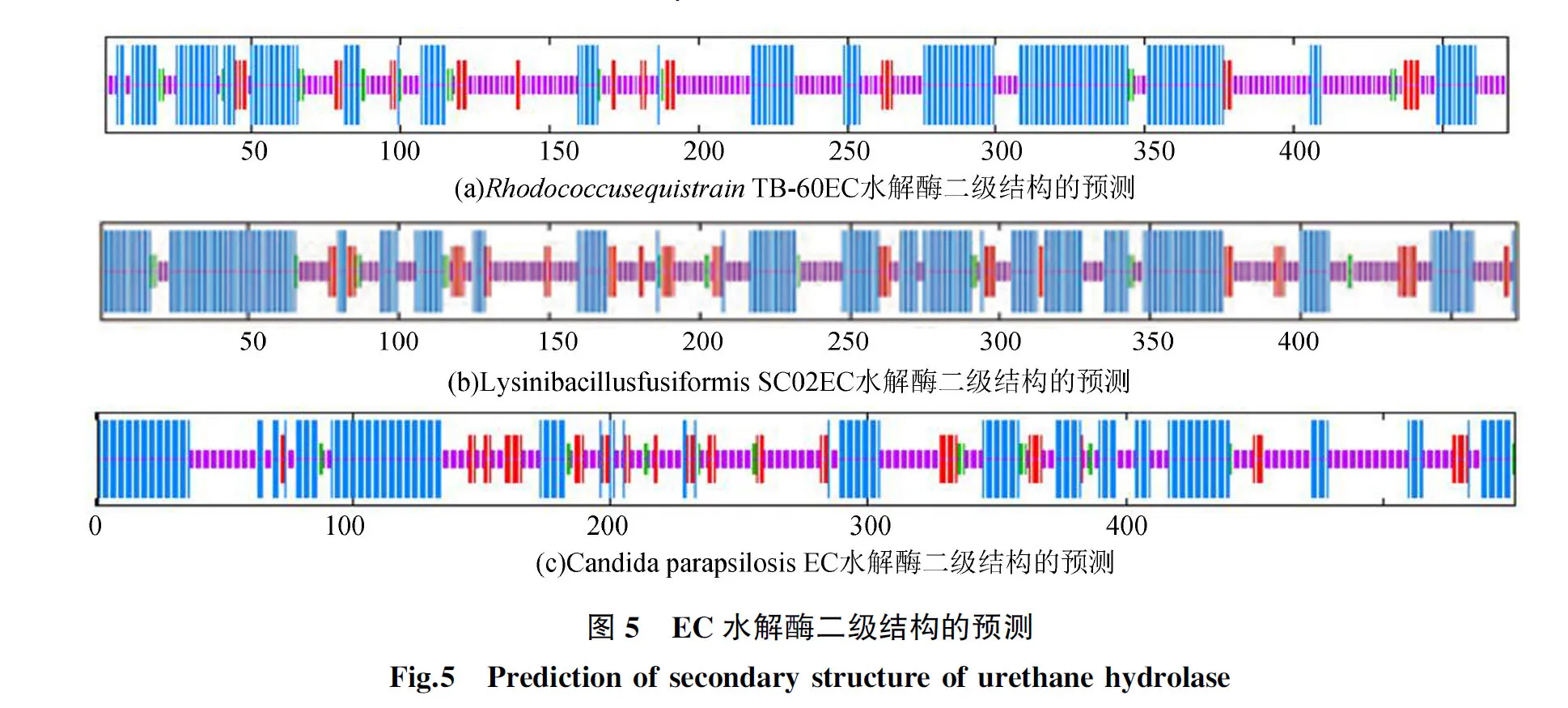
蛋白的二级结构一般由α-螺旋、β-链、β-转角和无规则卷曲等结构原件组成[19]。通过SOPMA在线工具对3株菌的EC水解酶的二级结构进行预测,结果(见图5)。RhodococcusequistrainTB-60的EC水解酶中α-螺旋占41.53%,延伸链占7.63%,β-转角占3.39%,无规则卷曲占47.46%;LysinibacillusfusiformisSC02的EC水解酶中α-螺旋占48.09%,延伸链占10.59%,β-转角占3.6%,无规则卷曲占37.71%;Candidaparapsilosis的EC水解酶中α-螺旋占39.34%,延伸链占11.07%,β-转角占3.45%,无规则卷曲占46.10%。
2.6 保守结构域分析
通过CDD和SMART对EC水解酶的保守结构域分析得出,来自于三种不同菌的EC水解酶都含有与酰胺酶家族相同的Amidase结构域(见图6)。

2.7 蛋白质三级结构的预测与评价
2.7.1 蛋白质三级结构
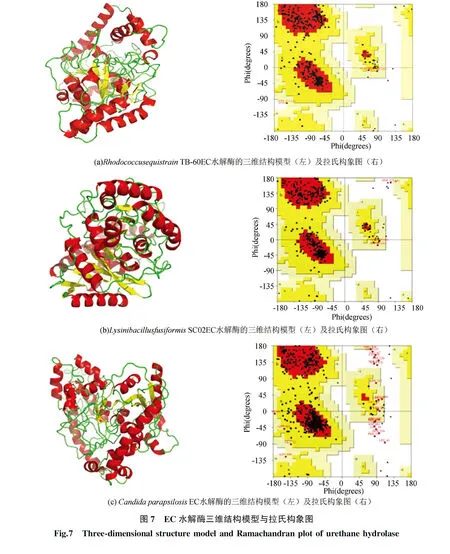
在NCBI上检索到的完整的EC水解酶的氨基酸序列有3条。利用NCBI上的blastp功能在蛋白质PDB数据库中分别获得与现存的EC水解酶序列相似度最高的氨基酸序列。RhodococcusequistrainTB-60的EC水解酶氨基酸序列与BacteriumCSBL00001的芳基酰基酰胺酶(PBD:4YJ1_A)同源性最高,为33.81%。LysinibacillusfusiformisSC02的EC水解酶氨基酸序列与PseudomonasaeruginosaPAO1的谷氨酰胺基转移酶A亚基(PDB:4WJ3_A)同源性最高,为39.47%。Candidaparapsilosis的EC水解酶氨基酸序列与Rattusnorvegicus的脂肪酸酰胺水解酶(PBD: 1MT5_A)同源性最高,为27.98%。由于使用SWISS-MODEL进行建模时,同源序列要大于30%,故对Candidaparapsilosis的EC水解酶进行建模时,使用I-TASSER。预测得到的三维结构中(见图7a、7b、7c),α-螺旋用红色表示,β-折叠用黄色表示,绿色为无规则卷曲,总体来看,EC水解酶的三维结构均以α-螺旋以及无规则卷曲为主,这预测结果与二级结构预测相对应。
2.7.2 蛋白质三维模型的评价
使用在线软件SAVES中的Verify_3D以及拉氏构象图对模型进行评价。RhodococcusequistrainTB-60的EC水解酶的Verify_3D评分为87.80%;LysinibacillusfusiformisSC02的EC水解酶的Verify_3D评分为89.44%;Candidaparapsilosis的EC水解酶的Verify_3D评分为84.39%。
拉氏构象图简称拉氏图(Ramachandran Plot),其作用是体现出氨基酸残基在拉氏图中的区域分布。拉氏构象图分为四个区,红色区域是最合适区域,该区域内氨基酸数目越多,则表示该蛋白模型的骨架结构越合理;黄色区域是允许区域;浅黄色区域是最大允许区;而白色区域是不允许区,该区域氨基酸的构象是不合理的。从图7a、7b、7c可知使用SWISS-MODEL以及I-TASSER所建的EC水解酶模型,处于允许区的氨基酸残基都大于90%,并且不允许区都小于5%。
2.8 蛋白质多序列比对分析
利用在线软件Clustal Omega中的多序列比对功能对3个EC水解酶与BacteriumCSBL00001的芳基酰基酰胺酶的氨基酸序列进行比对(见图8)。三个EC水解酶的序列相似度为29.17%。来自于真菌Candidaparapsilosis的EC水解酶氨基酸序列与两个来源于细菌的相似度较低,与RhodococcusequistrainTB-60序列相似度为14.49%,与LysinibacillusfusiformisSC02序列相似度为15.22%,而来源于细菌的两菌株氨基酸序列相似度为31.3%。从图中可见位于160位左右的氨基酸残基保守性较强,特别是GGSSGG,这些氨基酸残基在酰胺酶进化过程中具有较强的保守性。

图8 EC水解酶的多序列比对结果Fig.8 Results of multiple sequence alignment of urethane hydrolase
3 讨 论
EC作为2A类致癌物质,广泛存在于酒精饮料等发酵食品中。应用微生物酶法去消除EC具有直接、高效的特点。EC水解酶有三个完整的氨基酸序列得以鉴定,且所发现的EC水解酶在酸性或者乙醇存在条件下不稳定,不能在酒精饮料中得到广泛的应用。如果能利用分子生物学的手段对EC水解酶进行改造,使其能被应用于食品生产过程中,降低食品中EC的浓度,具有重要意义。
生物信息学作为一门交叉学科,利用数学、计算机科学、生命科学技术理论和工具,在生物科学领域的信息获取、加工、储存、分析等方面发挥着重要的作用。本研究利用生物信息学相关方法,在NCBI中查找到三条完整编码EC水解酶的基因序列,对其编码的氨基酸序列组成、基本理化性质进行分析,通过软件预测蛋白的亲疏水性、信号肽、跨膜域,更进一步预测了蛋白的二级以及三维结构。结果表明,三个蛋白序列都具有与酰胺酶家族相同的Amidase保守结构域,证明了目前已公布的EC水解酶都是酰胺酶,与刘庆涛所说的一致[6]。使用计算机进行分析蛋白质的稳定性时,蛋白质的不稳定系数是一个重要的表征。不稳定指数是对蛋白质在实验中的稳定性评估,当蛋白质的不稳定系数大于40时,推测该蛋白质为不稳定蛋白,当小于40时为稳定蛋白[20]。氨基酸序列理化性质分析表明,除RhodococcusequistrainTB-60的EC水解酶为较稳定蛋白,其余两种蛋白均表现为不稳定蛋白。在蛋白质中,氨基酸之间的亲/疏水性相互作用是形成其三级结构最重要作用力之一,亲疏水作用可以驱使蛋白质进行折叠,有利于蛋白质三级结构的稳定。亲疏水性预测结果显示,三个EC水解酶蛋白均属于亲水性蛋白。对蛋白的信号肽以及跨膜域分析表明,三株菌的EC水解酶都不含有跨膜域以及信号肽。蛋白质二级结构预测发现,EC水解酶都以α-螺旋、无规则卷曲为主。蛋白质空间结构预测,对其结构与功能的研究具有较为重要意义[21]。本研究通过同源建模的方式得到了EC水解酶的三维结构,除对Candidaparapsilosis的EC水解酶进行建模时,使用I-TASSER,其余两种均使用SWISS-MODEL进行建模。三维结构模型的合理性通过Verify_3D以及Ramachandran Plot进行验证。Verify_3D用于评估模型的三维结构与氨基酸一级序列结构的相容性,检测氨基酸侧链构象的合理性,SAVES获得的评分≥80%,表明侧链构象合理[22]。三个蛋白的三维结构的Verify_3D评分都大于80%,说明侧链构象合理。在Ramachandran Plot中,模型评价要求处于允许区的氨基酸大于90%,如果二面角中有高于90%的都位于一般允许区,则可表明其空间结构具有一定的稳定性[23-24]。三个蛋白质三维结构模型中处于允许区的氨基酸均大于90%,表明模型中所有氨基酸均形成了一个合理的二面角稳定构型。
4 结 论
1) EC水解酶基因编码472~551 aa,分子量在50~62 kD之间,理论等电点(pI)在5左右,均为酸性亲水性蛋白;
2) 目前EC水解酶无跨膜区和信号肽;
3) 二级结构预测结果显示EC水解酶的氨基酸都以α螺旋以及无规则卷曲为主,螺旋与折叠排列有序;
4) 目前发现的EC水解酶均属于酰胺酶家族;
5) 对EC水解酶进行三维模型的构建,预测结果质量评估均较好,并使用同源比对的方法,分析出EC水解酶高保守氨基酸残基。利用生物信息学相关软件对EC水解酶蛋白进行预测和分析,为挖掘新的EC水解酶、进一步研究EC与EC水解酶结合位点以及对EC水解酶的改造提供了理论依据。
