海兰鸡内源性白血病病毒位点序列鉴定与分析
2015-07-21陈孜孟崔治中
陈孜孟,董 宣,苏 帅,崔 宁,李 卓,崔治中*
(1.山东农业大学动物医学院,泰安 271018;2.国家海洋局第三海洋研究所,厦门 361005)
禽白血病病毒(avian leucosis virus,ALV)是反转录病毒科α-反转录病毒属的成员之一。根据病毒在不同种禽类的宿主范围,病毒间的干扰作用及病毒中和反应相关的囊膜蛋白抗原特性,禽白血病病毒分为A~J 10个亚群,A、B、C、D、E和J等6个亚群可感染鸡[1]。近年来王鑫等[2]也分离到了新的K亚群ALV。其中E亚群为内源性ALV,通常致病性很低或无致病性,其余均为对鸡有致病性的外源性ALV。内源性ALV是指整合进宿主细胞染色体基因组的可通过染色体垂直传播的ALV前病毒DNA及其可能产生的ALV病毒粒子。它可能只是病毒基因组的不完整片段,不会产生传染性病毒;但也可能是全基因组因而能产生有传染性的病毒粒子,不过这类病毒通常致病性很弱或没有致病性[3]。内源性的ALV-E虽然没有致病性,但它可干扰对外源性ALV感染的检测,给诊断和净化ALV 带来很大的困难[4-5]。
在内源性ALV这个大概念中,既包括在三十多年前就在鸡基因组上发现和报道的内源性白血病病毒(endogenous viruses,ev)位点[6],还有近二十多年中发现的中度重复性序列EAV(endogenous avian virus)[7]、ART-CH (avian retrotransposon from chicken genome)[8]及高度 重复 性序 列 CR1(chicken repeat 1)[9]。鸡的内源性反转录病毒只是在真核生物中存在的许多反转录序列的突出例子。
鸡的ev位点的遗传序列与E亚群ALV相关,几乎所有正常鸡都带有完整的或缺陷性的ALV-E基因组[10]。这些ev位点分布在鸡的体细胞和生殖细胞的不同的染色体上,并以孟德尔遗传法则遗传给它们的不同性别的后代[11-12]。迄今为止,已至少识别和鉴定出50个不同的ev位点,其中在白来航鸡中确认的有22个[13]。这些位点分别是在20世纪70、80年代通过RELP和分子杂交的方法从不同品种鸡鉴定和识别出来[6,14]。其中只有ev21这个位点是从海兰鸡发现的[12]。有些位点的染色体定位已经确定,有的尚待鉴定[15-18]。随着鸡基因组序列测定的完成[19],其他ev位点的定位也会逐渐完成。但是,迄今为止,大多数ev位点只是完成了染色体定位,尚未完成克隆和测序。要强调说明的是,这些特定位点是稳定地整合进鸡细胞基因组,因此能够按孟德尔法则遗传。
目前国内外对ALV鉴定报道,都是扩增其中的env片段或分段扩增三个基因片段后进行拼接得到的 ALV 序列[20-24]。在2011年X.Wang(王兴娜)等[25]从深海分离到一株超嗜热古菌,并重组表达了其Pol B DNA聚合酶并具有扩增长片段的能力(相关文章尚未发表)。本研究利用超嗜热古菌Thermococcus sp.4557来源重组表达的Pol B DNA聚合酶,通过一次PCR反应从海兰鸡中染色体基因组DNA扩增到4663bp的包含gag、pol、env部分基因片段和3′-LTR的内源性ALV-E位点,本文对其做了全面的比较分析。
1 材料与方法
1.1 实验材料
本实验室人工造病,感染ALV-J的海兰鸡颈部肿瘤组织和正常海兰鸡颈部组织提取的基因组DNA;DNA聚合酶由国家海洋局第三海洋研究所重组表达的从超嗜热古菌Thermococcus sp.4557来源的重组表达的Pol B DNA聚合酶;Top10感受态细胞由本实验室保存;质粒小提试剂盒购于北京索莱宝科技有限公司;通用型DNA纯化回收试剂盒购于天根生化科技(北京)有限公司;rTaq DNA聚合酶和pMD18-T购于TaKaRa公司。
1.2 长距离PCR
参照GenBank上发表的ALV毒株(NX0101[23],登录号为DQ115805;ev-1[26],登录号为 AY013303;JS11C1[24],登录号为 KF746200;SDAU09C2,登录号为 HM446005)序列,设计5′LTR右侧外的5′UTR区段设计上游引物,在3′LTR右端区段设计下游引物。设计简并引物如下,Primer F:5′-TTTGGTGACCCCGACGTGATAGTTAGGGAATAGTGGTCG-3′,Primer R:5′-TGAAGCCTTCCGCTTCATGCAGGTGTTCG-3′。使用从超嗜热古菌Thermococcus sp.4557来源的重组表达的Pol B DNA聚合酶,配制PCR扩增反应体系50μL:Tris(pH8.8)20mmol·L-1,MgSO42mmol·L-1,KCl 10mmol·L-1,(NH4)2SO410mmol·L-1,TritonX-1000.5‰,Trehalose 100mmol·L-1,dNTP 0.2mmol·L-1,BSA 20mg·L-1,F 50μmol·L-1,R 50μmol·L-1,Template DNA 100ng,Pol B DNA polymerase 1μL。反应条件:预变性95℃5 min;变性95℃30s,梯度退火72~52℃30s,延伸72℃5min,35个循环;后延伸72℃10min,4℃终止反应。感染ALV-J的海兰鸡颈部肿瘤组织提取的基因组DNA为试验组,正常海兰鸡颈部组织提取的基因组DNA为对照组。
1.3 重组载体的构建
将PCR产物电泳切胶回收,使用rTaq DNA聚合酶按照产品说明配制反应体系,将模板DNA改为切胶纯化的PCR产物,72℃延伸30min,使用天根通用型DNA纯化回收试剂盒对反应产物进行纯化,产物克隆至pMD18-T,转化Top10感受态细胞,挑取单菌落,摇菌提取质粒,经SalⅠ单酶切后进行琼脂糖凝胶电泳得到约7000bp大小条带的质粒,送上海美吉生物进行核苷酸序列测定。
1.4 基因组序列的比较分析
使用Genetyx和Chromas2软件测序结果进行剪辑和拼接,使用MegAline软件对测序拼接结果与国内外已发表的不同亚群ALV基因组序列进行比较分析。
2 结 果
2.1 ALV基因组的扩增
提取的组织基因组经过NanoDrop2000超微量分光光度计检测后,将基因组样品稀释到100 ng·μL-1作为模板使用。感染ALV-J海兰鸡颈部肿瘤组织基因组扩增到约4.3kb大小的DNA片段(图1A)。为了确定它与外源性白血病病毒cDNA的关系,又使用正常海兰鸡颈部组织提取基因组做模板进行扩增,也扩增到同样约4.3kb大小的DNA片段(图1B)。
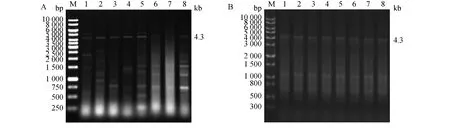
图1 ALV-E基因组扩增Fig.1 PCR amplification of ALV-E
2.2 重组载体的构建
将不同模板扩增的4.3kb大小的DNA片段进行切胶回收,使用NanoDrop2000超微量分光光度计检测后,将扩增的DNA末端使用rTaq进行3′端加A后,进行纯化,连接pMD18-T,转化Top10感受态细胞,挑取单克隆,摇菌提取质粒后,使用SalⅠ单酶切,选择得到约7000bp大小DNA条带的克隆,送上海美吉生物测序,确定含有4300bp的内源性ALV部分基因组序列,定名为UL-E1。
2.3 对测序序列的分析
使用Genetyx和Chromas2对测序结果进行拼接和校正,得到完整的序列。对以ALV感染海兰鸡颈部肿瘤组织基因组和无外源性ALV感染的正常海兰鸡颈部组织基因组为模板分别扩增的序列做了比较分析,二者大小都为4663bp,相似性为100%,表明该片段来自正常鸡细胞基因组的内源性ALV,而与感染的外源性ALV无关。
将测序拼接得到的基因组序列与NCBI网站上的同源序列进行BLAST,发现与许多白血病病毒的基因组序列相似性最高达99%,在该内源性ALV片段中,LTR全长274bp,3个主要结构基因gag、pol和env片段的大小分别为2013、728和1046bp,与完整的ALV基因组的相应基因相比都有严重缺失(图2)。其中与在原鸡4号染色体上的ev位点内源性 ALVE-B10[27](GenBank 登录号:KC610516)同源性最高,但是相比ALVE-B10基因组序列缺失严重,分别在gag、pol、env三个主要功能基因中位置缺失了93、1960和805个碱基。
2.4 与原鸡的基因组其他序列的比较
在GenBank上找到原鸡的基因组序列,HL-E1序列直接与原鸡基因组序列进行比对,在1号染色体(GenBank登录号:NC006088)第32561736—32568956(测序gap)碱基位置之间找到了同源序列,相似性99.2%。把这段序列拷贝下来,使用Genetyx进行比对发现,相比原鸡1号染色体序列在32563954—32563686、32564811—32566771和32567340—32568147碱基位置分别缺失了93、1960和808个碱基。
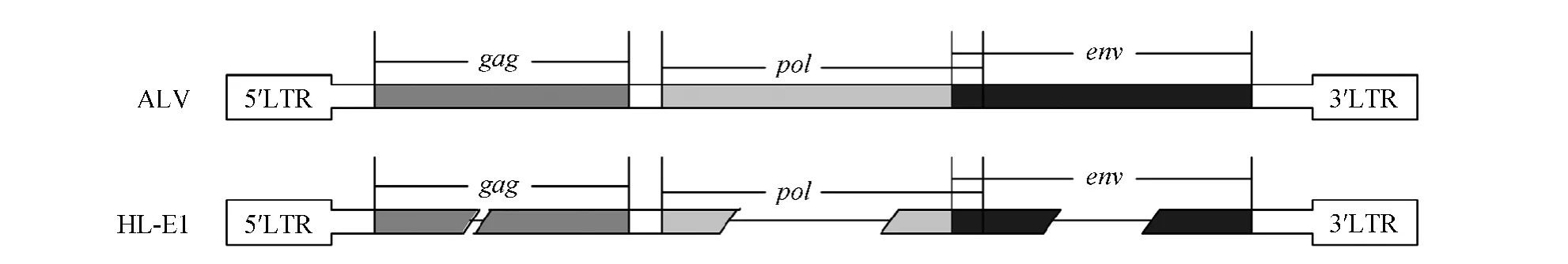
图2 HL-E1结构示意Fig.2 Schematic illustration of HL-E1
2.5 与其他亚群白血病病毒全基因组序列比较分析
HL-E1全基因组核苷酸序列与 ALV-A(SDAU09C1,登录号为HM452339)、ALV-B(SDAU09C2,登录号为 HM446005)、ALV-E(ev-1[26],登录号为 AY013303)、ALV-J(NX0101[23],登录号为 DQ115805)和 ALV-K[2](JS11C1[24],登录号为KF746200)的代表毒株进行序列比较,结果显示:该内源性ALV片段与几个不同亚群的代表株的完整病毒粒子中的基因组相比,其gag、pol、env都发生了严重缺失(表1),其env基因片段与E亚群ev-1的env基因相似性最高,达96.8%,HL-E1的LTR与ev-1和TW-3593的LTR长度相同,比较之下HL-E1的基因组大小明显小于其他ALV亚群(表1)。此外,通过对比HL-E1存在的三个缺失基因序列对应其他亚群的同源序列,统计了HL-E1与其他亚群三个主要功能基因和LTR的相似性(表2)。
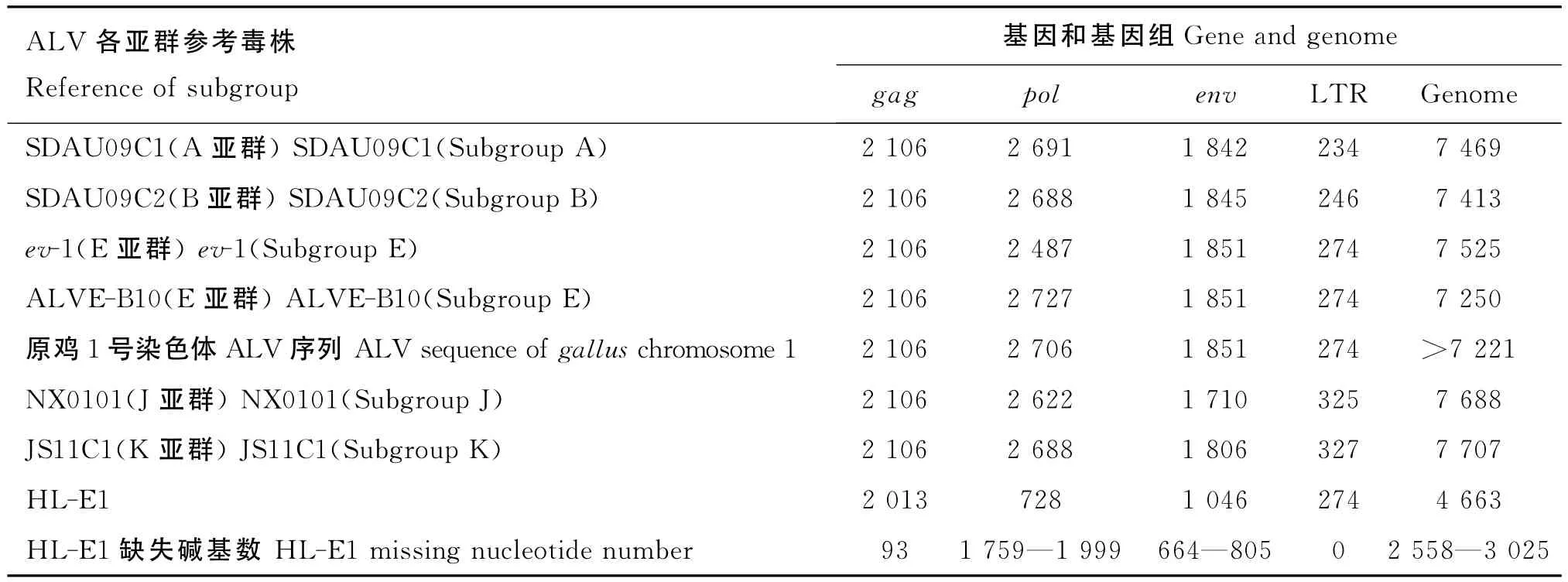
表1 HL-E1与各亚群外源性ALV毒株基因组和鸡基因组内源性ALV成分的大小比较Table 1 Comparison of HL-E1strains with other subgroups ALV genes and genome size bp

表2 HL-E1与各亚群外源性ALV毒株基因组和鸡基因组内源性ALV成分序列相似性比较Table 2 Comparison of HL-E1strains with other subgroups ALV three genes and LTR homologous %
3 讨 论
利用自行研制的超嗜热古菌Thermococcus sp.4557来源重组表达的Pol B DNA聚合酶能扩增长片段DNA的特点(待发表),本研究最初试图通过一次PCR从鸡的急性肉瘤病料基因组DNA中扩增缺陷型急性致肿瘤ALV的全基因组,并得到了一个4663bp的片段。但在对照试验中,用相同的酶和引物,从未感染ALV的健康海兰鸡的细胞基因组DNA也扩增到序列完全相同的片段HL-E1,这表明该片段实际上只是正常鸡基因组的一个组成部分。序列分析表明,对该片段全长4663bp,分别包含gag、pol、env三个基因的部分序列及其3′-LTR(表1)。在HL-E1序列中LTR全长274bp;三个主要ORF编码的基因gag、pol、env的长度分别为2013、728和1046bp,与不同亚群的完整的ALV基因组上这三个基因相比都发生了很大的缺失。该片段env基因和LTR都与鸡基因组上经典的ev1位点的相应基因有96.8%和97.4%的相似性,而与其他亚群的外源性ALV的相似性较低(表2)。这表明这是整合进海兰鸡染色体基因组中的一个内源性E亚群ALV片段构成的ev位点。虽然到目前为止已分别从不同品系的正常鸡基因组识别和鉴定出50个不同的ev位点[13],但这些位点分别是在20世纪70、80年代通过RELP和分子杂交的方法从不同品种鸡鉴定和识别出来[14,15]。这些ev位点既有E亚群ALV完整的序列,如ALVE-B10等[18],还有很多都是缺陷型的不完整基因组,其中只有少数几个有序列报道,如ev1。到目前为止,从海兰鸡基因组还只有发现和报道了ev21这个位点[12],这是与鸡的快慢羽性状相关基因紧密连锁的一个ev位点,它包括了完整的E亚群ALV全基因组。本研究克隆鉴定的HL-E1这个基因位点是不完整片段,显然这是不同于ev21的海兰鸡基因组上的又一个ev位点。
将HL-E1的序列与原鸡的基因组草图序列比对时,只在原鸡的1号染色体的第32561736—32568956(此端为测序gap)碱基之间找到了同源序列,相似性99.2%,HL-E与原鸡的此段同源序列相比也在1859—1951、3076—5036和5605—6412位置缺失了93、1960和808个碱基,其他位点没有找到同源序列。但该序列与从原鸡4号染色体扩增到的ALVE-B10(内源性E亚群位点)的同源性更高(表1和表2),根据我们现有的研究结果还很难推断本研究扩增得到的HL-E1的位点是在海兰鸡的哪一条染色体上。这一内源性ALV-E位点是否会影响相应品系鸡的某种遗传性状也有待进一步研究。
4 结 论
使用超嗜热古菌Thermococcus sp.4557来源重组表达的Pol B DNA聚合酶通过一次PCR反应成功扩增到了位于海兰鸡染色体基因组的一个新的ev位点HL-E1。其LTR全长274bp,3个主要结构基因gag、pol和env片段的大小分别为2013、728和1046bp,与完整的ALV基因组的相应基因相比都有严重缺失。
(References):
[1]COFFIN J M.Structure and Classification of Retroviruses[M].New York:Plenum Press,1992,1:19-49.
[2]王 鑫,赵 鹏,崔治中.我国地方品种鸡分离到的一个禽白血病病毒新亚群的鉴定[J].病毒学报,2012,28(6):609-614.WANG X,ZHAO P,CUI Z Z.Identification of a new subgroup of avian leukosis virus isolated from Chinese indigenous chicken breeds[J].Chinese Journal of Virology,2012,28(6):609-614.(in Chinese)
[3]SAIF Y M,BARNES H J,GLISSON J R,et al.Disease of poultry[M].Iowa State University Press,2003:64-78.
[4]SPENCER J L,GILKA F,GAVORA J S,et al.Distribution of lymphoid leukosis virus and p27group-specific antigen in tissues from laying hens[J].Avian Dis,1984,28(2):358-373.
[5]CRITTENDEN L B,SMITH E J.A comparision of test materials for differentiating avian leukosis virus group-specific antigens of exogenous and endogenous origin[J].Avian Dis,1984,28(4):1057-1070.
[6]ASTRIN S M.Endogenous viral genes of the White Leghorn chicken:common site of residence and sites associated with specific phenotypes of viral gene expression[J].Proc Natl Acad Sci U S A,1978,75(12):5941-5945.
[7]DUNWIDDIE C T,RESNICK R,BOYCE-JACINO M,et al.Molecular cloning and characterization of gag-,pol-,and env-related gene sequences in the evchicken[J].J Virol,1986,59(3):669-675.
[8]SACCO M A,FLANNERY D M,HOWES K,et al.Avian endogenous retrovirus EAV-HP shares regions of identity with avian leukosis virus subgroup J and the avian retrotransposon ART-CH[J].J Virol,2000,74(3):1296-1306.
[9]STUMPH W E,HODGSON C P,TSAI M J,et al.Genomic structure and possible retroviral origin of the chicken CR1repetitive DNA sequence family[J].Proc Natl Acad Sci U S A,1984,81(21):6667-6671.
[10]HUDA A,POLAVARAPU N,JORDAN I K,et al.Endogenous retroviruses of the chicken genome[J].Biol Direct,2008,3:9.
[11]BACON L D,FADLY A M,CRITTENDEN L B.Absence of influence on immune competence by the sexlinked gene(K)determining slow feathering in White Leghorn chickens[J].Avian Dis,1986,30(4):751-760.
[12]BACON L D,SMITH E,CRITTENDEN L B,et al.Association of the slow feathering(K)and an endogenous viral(ev21)gene on the Z chromosome of chickens[J].Poult Sci,1988,67(2):191-197.
[13]CRITTENDEN L B.Retroviral elements in the genome of the chicken:implications for poultry genetics and breeding[J].Crit Rev Poult Biol,1991,3(2):73-109.
[14]SMITH E J,CRITTENDEN L B.Endogenous viral genes in a slow-feathering line of white leghorn chickens[J].Avian Pathol,1986,15(3):395-406.
[15]BENKEL B F.Locus-specific diagnostic tests for endogenous avian leukosis-type viral loci in chickens[J].Poult Sci,1998,77(7):1027-1035.
[16]SMITH A,BENKEL B F.Novel avian leukosis virusrelated endogenous proviruses from layer chickens:Characterization and development of locus-specific assays[J].Poult Sci,2009,88(8):1580-1585.
[17]YANG J,YU Y,YAO J,et al.Molecular identification of avian leukosis virus subgroup E loci and tumor virus B locus in Chinese indigenous chickens[J].Poult Sci,2011,90(4):759-765.
[18]RUTHERFORD K,BENKEL B F.Characterization of insertion sites and development of locus-specific assays for three broiler-derived subgroup E avian leukosis virus proviruses[J].Avian Pathol,2013,42(4):373-378.
[19]International Chicken Genome Sequenicing Consortium.Sequence and comparative analysis of the chicken genome provide unique perspectives on vertebrate evolution[J].Nature,2004,432(7018):695-716.
[20]徐海鹏,孟凡峰,董 宣,等.种蛋中内源性禽白血病病毒的检测和鉴定[J].畜牧兽医学报,2014,45(8):1317-1323.XU H P,MENG F F,DONG X,et al.Detection and identification of an endogenous subgroup E avian lekosis virus in a chicken breeder embroy[J].Acta Veterinaria et Zootechnica Sinica,2014,45(8):1317-1323.(in Chinese)
[21]刘超男,高玉龙,高宏雷,等.J亚群与E亚群禽白血病自然重组病毒的全基因组序列分析[J].中国预防兽医学报,2009,31(12):978-981.LIU C N,GAO Y L,GAO H L,et al.Molecular characterization of a natural recombinant subgroup J avian leucosis virus with subgroup E partial env gene[J].Chinese Journal of Preventive Veterinary Medicine,2009,31(12):978-981.(in Chinese)
[22]孔义波,张兴晓,姜世金,等.SPF鸡胚中内源性白血病病毒全基因序列鉴定与分析[J].病毒学报,2008,24(1):53-58.KONG Y B,ZHANG X X,JIANG S J,et al.Sequence analysis for the complete provial genome of endogenous avian leukosis virus strian SD0501[J].Chinese Journal of Virology,2008,24(1):53-58.(in Chinese)
[23]CUI Z,SUN S,WANG J.Reduced serologic response to Newcastle disease virus in broiler chickens exposed to a Chinese field strain of subgroup J avian leukosis virus[J].Avian Dis,2006,50(2):191-195.
[24]CUI N,SU S,CHEN Z,et al.Genomic sequence analysis and biological characteristics of a rescued clone of avian leukosis virus strain JS11C1,isolated from indigenous chickens[J].J Gen Virol,2014,95(Pt 11):2512-2522.
[25]WANG X,GAO Z,XU X,et al.Complete genome sequence of Thermococcus sp.strain 4557,a hyperthermophilic archaeon isolated from a deep-sea hydrothermal vent area[J].J Bacteriol,2011,193(19):5544-5545.
[26]JOHNSON J A,HENEINE W.Characterization of endogenous avian leukosis viruses in chicken embryonic fibroblast substrates used in production of measles and mumps vaccines[J].J Virol,2001,75(8):3605-3612.
