脊椎动物NF-κB1/2基因亚家族分子进化和功能分歧分析
2015-07-21王继文刘贺贺孙文强王郁石黄惠兰
甘 翔,王继文,刘贺贺,李 亮,孙文强,王郁石,张 涛,陈 达,黄惠兰
(四川农业大学,畜禽遗传资源发掘与创新利用四川省重点实验室,成都 611130)
NF-κB1与 NF-κB2共同隶属于 NF-κB基因家族,分别编码p50与p52的前体蛋白p105和p100。p105与p100是核转录因子,在二聚体化后能与免疫球蛋白轻链(Immunoglobulin light chain,MYLC)基因增强子cDNA序列(5′-GGGACTTTCC-3′)特异性结合并调控其转录,从而启动和调节众多免疫和炎症反应[1-2]。NF-κB1/2拥有Rel homology domain(RHD)、IPT domain(IPT)、ankyrin repeats(ANK)和 Death domain(DD)4个结构域[3]。RHD由大约200个氨基酸残基组成,拥有核定位信号序列,是与DNA结合的关键区域,也是与抑制剂作用的重要区域;ANK可作为NF-κB家族的抑制剂,通过与RHD结合来使其滞留在细胞质并保持静息态,ANK蛋白的磷酸化、泛素化、降解释放RHD的过程也就是NF-κB蛋白的激活过程[4];而IPT和DD则与NF-κB蛋白二聚体的稳定性与活性有着密切的联系[5]。
研究表明,NF-κB基因的原始结构起源于后生动物以前,在进化过程中分化为 NF-κB1和 NF-κB2[6]。利用模式脊椎动物的 NF-κB1/2氨基酸序列构建进化树,发现它们分成两支,且完全没有交叉的迹象,说明两者在进化上出现了明显的分歧,暗示它们在结构上存在一定差异。另外,在脊椎动物中,NF-κB1/2蛋白的功能也表现出了一定的差异。有文献报道[7-8],不同的 NF-κB1/2蛋白二聚体对于相应的κB位点会显示出不同的亲和力与激活能力。不仅如此,两者的ANK抑制结构对于不同的二聚体也可能显示出不同的抑制强度。还有报告[7-9]称,许多刺激信号如 TNF-α、IL-1、LPS等对 NF-κB1和NF-κB2氨基酸序列的刺激能力与刺激途径也有所差异。在小鼠中,敲除NF-κB1会导致B细胞的多种功能缺陷,而敲除NF-κB2则会引起淋巴结构改变,影响上皮发育。另外,两者的敲除反应都会一定程度上导致胃黏膜增生,但NF-κB2对胃黏膜的影响程度要大于NF-κB1[4]。当前的研究已经基本定义了NF-κB1/2基因的起源,也明确了 NF-κB家族在脊椎动物中的功能差异,但对造成这些差异的原因还没有明确的定义。不过可以推断,NF-κB1/2的 RHD、IPT、ANK 与 DD 在 NF-κB1与 NF-κB2基因亚家族分子进化特征的形成和分子功能分歧的产生方面扮演了重要的角色。
本研究选择模式脊椎动物的NF-κB1/2基因与蛋白质序列,针对NF-κB1/2的RHD、IPT、ANK和DD 4个结构域进行生物信息学分析,旨在揭示引起NF-κB1/2功能分化的部分原因,并阐述 NF-κB1/2在脊椎动物中进化、变迁的基本趋势。
1 材料与方法
1.1 序列数据的收集与下载
序列比对与核苷酸替代率分析所用的NF-κB1/2氨基酸、核苷酸序列均下载自NCBI数据库(http://www.ncbi.nlm.nih.gov/)。序列比对使用了包括人类(Homo sapiens)、大鼠(Rattus norvegicus)、小鼠(Mus musculus)、原鸡(gallus gallus)、非洲爪蟾(Xenopus(Silurana)tropicalis)、美国短吻鳄 (Alligator mississippiensis)、二 色热带鱼(Stegastes partitus)7种具有代表性的模式动物的NF-κB1/2的氨基酸序列。而核苷酸替代率分析则选用了鱼类、两栖类、爬行类、鸟类和海陆哺乳类共计20余种脊椎动物的 NF-κB1/2核苷酸序列[10]。
1.2 序列比对与系统发育树的构建
使用ClustalW将下载序列对齐,使用DAMBE等软件进行格式处理,得到的结果输入MEGA 6.0软件,使用邻接法(Neighbor-Joining,NJ),经过自展法(Bootstrap)1000次重复验证,以保证树拓扑结构的可靠性,分别构建NF-κB1/2氨基酸全序列及其各个结构域的氨基酸序列系统发育树[11-12]。
1.3 NF-κB1/2蛋白的结构域预测
使用Pfam 在线数据库(http://pfam.sanger.ac.uk/)对 NF-κB1/2蛋白进行结构预测。
1.4 进化速率计算检验与分析
使用DAMBE等软件处理序列文本格式,使用MEGA6.0,ClustalW 进行向导树的构建[13]。将处理结果文件输入paml软件包,codeml工具[14-15],分别选取模型A:所有的分支有一个ω,模型B:所有的分支有相同的ω且等于1,模型H:每一支都有自己的ω,选择最优模型从而得出各个结构域的整体ω值。每个位点ω的计算也使用codeml工具,NS-site参数为0、1、2、7、8,在生成结果中优先选用BEB模型,从而得到 NF-κB1/2的 RHD、ANK 和DD 中各个位点的 ω值[16-17]。
2 结 果
2.1 基于 NF-κB1/2与结构域 RHD、IPT、ANK和DD氨基酸序列的系统发育树
如图1a所示,进化树中 NF-κB1/2分为两大支,进化趋势与物种进化趋势一致。由鱼类至两栖类之后,系统发育树再分为两支,其中一支为爬行类与鸟类,另一支为哺乳类。树中NF-κB1/2两个成员在整个脊椎动物进化历程中并没有出现任何的交织,这表明NF-κB基因家族早在脊椎动物与非脊椎动物分歧时,就已经分化成为 NF-κB1和 NF-κB2,然后开始独立进化。
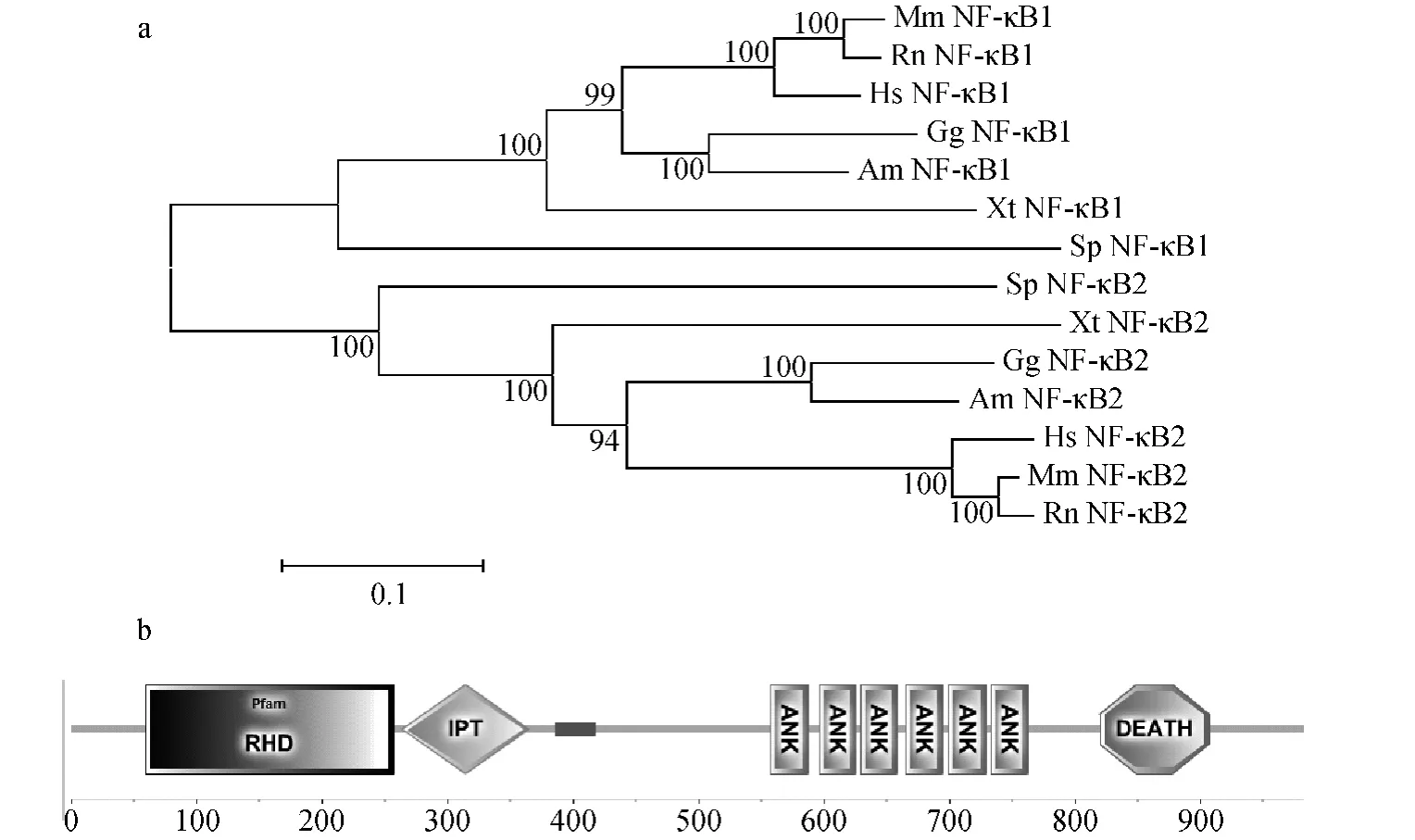
图1 NF-κB1/2氨基酸序列的系统发育树与保守结构域Fig.1 Phylogenetic tree of NF-κB1/2and their conserved domains
脊椎动物 NF-κB1/2两个成员都包含 RHD、ANK、IPT和DD 4个结构域(图1b),由它们的氨基酸序列所构建的系统发育树(图2)与基于全序列构建的发育树基本一致。
2.2 基于 NF-κB1/2 结构域(RND、IPT、ANK 和DD)的序列分析
图3中,NF-κB1/2之间在RHD上存在较多保守区域,但在序列的60~66、108~117、157~164位氨基酸区段(图中方框标出)也发现了它们各自的特征序列。在IPT区域上,两个基因在相应物种间都表现出较高的同源性。而在ANK区域上,无论在直系同源还是旁系同源结构中,该区域都出现了较大差异。另外,相对于其他3个结构域,DD区域的旁系同源结构之间存在很大的差异,而直系同源结构则表现出较高的同源性。
2.3 NF-κB1/2各保守结构域选择压力比较
Codeml中各模型比对结果显示,NF-κB1的ANK、RHD和 DD,NF-κB2的 ANK 与 RHD都更适用于H模型,检测结果为极显著;而NF-κB1/2的IPT,NF-κB2的DD更适用于A模型,检测结果也极显著。
分别比较了 NF-κB1/2的 RHD、ANK、IPT与DD区域的选择压力,结果(表1)表明,在鱼类及两栖动物上,NF-κB1/2的 RHD,NF-κB1的 ANK 显示出较高的ω值;在爬行动物与鸟类上,NF-κB1/2的ANK与NF-κB1的DDω值较大;而在哺乳动物中,大多数结构域的ω值均低于0.1。
而对NF-κB1/2氨基酸各位点选择压力(图4)分析表明,RHD、IPT、ANK与DD序列的ω值均小于1.2,且在所分析脊椎动物中,4个结构域均没有出现处于显著水平的正选择位点。NF-κB1/2的RHD区域大多数位点ω值在0.1以下。相对于RHD、IPT与DD区域,ANK区域则拥有较高的进化速率;NF-κB1的IPT绝大多数位点ω值较低,而NF-κB2的IPT所有位点的ω值均在0.01以下;NF-κB1/2的DDω曲线存在较大差异,NF-κB1的DD各个位点的ω值显示出较大的波动。
3 讨 论
NF-κB1/2属于 NF-κB基因家族中的亚家族,作为转录因子,能够诱导机体多种正向调控基因的转录,在动物机体免疫反应、骨骼肌发育中发挥重要作用[1]。研究表明,NF-κB基因的原始结构起源于后生动物以前,在进化过程中分化为NF-κB 1和NF-κB2[6]。通过构建NF-κB1/2 的进化树,发现NF-κB基因家族早在脊椎动物与非脊椎动物发生分歧前,就已经分化成为 NF-κB1和 NF-κB2,然后开始独立进化,这与其他报道[18]得出的结论一致。
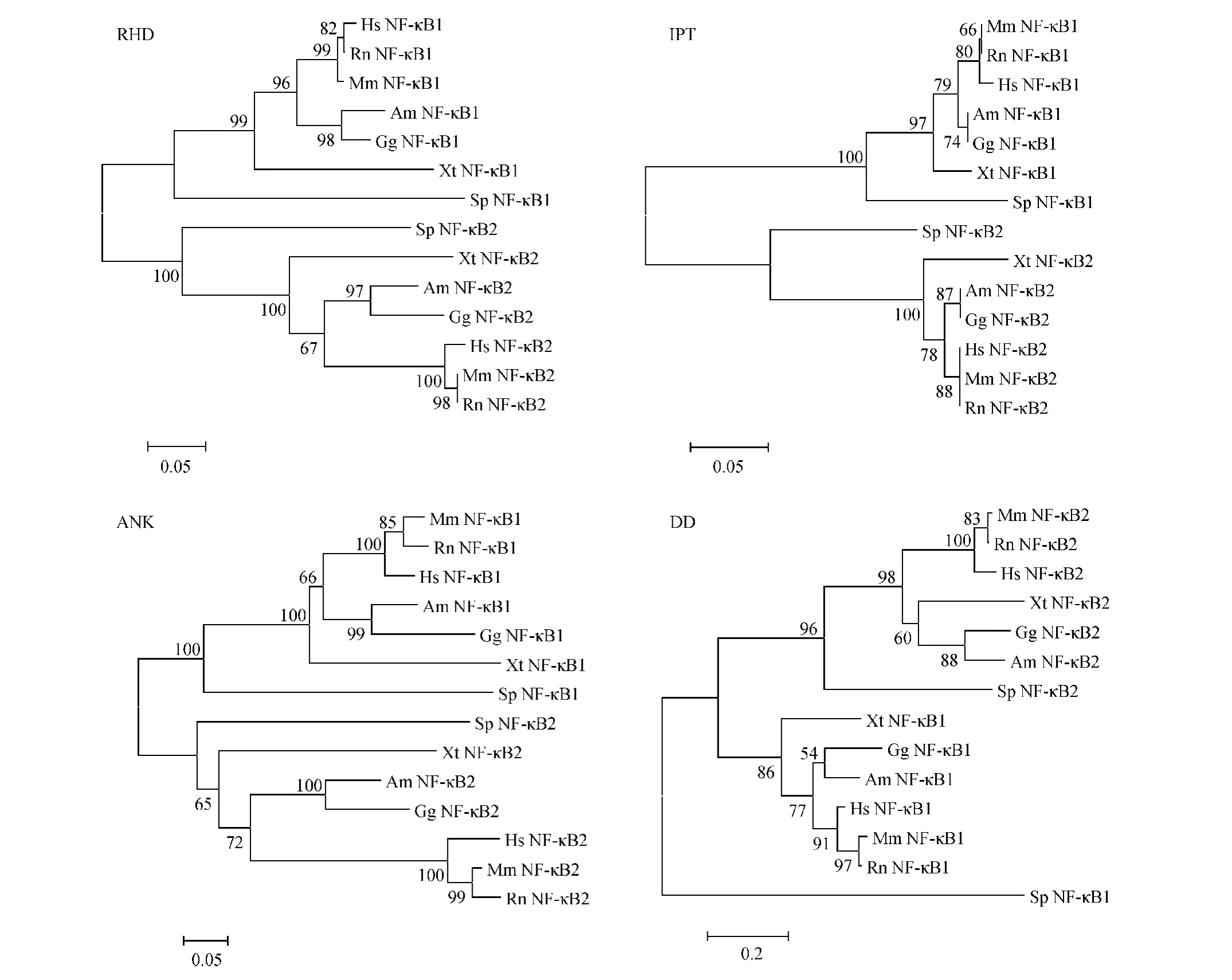
图2 NF-κB1/24个结构域RHD、IPT、ANK与DD氨基酸序列的系统发育树Fig.2 The phylogenetic trees of RHD,IPT,ANK and DD of NF-κB1/2based on their amino acid sequences
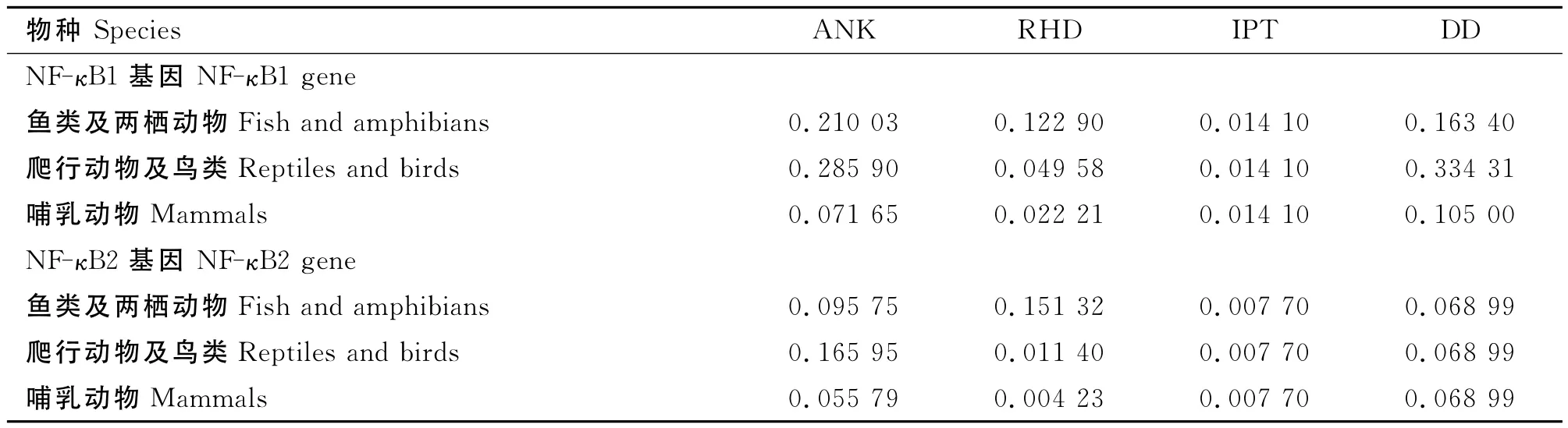
表1 NF-κB1/2基因各结构域在脊椎动物各个群类中的ω均值Table 1 The average value ofωof domains of NF-κB1/2genes in different vertebrate groups

图3 NF-κB1/2的RHD、IPT、ANK和DD保守结构域区域比对Fig.3 Alignment of RHD,IPT,ANK and DD conserved domains of NF-κB1/2
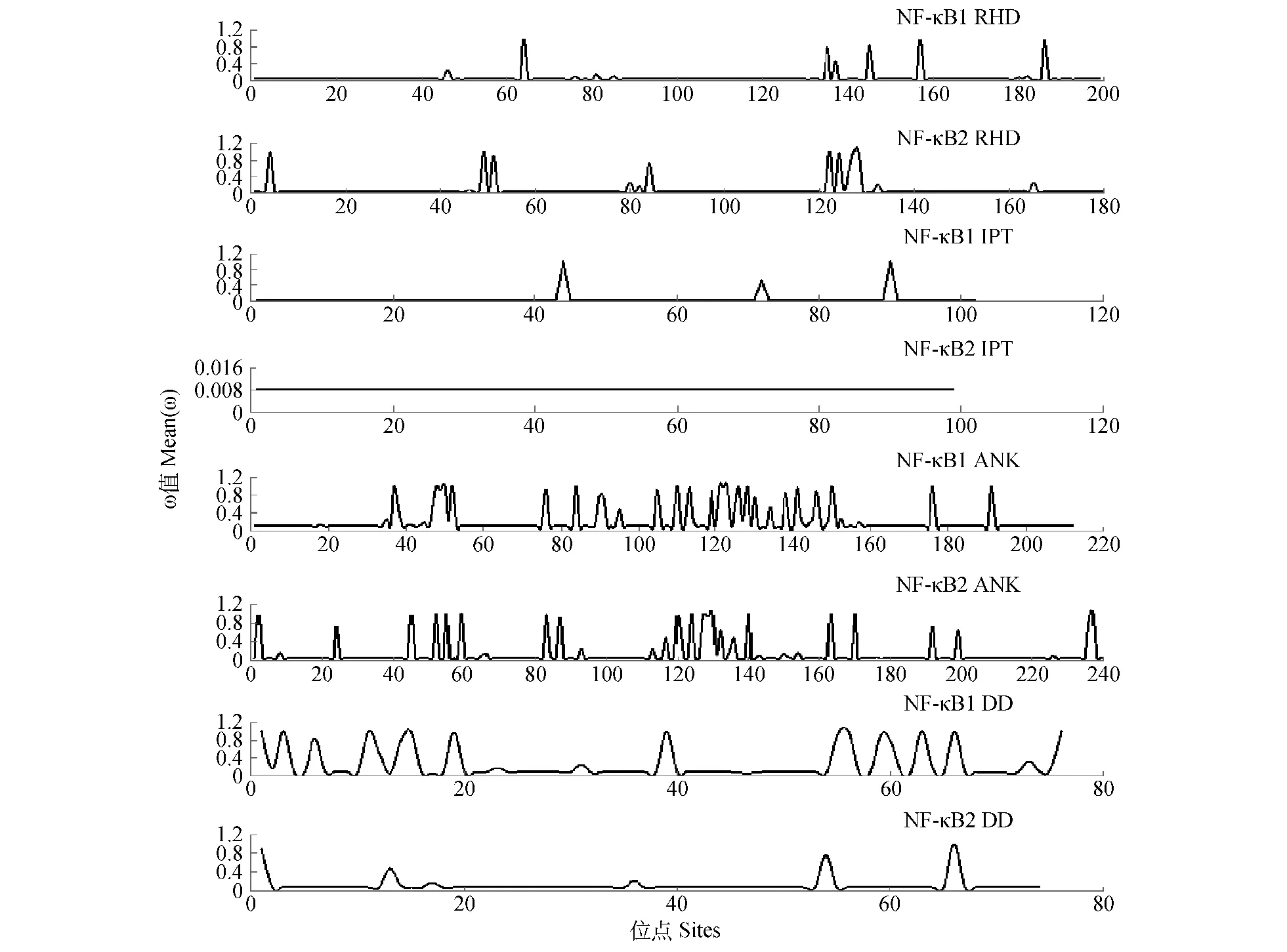
图4 NF-κB1/2RHD、IPT、ANK与DD结构域各位点选择压力Fig.4 The selection pressure of RHD,IPT,ANK and DD conserved domains of NF-κB1/2
RHD、IPT、ANK 与 DD 是 NF-κB1/2亚家族成员重要的典型结构域,早在两侧对称动物时期4个结构域就已经形成[8],且这样的序列结构一直持续到了哺乳动物[8,18]。可以推断,RHD、IPT、ANK与DD在NF-κB1与NF-κB2基因亚家族分子进化特征的形成和分子功能分歧的产生方面扮演了重要的角色。本研究构建了各个结构域氨基酸系统发育树,发现由它们的氨基酸序列构建的系统发育树与基于全序列构建的发育树形态基本相同,说明NF-κB1和 NF-κB2分歧后,NF-κB1和 NF-κB2各个结构域的进化趋势与其整体序列基本一致。
选择压力分析能使人们对NF-κB1/2在进化中所受到的环境阻力与其序列位点的变异情况有更深入的认识。因此,为了更进一步探索NF-κB1/2在脊椎动物阶段的进化历程,笔者又分别分析了NF-κB1/2各个结构域在不同动物类群中(鱼类、鸟类、哺乳动物)的选择压力,发现不同种类动物NF-κB1/2保守结构域 ω值存在差异,说明 NF-κB1/2各个结构域在不同的进化阶段存在着明显不同的进化历程。而在对NF-κB1/2各个结构域序列位点的分析中,发现NF-κB1/2整体序列相对保守,不存在显著的正选择位点。不过NF-κB2的DD所承受的选择压力却明显大于NF-κB1的DD,这很可能是由于NF-κB2的DD所含有的水解抑制区(Processing inhibitory domain,PID)在生理过程中的重要作用所导致[19]。
另外,对脊椎动物 NF-κB1/2 的 RHD、IPT、ANK和DD与直系、旁系同源结构的同源性分析显示,NF-κB1/2的序列发生了各自独立的进化,且这样的独立进化在RHD中表现的尤为显著,暗示NF-κB1/2在功能上的分工越来越明确。而IPT表现的高保守性与它在生理过程中的重要功能相吻合[5,20]。ANK相对于其他3个结构域在脊椎动物的NF-κB1/2间拥有显著的差异,推测它可能是导致NF-κB1/2在脊椎动物阶段序列、功能差异形成的重要因素。而DD仅在NF-κB1/2之间存在较大的差异,且这样的差异在鱼类中就已经表现出来了,这不仅说明了它在脊椎动物阶段中的保守性,也暗示了NF-κB1/2的DD分歧的年代较为久远,甚至可能与NF-κB1/2基因的分歧有一定关联,但是因为NF-κB1/2的分化早于脊椎动物而晚于两侧对称动物,期间相关的动物序列素材较少,因此人们无法进行确切的论证。
综上,在脊椎动物的各个类群中,NF-κB1/2承受着不同的选择压力,它们的4个结构域也表现出了不同的变异程度与特点。NF-κB1/2在脊椎动物中的进化与功能分歧主要由ANK的序列变异造成,而NF-κB1/2在脊椎动物之前的分歧很可能与DD有着密切的联系。
(References):
[1]VALLABHAPURAPU S,KARIN M.Regulation and function of NF-κB transcription factors in the immune system[J].Annu Rev Immunol,2009,27(1):693-733.
[2]IWANASZKO M,BRASIER A R,KIMMEL M.The dependence of expression of NF-κB-dependent genes:statistics and evolutionary conservation of control sequences in the promoter and in the 3′UTR[J].BMC Genomics,2012,13:182.
[3]GUTIERREZ H,DAVIES A M.Regulation of neural process growth,elaboration and structural plasticity by NF-kB[J].Cell,2011,34(6):316-325.
[4]BAEUERLE P A,BALTIMORE D.NF-κB:ten years after[J].Cell,1996,87(1):13-20.
[5]BAKKAR N,GUTTRIDGE D C.NF-κB signaling:a tale of two pathways in skeletal myogenesis[J].Physiol Rev,2010,90(2):495-511.
[6]GAUTHIER M,DEGNAN B M.The transcription factor NF-κB in the demosponge Amphimedon queenslandica:insights on the evolutionary origin of the Rel homology domain[J].Dev Genes Evol,2008,218(1):23-32.
[7]SIMEONIDIS S,STAUBER D,CHEN G,et al.Mechanisms by which IκB proteins control NF-κB activity[J].Proc Natl Acad Sci U S A,1999,96(1):49-54.
[8]SAVINOVA O V,HOFFMANN A,GHOSH G.The Nfkb1and Nfkb2proteins p105and p100function as the core of high-molecular-weight heterogeneous complexes[J].Mol Cell,2009,34(5):591-602.
[9]WHITESIDE S T,ISRAEL A.IκB proteins:structure,function and regulation[J].Semin Cancer Biol,1997,8(2):75-82.
[10]HUANG Y H,TEMPERLEY N D,REN L M,et al.Molecular evolution of the vertebrate TLR1gene family-a complex history of gene duplication,gene conver-sion,positive selection and co-evolution[J].BMC Evol Biol,2011,11(1):149.
[11]LU M X,HUA J,CUI Y D,et al.Five small heat shock protein genes from Chilo suppressalis:characteristics of gene,genomic organization,structural analysis,and transcription profiles[J].Cell Stress Chaperones,2014,19(1):91-104.
[12]QIAN Y H,WU H Y,JI X Y,et al.Mitochondrial genome of the stonefly kamimuria wangi(Plecoptera:Perlidae)and phylogenetic position of plecoptera based on mitogenomes[J].PLoS ONE,2014,9(1):e86328.
[13]徐怀亮.7个灵长类物种PRSS12基因编码区序列测定与进化分析[J].东北林业大学学报,2010,38(4):85-88.XU H L.Sequencing and phylogenetic analysis of PRSS12coding region of seven primate[J].Journal of Northeast Forestry University,2010,38(4):85-88.(in Chinese)
[14]PICONE B,CHRISTOFFELS A.Molecular evolution of key receptor genes in primates and non-human primates[J].J Genet Genomics,2014,2(4):57-61.
[15]ARENAS M,POSADA D.Simulation of genome-wide evolution under heterogeneous substitution models and complex multispecies coalescent histories[J].Mol Biol Evol,2014,31(5):1295-1301.
[16]ZHAO H,ZHOU Y,PINTO C M,et al.Evolution of the sweet taste receptor gene Tas1r2in bats[J].Mol Biol Evol,2010,27(11):2642-2650.
[17]YU L,JIN W,ZHANG X,et al.Evidence for positive selection on the leptin gene in Cetacea and Pinnipedia[J].PloS ONE,2011,6(10):e26579.
[18]HUGUET C,CREPIEUX P,LAUDET V.Rel/NF-kappa B transcription factors and I kappa B inhibitors:evolution from a unique common ancestor[J].Oncogene,1997,15(24):2965-2974.
[19]李 贤,刑 达,陈小佳.核因子KB抑制蛋白家族的研究进展[J].中国病理生理杂志,2006,22(12):2480-2484.LI X,XING D,CHEN X J.Advances of nuclear factor KB of inhibit protein family[J].Chinese Journal of Pathophysiology,2006,22(12):2480-2484.(in Chinese)
[20]CRAMER P,MÜLLER C W.A firm hand on NFκB:structures of the IκBα-NFκB complex[J].Structure,1999,7(1):R1-R6.
