合成生物学技术研究进展
2015-07-19吕永坤堵国成陈坚周景文
吕永坤 堵国成 陈坚 周景文
(江南大学,无锡 214122)
合成生物学技术研究进展
吕永坤 堵国成 陈坚 周景文
(江南大学,无锡 214122)
合成生物学是一门将生物学工程化的应用学科,目的在于将生命元件标准化和模块化;也可用于生命起源等基础理论研究。对合成生物学进行了较为详细地介绍,主要综述了合成生物学领域新出现的方法及应用。
合成生物学;生物功能元件;无细胞合成生物学;最小基因组;定向进化;基因组编辑
合成生物学自诞生之日起就受到人们的广泛关注。2006年,由美国国家科学院资助的合成生物学工程研究中心成立;2007年,欧洲委员会启动了18项合成生物学领域的主要研究项目;英国也于2007年将合成生物学列为高优先级资助研究领域[1]。2008年,时代周刊将“创造生命”列为十大科学发现之一;2009年,英国皇家工程学院对合成生物学及其应用前景和影响进行了详细介绍。同时,欧美纷纷向合成生物学研究领域投入巨资。另一方面,合成生物学的市场也在迅速扩张。BCC Research的研究报告认为,2012年和2013年合成生物学总的市场份额分别为21亿和27亿美元;到2018年,该市场份额将达到118亿美元,且2013-2018五年间的复合年增长率为34.4%。
合成生物学起源于工程应用,其目的在于简化复杂的自然生物系统,将其变为简单、可靠、质量可控的模块或者零件。这些模块或零件可以使用计算机辅助设计(Computer aided design,CAD)来进行精确的预测和设计,可以进行不同的组合以获得预期的功能,并且可以在工业规模进行生产制造[2]。合成生物学的快速发展得益于基因合成和测序等技术的进步。人工合成DNA价格的持续下降,使得经过密码子优化的基因、启动子及其他调控元件的使用更加广泛,甚至出现了完全由人工合成的基因组[3]。高通量测序技术的快速发展使得测序的速度空前提高,测序成本也不断下降,不断有新的物种基因组数据被公开。基因元件功能表征和标准化是设计和构建具有新特征生物的关键步骤,越来越多的基因元件被构建和测试。
1 合成生物学及其起源
目前对于合成生物学有多种不同的定义,较普遍的观点是,合成生物学的目标是设计和操纵生物基零件、装置和系统,创造新功能甚至新物种,也可以对已有的天然生物系统进行重新设计。合成生物学是一门应用工程学原理进行系统设计的应用学科[9]。要理解合成生物学首先需要对其起源进行研究。关于合成生物学的起源目前有三种不同的看法:高通量化学合成DNA的实现;第一个基因计时器[10]和基因电路开关的构建[11];以合成的基因元件构建完整的代谢通路。
如果将DNA的高通量化学合成作为合成生物学的开端,那么合成生物学其实是一种新的合成技术,为代谢途径的构建和调控提供了一种简单廉价的方法。化学法合成基因的长度和类型不断扩展,如今已经可以实现小型基因组的合成[12]。如果以第一个基因计时器或基因电路开关的构建作为合成生物学的开端,那么合成生物学其实是一种基础生物学问题研究的新方法。在这种观点下,细胞被视为一台设备,化学合成的DNA赋予其感受环境变化并以此调整自身的功能、生理和突变率[13]。这类基因控制元件有许多应用,可用以设计医学治疗的装置[14]。由于存在巨大的工业化潜力,合成生物学在代谢途径设计和构建中的应用可能是以上三种情况中最受关注的。然而,从理论的角度来讲,该领域并无太大贡献。因为研究者会认为,大多数所谓的合成生物学不过是在代谢工程中引入化学合成的DNA而已。
2 生物功能元件
正如电子工程相对于物理学一样,合成生物学可以理解为生物学的工程化,而生物功能元件正是该工程领域的基本单元。通过理性设计,将不同功能特征的元件进行组合,可以得到具有新功能或新表型的生物体。实现这一目标需要依赖于对生物元件功能的表征、标准化、组装和调试,以及对基因回路、底盘和整个体系的设计。
2.1 生物功能元件的设计、合成和功能表征
生命形态的多样性,决定了生命系统可以合成几乎所有现存有机化合物;遗传信息的可编辑性,又提供了创造新的生命形态的可能性。生物功能元件是合成生物学的基石,是具有特定功能的最小生物元件,可以是氨基酸序列或核苷酸序列。不同的功能元件可以进一步被组合成具有更加复杂功能的装置和系统。理论上,通过对生物功能元件的设计、组合,可以实现任何有机化合物和新物种的合成。另一方面,非天然氨基酸的使用、新核酸的开发及随之而来的遗传密码子表的扩展,又可以进一步扩展化合物和新物种合成的范围。随着DNA合成技术的发展和商业化,基因元件合成的保真性和通量不断提高,同时成本迅速下降,这为基因元件的大规模合成提供了基础。另一方面,高通量筛选技术的普及,使得人们可以对大量基因元件进行高通量筛选和功能表征。
2.2 生物功能元件的标准化
随着合成生物学的诞生,人们即在尝试将生物功能元件标准化。1996年,Rebatchouk等[15]尝试通过克隆技术建立一个标准的基因元件清单,但该研究并未立即引起广泛的兴趣。来自麻省理工学院的Kight于2003年提出了“生物砖”(BioBrick)的概念。随后,大量的研究机构开始使用标准的生物砖元件来构建新的生物装置和生物系统。生物砖是进行生物功能元件标准化的重要尝试,可以将其理解为积木的单元,通过这些积木不同方式的组合,我们可以组装出各种不同的结构[16]。通常生物砖元件应该包括基因的编码序列和调控序列,如启动子、核糖体结合位点和终止子等。由麻省理工学院于2003年成立的标准生物元件库(Registry of Standard Biological Parts)目前收集了3 400件基因元件,用于组装生物装置和生物系统。这些标准的基因元件来源于学术研究机构、科学家个人和每年参加国际遗传工程机器设计竞赛(International genetically engineered machine competition,iGEM)的学生团队。其中的每个生物元件都有自己的编码,还包括其序列、创建者、功能及使用者等信息。这些信息都是公开的,可供免费使用。
2.3 生物功能元件的组装
合成生物学的特点在于,可以对标准化功能元件进行组装,以获得不同功能的装置和系统等。但是,由于生命系统的复杂性及人类认知的局限性,合成生物学往往不能像电气工程等其它现代工程学科一样,将特定功能的基因元件进行简单的组装,就可以获得预期的结果[16]。在此过程中,需要对元件与元件之间、元件与装置之间以及装置与装置之间进行适配和优化;同时,元件、装置与底盘之间的兼容性也是重要的影响因素。在此过程中,高通量的测试方法和系统生物学的分析方法等都是进行适配和优化的有效方法。利用计算机辅助的动态模拟,在构建之前对系统进行建模、预测和优化,这样可以减少系统测试的工作量,加快构建的速度[9]。如可以对各种组学数据进行整合、构建接近真实的生物系统模型、预测系统的功能及对环境扰动的响应,从而给出优化方案。
3 无细胞合成生物学
合成生物学试图使用工程学方法来缩短设计-构建-验证的工作周期,因为目前人工构建细胞的方法工作量极其巨大,且成本较高[17]。另一方面,人们对复杂生命活动认知的局限性,原有部分和合成(外来)部分之间的干扰,合成途径的动态监控,这些都是目前胞内合成生物学(in vivo synthetic bilogy)所面临的挑战[16],如表1所示[18]。由于不需要为细胞提供生长所需的辅助环境,无细胞合成生物学(cell-free synthetic biology)为解决上述问题提供了有力的支撑。无细胞合成生物学研究的是,不需要完整活细胞的条件下进行的复杂的生物学反应过程,这些过程是在体外进行的[19,20]。相对于胞内合成生物学,无细胞合成生物学的特点是,它是一个开放的反应体系。相对于胞内合成生物学,无细胞合成生物学具有许多优势,如表1所示[21]。
作为基础研究和应用研究工具,无细胞合成生物学已被研究了数十年,然而直到现在无细胞合成生物学用于工业生产才具有商业可行性,包括医药、代谢产物和非天然产物[22,23]。无细胞体外反应体系可以划分为粗提物无细胞反应体系(Crude extract cell-free systems,CECFs)和合成酶途径(Synthetic enzymatic pathways,SEPs)[17]。在选择反应体系时,首先考虑的因素是需要保留的细胞“看家”功能(如持续提供能量)。构建SEPs所面临的主要挑战是人们对基础生物学功能认知的局限性;而CECFs则经常发生不想要的反应或者产生某些未知产物。时间和成本也是需要考虑的因素。例如,在无细胞蛋白质合成过程中,重构SEP需要涉及能量代谢、呼吸、转录、翻译和蛋白质折叠等,对于大多数商业应用,蛋白质的折叠都是价格高昂的。尽管SEPs和CECFs的应用有所重叠(图1),但依然需要谨慎权衡二者之间的平衡[17]。以下为无细胞合成生物学的主要应用。

表1 胞内合成生物学所面临的挑战
3.1 蛋白质合成
无细胞蛋白质合成(Cell-free protein synthesis,CFPS)是无细胞合成生物技术最常见的应用。CFPS系统可以利用酶转化细菌、植物或者动物细胞提取物。当在系统中提供必要的氨基酸和能量等时,这些活化的生物催化剂就可以催化氨基酸向多肽的聚合。大肠杆菌CFPS系统高效的蛋白质合成能力(>1 g/L),使得商业化生产个性化蛋白质药物[24]、疫苗[25]和难以胞内合成的蛋白质(如水解酶类)[26]成为可能。
3.2 代谢产物合成
在过去的研究中,人们开始意识到完整的代谢途径也能够在CECF系统中实现,包括中心代谢、三羧酸循环和氧化磷酸化等。根据这一思路,人们正在努力通过控制CECF生产代谢产物。Panke等[27]设计的CECF多酶催化系统,以葡萄糖为底物合成二羟丙酮磷酸(DHAP)。由于DHAP的不稳定性,可以直接向反应体系中添加丁醛和兔肌肉醛缩酶,将DHAP转化为一种稳定形式。相对于CECFs,SEPs系统多用于小分子的生产[28]。SEPs的优势在于理性设计生物合成网络时的灵活性。Wang等[29]以纤维二糖为底物,通过一个12个酶的SEP系统合成了NADPH形式的氢。其中的纤维素二糖主要由酶解纤维素和稀酸预处理生物质水解物得到。在有酸处理的水解物存在的情况下,大肠杆菌是不能生长的。这一应用实例显示了无细胞合成技术的优势,即不需要满足细胞生长的需求。
3.3 基因元件的构建
相对于在体内构建基因元件,体外方法具有独特的优势:反应条件的可控性和可预测性;大量的已知部件加快原型构建的速度;体外基因元件可以转化为真正的基因模块。得益于体外基因元件构建的快速发展,原型构建可以在1个工作日内完成;由4个基因开关组成的原型则可以在8 h内完成[30]。人们开发了诸多无细胞元件,包括逻辑门、存储单元和振荡器。其中,逻辑门包括AND、OR、NOR、XOR、NAND和NOT等;存储单元有1-输入双推和2-输入开关,以及诸多振荡器。这些无细胞基因元件可以用于组装最小人工细胞和细胞样微装置[18]。
3.4 扩展生命的化学结构
非天然氨基酸(Unnatural amino acid,uAA)掺入是无细胞合成生物学的又一强大工具,因为其允许其他的化学成分掺入到蛋白质中,从而可以扩展生命的化学结构。目前有两种途径可以实现uAA的掺入[31,32]。(I)残基置换法:天然氨基酸类似物能够被细胞内原有机制所识别,从而可以将天然氨基酸置换为相应的氨基酸类似物。该方法需要营养缺陷型宿主,且仅限于天然氨基酸类似物。(II)密码子重置或终止密码子消除法:该方法依赖于突变氨酰基-tRNA合成酶/tRNA对(Mutated aminoacyltRNA synthetase/tRNA,aaRS/tRNA)与uAAs的正交氨酰化,运送uAAs至蛋白质并被掺入。这两种方法均在无细胞合成系统中获得了成功。将新的化学物质引入蛋白质的特定位点具有很多种重要的应用,包括将药物连接到抗体上用于临床治疗[33],将蛋白质按特定方向固定到非生物材料表面[34],生产功能性病毒样颗粒[35],探针和标记物掺入[36]等。
另一方面,研究者通过开发新的核酸来扩展遗传密码子表[20]。其中,Benner等通过重新定位胞嘧啶和鸟嘌呤中氢键的供体和受体,获得了新的碱基对[异胞嘧啶:异鸟嘌呤(iso-C:iso-G)]。这导致了第65个密码子——反密码子对的出现,从而可以使uAAs在蛋白质体外合成过程中高效掺入。Benner等[37]对其工作进行了进一步的优化,创造了人工扩展遗传信息系统(Artificially expanded genetic information system,AEGIS)。通过重置碱基上氢键供体和受体基团,将碱基数量由4个增加至12个。其中,碱基Z和P成为研究的焦点,二者不易受到氧化和差向异构的影响。在6碱基遗传密码子表(G∶C、A∶T、Z∶P)的基础上,Benner等[38]进一步通过PCR反应扩增出了各种各样的序列,其中包括多个连续非天然Z和P的序列。这是令人激动的成果,它说明这种拥有216种可能密码子的非天然的遗传密码子表(ATGCZP)有可能被用于在体外合成生命[20]。
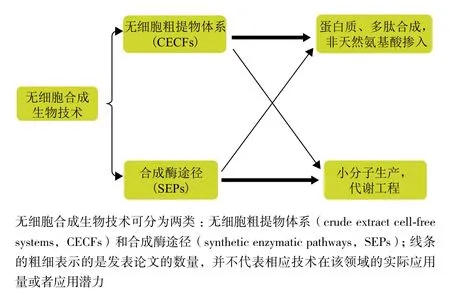
图1 无细胞合成生物学的分类及应用
4 最细小基因组和最小细胞
构建人工细胞的关键是获得最小基因组。最小基因组指的是在最优生存条件下,细胞维持生存所需要的最小的基因集合[39]。最小基因组可以作为特定功能的基因的载体被转移到细胞底盘中,最终构建具有特定功能的最小人工细胞。细胞底盘指的是包含代谢系统的容器或分隔的空间,其中不包括遗传信息或基因组。如果将基因组视为细胞的软件系统,则底盘指的是细胞的硬件系统。最小细胞指的是,一定环境条件下,能够维持生命活动的最小细胞单元,也可以理解为由最小基因组控制的自主复制细胞[39]。最小细胞不仅具有重要的应用价值,而且可用于研究地球生命的起源。值得注意的是,最小基因组和最小细胞均为相对的概念,因物种及其生存环境不同而异[40]。例如,当对两个细菌的基因组进行比较时,得到的最小基因组由256个基因组成;当对100个基因组进行比较时,最小基因组的基因数减少至63;当对1 000个基因组进行比较时,最小基因组的基因数为0[41]。又如,寄生或共生微生物的某些基本生命活动(如氨基酸、维生素合成等)相关的功能基因在进化过程中被简化掉了,因为这些微生物可以从寄主中直接获取这些物质;另一方面,与其同源的非寄生微生物则保留有这些功能基因[42]。
最小基因组的研究方法包括比较基因组学方法、转座子饱和突变法、反义RNA技术、单基因特异性突变法和基因组简化法等[40]。这些方法均存在不足之处,目前无明确、统一的研究结果。在最小基因组研究初期,人们期望通过扩大研究的物种范围,获得一个相对于所有物种通用的最小基因组。然而,这种普适最小基因组可能并不存在[39]。有以下几方面的原因。(1)在比较基因组学研究中,当物种及其环境多样性足够大时,人们所获得的最小基因组的基因数为0,即并不存在所有物种必需基因的交集[41]。(2)某些生命必需的功能,在不同物种中由非同源的基因所编码,在同一物种中也可能由多个基因发挥同一功能。如短RNA残留物的降解被认为是必需功能,该功能由nanoRNases负责。nanoRNases在不同物种中有不同的来源(orn来源于Escherichia coli,nrnA和nrnB来源于Bacillus subtilis,nrnC来源于Bartonella birtlesii),且orn与nrnA或nrnB在序列上几乎没有相似性[43];在Bacillus subtilis中则由两个不同基因(nrnA、nrnB)所编码,其中一个基因的失活不能导致细胞的死亡,因此可能被误认为非必需基因;而在E. coli中,nanoRNases只能由orn编码,因此无疑是必需的。(3)理论上,通用最小基因组概念的前提是所有生命起源于一个“最晚出现的共同祖先”(last universal commen ancestor,LUCA)[44],即所有遗传信息起源于同一套基因组。然而,LUCA事实上是一个初始细胞的群体,它们的基因组分别是古生菌、真细菌和真核生物的遗传基础。基因组的发展是在细胞进化的后期,在此之前已经出现了DNA复制、脂类合成和RNA降解相关的酶类的多样性[45]。(4)以必需基因的集合作为最小基因组,那么最小细胞只能在最优条件下生存,而不能持续生存,尤其是当环境条件有所波动的情况下。
Acevedo-Rocha等[39]分析认为,通用最小基因组研究不成功的原因在于以必需基因的集合作为最小基因组;并提出以持续基因(Persistent gene)的概念来替代必需基因。持续基因指的是位于前导链上优先表达的基因,在细胞的长期繁殖(必需基因的功能)或细胞的自我维持、修复中发挥作用。基因组应当被划分为两部分:Paleome和Cenome。Paleome包括持续基因,与生长、复制、转录、翻译、维持和衰老等关键功能有关。Paleome由大约500个持续基因组成,这些基因的功能包括:核心代谢,氨基酸、核苷酸、辅酶、脂类合成,细胞分裂,氨酰-tRNA合成,转录和翻译。Paleome可被进一步划分为持续必需基因和持续非必需基因。这些持续非必需基因被认为是可以省却的,但实际上其与细胞的维持与压力响应有关,消除后将导致细胞在环境波动时死亡。Cenome由非持续基因组成,这些基因负责细胞对特定环境的适应。因此,Paleome负责细胞在最优条件下存活,而Cenome负责细胞在特定自然条件下的长期生存。Cenome在同一物种的不同个体之间变换很大。例如,某E. coli的1 500个非持续基因(Cenome)与500个持续基因(Paleome)组成了该菌株的核心基因组(2 000个基因),而所有E. coli菌株的Cenome共包括18 000个基因[46]。
基因持续的概念在合成人工细胞时非常重要,因为按照必需基因概念构建的细胞只能在最优的环境条件下存活;而按照持续基因概念设计的细胞则可以在特定环境下长期生存,并能应对环境的波动。根据持续基因概念合成人工细胞应当包括如下步骤:(1)对细胞需要应对的环境条件进行评估;(2)根据环境条件对细胞进行理性设计,包括去除部分非持续基因、添加特定功能的基因,得到由paleome、简化的cenome和特定功能基因组成的人工基因组;(3)基因的合成与扩增;(4)人工基因组在Saccharomyces cerevisiae中组装;(5)人工基因组转移到合适的底盘中(图2)。
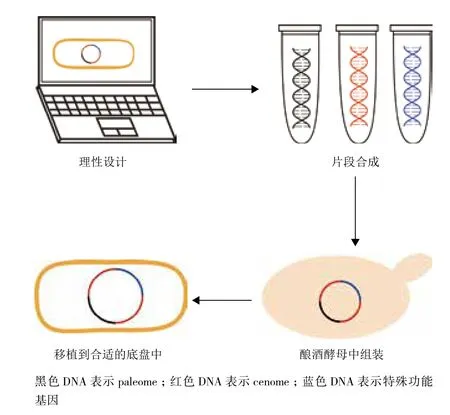
图2 特定环境下具有特殊功能的最小细胞的合成策略
5 基因组编辑方法
在许多研究中,人们发现质粒分离、质粒上基因表达的不精确性以及质粒保持造成的代谢负担等问题随着质粒拷贝数的增加而加剧[47]。在基因组上整合表达显然可以解决这些问题,且避免了抗生素和诱导剂等的使用[48]。由于细胞系统几乎所有的功能都由基因组编码,因此理论上基因组编辑可以重构生命系统[49]。在合成生物学的应用中,基因组编辑是异源合成途径的重构及模块优化的基本操作手段。传统的定点重组酶系统如Cre/lox、PFlp/FRT和φC31等能够实现对基因组的编辑,然而这些方法存在诸多不足,如重组效率低,筛选过程复杂,存在不利突变的可能性等。近年来出现的锌指核酸酶(Zinc-finger nucleases,ZFN)、转录激活因子样效应生物核酸酶(Transcription activator-like effector nucleases,TALEN)和成簇的规律间隔的短回文重复序列(Clustered regularly int-erspaced short palindromic repeats,CRISPR)/Cas9系统使得人们能够高效、便捷且廉价地对基因组进行精确编辑。研究者既可以通过常规的分子生物学手段在较短的时间内构建这些系统,也可以购买商业化的试剂盒或者向生产商购买订制的产品。
5.1 ZFN和TALEN
ZFN是第一个使用定制DNA结合结构域的核酸内切酶。ZFN是异源二聚体,其中每个亚基含有一个锌指结构域和一个FokI核酸内切酶结构域。Cys2-His2锌指结构域是真核生物中最常见的DNA结合基元。每个锌指结构域在其保守的ββα构型中包含30个氨基酸,其中α螺旋表面的氨基酸识别基因组DNA上3-4个连续的核苷酸序列。经过设计和改造的锌指结构域已经可以识别所有64个核苷酸三联体;另一方面,锌指结构域可以由连接肽串联,从而可以识别9-18 nt的DNA序列。经过上述改造,锌指蛋白理论上可以识别任意基因组上的任意目标序列[50,51]。Fok I为非特异性核酸内切酶,但锌指结构域赋予其切割位点的特异性;Fok I结构域必须二聚化才有活性,确保必须存在两个相邻的DNA结合事件才能实现双链断裂,从而增加了切割特异性。在双链断裂后,细胞试图修复它。最简单的方法是非同源末端接合(Nonhomologous end joining,NHEJ),其中细胞基本上磨平断裂DNA的两端,再将其彼此拉近,这往往产生移码。另一种方法是同源定向修复(Homology-directed repair,HDR)。细胞试图利用另一条染色体上对应的DNA序列作为模板来修复断裂。也可以通过提供特定的外源模板,对断裂位点进行编辑。
TALEN也是二聚的转录因子/核酸酶,由转录激活因子样效应物(Transcription Activator-like Effector,TALE)与FokI内切酶结构域融合而成。TALE是植物病原菌Xant-homonas的天然蛋白,其DNA结合结构域由一系列结构重复而成。每个重复结构包括33-35个氨基酸,且只能识别1个碱基。与锌指结构域一样,TALE也通过重复结构的串联来识别特定的DNA序列;且通过不同的串联组合,研究人员可靶定他们想要的任何序列[52]。TALE重复结构的串联可以在数天内快速构建[53]。限于三联核酸识别模式,ZFN构建困难且成本较高,而TALEN和CRISPR/Cas系统在这方面则具有相对优势。
5.2 CRISPR/Cas9
CRISPR/Cas是细菌和古细菌在长期演化过程中形成的一种适应性免疫防御系统,可用来对抗入侵的病毒及外源DNA。CRISPR/Cas通过将入侵噬菌体和质粒DNA的片段整合到CRISPR中,并利用相应的CRISPR RNAs(crRNAs)来指导同源序列的降解,从而提供免疫性[54]。目前,在基因组定向编辑方面,运用最广泛的是源自于生脓链球菌(Streptococcus hyogenes)的Type II CRISPR/Cas9系统。此系统的工作原理是crRNA(CRISPR-derived RNA)通过碱基配对与tracrRNA(Trans-activating RNA)结合形成tracrRNA/crRNA复合物,此复合物引导核酸酶Cas9蛋白在与crRNA配对的基因组序列靶位点剪切双链DNA,形成双链缺口(Double strand break,DSB)。然后细菌通过非同源重组NHEJ或同源重组HDR两种方式修复DSB[55]。近来,研究表明,通过人工设计的sgRNA(Short guide RNA)也可以引导Cas9对DNA的定点切割(图3-A)[56]。
研究发现,Cas9蛋白不具有特异性,只需要一个特异识别基因组靶序列的sgRNA,即能实现对基因组靶序列核酸酶定向切割。人工合成的sgRNA一般包括3个部分(图3-B):靶序列互补配对区(Base-pairing region,20 nt)、Cas9蛋白结合区(Cas9 handle Region,42 nt)、转录终止区(S. hyogenes terminator,40 nt)。sgRNA引导Cas9蛋白识别基因组上的PAM(Protospacer adjacent motif)序列(S. hyogenes Cas9识别序列为NGG),并与PAM序列的5'端20个核苷酸序列互补配对,然后Cas9蛋白在PAM序列上游5'端第3个碱基处切割形成DSB[57]。由于CRISPR/Cas系统在基因组编辑上的高效、简便,这一新技术目前被广泛使用,并已成功运用于肺炎链球菌、大肠杆菌[58]、果蝇[59]、人类、小鼠、斑马鱼等[60]物种的基因组编辑。
6 定向进化
虽然我们已经可以从头合成完整的细菌基因组及部分真核生物基因组,然而对于生命系统的认知依然有限,这使得我们在面对一些问题时并无太多策略。如异源表达蛋白质溶解度低、热稳定性差,合成途径中多个基因的协调性问题,在异源环境中基因元件功能的优化,基因组优化以使合成生命获得预设的表型等[61]。定向进化并不需要我们预先了解太多生命系统的相关信息,从而成为解决这些问题的有力工具。本节主要介绍定向进化在合成生物学领域的应用及其新发展。
6.1 定向进化在合成生物学中的应用
定向进化指的是在实验室条件下创造突变,并对突变文库施加筛选压力,从而筛选出具有期望表型的突变体[62]。定向进化实验步骤包括创造随机突变,将突变基因在合适的宿主中表达,以适当的筛选条件筛选有利突变。重复上述步骤,经过不断的循环有利突变在较短的时间内快速积累,并可被用于后续操作以解决上述问题。定向进化可以在单分子水平实施,也可以在合成途径和调控网络水平实施,甚至在细胞整体水平实施以直接获取某些特定表型[61]。
6.1.1 改善酶的催化特性 酶既是生命的基本元素,也可被单独用于生物催化,来合成医药、化学品和能源。能够在生产中应用的酶应当具有如下特性,催化活性高、底物特异性强、稳定。定向进化非常适合于提高酶的上述特性。PhlD被用于藤黄酚的生物催化合成,但PhlD的催化活性和稳定性均较差,成为限制其应用的重要因素。通过序列分析,Zha等[63]找到了PhlD的52个同源基因,并构建了容量为1.2×1011的突变体文库。使用高通量筛选技术,筛选到了酶活性和稳定性均提高的突变体。来源于Methanococcus jannaschii的柠苹酸合酶(citramalate synthase,CimA)在E. coli中表达活性较低。柠苹酸途径能够使L-异亮氨酸缺陷型E. coli在基本培养基上生长,且细胞生长速率与柠苹酸途径的活性正相关。根据这一原理对CimA在L-异亮氨酸缺陷型E. coli中进行定向进化。经过6轮定向进化实验,得到了1个活性提高且抗反馈抑制的CimA突变体。突变体使正丙醇和正丁醇的生产效率分别提高了9倍和22倍[64]。
6.1.2 非天然氨基酸掺入 当使用密码子重置法将非天然氨基酸掺入蛋白质时,需要3个元件,即终止密码子、tRNA和氨酰-tRNA合酶(aaRS)[65]。通过活性位点的改变,可以改变aaRS携带氨基酸的特异性,从而实现非天然氨基酸的掺入。通过定向进化,已经有70多种不同结构的非天然氨基酸掺入到细菌、酵母和哺乳动物的蛋白质中[32]。虽然非天然氨基酸的掺入可以使蛋白质的性质和功能发生变化,但同时也常常导致蛋白质活性和稳定性的下降。定向进化不仅可用于提高天然蛋白质的性质,也可用于提高携带有非天然氨基酸的蛋白质的活性和稳定性[66]。
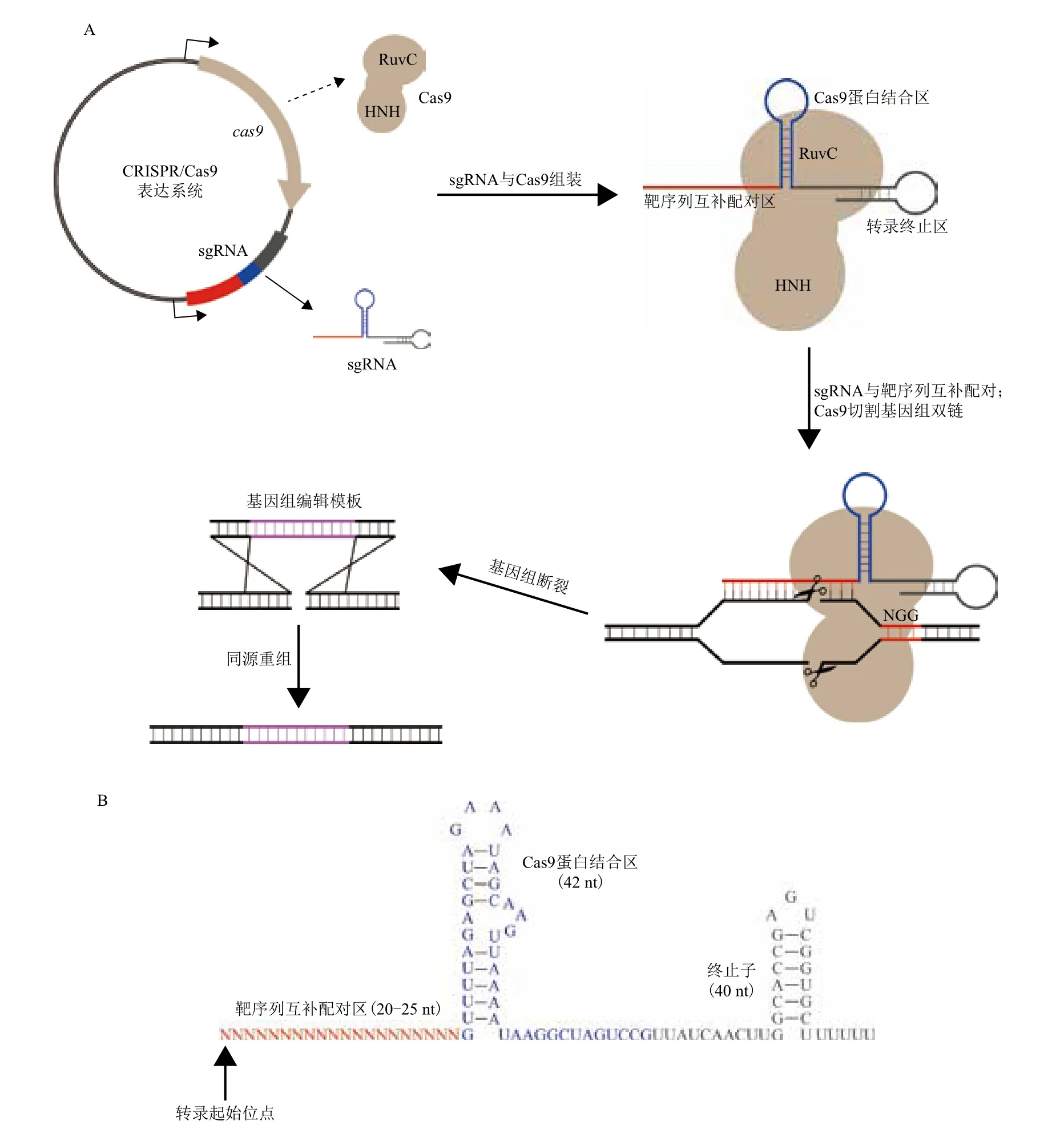
图3 Type II CRISPR/Cas9系统基因组编辑示意图
6.1.3 基因表达水平调控 定向进化可以用于基因表达水平改造,如通过改造启动子和构建操纵子来调控基因表达。对组成型启动子PL-λ的衍生物进行易错PCR,获得突变体文库。将突变体亚克隆至报告载体的绿色荧光蛋白GFP基因上游,并转化E.coli。通过筛选,获得了22个不同荧光信号强度的突变体。以这些突变体来考察磷酸烯醇式丙酮酸羧化酶对细胞生长和脱氧木酮糖-5-磷酸合成酶对番茄红素产量的影响。另外,定向进化手段改造启动子也可以用于S. cerevisae。为了同时调控多个相关基因,而又不分别对各自的启动子进行改造,构建操纵子是比较方便的手段之一。为了调控操纵子内多个基因的表达水平,需要构建和筛选可调基因间区(Tunable intergenic region,TIGRs)文库[67]。TIGR包括一系列控制元件,包括mRNA二级结构、RNA酶切割位点和核糖体结合位点(RBS)分隔序列(图4)。以红色荧光蛋白RFP和绿色荧光蛋白GFP作为报告系统,TIGRs可以使两个报告基因的相对表达强度改变100倍。通过TIGR方法来平衡甲羟戊酸合成途径中的3个基因的表达水平,可以使甲羟戊酸在E. coli中的产量提高7倍。
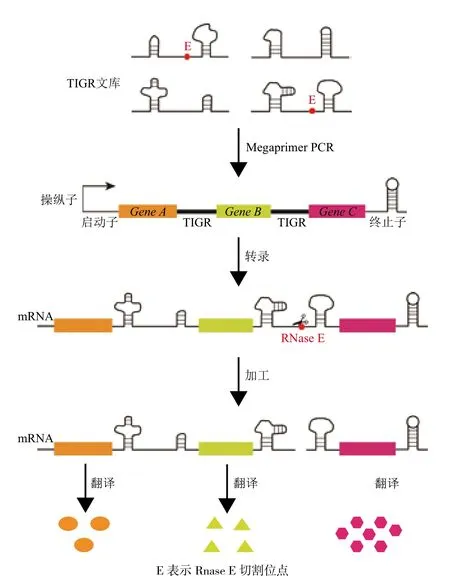
图4 TIGR法调控操纵子中多基因表达水平[67]
6.2 定向进化的新进展
虽然定向进化是解决合成生物学问题的有力工具,但是其也受到文库大小、筛选效率和进化实验所需时间等因素的限制。不过,不断出现的新方法已经可以用于缓解定向进化的这些限制,如智能文库的构建、体内定向进化等。
6.2.1 智能文库构建 在突变体文库构建过程中,传统的定向进化使用的是随机突变的策略。在随机突变产生的大量突变体中,有利突变往往并不占多数,但文库容量的增加又增加了筛选的难度。而智能文库指的就是容量较小且富含有益突变的突变体文库。重构的进化适应途径(Reconstructed evolutionary adaptive path,REAP)分析是构建智能文库的方法之一[68]。REAP被用于扩展DNA聚合酶的底物谱。通过分析一系列DNA聚合酶在进化过程中的保守位点和可变位点,找到进行基因突变的位点。通过对93个突变体的筛选,得到两个能够高效利用新底物的突变体。相对于传统的随机突变文库,从智能文库中筛选出有利突变的效率极大地提高。当需要进化的对象的遗传信息未知时,可以使用另一种被称为截短的宏基因组基因特异性PCR的方法[69]。该方法将环境中的DNA按照功能进行筛选,接着使用一套引物扩增其中具有一定同源性的基因,从而得到一个具有功能和遗传特异性的突变体文库。
6.2.2 体内定向进化 体内定向进化策略可以简化定向进化的实验操作,并减少人为因素的干扰。噬菌体辅助持续进化(Phage-assisted continuous evolution,PACE)就是很好的例子[70]。在PACE实验中,E. coli持续流经一个装有复制性噬菌体的装置,复制性噬菌体携带有目的基因。噬菌体要产生具有侵染力的后代需要pIII蛋白质,而研究者将噬菌体合成pIII蛋白质的能力与目标蛋白质的功能相偶联。因此,当噬菌体感染流经的E. coli,并编码具有功能的目标蛋白质时,可以产生pIII,进而产生具有侵染能力的后代噬菌体;当噬菌体编码的蛋白质不具有功能时,pIII不能被合成,进而不能产生具有侵染能力的后代噬菌体。期间,噬菌体持续受到突变,并产生新的突变体。具有侵染能力的后代噬菌体不断繁殖,而无侵染能力的后代噬菌体被洗出。最终,有利突变随着具有侵染能力的噬菌体的富集而得到富集(图5)。
基于边突变边进化的原则,李寅等提出了基于基因组复制工程的连续进化(Genome replication Engineering assisted continuous evolution,GREACE)的概念[71]。GREACE的原理是通过在微生物细胞中引入一系列经遗传修饰的DNA聚合酶校正元件,使细胞进入一种高突变率的状态,从而在筛选压力条件下加速进化过程。经过一段时间的进化后,将遗传改造的校正元件从细胞中移除,这时细胞就能维持其基因型的稳定,同时能将这种优势表型遗传下去。
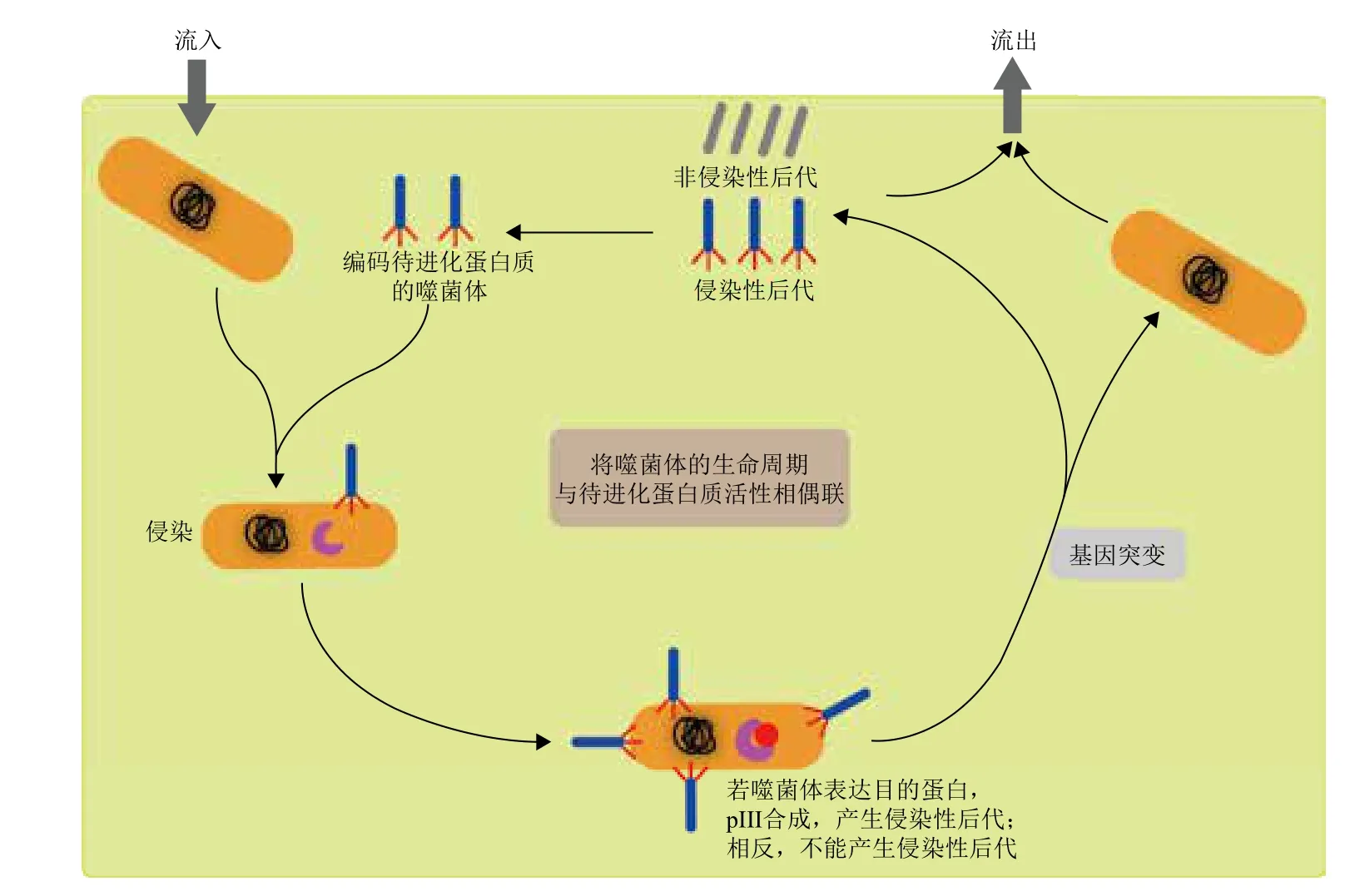
图5 PACE原理示意图
7 合成生物学的应用
最初合成生物学发展的驱动力来源于对可再生性生物燃料的追求,人们期望通过合成自然界中不存在的生物,来将大量存在的糖类和纤维素物质转化燃料。现如今合成生物学的应用已经扩展至很多领域领域,如生物燃料生产、天然产物合成、生物医药、合成新物种等。
7.1 生物燃料
石化资源的不断枯竭及其燃烧产生的污染迫使人们寻求新的可替代清洁能源,通过微生物大规模生产生物燃料就是其中之一。如生物乙醇已经在部分国家实现商业化应用,而丁醇、异丁醇、生物柴油等物理化学性状更优的生物燃料的应用也在探索中[72,73]。合成生物学使得人们可以将生物燃料的合成代谢途径异位重构至性状优良的微生物中,如Escherichia coli、Lactobacillus、Pseudomonas putida和Bacillus subtilis等。相对于传统的生产菌,这些工程菌具有很多优势,如生长迅速、营养要求简单、易于遗传改造和代谢副产物少等[74]。不过,生物燃料对微生物自身普遍具有毒害作用[75],这也是人工合成微生物在高效生产生物燃料的过程中所面临的最大挑战。突变和遗传改造等策略已经被应用于构建生物燃料耐受性菌株[76,77],且生产过程表明其耐受性明显提高。但是,人工合成微生物的生物燃料耐受性机制并不十分清楚,这成为阻碍理性设计耐受性更强微生物的最大障碍[74]。Kim等[78]和Lee等[79]针对这一基础问题展开了研究,以期为提高生物燃料耐受性提供更加理性的策略。
7.2 天然产物
来源于动物、植物、真菌和细菌的次级代谢产物的性质与化学法合成的化合物差异很大。天然产物有着长期作为药物使用的历史,但是它们往往难以得到单一的化合物,且来源有限。与高通量测序技术及质谱分析技术等相结合,合成生物学将外源的代谢途径引入易于遗传改造的模式微生物中,实现了天然产物的高效生产。其中,代表性的天然产物包括青蒿素、黄酮类化合物等。Keasling及其团队将青蒿中的紫穗槐-4,11-二烯合酶基因(ADS)导入大肠杆菌中表达,同时用酵母萜类合成途径代替大肠杆菌萜类合成途径,首次在微生物体内合成出青蒿素的第一个关键前体——紫穗槐-4,11-二烯[80]。而后他们又将ADS基因连同细胞色素P450单氧化酶基因(CYP71AV1)及其还原酶基因(CPR)同时导入酿酒酵母中表达,培育出世界上第一株生产青蒿酸的酵母工程菌[81]。国内江南大学研究团队通过人工合成、密码子优化及异源表达黄酮类化合物合成相关基因,实现了多种黄酮类化合物在大肠杆菌中的合成;通过模块化策略,使圣草酚的产量达到107 mg/L[82]。
7.3 医药领域
合成生物学在医药领域的应用主要有体内文库构建、药物发现、药物合成、药物输送和药物优化等方面[83]。(1)体内文库构建:将待筛选的化合物在细胞中表达,该文库可以随着细胞的培养而得到保存和扩增。(2)药物发现:为了充分发挥体内文库的优势,可以利用颜色信号或者可筛选的遗传表型等胞内分析方法来检测和筛选阳性克隆。不断增加的生物传感器可以作为检测工具。(3)药物合成:通过合成代谢途径的异位重构,一些重要的天然产物已经被成功合成,如萜类、聚酮化合物、非核糖体多肽、生物碱等。合成生物学还可以对这些合成途径进行精确调控。(4)药物输送:通过合成生物学方法,可以理性设计微生物使其能够作为药物输送的载体,且具有安全性。通过活体接种,这些携带药物(如益生菌、抗肿瘤药物等)的微生物侵袭特定的靶点(如肿瘤)[84],在释放前药之后自毁。(5)药物优化:蛋白质等药物的修饰可以使其具有更好药效和更长半衰期,通过在大肠杆菌中构建翻译后修饰系统,使得大肠杆菌能够生产经过特定修饰的重组药物蛋白[85]。
7.4 新物种合成
2010年,Gibson等[3]将一个1.08 Mb的Mycoplasma mycoides人工合成染色体转移到M. capricolum受体细胞中,创造出一个全新的M. mycoides细胞。该细胞完全由人工合成的基因组控制,并具有预期的表型特征,且能够自主复制。这被认为是第一个人工合成细胞。不过,该细胞只能在最理想的环境条件下存活和自主复制,而不是在真正的自然环境中。关于真正的生命形式有不同的定义,因此,对于合成生命也就有不同的看法。一般的观点认为,单细胞生命不仅仅包括其自身的生化和生理特性,还应当包括其所生存的环境条件,如主要的物质来源和能量来源、渗透压、离子强度等[86]。自下而上的细胞合成一般需要满足三方面的特征:遗传信息、代谢和自组织。这三个特征对于具有自主复制和进化功能的合成生命是必不可少的[87]。虽然目前尚不能合成同时具有上述三个特征的生命形式,但合成生物学已经分别在这三方面取得了一定的进展[86]。
8 展望
虽然合成生物学研究已经取得较大进展,且有着很好的应用前景,但依然面临诸多挑战。2010年,Roberta Kwok在Nature杂志上撰文认为,合成生物学需要面对五大难题[9,16]:(1)大部分生物元件的特征尚不明确,我们不清楚它们在不同底盘或者不同实验条件下的表型特征,甚至不知道它们能用来干什么;(2)功能回路的不可预期,即我们不能像其他现代工程技术一样,将基因零件简单的组装起来就能获得预期的功能;(3)系统过于复杂,即随着系统的不断扩大,构建和测试变得非常困难;(4)不兼容性,即外源的基因元件和底盘之间往往不兼容;(5)变异对于系统的影响,胞内分子具有波动性或噪音,这将影响细胞的功能,而这些随机变异的积累将最终摧毁回路。生命是复杂而精密的系统,从转录调控、翻译后修饰到代谢网络之间的协调,以及细胞与细胞、细胞与外界环境之间的互动,这些都需要在特定的时间和空间上发生。对于生命认知的局限性是限制人们理性设计、构建和测试合成生命的最大障碍。人们对于不同元件、装置和系统的构建和测试,使得生物合成相关的数据迅速扩增。对这些数据进行系统的整合,形成对生命系统较为完整的认知,是需要解决的问题和合成生物学的发展方向[1]。
[1] Zhang L, Chang S, Wang J. Synthetic biology:From the first synthetic cell to see its current situation and future development[J]. Chinese Science Bulletin, 2011, 56(3):229-237.
[2] Mitchell W. Natural products from synthetic biology[J]. Current Opinion in Chemical Biology, 2011, 15(4):505-515.
[3] Gibson DG, Glass JI, Lartigue C, et al. Creation of a bacterial cell controlled by a chemically synthesized genome[J]. Science, 2010, 329(5987):52-56.
[4] Bailey JE. Toward a science of metabolic engineering[J]. Science, 1991, 252(5013):1668-1675.
[5] Stephanopoulos G, Vallino JJ. Network rigidity and metabolic engineering in metabolite overproduction[J]. Science, 1991, 252(5013):1675-1681.
[6] Xie LZ, Wang DIC. High cell density and high monoclonal antibody production through medium design and rational control in a bioreactor[J]. Biotechnology and Bioengineering, 1996, 51(6):725-729.
[7] Hamilton SR, Bobrowicz P, Bobrowicz B, et al. Production of complex human glycoproteins in yeast[J]. Science, 2003, 301(5637):1244-1246.
[8] Stephanopoulos G. Challenges in engineering microbes for biofuels production[J]. Science, 2007, 315(5813):801-804.
[9] Kitney R, Freemont P. Synthetic biology-the state of play[J]. Febs Letters, 2012, 586(15):2029-2036.
[10] Elowitz MB, Leibler S. A synthetic oscillatory network of transcriptional regulators[J]. Nature, 2002, 403(6767):335-338.
[11] Gardner TS, Cantor CR, Collins JJ. Construction of a genetic toggle switch in Escherichia coli[J]. Nature, 2000, 403(6767):339-342.
[12] Smith HO, Hutchison CA, Pfannkoch C, Venter JC. Generating a synthetic genome by whole genome assembly:phi X174 bacteriophage from synthetic oligonucleotides[J]. Proceedings of the National Academy of Sciences of the United States of America, 2003, 100(26):15440-15445.
[13] Good MC, Zalatan JG, Lim WA. Scaffold proteins:Hubs for controlling the flow of cellular information[J]. Science, 2011, 332(6030):680-686.
[14] Weber W, Fussenegger M. The impact of synthetic biology on drug discovery[J]. Drug Discovery Today, 2009, 14(19-20):956-963.
[15] Rebatchouk D, Daraselia N, Narita JO. NOMAD:A versatile strategy for in vitro DNA manipulation applied to promoter analysis and vector design[J]. Proceedings of the National Academy of Sciences of the United States of America, 1996, 93(20):10891-10896.
[16] Kwok R. Five hard truths for synthetic biology[J]. Nature, 2010, 463(7279):288-290.
[17] Hodgman CE, Jewett MC. Cell-free synthetic biology:Thinking outside the cell[J]. Metabolic Engineering, 2012, 14(3):261-269.
[18] Smith MT, Wilding KM, Hunt JM, et al. The emerging age of cellfree synthetic biology[J]. Febs Letters, 2014, 588(17):2755-2761.
[19] Swartz JR. Transforming biochemical engineering with cell-free biology[J]. Aiche Journal, 2012, 58(1):5-13.
[20] Harris DC, Jewett MC. Cell-free biology:Exploiting the interface between synthetic biology and synthetic chemistry[J]. Current Opinion in Biotechnology, 2012, 23(5):672-678.
[21] Forster AC, Church GM. Synthetic biology projects in vitro[J]. Genome Research, 2007, 17(1):1-6.
[22] Lopez-Gallego F, Schmidt-Dannert C. Multi-enzymatic synthesis[J]. Current Opinion in Chemical Biology, 2010, 14(2):174-183.
[23] Zawada JF, Yin G, Steiner AR, et al. Microscale to manufacturing scale-up of cell-free cytokine production-A new approach for shortening protein production development timelines[J]. Biotechnology and Bioengineering, 2011, 108(7):1570-1578.
[24] Kanter G, Yang J, Voloshin A, et al. Cell-free production of scFv fusion proteins:an efficient approach for personalized lymphoma vaccines[J]. Blood, 2007, 109(8):3393-3399.
[25] Bundy BC, Franciszkowicz MJ, Swartz JR. Escherichia coli-based cell-free synthesis of virus-like particles[J]. Biotechnology and Bioengineering, 2008, 100(1):28-37.
[26] Boyer ME, Stapleton JA, Kuchenreuther JM, et al. Cell-free synthesis and maturation of[FeFe]hydrogenases[J].Biotechnology and Bioengineering, 2008, 99(1):59-67.
[27] Bujara M, Schuemperli M, Billerbeek S, et al. Exploiting cellfree systems:Implementation and debugging of a system of biotransformations[J]. Biotechnology and Bioengineering, 2010, 106(3):376-389.
[28] Zhang YHP. Production of biocommodities and bioelectricity by cell-free synthetic enzymatic pathway biotransformations:Challenges and opportunities[J]. Biotechnology and Bioengineering, 2010, 105(4):663-677.
[29] Wang Y, Huang W, Sathitsuksanoh N, et al. Biohydrogenation from biomass sugar mediated by in vitro synthetic enzymatic pathways[J]. Chemistry & Biology, 2011, 18(3):372-380.
[30] Sun ZZ, Yeung E, Hayes CA, et al. Linear DNA for rapid prototyping of synthetic biological circuits in an Escherichia coli based TX-TL cell-free system[J]. Acs Synthetic Biology, 2014, 3(6):387-397.
[31] Singh-Blom A, Hughes RA, Ellington AR. Residue-specific incorpora-tion of unnatural amino acids into proteins in vitro and in vivo[M/OL]//Samuelson JC. Enzyme Engineering, Methods in Molecular Biology. Humana Press, 2013, 978: 93-114.
[32] Liu CC, Schultz PG. Adding new chemistries to the genetic code[J]. Annual Review of Biochemistry, 2010, 79(1):413-444.
[33] Zimmerman ES, Heibeck TH, Gill A, et al. Production of sitespecific antibody-drug conjugates using optimized non-natural amino acids in a cell-free expression system[J]. Bioconjugate Chemistry, 2014, 25(2):351-361.
[34] Smith MT, Wu JC, Varner CT, Bundy BC. Enhanced protein stability through minimally invasive, direct, covalent, and sitespecific immobilization[J]. Biotechnology Progress, 2013, 29(1):247-254.
[35] Smith MT, Hawes AK, Bundy BC. Reengineering viruses and viruslike particles through chemical functionalization strategies[J]. Current Opinion in Biotechnology, 2013, 24(4):620-626.
[36] Loscha KV, Herlt AJ, Qi R, et al. Multiple-site labeling of proteins with unnatural amino acids[J]. Angewandte Chemie-International Edition, 2013, 51(9):2243-2246.
[37] Yang Z, Hutter D, Sheng P, et al. Artificially expanded genetic information system:a new base pair with an alternative hydrogen bonding pattern[J]. Nucleic Acids Research, 2006, 34(21):6095-6101.
[38] Yang Z, Chen F, Alvarado JB, Benner SA. Amplification, mutation, and sequencing of a six-letter synthetic genetic system[J]. Journal of the American Chemical Society, 2011, 133(38):15105-15112.
[39] Acevedo-Rocha CG, Fang G, Schmidt M, et al. From essential to persistent genes:a functional approach to constructing synthetic life[J]. Trends in Genetics, 2013, 29(5):273-279.
[40] Juhas M, Eberl L, Glass JI. Essence of life:essential genes of minimal genomes[J]. Trends in Cell Biology, 2011, 21(10):562-568.
[41] Lagesen K, Ussery DW, Wassenaar TM. Genome update:the 1000th genome - a cautionary tale[J]. Microbiology-Sgm, 2010, 156:603-608.
[42] McCutcheon JP. The bacterial essence of tiny symbiont genomes[J]. Current Opinion in Microbiology, 2010, 13(1):73-78.
[43] Liu MF, Cescau S, Mechold U, et al. Identification of a novel nanoRNase in Bartonella[J]. Microbiology-Sgm, 2012, 158:886-895.
[44] Ouzounis CA, Kunin V, Darzentas N, Goldovsky L. A minimal estimate for the gene content of the last universal common ancestor - exobiology from a terrestrial perspective[J]. Research in Microbiology, 2006, 157(1):57-68.
[45] Kim KM, Caetano-Anolles G. The evolutionary history of protein fold families and proteomes confirms that the archaeal ancestor is more ancient than the ancestors of other superkingdoms[J]. Bmc Evolutionary Biology, 2012, 12:13.
[46] Lukjancenko O, Wassenaar TM, Ussery DW. Comparison of 61 sequenced Escherichia coli genomes[J]. Microbial Ecology, 2010, 60(4):708-720.
[47] Anthony JR, Anthony LC, Nowroozi F, et al. Optimization of the mevalonate-based isoprenoid biosynthetic pathway in Escherichia coli for production of the anti-malarial drug precursor amorpha-4, 11-diene[J]. Metabolic Engineering, 2009, 11(1):13-19.
[48] Yadav VG, De Mey M, Lim CG, et al. The future of metabolic engineering and synthetic biology:Towards a systematic practice[J]. Metabolic Engineering, 2012, 14(3):233-241.
[49] Perez-Pinera P, Ousterout DG, Gersbach CA. Advances in targeted genome editing[J]. Current Opinion in Chemical Biology, 2012, 16(3-4):268-277.
[50] Kim S, Lee MJ, Kim H, et al. Preassembled zinc-finger arrays for rapid construction of ZFNs[J]. Nature Methods, 2011, 8(1):7.
[51] Bhakta MS, Henry IM, Ousterout DG, et al. Highly active zincfinger nucleases by extended modular assembly[J]. Genome Research, 2013, 23(3):530-538.
[52] Miller JC, Tan S, Qiao G, et al. A TALE nuclease architecture for efficient genome editing[J]. Nature Biotechnology, 2011, 29(2):143-148.
[53] Gaj T, Gersbach CA, Barbas CF III. ZFN, TALEN, and CRISPR/ Cas-based methods for genome engineering[J]. Trends in Biotechnology, 2013, 31(7):397-405.
[54] Bhaya D, Davison M, Barrangou R. CRISPR-Cas systems in bacteria and archaea:versatile small RNAs for adaptive defense and regulation[J]. Annual Review of Genetics, 2011, 45:273-297.
[55] Gasiunas G, Siksnys V. RNA-dependent DNA endonuclease Cas9 of the CRISPR system:Holy Grail of genome editing?[J]. Trends in Microbiology, 2013, 21(11):562-567.
[56] Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337(6096):816-821.
[57] Ran FA, Hsu PD, Wright J, et al. Genome engineering using the CRISPR-Cas9 system[J]. Nature Protocols, 2013, 8(11):2281-2308.
[58] Jiang W, Bikard D, Cox D, et al. RNA-guided editing of bacterial genomes using CRISPR-Cas systems[J]. Nature Biotechnology, 2013, 31(3):233-239.
[59] Yu Z, Ren M, Wang Z, et al. Highly Efficient genome modifications mediated by CRISPR/Cas9 in Drosophila[J]. Genetics, 2013, 195(1):289-291.
[60] Gilbert LA, Larson MH, Morsut L, et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes[J]. Cell, 2013, 154(2):442-451.
[61] Cobb RE, Sun N, Zhao H. Directed evolution as a powerful synthetic biology tool[J]. Methods, 2013, 60(1):81-90.
[62] Cobb RE, Si T, Zhao H. Directed evolution:an evolving and enabling synthetic biology tool[J]. Current Opinion in Chemical Biology, 2012, 16(3-4):285-291.
[63] Zha W, Rubin-Pitel SB, Zhao H. Exploiting genetic diversity by directed evolution:molecular breeding of type III polyketide synthases improves productivity[J]. Molecular BioSystems, 2008, 4(3):246-248.
[64] Atsumi S, Liao JC. Directed evolution of Methanococcus jannaschii citramalate synthase for biosynthesis of 1-propanol and 1-butanol by Escherichia coli[J]. Applied and Environmental Microbiology, 2008, 74(24):7802-7808.
[65] Filipovska A, Rackham O. Building a parallel metabolism within the cell[J]. Acs Chemical Biology, 2008, 3(1):51-63.
[66] Liu CC, Mack AV, Brustad EM, et al. Evolution of proteins with genetically encoded “Chemical warheads”[J]. Journal of the American Chemical Society, 2009, 131(28):9616-9617.
[67] Pfleger BF, Pitera DJ, Smolke CD, Keasling JD. Combinatorial engineering of intergenic regions in operons tunes expression of multiple genes[J]. Nat Biotech, 2006, 24(8):1027-1032.
[68] Chen F, Gaucher EA, Leal NA, et al. Reconstructed evolutionary adaptive paths give polymerases accepting reversible terminators for sequencing and SNP detection[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(5):1948-1953.
[69] Wang Q, Wu H, Wang A, et al. Prospecting metagenomic enzyme subfamily genes for DNA family shuffling by a novel PCR-based approach[J]. Journal of Biological Chemistry, 2010, 285(53):41509-41516.
[70] Dickinson BC, Packer MS, Badran AH, Liu DR. A system for the continuous directed evolution of proteases rapidly reveals drugresistance mutations[J]. Nat Commun, 2014, 5:5352.
[71] Luan G, Cai Z, Li Y, Ma Y. Genome replication engineering assisted continuous evolution(GREACE)to improve microbial tolerance for biofuels production[J]. Biotechnology for Biofuels, 2013, 6(1):137.
[72] Connor MR, Atsumi S. Synthetic biology guides biofuel production[J]. Journal of Biomedicine and Biotechnology, 2010, doi:10. 1155/2010/541698.
[73] Wang B, Wang J, Zhang W, Meldrum DR. Application of synthetic biology in cyanobacteria and algae[J]. Frontiers in Microbiology, 2012, 3:344.
[74] Jin H, Chen L, Wang J, Zhang W. Engineering biofuel tolerance in non-native producing microorganisms[J]. Biotechnology Advances, 2014, 32(2):541-548.
[75] Ramos JL, Duque E, Gallegos MT, et al. Mechanisms of solventtolerance in gram-negative bacteria[J]. Annual Review of Microbiology, 2002, 56:743-768.
[76] Lo TM, Teo WS, Ling H, et al. Microbial engineering strategies to improve cell viability for biochemical production[J]. Biotechnology Advances, 2013, 31(6):903-914.
[77] Shao X, Raman B, Zhu M, et al. Mutant selection and phenotypic and genetic characterization of ethanol-tolerant strains of Clostridium thermocellum[J]. Applied Microbiology and Biotechnology, 2011, 92(3):641-652.
[78] Kim HJ, Turner TL, Jin YS. Combinatorial genetic perturbation to refine metabolic circuits for producing biofuels and biochemicals[J]. Biotechnology Advances, 2013, 31(6):976-985.
[79] Lee SJ, Lee SJ, Lee DW. Design and development of synthetic microbial platform cells for bioenergy[J]. Frontiers in Microbiology, 2013, 4:92.
[80] Martin VJJ, Pitera DJ, Withers ST, et al. Engineering a mevalonate pathway in Escherichia coli for production of terpenoids[J]. Nature Biotechnology, 2013, 21(7):796-802.
[81] Ro DK, Paradise EM, Ouellet M, et al. Production of the antimalarial drug precursor artemisinic acid in engineered yeast[J]. Nature, 2006, 440(7086):940-943.
[82] Zhu S, Wu J, Du G, et al. Efficient synthesis of eriodictyol from L-tyrosine in Escherichia coli[J]. Applied and Environmental Microbiology, 2014, 80(10):3072-3080.
[83] Neumann H, Neumann-Staubitz P. Synthetic biology approaches in drug discovery and pharmaceutical biotechnology[J]. Applied Microbiology and Biotechnology, 2010, 87(1):75-86.
[84] Nguyen VH, Kim HS, Ha JM, et al. Genetically engineered Salmonella typhimurium as an imageable therapeutic probe for cancer[J]. Cancer Research, 2010, 70(1):18-23.
[85] Neumann H, Hancock SM, Buning R, et al. A Method for genetically installing site-specific acetylation in recombinant histones defines the effects of H3 K56 acetylation[J]. Molecular Cell, 2009, 36(1):153-163.
[86] Caschera F, Noireaux V. Integration of biological parts toward the synthesis of a minimal cell[J]. Current Opinion in Chemical Biology, 2014, 22:85-91.
[87] Noireaux V, Maeda YT, Libchaber A. Development of an artificial cell, from self-organization to computation and selfreproduction[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(9):3473-3480.
Advances in Synthetic Biology
Lü Yongkun Du Guocheng Chen Jian Zhou Jingwen
(Jiangnan University,Wuxi 214122)
Synthetic biology is an applied discipline that introduces engineering into biology. The aim of synthetic biology is to standardize and modularize biological parts. It can also be applied in the basic researches,such as the research of life origin. This review gives a detailed introduction to the synthetic biology,especially the new methods and applications.
synthetic biology;biological part;cell-free synthetic biology;minimal genome;directed evolution;genome editing
10.13560/j.cnki.biotech.bull.1985.2015.03.017
2015-03-30
国家自然科学基金项目(31370130),江南大学自主科研计划重点项目(JUSRP51307A)
吕永坤,男,博士研究生,研究方向:合成生物学;E-mail:lykun2012@sina.com
周景文,男,博士,教授,研究方向:合成生物学、代谢工程;E-mail:zhoujw1982@jiangnan.edu.cn
