基于DNA自组装过程的纳米结构研究
2015-07-19俞洋李江张钊樊春海
俞洋李江张钊樊春海
(1.上海科技管理干部学院电子信息系,上海 201800;2.中国科学院上海应用物理研究所,上海 201800;3. 丹麦奥胡斯大学化学系,奥胡斯 8000)
基于DNA自组装过程的纳米结构研究
俞洋1李江2张钊3樊春海2
(1.上海科技管理干部学院电子信息系,上海 201800;2.中国科学院上海应用物理研究所,上海 201800;3. 丹麦奥胡斯大学化学系,奥胡斯 8000)
基于DNA自组装的纳米结构在近年来取得了巨大的发展。回顾了DNA纳米结构的原理和发展历程,介绍了DNA纳米结构的特点和优势,对DNA纳米结构在生物检测、纳米反应器、可控排布、纳米机器人和药物递送领域的新进展和应用进行了综述,并对DNA纳米技术的未来进行了展望。
DNA折纸;纳米机器;纳米药物
DNA作为遗传物质是生命体得以延续与传承的重要组成部分,有趣的是,随着纳米技术的发展,研究者也逐渐认识到DNA也是一种典型的纳米材料。它的双螺旋直径约为2 nm,螺距为3.4-3.6 nm,而每个碱基的长度约为0.34 nm[1]。双链DNA是一个刚性的高分子材料,其回转长度达50 nm;而单链DNA则具有较大的灵活性。特别是DNA具有出色的可编码性,可以根据碱基配对原则进行比较精确的设计。这些特征使我们可以构造出变化多样的纳米结构[2]。纽约大学的Nadrian Seeman教授在20世纪80-90年代就提出可以用DNA分子组装出纳米图形,并引领了整个DNA纳米技术领域的发展[3]。
DNA纳米技术在近20年来得到了迅猛的发展[4,5]。构造自组装DNA纳米结构有两种主要方法,包括Seeman课题组发展的瓦块(tile)自组装[6,7]和Paul Rothemund博士发明的DNA折纸术(DNA origami)[8]。本文将主要回顾DNA自组装纳米结构的设计、制造及应用。
1 DNA自组装纳米技术简介
构造自组装DNA纳米结构主要有两大类方法。1998年,Seeman课题组发展了瓦块(tile)自组装,使我们可以在原子力显微镜(Atomic force microscope,AFM)下看到了DNA构造的二维网格结构[6,7]。经过多年发展,利用瓦块自组装可以构造出丰富多彩的纳米结构,包括一维线性排列[9]、二维平面网格、可寻址阵列、纳米纤维、三维多面体乃至三维晶体[10]等。但是,这种类型的结构也存在一些不足。例如,它们的组装依赖多条DNA链间的精确互补配对,对各链的化学计量比和热力学性质要求较严格,组装过程相对繁琐费时;而且,由于受到瓦块单元的限制,它们的最终结构的复杂程度也是很有限的。
2006年,加州理工学院的Paul Rothemund博士发明了DNA折纸术(DNA origami),这一里程碑式的技术使制作复杂DNA自组装结构的能力得到极大提升[8]。DNA折纸术本质上是一种成核自组装(Nucleation assembly),这不同于瓦块自组装的层次自组装(Hierarchical assembly)。其整个组装过程是围绕若干成核点(或称成核链)一次进行而不分步的。DNA成核自组装的思想由来已久,然而当Rothemund基于这一思想,提出、设计、并实现了DNA折纸术[8],研究者马上发现DNA折纸术生成图形的复杂度较瓦块自组装至少提高了一个数量级,而且其编码便捷、实验简单、反应快速,因而受到了极大的关注。所谓“DNA折纸术”,就是把一根长的DNA单链像折一张纸一样,绑定并铺展成某个特定形状。事实上,“折纸”并非很恰切的比喻,这种技术更为类似织毛衣的过程(图1-A)。Rothemund使用的这根“毛线”(即成核长链)是M13mp18噬菌体的单链DNA,有7 249个碱基。Rothemund将这条成核链称为骨架链(Scaffold),而用来绑定骨架链的互补短链则称为订书钉链(Staple)。Rothemund设计的订书钉链大多数为32个碱基,以8个碱基为一个单元。整个骨架链为200多条订书钉链完全互补为双链,从而凝固成刚性的结构。DNA折纸术产生的图形复杂度远远超过了以前的绝大多数DNA自组装结构。其像素点数目较之前的瓦块自组装至少高10倍;单个折纸术图形分子量为4.7 MDa,超越了自然界最复杂的自组装机器——真核生物核糖体(4.2 MDa);而且折纸术可以一次产生500亿个拷贝的图形。
Rothemund在这篇里程碑论文中展示的图形均为对称结构(图1-B)。随后,上海交通大学和中国科学院上海应用物理研究所的研究人员用DNA折纸术构造了第一个非对称结构,即中国地图模拟图[11](图1-C),丹麦奥胡斯大学则设计出了尾部可以活动的海豚形状(奥胡斯大学的校徽,图1-D)[12]。2009年,多个课题组接连报道了三维DNA折纸术方面进展。Andersen等折出了一个六面体的空心盒子,而且其中一个面还能通过链置换反应人为控制开闭(图1-E)[13];Ke等[14]也用DNA折纸术得到了一个立体结构——空心四面体(图1-F)。Kuzuya等[15]根据前者得到了一个空心立方体,而且通过选择性加入跨面订书钉链,可以控制立方体的整体构型。哈佛大学的William Shih小组在提出了“蜂窝褶状模型”(Honeycomb-pleated),构造了诸多三维图形,包括块状巨石(多面体)、方形螺帽(有孔三维)、桥式结构(细线连接)、细颈瓶(同向嵌套结构)、插槽交叉(垂直向嵌套结构)、堆积交叉(垂直向堆积结构)等(图1-G)[16]。他们通过控制交叉处的间距,进一步制造出整体扭曲或弯折的三维图形[17]。颜灏的研究组[18]更是展示了精美的曲面三维纳米花瓶结构(图1-H)。
2012年,哈佛大学的殷鹏与Shih教授的研究组发表了一种类似于瓦块组装技术的DNA“积木”组装技术[19]。这种技术以DNA单链为“积木”,每个积木上不同区域的序列可以与另外的“积木”上的对应序列互补杂交。并且,由于DNA螺旋转角的确定性,这样的单链积木在组装之后的构型也是确定的。这样,在经过精心的序列设计之后,研究者们可以像组装乐高积木一样将这些DNA单链组装为各种三维几何体,包括三维的英文字母等有着复杂表面的立体结构,展现了这一技术在几何构型上的几乎无限的可能。这一方法不再需要多链组装的结构单元,每一条DNA单链即是一个结构单元,最终结构可由一步退火完成;同时,这种结构也是一种“去中心化”的结构,不再受到DNA折纸术中骨架链的长度和序列限制,在结构设计上更为自由。
2 DNA纳米技术的应用
DNA纳米技术不仅可以产生漂亮的图形,而且还在很多领域具有重要应用价值。例如,Shih课题组[20]将DNA折纸术形成的纳米管作为介质,可以在用核磁共振(NMR)测定膜蛋白结构中产生积极作用。Steinhauer等[21]基于荧光标记的DNA折纸图形发展出用于校准超分辨率显微镜的工具尺。Rothemund与IBM公司合作将DNA折纸术与光刻技术结合起来,可以将折纸图形定向固定在宏观基底上,实现了在宏观上对电子器件的纳米级精确排布[22,23]。DNA折纸术最大的优点是所有位点都可以在其产生的结构上直接寻址,因而可以作为纳米尺度可控排布的理想模板。以下将重点介绍基于这种可控排布的若干应用。
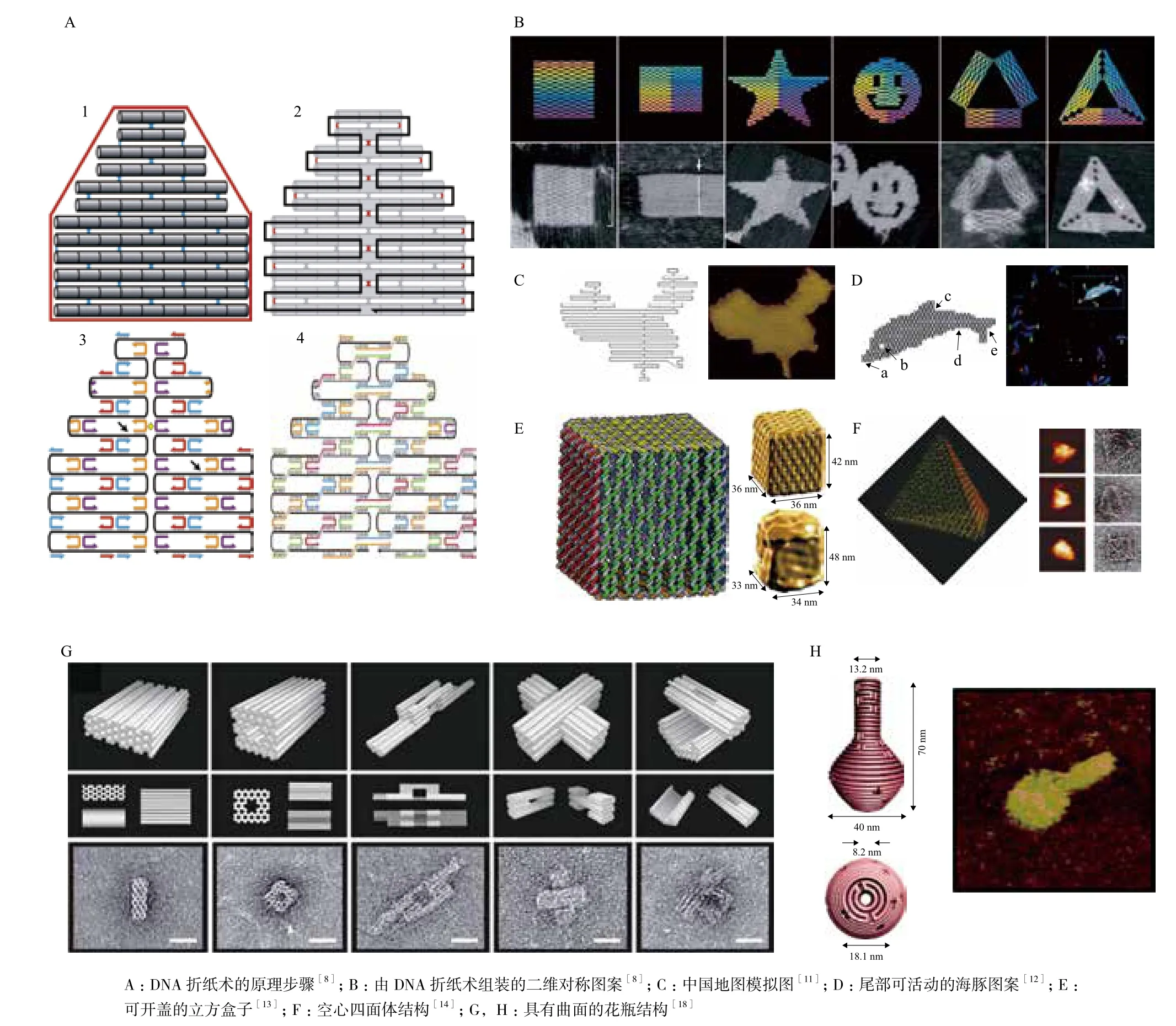
图1 DNA折纸术的研究进展
2.1 生物分子检测
生物分子的检测对于疾病早期诊断、食品安全乃至反恐方面都有着重要的意义。然而,常规方法难以实现在纳米尺度的生物检测。DNA折纸术则提供了一种可以在纳米尺度操控生物分子识别的新方法。2008年,Ke等[24]开发了DNA折纸芯片,通过V字形探针的设计方式,能够在AFM下直接检测到核酸杂交结果,从而实现对小RNA的灵敏检测(图2-A)[25]。Zhang等[26]以DNA中国地图模拟图为基底,制备了新型的无标记折纸芯片。这种芯片由于本身是非对称图形,所以无需添加“索引”。
核酸适配体(Aptamer)是一类利用指数富集配基系统进化(Systematic evolution of ligands by exponential enrichment,SELEX)技术,从随机寡核苷酸文库中筛选获得的能够与靶分子特异结合的短单链寡核苷酸配体。由于aptamer本身也是核酸构成的,所以在DNA自组装结构中应用非常方便。颜灏课题组用核酸适配体实现了多种蛋白在同一个DNA折纸模板上特异性可编码排布[27]。Rinker等[28]则通过控制核酸适配体的间距离,实现了高亲和力的多价结合(Multivalent binding)。Lin等[29]把DNA基底上的核酸适配体用于检测,可以通过荧光信号检测溶液中的特异蛋白。之后他们通过竞争机制造成DNA结构荧光颜色的变化,并用调色的办法来形成多种颜色作为条形码,从而一次检验多种蛋白[30]。他们还尝试用杂交链式反应(Hybridization chain reaction,HCR)对信号进行放大,成功使检测灵敏度提高了两个数量级[31]。2009年,樊春海研究组[32]构建了载带有针对不同靶标分子的核酸适配体的DNA四面体“逻辑门”探针(图2-B),当检测样本中出现一种或多种靶标分子时,四面体的构象会因为核酸适配体的构型变化而发生相应变化,从而产生不同的荧光共振能量转移(Fluorescent resonance energr transfer,FRET)效应,实现相当于与门、或门、与非门等逻辑门的逻辑判定。并且,该逻辑门检测策略还可用于对细胞内的靶标分子的检测。
2.2 DNA纳米机器人
发展可以在纳米尺度运动的机器人是纳米技术的目标之一。DNA折纸术同样提供了一个强大的平台用于纳米机器人的研究。自20世纪90年代以来,已有多种动态DNA纳米机器被相继报道[33,34]。它们还可采用金属离子[35]、质子[36,37]、DNA链或酶反应[38-40]驱动。例如,Gu等[41]把两种DNA纳米结构元件插入带两个卡槽的DNA折纸图形中,于是两个元件就有4种可能的状态组合。他们设计出在每种组合下两个器件都能共同捕获一个特异的识别分子,从而用AFM确认了这个动态系统的正常工作(图2-C)。之后,Gu等[42]在一个阶梯状折纸基底上依次放3个卡槽,并设计了一个三臂四足的机器人。机器人在基底上运动,行进过程中通过判断3个卡槽的状态来决定是否接纳对应的货物。他们用3种纳米金粒子作为货物,并在透视电子显微镜(Transmission electron microscope,TEM)下观察纳米金的排布形状,结果显示能特异的得到8种货物组合的任意一种(图2-D)。这样就形成了一个迷你的纳米组装生产线。DNA纳米机器人中的另一个经典工作是一种在折纸图形表面行走的分子蜘蛛[43]。该“蜘蛛”的模型是由Pei等[44]开发出来,它的“身子”是一个四价链霉亲和素,三只脚的序列包含一段核酶,核酶对杂交基底的切割是“蜘蛛”行进的关键。研究者在折纸术上构造了一系列连续的路径,然后用AFM来捕捉“蜘蛛”的移动,这可以说是一个真正意义上的分子机器人[45]。
2.3 DNA纳米反应器
化学反应的速率在很大程度上取决于溶液中分子的碰撞几率。因此,如果能够将两个分子或基团的空间位置拉近,形成一种反应中心或活性域,那么就可以极大提高化学反应的效率。用DNA纳米结构作为模板来组织引导化学反应(DNA-directed/ templated reaction),从而构建纳米生物反应器,是一个新兴的研究方向[46-48]。
丹麦奥胡斯大学CDNA中心的Gothelf组开发了一种称为线型DNA模块(linear/tripoidal oligonucleotide functionalized modules,LOM) 或 三 岔 型NDA模 块(tripoidal olgonucleotide functinalized module,TOM)。LOM的核心是一段3苯环串联[49](也可以5苯环[50])的有机结构,两端两个苯环上面各有一个半salen的结构域,此外还各连有一条DNA单链。如果把每个LOM看做一个构件,那么构件间就通过两侧的DNA链互补连接,形成一维线性产物。由于设计的互补链修饰位置是相反的,所以在该产物中,两个半salen结构域恰好“顶在一起”,此时当加入锰盐和乙二胺,将发生metal-salen反应,使得瓦块间不仅依靠DNA杂交相连,而且苯环部分也“粘”在了一起。研究发现,此时即使把DNA链切断(通过引入二硫键),线性结构依然牢固[51]。这样,整个过程就相当于借助DNA的配对来诱使其他有机结构的聚合,而且由于苯环骨架和metalsalen配合物都是共面的,这为将来制造纳米线路提供了新的思路[52]。TOM与LOM原理相同,只不过TOM是个三臂结构,能够形成更复杂的二维组装产物[53]。另外,核心的metal-salen反应也可以替换为酸性环境的还原胺化反应(Reductive amination,产物为Tetrahydrosalen),产物甚至更稳定[54]。除了LOM/TOM模块,该小组最近还用DNA双链[55]或4螺旋束tile(4HB)[56]来控制有机棒(Organic rods)的耦合,其距离已精确到双螺旋内的两条单链。
以色列希伯来大学的Willner课题组在金电极上伸出单链DNA作为模板,然后在中间和末端的两个位置分别结合连有不同反应基团的两条DNA。不同的基团排列顺序、不同的基团与电极间距离,导致了电流强度的改变,据此检测和验证结果。参与的反应包括葡萄糖氧化酶(glucose oxidase,GOx)-二茂铁、GOx-MP11(微过氧化物酶-11)等生物电催化反应[57],CdS纳米颗粒-relay(类似“继电器”作用的分子)、光敏剂(Ru2+)-relay等光电化学反应[58],荧光基团与QD的FRET反应[59]等。有的还通过加入酶等实现了可逆反应[59]。他们的另一个工作是用DNA自组装得到特定的结构,然后在相邻孔洞内修饰不同辅酶(实际使用两套辅酶对,分别是GOx-HRP和GDH-NAD+)。由于辅酶对离得很近,所以能够形成级联反应,反应效率比没有DNA模板时高出很多,说明DNA网格确实起着拉近距离的作用(图2-E)[60]。他们又把这套体系移植到DNA纳米线上,纳米线是先由4条DNA组成圆形结构,然后借助可卡因分子组装而成。相邻tile上分别修饰GOx和HRP,其反应效率比没有DNA模板或可卡因时高6倍以上[61]。
在Gothelf与樊春海研究小组的合作研究中[62],他们则用DNA折纸结构作为模板固定GOx与HRP。这一结构不但能精确控制两种酶分子之间的距离,还能在展开的二维平面与卷曲的三维管状结构之间变换(图2-F)。研究结果表明,这样的结构变化能明显影响到双酶系统反应的效率。当酶分子被卷入DNA纳米管结构内部后,反应效率明显增强。研究者们认为这是对自然界细胞内部受限空间内发生的生化反应的一种模拟,在这样的限域空间内,酶分子与反应各组分的局域浓度较高,因此,尽管其在宏观尺度上的平均浓度很低,但也能获得较高的反应效率。根据这样的机制和策略,人们可以更好地效法自然界设计和构建高效的纳米生物反应器。
2.4 无机纳米材料可控排布
DNA纳米结构具有精确可控的优势,然而DNA本身缺乏明显的光电效应,因而难以在光学或电子学领域发挥作用。幸运的是,DNA纳米结构可以引导具有光电效应的无机纳米粒子的精确排布,从而制备出具有可控光电性质的纳米结构。
2.4.1 纳米金粒子 纳米金粒子(Au nanoparticle)在水中形成的分散系俗称胶体金,金颗粒可与氨基发生非共价的静电吸附而牢固结合,与巯基之间形成很强的Au-S键,后者也是最常用的纳米金与DNA的结合方式。2004年,Seeman小组率先用杂交法实现了纳米金的条纹排列[63],之后他们又完成了两种大小纳米金的有序交叉排列[64]。利用类似的方法,Hung等[23]和Gerdon等[65]想在“已特异吸附在固相表面的折纸图形”上进一步修饰纳米金时,Ding等[66]和Pal等[67]在DNA折纸三角形上线性排列纳米金。Sharma等[68]则把硫醇修饰的DNA改为用酯化的硫辛酸修饰,从而与金的结合由一个硫键变为了两个硫键,进而显著提高了这种方法的产率。他们在折纸术矩形的两个位置特异的连接了两个金球,效率高达90%以上。
除了可以在DNA模板上排布金球,还能实现在DNA上生长金球。Stearns等利用无机结合肽(Inorganic binding peptides,IBPs)对金离子的成核作用,先用杂交法把IBP连接到纳米管上,再加入AuCl4使成核,最终也得到了纳米金颗粒的一维排布[69]。纳米金不但能被DNA排布,有时候它还会反作用于自组装图形。2009年,Sharma等[70]在DX lattice中修饰纳米金,由于所使用的金球直径大于DNA结构上金球连接位点的平面距离,于是金球的斥力使得平面array卷曲成纳米管状,且通过控制金球大小可以控制得到的纳米管构型(图2-G)。
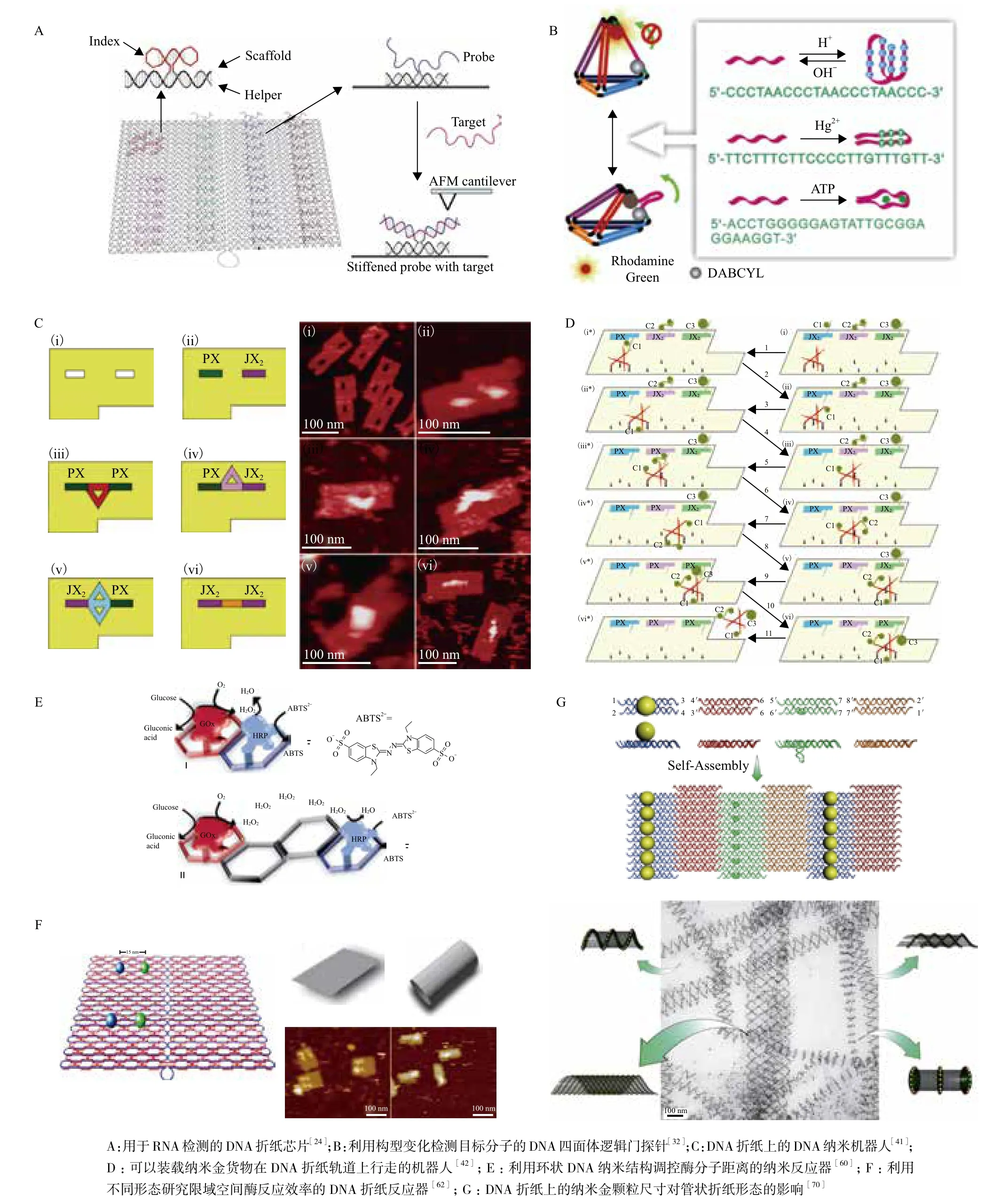
图2 DNA纳米结构的代表性应用
2.4.2 碳纳米管 碳纳米管(Carbon nanotubes)是由一层或多层石墨按照一定方式卷曲而成的具有管状结构的纳米材料。自从1991年日本科学家Sumio Iijima发现碳纳米管以来[71],碳纳米管以其优异的热学、力学以及光电特性[72]受到了多个领域研究者的广泛关注[73,74],并实现了多种应用[75,76]。对于特定的碳纳米管,控制其属性甚至功能化的关键,在于对其进行特定的组装与排布。用DNA来识别、筛选、组装、排列碳纳米管,需要解决把DNA与CNT连接起来的问题。2002年,Dwyer等[77]发明了在DNA末端修饰氨基,再与碳纳米管末端连接的方法[78]。他还据此提出了用DNA排布碳纳米管的通用思路、设计和算法。碳纳米管除了末端可以修饰DNA,其侧壁也是很好的结合部位。例如,Han和Deng等[79]在碳纳米管上缠绕了修饰有硫醇的DNA,然后加入不同大小的纳米金,使金一维排列在碳纳米管上。
2009年,Maune等[80]在一个连有tile长带的折纸矩形上特异交叉排布了两根碳纳米管。他们先用保护链把碳纳米管上用于杂交的序列局部封闭,然后与带有探针的矩形DNA折纸混合,可以使碳纳米管结合在折纸基底上。他们发现用这种方法可以产生具有场效应晶体管(FET)特性的器件。这种用DNA排布碳纳米管来制作纳米电路的方法尽管目前效率不高,但其精确性和并行性却是传统光刻技术所不能比拟的。
2.5 药物递送
众所周知,在当今人类面对癌症等多种重大疑难疾病的挑战时,传统的药物存在着诸多亟需克服的缺点,而新药的开发又代价高昂。为了改善这些既有药物的性能,发展合适的药物递送系统就成为了当前生物医药领域的迫切需求和研究热点。近几十年来,随着纳米技术的兴起,多种纳米材料都被用于药物递送的研究,如阳离子脂质体、聚合物、各种金属纳米颗粒、碳纳米管等。但是,这类载体通常存在不容忽视的安全问题,如细胞毒性和脏器累积毒性等。而DNA纳米结构作为近年来兴起的一类新型的纳米材料,在生物医学领域也有着其它材料不可比拟的优势:(1)DNA本身作为生命的遗传物质材料普遍存在于生物体内,而外源DNA在生物体内则可被降解利用,因此几乎没有生物相容性、细胞毒性和累积毒性的问题。(2)DNA分子合成和化学修饰技术已经较为成熟和商业化,便于我们在DNA纳米结构上进行各种功能化修饰,从而拓展DNA纳米结构的功能,并且能在同一个结构上实现多种功能的组合。作为药物载体,这样的特点有利于实现药物靶向、多药物协同载运和药物控制释放等功能。(3)更重要的是,由于DNA基于碱基互补配对带来的精确性和可编程性,我们可以对DNA纳米结构的外形、尺寸、价态等性状进行精确调控,获得高度定制化和有序化的“智能”载体,最终实现诊疗一体化的“纳米医生”。
但是,DNA纳米结构用作药物载体还有一大关键问题,带负电荷的DNA分子在形成纳米结构之后,能否穿过同样带负电荷的细胞膜进入细胞。在常规的细胞转染核酸分子的操作中,我们通常需要带正电荷的转染试剂(主要是阳离子脂质体聚合物)帮助外源核酸分子被细胞摄取。但是,2011年,Turberfield团队[81]和樊春海研究组[82]都观察到,纳米DNA四面体结构可在不需要借助任何特异配体或转染试剂的情况下被活细胞摄取。Sleiman团队[83]也报道了滚环扩增(RCA)组装的管状DNA纳米结构有较高的细胞摄取率。这些研究表明,与非结构化的线性核酸分子不同,有一定刚性和密实度的DNA纳米结构本身可被细胞主动摄取。樊春海研究组对荧光标记的DNA四面体为研究对象进行了单颗粒追踪,观察了它被哺乳细胞摄取的过程,说明了该类型DNA纳米结构可以通过能量依赖的细胞主动摄取进入细胞[84]。这些研究结果为DNA纳米结构直接作为药物递送载体的可能性提供了支持。以下介绍DNA纳米结构在药物递送领域的一些应用。
2.5.1 小分子药物递送 小分子药物,尤其是像阿霉素、顺铂、紫杉醇这样的肿瘤治疗药物,通常对机体正常组织和细胞也有很高的毒性,副作用极强。并且,反复使用这类药物容易使目标肿瘤细胞产生多药耐药性(MDR),降低药物的治疗效果。因此,发展这类针对小分子药物的药物递送系统,改善这类药物的缺陷就显得很有意义。例如,阿霉素就是这类药物中的一个典型代表,它可以通过嵌入DNA双螺旋结构抑制其复制和表达,因此对肿瘤细胞有明显的杀伤效果。然而,它的选择性较差,有明显的毒副作用,也容易因为细胞的耐药性而失去效果。值得注意的是,由于阿霉素具有可嵌入DNA双链的特性,DNA纳米结构就显得尤其适合用于阿霉素转运。在这一研究方向,丁宝全教授的研究组已经取得了较大的进展[85,86],他们利用DNA折纸结构实现了对阿霉素分子的高效装载(图3-A)。研究表明,DNA折纸结构可以将阿霉素分子释放到具有耐药性的肿瘤细胞内,从而杀死那些在同等浓度条件下游离阿霉素分子无法杀死的肿瘤细胞。并且,在动物实验中,这些载带阿霉素的DNA折纸结构可以通过肿瘤的“穿透性与滞留性增强”(EPR)效应聚集在小鼠的肿瘤部位,从而有效地抑制肿瘤生长。而Hogberg团队[87]则研究了不同扭转角度的DNA折纸结构转运阿霉素到3种不同的乳腺癌细胞系中的效果。他们发现,通过调节DNA结构的扭转角度可以调节药物的载带量及药物的释放动力学,实现可预定义的药物释放。
2.5.2 核酸分子递送 与小分子药物相比,核酸分子药物,如CpG(“胞嘧啶-磷酸-鸟嘌呤”)核酸、反义核酸(如反义DNA、小干扰RNA、微小RNA等)、核酸适配体(Aptamer)等,因其较高的选择性和较低的副作用,显示出了良好的临床应用前景。这些核酸药物的传统递送策略包括对核酸分子进行化学修饰、利用阳离子转染试剂帮助其进入细胞等。近年来,用DNA纳米结构来载带核酸分子也了吸引研究者们的关注。因为,载体与载荷都是核酸分子,它们之间只需要通过碱基互补配对就可以很便利地进行杂交连接,这就省却了将载体和载荷分子进行化学偶联的麻烦。与通过非共价吸附载带药物的方式相比,这样的连接又可以对核酸药物分子的空间排布进行更精确的控制,使其作用更具确定性。例如,研究发现,未甲基化的“胞嘧啶-磷酸-鸟嘌呤”(CpG)序列的寡核苷酸可被哺乳动物免疫细胞识别并激活免疫应答反应,因此可以作为一种有效的免疫激活药物或疫苗佐剂用于各种免疫相关疾病(包括传染疾病、癌症等)的治疗。然而,在生理条件下,天然的CpG寡核苷酸会很容易被核酸酶降解,也很难直接进入细胞达到目标位置发挥作用。因此,近年来,研究者们报道了多种利用DNA纳米结构载带CpG寡核苷酸的策略。这其中包括:Nishikawa等采用的Y-形[88]、X-形等“多足形”DNA结构[89]和以这些结构为单元组装的更大尺度的树枝状结构[90]乃至水凝胶结构[88],樊春海研究组[82]和颜灏研究组[91]相继报道的DNA四面体结构(图3-B),以及更大尺度的基于DNA折纸术的纳米管[92]、纳米带[93]等结构。这些研究工作的结论表明,与游离的CpG核酸分子相比,引入DNA纳米结构之后,CpG核酸的稳定性普遍增加了,免疫细胞对于CpG核酸的摄取效率显著提高了,对于细胞的免疫激活能力也大大增强了。这种性能改变与DNA纳米结构带来的尺寸变化相关。因此,DNA纳米结构对于CpG核酸而言无疑是一种较为理想的载体。此外,DNA纳米载体在载带CpG核酸的同时还可以共载运其它的药物分子,如疫苗[91](图3-C)和阿霉素[88],发挥CpG免疫佐剂的功能来强化其它药物的效力,获得了比单独使用这些药物更好的效果,为多药物共递送和联合治疗提供了借鉴。
另外一类引起广泛重视的核酸药物是小干扰RNA(siRNA),这类RNA分子可以有选择性且高效率地沉默目标基因的表达,即RNA干扰(RNAi)作用,因此在基因治疗领域被寄予厚望。但是,未经化学修饰的RNA分子同样存在极易被降解、体内半衰期很短,难以进入细胞等难题。2012年,来自哈佛医学院的Anderson教授等研究者们成功地将DNA四面体作为载体载带siRNA进入小鼠体内的肿瘤细胞当中,并实现了靶向基因的沉默(图3-D)。在他们的策略当中,siRNA通过序列互补杂交连接到四面体侧面伸出的DNA手臂链上,其诱导RNA干扰的效果依然不受影响,说明了DNA纳米结构用于siRNA载带的可行性。
2.5.3 “智能化”药物递送 除了起到保护并帮助药物分子到达靶位点的作用之外,理想的药物递送载体还应该更加“智能化”,能够实现药物靶向递送并根据环境因素或外部刺激控制药物释放,最终成为“诊断-治疗一体化”的“纳米医生”。近年来,DNA自组装纳米结构在这一领域也逐渐崭露头角。
载体的主动靶向性,通常是通过在载体上偶联适当的配体,如多肽、抗体、核酸适体等实现的。这些配体能与靶细胞内部或表面的一些高表达的受体结合,从而使得载体在该细胞处有选择性地富集并发挥作用。因为DNA纳米结构的高度可编程性和可寻址性,DNA纳米结构可以精确控制靶向配体在其结构上的空间排布、朝向和密度等参数,因此尤其适合用于靶向性载药的研究。在DNA四面体用于siRNA递送的研究工作中[94],研究者们引入了叶酸分子作为靶向配体,因为叶酸受体在许多癌细胞系中都是过量表达的。研究结果表明,这些叶酸修饰的DNA载体展现出很强的靶向效应,能够有选择性地在肿瘤中富集并产生基因沉默效果;而未修饰叶酸分子或者叶酸分子朝向不合适的载体则不能发挥预期的效果。谭蔚弘教授的研究组[95,96]则利用带有核酸适配体序列的DNA纳米自组装结构载运阿霉素或反义核酸,使药物获得了针对特定的白血病细胞株的靶向性(图3-E)。
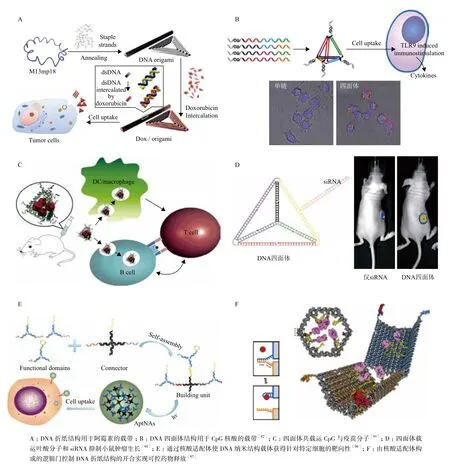
图3 DNA纳米自组装结构在药物递送领域的应用研究
另一方面,依赖单一的受体-配体相互作用带来的靶向性,其选择性往往是不足的,这就需要引入多个判定条件增加靶向的可靠性。于是,“逻辑门”对多个条件进行判定的原理就可以被研究者们用到靶向可控的药物释放上,这就是“智能药物”的思路。而DNA纳米结构的众多优点也为构造这样的智能递送系统提供了可能。2012年,Church研究组[97]的研究者们利用一种可开合的DNA纳米匣子实现了由逻辑门控制的药物载体。这个匣子由两个包含了不同核酸适配体的“锁扣”控制,只有当环境中这两种核酸适配体所针对的靶标分子——相当于两把钥匙——同时存在并触发核酸适配体的构型变化时,两个锁扣才能打开,从而使匣子内部的载荷分子得以暴露在外(图3-F)。这样,研究者们获得了一种由逻辑“与”门控制的,只有当两种条件同时满足时才能触发药物分子释放的智能药物载体。这种开合自如的出众特色得益于DNA纳米结构的精确可控性,这一成果展示了DNA纳米结构在智能化药物领域的巨大潜力。
3 结语
综上可见,DNA纳米技术在近20年间获得了飞跃式的发展。从技术演进趋势来看,基于DNA自组装人工构建的结构已经从简单的一维或无定型结构发展到了精确可定义的复杂三维结构,从静态结构发展到了可控的动态结构,研究者们也从最初单纯的结构设计逐渐向功能探索方向深入。在对于DNA纳米结构的功能研究上,从对其它材料或分子的空间排布的控制,到纳米反应器的构建,再到智能化的分子诊断和药物递送,DNA纳米技术也走过了一条优美的发展路径。目前,DNA纳米技术的广泛应用还面临一些挑战。例如,DNA大规模合成的成本仍然偏高,组装复杂DNA纳米结构的纯度和产率还有待提高[98]。但是,我们认为,凭借化学合成技术和生物技术的发展进步,DNA微阵列合成术[99]和细胞内的DNA生物合成[100]这样的手段可以为DNA纳米结构的组装提供新的方法,改进后的电泳[101]、超速离心[102]和高效液相色谱等手段[103]也可以用于优化DNA纳米结构的产率,更加精巧、复杂、智能化的DNA纳米结构还将层出不穷。并且为克服这些挑战付出的努力也是值得的,因为DNA纳米结构的尺度正好处于生物大分子相互作用产生复杂生命活动的尺度范围内,在可以预见的未来,我们相信DNA纳米技术本身也还将更加紧密地与生物医学、系统生物学与合成生物学相互交叉结合,使人们通过对DNA结构的设计更加深入地介入到生命活动最基本的层级当中,从而帮助人们更加深刻地理解生命活动的机制。
[1]Seeman NC. Nanotechnology and the double helix[J]. Sci Am, 2004, 290(6):64-75.
[2]Somoza Á. Evolution of DNA origami[J]. Angew Chem Int Ed, 2009, 48(50):9406-9408.
[3]Weiss PS. A conversation with Prof. Ned Seeman:founder of DNA nanotechnology[J]. ACS Nano, 2008, 2(6):1089-1096.
[4]Seeman NC. Structural DNA nanotechnology:growing along with Nano Letters[J]. Nano Lett, 2010, 10(6):1971-1978.
[5]LaBean TH. Nanotechnology:Another dimension for DNA art[J]. Nature, 2009, 459(7245):331-332.
[6]Yang X, Wenzler LA, Qi J, et al. Ligation of DNA triangles containing double crossover molecules[J]. J Am Chem Soc, 1998, 120(38):9779-9786.
[7]Winfree E, Liu F, Wenzler LA, et al. Design and self-assembly of two-dimensional DNA crystals[J]. Nature, 1998, 394(6693):539-544.
[8]Rothemund PWK. Folding DNA to create nanoscale shapes and patterns[J]. Nature, 2006, 440(7082):297-302.
[9]Kuzuya A, Wang R, Sha R, et al. Six-helix and eight-helix DNA nanotubes assembled from half-tubes[J]. Nano Lett, 2007, 7(6):1757-1763.
[10]Zheng JP, Birktoft JJ, Chen Y, et al. From molecular to macroscopic via the rational design of a self-assembled 3D DNA crystal[J]. Nature, 2009, 461(7260):74-77.
[11]Qian L, Wang Y, Zhang Z, et al. Analogic China map constructed by DNA[J]. Chin Sci Bull, 2006, 51(24):2973-2976.
[12]Andersen ES, Dong M, Nielsen MM, et al. DNA origami design of dolphin-shaped structures with flexible tails[J]. ACS Nano, 2008, 2(6):1213-1218.
[13]Andersen ES, Dong M, Nielsen MM, et al. Self-assembly of a nanoscale DNA box with a controllable lid[J]. Nature, 2009, 459(7243):73-76.
[14]Ke Y, Sharma J, Liu M, et al. Scaffolded DNA origami of a DNA tetrahedron molecular container[J]. Nano Lett, 2009, 9(6):2445-2447.
[15]Kuzuya A, Komiyama M. Design and construction of a box-shaped 3D-DNA origami[J]. Chem Commun, 2009, 28:4182-4184.
[16]Douglas SM, Dietz H, Liedl T, et al. Self-assembly of DNA into nanoscale three-dimensional shapes[J]. Nature, 2009, 459(7245):414-418.
[17]Dietz H, Douglas SM, Shih WM. Folding DNA into twisted and curved nanoscale shapes[J]. Science, 2009, 325(5941):725-730.
[18]Han D, Pal S, Nangreave J, et al. DNA origami with complex curvatures in three-dimensional space[J]. Science, 2011, 332(6027):342-346.
[19]Ke Y, Ong LL, Shih WM, et al. Three-dimensional structures selfassembled from DNA bricks[J]. Science, 2012, 338(6111):1177-1183.
[20]Douglas SM, Chou JJ, Shih WM. DNA-nanotube-induced alignment of membrane proteins for NMR structure determination[J]. Proc Natl Acad Sci, 2007, 104(16):6644-6648.
[21]Steinhauer C, Jungmann R, Sobey TL, et al. DNA origami as a nanoscopic ruler for super-resolution microscopy[J]. Angew Chem, Int Ed, 2009, 48(47):8870-8873.
[22]Kershner RJ, Bozano LD, Micheel CM, et al. Placement and orientation of individual DNA shapes on lithographically patterned surfaces[J]. Nat Nanotechnol, 2009, 4(9):557-561.
[23]Hung AM, Micheel CM, Bozano LD, et al. Large-area spatially ordered arrays of gold nanoparticles directed by lithographically confined DNA origami[J]. Nat Nanotechnol, 2010, 5(2):121-126.
[24]Ke Y, Lindsay S, Chang Y, et al. Self-assembled water-soluble nucleic acid probe tiles for label-free RNA hybridization assays[J]. Science, 2008, 319(5860):180-183.
[25] Kaganman I. An origami chip of DNA[J]. Nat Methods, 2008, 5(3):222.
[26]Zhang Z, Wang Y, Fan C, et al. Asymmetric DNA origami for spatially addressable and index-free solution-phase DNA chips[J]. Adv Mater, 2010, 22(24):2672-2675.
[27]Chhabra R, Sharma J, Ke Y, et al. Spatially addressable multiprotein nanoarrays templated by aptamer-tagged DNA nanoarchitectures[J]. J Am Chem Soc, 2007, 129(34):10304-10305.
[28]Rinker S, Ke Y, Liu Y, et al. Self-assembled DNA nanostructures for distance-dependent multivalent ligand-protein binding[J]. Nat Nanotechnol, 2008, 3(7):418-422.
[29]Lin C, Katilius E, Liu Y, et al. Self-assembled signaling aptamer DNA arrays for protein detection[J]. Angew Chem Int Ed, 2006, 45(32):5296-5301.
[30]Lin C, Liu Y, Yan H. Self-assembled combinatorial encoding nanoarrays for multiplexed biosensing[J]. Nano Lett, 2007, 7(2):507-512.
[31]Lin C, Nangreave JK, Li Z, et al. Signal amplification on a DNA-tile-based biosensor with enhanced sensitivity[J]. Nanomed, 2008, 3(4):521-528.
[32]Pei H, Liang L, Yao G, et al. Reconfigurable three-dimensional DNA nanostructures for the construction of intracellular logic sensors[J]. Angew Chem Int Ed, 2012, 51(36):9020-9024.
[33]Krishnan Y, Simmel FC. Nucleic acid based molecular devices[J]. Angew Chem Int Ed, 2011, 50(14):3124-3156.
[34]Wang F, Lu CH, Willner I. From cascaded catalytic nucleic acids to enzyme-DNA nanostructures:controlling reactivity, sensing, logic operations, and assembly of complex structures[J]. Chem Rev(Washington, DC, U S), 2014, 114(5):2881-2941.
[35]Mao CD, LaBean TH, Reif JH, et al. Logical computation using algorithmic self-assembly of DNA triple-crossover molecules[J]. Nature, 2000, 407(6803):493-496.
[36] Liu D, Balasubramanian S. A proton-fuelled DNA nanomachine[J]. Angew Chem Int Ed, 2003, 42(46):5734-5736.
[37]Wang C, Huang Z, Lin Y, et al. Artificial DNA nano-spring powered by protons[J]. Adv Mater(Weinheim, Ger), 2010, 22(25):2792-2798.
[38]Tian Y, He Y, Chen Y, et al. A DNAzyme that walks processively and autonomously along a one-dimensional track[J]. Angew Chem Int Ed, 2005, 44(28):4355-4358.
[39]Wickham SFJ, Bath J, Katsuda Y, et al. A DNA-based molecular motor that can navigate a network of tracks[J]. Nat Nanotechnol, 2012, 7(3):169-173.
[40]Wickham SFJ, Endo M, Katsuda Y, et al. Direct observation of stepwise movement of a synthetic molecular transporter[J]. Nat Nanotechnol, 2011, 6(3):166-169.
[41]Gu H, Chao J, Xiao SJ, et al. Dynamic patterning programmed by DNA tiles captured on a DNA origami substrate[J]. Nat Nanotechnol, 2009, 4(4):245-248.
[42]Gu H, Chao J, Xiao SJ, et al. A proximity-based programmable DNA nanoscale assembly line[J]. Nature, 2010, 465(7295):202-205.
[43]Lund K, Manzo AJ, Dabby N, et al. Molecular robots guided by prescriptive landscapes[J]. Nature, 2010, 465(7295):206-210.
[44]Pei R, Taylor SK, Stefanovic D, et al. Behavior of polycatalytic assemblies in a substrate-displaying matrix[J]. J Am Chem Soc, 2006, 128(39):12693-12699.
[45]Smith LM. Nanotechnology:Molecular robots on the move[J]. Nature, 2010, 465(7295):167-168.
[46]Li X, Liu DR. DNA-templated organic synthesis:nature’s strategy for controlling chemical reactivity applied to synthetic molecules[J]. Angew Chem Int Ed, 2004, 43(37):4848-4870.
[47]Gartner ZJ, Tse BN, Grubina R, et al. DNA-templated organic synthesis and selection of a library of macrocycles[J]. Science, 2004, 305(5690):1601-1605.
[48]Kanan MW, Rozenman MM, Sakurai K, et al. Reaction discovery enabled by DNA-templated synthesis and in vitro selection[J]. Nature, 2004, 431(7008):545-549.
[49]Nielsen M, Thomsen AH, Clo E, et al. Synthesis of linear and tripoidal oligo(phenylene ethynylene)-based building blocks for application in modular DNA-programmed assembly[J]. J Org Chem, 2004, 69(7):2240-2250.
[50]Blakskjaer P, Gothelf KV. Synthesis of an elongated linear oligo(phenylene ethynylene)-based building block for application in DNA-programmed assembly[J]. Org Biomol Chem, 2006, 4(18):3442-3447.
[51]Brown RS, Nielsen M, Gothelf KV. Self-assembly of aluminiumsalen coupled nanostructures from encoded modules with cleavable disulfide DNA-linkers[J]. Chem Commun, 2004, 13:1464-1465.
[52]Gothelf KV, Brown RS. A modular approach to DNA-programmed self-assembly of macromolecular nanostructures[J]. Chemistry, 2005, 11(4):1062-1069.
[53]Gothelf KV, Thomsen A, Nielsen M, et al. Modular DNA-programmed assembly of linear and branched conjugated nanostructures[J]. J Am Chem Soc, 2004, 126(4):1044-1046.
[54]Nielsen M, Dauksaite V, Kjems J, et al. DNA-directed coupling of organic modules by multiple parallel reductive aminations and subsequent cleavage of selected DNA sequences[J]. Bioconjug Chem, 2005, 16(4):981-985.
[55]Andersen CS, Yan H, Gothelf KV. Bridging one helical turn in double-stranded DNA by templated dimerization of molecular rods[J]. Angew Chem Int Ed, 2008, 47(30):5569-5572.
[56]Andersen CS, Knudsen MM, Chhabra R, et al. Distance dependent interhelical couplings of organic rods incorporated in DNA 4-helix bundles[J]. Bioconjug Chem, 2009, 20(8):1538-1546.
[57]Piperberg G, Wilner OI, Yehezkeli O, et al. Control of bioelectrocatalytic transformations on DNA scaffolds[J]. J Am Chem Soc, 2009, 131(25):8724-8725.
[58]Tel-Vered R, Yehezkeli O, Yildiz HB, et al. Photoelectrochemistry with ordered CdS nanoparticle/relay or photosensitizer/relay dyads on DNA scaffolds[J]. Angew Chem, Int Ed, 2008, 47(43):8272-8276.
[59]Elbaz J, Tel-Vered R, Freeman R, et al. Switchable motion of DNA on solid supports[J]. Angew Chem Int Ed, 2009, 48(1):133-137.
[60]Wilner OI, Weizmann Y, Gill R, et al. Enzyme cascades activated on topologically programmed DNA scaffolds[J]. Nat Nanotechnol, 2009, 4(4):249-254.
[61]Wang ZG, Wilner OI, Willner I. Self-assembly of aptamer-circular DNA nanostructures for controlled biocatalysis[J]. Nano Lett, 2009, 9(12):4098-4102.
[62]Fu YM, Zeng DD, Chao J, et al. Single-step rapid assembly of DNA origami nanostructures for addressable nanoscale bioreactors[J]. J Am Chem Soc, 2013, 135(2):696-702.
[63]Le JD, Pinto Y, Seeman NC, et al. DNA-templated self-assembly of metallic nanocomponent arrays on a surface[J]. Nano Lett, 2004, 4(12):2343-2347.
[64]Pinto YY, Le JD, Seeman NC, et al. Sequence-encoded selfassembly of multiple-nanocomponent arrays by 2D DNA scaffolding[J]. Nano Lett, 2005, 5(12):2399-2402.
[65]Gerdon AE, Oh SS, Hsieh K, et al. Controlled delivery of DNA origami on patterned surfaces[J]. Small, 2009, 5(17):1942-1946.
[66]Ding B, Deng Z, Yan H, et al. Gold nanoparticle self-similar chain structure organized by DNA origami[J]. J Am Chem Soc, 2010, 132(10):3248-3249.
[67]Pal S, Deng Z, Ding B, et al. DNA-origami-directed self-assembly of discrete silver-nanoparticle architectures[J]. Angew Chem Int Ed, 2010, 49(15):2700-2704.
[68]Sharma J, Chhabra R, Andersen CS, et al. Toward reliable gold nanoparticle patterning on self-assembled DNA nanoscaffold[J]. J Am Chem Soc, 2008, 130(25):7820-7821.
[69]Stearns LA, Chhabra R, Sharma J, et al. Template-directed nucleation and growth of inorganic nanoparticles on DNA scaffolds[J]. Angew Chem Int Ed, 2009, 48(45):8494-8496.
[70]Sharma J, Chhabra R, Cheng A, et al. Control of self-assembly of DNA tubules through integration of gold nanoparticles[J]. Science, 2009, 323(5910):112-116.
[71]Iijima S. Helical microtubules of graphitic carbon[J]. Nature, 1991, 354(6348):56-58.
[72]Ouyang M, Huang JL, Lieber CM. Scanning tunneling microscopy studies of the one-dimensional electronic properties of singlewalled carbon nanotubes[J]. Annu Rev Phys Chem, 2002, 53:201-220.
[73] Dai H. Carbon nanotubes:synthesis, integration, and properties[J]. Acc Chem Res, 2002, 35(12):1035-1044.
[74] Terrones M. Science and technology of the twenty-first century:synthesis, properties, and spplications of carbon nanotubes[J]. Annual Review of Materials Research, 2003, 33:419-501.
[75]Kostarelos K, Bianco A, Prato M. Promises, facts and challenges for carbon nanotubes in imaging and therapeutics[J]. Nat Nanotechnol, 2009, 4(10):627-633.
[76]Cao Q, Rogers JA. Ultrathin films of single-walled carbon nanotubes for electronics and sensors:A review of fundamental and applied aspects[J]. Adv Mater, 2009, 21(1):29-53.
[77]Dwyer C, Guthold M, Falvo M, et al. DNA-functionalized singlewalled carbon nanotubes[J]. Nanotechnology, 2002, 13(5):601-604.
[78]Dwyer C, Johri V, Cheung M, et al. Design tools for a DNA-guided self-assembling carbon nanotube technology[J]. Nanotechnology, 2004, 15(9):1240-1245.
[79]Han X, Li Y, Deng Z. DNA-wrapped single-walled carbon nanotubes as rigid templates for assembling linear gold nanoparticle arrays[J]. Adv Mater, 2007, 19(11):1518-1522.
[80]Maune HT, Han SP, Barish RD, et al. Self-assembly of carbon nanotubes into two-dimensional geometries using DNA origami templates[J]. Nat Nanotechnol, 2010, 5(1):61-66.
[81]Walsh AS, Yin H, Erben CM, et al. DNA cage delivery to mammalian cells[J]. ACS Nano, 2011, 5(7):5427-5432.
[82]Li J, Pei H, Zhu B, et al. Self-assembled multivalent DNA nanostructures for noninvasive intracellular delivery of immunostimulatory CpG oligonucleotides[J]. ACS Nano, 2011, 5(11):8783-8789.
[83]Hamblin GD, Carneiro KMM, Fakhoury JF, et al. Rolling circle amplification-templated DNA nanotubes show increased stability and cell penetration ability[J]. J Am Chem Soc, 2012, 134(6):2888-2891.
[84]Liang L, Li J, Li Q, et al. Single-particle tracking and modulation of cell entry pathways of a tetrahedral DNA nanostructure in live cells[J]. Angew Chem Int Ed, 2014, 53(30):7745-7750.
[85]Jiang Q, Song C, Nangreave J, et al. DNA origami as a carrier for circumvention of drug resistance[J]. Journal of the American Chemical Society, 2012, 134(32):13396-13403.
[86]Zhang Q, Jiang Q, Li N, et al. DNA origami as an in vivo drug delivery vehicle for cancer therapy[J]. ACS Nano, 2014, 8(7):6633-6643.
[87]Zhao YX, Shaw A, Zeng XH, et al. DNA origami delivery system for cancer therapy with tunable release properties[J]. ACS Nano, 2012, 6(10):8684-8691.
[88]Nishikawa M, Mizuno Y, Mohri K, et al. Biodegradable CpG DNA hydrogels for sustained delivery of doxorubicin and immunostimulatory signals in tumor-bearing mice[J]. Biomaterials, 2011, 32(2):488-494.
[89]Mohri K, Nishikawa M, Takahashi N, et al. Design and development of nanosized DNA assemblies in polypod-like structures as efficient vehicles for immunostimulatory CpG motifs to immune cells[J]. ACS Nano, 2012, 6(7):5931-5940.
[90]Rattanakiat S, Nishikawa M, Funabashi H, et al. The assembly of a short linear natural cytosine-phosphate-guanine DNA into dendritic structures and its effect on immunostimulatory activity[J]. Biomaterials, 2009, 30(29):5701-5706.
[91]Liu X, Xu Y, Yu T, et al. A DNA nanostructure platform fordirected assembly of synthetic vaccines[J]. Nano Lett, 2012, 12(8):4254-4259.
[92]Schuller VJ, Heidegger S, Sandholzer N, et al. Cellular immunostimulation by CpG-sequence-coated DNA origami structures[J]. ACS Nano, 2011, 5(12):9696-9702.
[93]Ouyang X, Li J, Liu H, et al. Rolling circle amplificationbased DNA origami nanostructrures for intracellular delivery of immunostimulatory drugs[J]. Small, 2013, 9(18):3082-3087.
[94]Lee H, Lytton-Jean AKR, Chen Y, et al. Molecularly self-assembled nucleic acid nanoparticles for targeted in vivo siRNA delivery[J]. Nat Nano, 2012, 7(6):389-393.
[95]Zhu G, Zheng J, Song E, et al. Self-assembled, aptamer-tethered DNA nanotrains for targeted transport of molecular drugs in cancer theranostics[J]. Proceedings of the National Academy of Sciences, 2013, 110(20):7998-8003.
[96]Wu C, Han D, Chen T, et al. Building a multifunctional aptamerbased DNA nanoassembly for targeted cancer therapy[J]. J Am Chem Soc, 2013, 135(49):18644-18650.
[97]Douglas SM, Bachelet I, Church GM. A logic-gated nanorobot for targeted transport of molecular payloads[J]. Science, 2012, 335(6070):831-834.
[98]Pinheiro AV, Han DR, Shih WM, et al. Challenges and opportunities for structural DNA nanotechnology[J]. Nat Nanotechnol, 2011, 6(12):763-772.
[99]Kosuri S, Eroshenko N, LeProust EM, et al. Scalable gene synthesis by selective amplification of DNA pools from high-fidelity microchips[J]. Nat Biotechnol, 2010, 28(12):1295-U1108.
[100]Lin CX, Rinker S, Wang X, et al. In vivo cloning of artificial DNA nanostructures[J]. Proc Natl Acad Sci USA, 2008, 105(46):17626-17631.
[101] Bellot G, McClintock MA, Lin C, et al. Recovery of intact DNA nanostructures after agarose gel-based separation[J]. Nat Methods, 2011, 8(3):192-194.
[102] Lin C, Perrault SD, Kwak M, et al. Purification of DNA-origami nanostructures by rate-zonal centrifugation[J]. Nucleic Acids Res, 2013, 41(2):e40.
[103] Xing S, Jiang D, Li F, et al. Constructing higher-order DNA nanoarchitectures with highly purified DNA nanocages[J]. ACS Appl Mater Interfaces, 2014, doi:10. 1021/am505592e.
Advances on Self-Assembled DNA Nanostructures
Yu Yang1Li Jiang2Zhang Zhao3Fan Chunhai2
(1. Department of Electronic Information,Shanghai Institute of Scientific Management,Shanghai 201800;2. Shanghai Institute of Applied Physics,Chinese Academy of Sciences,Shanghai 201800;3. Department of Chemistry,Aarhus University,Aarhus 8000,Denmark)
Studies on self-assembled DNA nanostructures have achieved great progress in recent decades. In this article, we introduced the general principles of DNA nanostructures and the history of their development. Their features and advantages are also summarized. Their applications in biosensing, nanoreactors, nanoscale spatial arrangement, nanorobots, and drug delivery have been reviewed. The future of DNA nanotechnology has also been prospected.
DNA origami;nanomachine;nanodrug
10.13560/j.cnki.biotech.bull.1985.2015.03.011
2015-02-04
国家自然科学基金项目(B050902)
俞洋,女,硕士,研究方向:DNA计算及DNA纳米技术;E-mail:daizy1111@hotmail.com
樊春海,男,博士,研究方向:生物传感、生物成像及DNA纳米技术;E-mail:fchh@sinap.ac.cn
