耐复方新诺明嗜麦芽窄食单胞菌耐药基因检测及同源性分析
2015-06-05伍启康吴奎海崔东岚
伍启康,吴奎海,崔东岚
(佛山市第一人民医院检验科,广东 佛山528000)
嗜麦芽窄食单胞菌是一种广泛存在于自然界和医院环境的条件致病菌,随着广谱抗生素和免疫抑制剂的广泛使用,以及侵入性治疗技术的不断应用,该菌已成为院内感染的重要病原菌之一,在非发酵菌的分离率中仅次于鲍曼不动杆菌和铜绿假单胞菌[1],复方新诺明是临床抗嗜麦芽窄食单胞菌感染的首选用药,但近几年,临床经常能分离到耐复方新诺明的嗜麦芽窄食单胞菌,其耐药性常和I类整合子中的sul1基因[2]以及ISCR2携带的sul2相关[3]。本文主要对耐复方新诺明嗜麦芽窄食单胞菌株相关基因进行检测,并对其进行基因分型。
1 材料与方法
1.1 菌株来源 我院2013年1月至2013年12月共分离获得126株嗜麦芽窄食单胞菌,其中对复方新诺明耐药的有22株,剔除同一患者相同部位7d内的重复分离株。
1.2 细菌鉴定和药敏试验 法国生物梅里埃公司的VITEK2-compact仪器及其配套GN鉴定卡进行菌种鉴定,药敏试验采用K-B法。依据临床实验室标准化委员会(CLSI)2013年标准判断结果,质控菌株为大肠埃希菌ATCC25922和铜绿假单胞菌A TCC27853。
1.3 主要仪器和试剂 德国Eppendorf公司的PCR扩增仪器,Bio-Rad公司的凝胶仪,TaqDNA聚合酶﹑dNTPs、DNA Marker、细菌基因组DNA提取试剂盒均为TaKaRa公司产品。复方新诺明药敏纸片购自英国OXOID公司,
1.4 PCR扩增 DNA的提取参考试剂盒说明书,-40℃保存。相关基因的PCR反应体系均为20μl体积,其中含 Mg2+的 10×buffer 2μl,dNTP 200μmol/L,上下游引物 0.2μmol/L,DNA 模板 1μl,TaqDNA 聚合酶1U,加入灭菌去离子水至20μl。所扩增的sul1、sul2、ISCR1、ISCR2 阳性菌株均为本实验 PCR扩增阳性并测序验证,具体的引物序列和退火温度见表1。

表1 PCR反应的引物序列
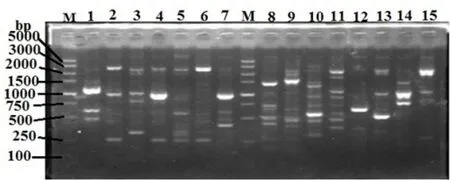
图1 部分耐复方新诺明嗜麦芽窄食单胞菌ERIC-PCR电泳结果
1.5 PCR产物序列分析 sul1和整合子I阳性产物送华大基因科技公司进行纯化并双向测序,利用DNAstar软件对测序结果进行序列校正,在Gen-Bank中用Blastn进行核酸同源性搜索,分析基因类型。
1.6 ERIC-PCR 对耐复方新诺明的嗜麦芽窄食单胞菌进行基因分型,分析其遗传相关性。引物ERIC-2:AAGTAAGTGACTGGGGTGAGCG[6]。反应条件为 95℃预变性 5min;94℃变性 1 min,26℃退火 2min,72℃延 伸 1min,4 个 循 环 ;94℃变 性 1 min,40℃退火 1min,72℃延伸 1min,40 个循环;72℃延伸 10min。
2 结果
2.1 药敏试验 复方新诺明的耐药率为17.4%(22/126),中介为4.0%(5/126),敏感率为 78.6%(99/126)。
2.2 PCR扩增 在126株嗜麦芽窄食单胞菌中,sul2和ISCR2均阴性。在22株耐复方新诺明菌株中,15株I类整合子基因盒和sul1均为阳性,有1株仅sul1阳性。在104株复方新诺明中介或敏感菌株中,sul1和I类整合子基因盒检测均为阴性。
2.3 I类整合子基因盒 15株I类整合子基因盒测序结果:dfrA17,aadA5同时阳性9株,aacA4、blaIMP-25、blaOXA-30、catB3 同 时 阳 性 1 株 ,aadB、aadA2同时阳性 1株,dfrA15阳性 4株,qacF阳性1株。
2.4 ERIC-PCR 电泳条带集中分布在250~3000bp之间,分21个基因型,部分结果见图1。
3 讨论
整合子因其在细菌耐药基因水平转移中的重要作用而受到研究者的关注,I类整合子在临床上最常见,结构由3个部分组成:5’保守末端(5’conserved segment,5’CS)、 3’ 保守末端(3’conserved segment,3’CS)和两者之间的可变区(variable region)。3’CS包括3个开放读码框(ORF):磺胺耐药基因 (sul1),季铵盐化合物及溴乙锭的耐受基因 (qacE△1)及功能不明的ORF5[7]。可变区带有不同数量的基因盒,大部分是编码各种抗生素抗性的耐药基因盒。本研究结果表明,耐复方新诺明嗜麦芽窄食单胞菌I类整合子携带率较高,达到68%(15/22)。从基因盒携带的耐药基因来看,包括耐甲氧苄啶类 (dfrA17,dfrA15)、耐氨基糖苷类(aadA5,aacA4,aadB ,aadA2)、耐氯霉素(catB3)、β-类酰胺酶类(blaIMP-25,blaOXA-30)、耐消毒剂(qacF)。 Huang 等[8]通过基因敲除的方法,发现I类整合子能明显增加复方新诺明的耐药性,但消毒剂的MIC浓度未见明显改变,可能是qacF基因产生的耐药作用较小,主要与菌体外膜通透性有关。嗜麦芽窄食单胞菌I类整合子携带的氨基糖苷类耐药基因可以增加氨基糖类的MIC浓度,但其染色体携带的Aph(3’)-IIc、aac(6′)-Ib′、SmeZ 、SmeJK 可导致高水平氨基糖类耐药[9,10]。ISCR2属于插入序列一种,通过滚换复制转移邻近序列,是一个强有力的移动系统,携带多重耐药编码基因[11,12],如SXT、质粒介导的氯霉素和磺胺类药物耐药,常与sul2基因相关。在126菌株中未检出基因ISCR2、sul2,可能是由于地区和菌种差异,sul2在欧洲、南美地区或肠杆菌科细菌的检出率相对较高[3]。在6株耐复方新诺明菌株中,4种耐药基因均未检出,可能存在其他耐药机制,如外排泵、生物膜作用[13-15]。
ERIC-PCR是以肠杆菌科基因间重复序列为引物进行 PCR,扩增出多态性的DNA图谱。它快速、经济、简便、重复性较好,其分辨率与脉冲场凝胶电泳(PFGE)高度一致[16]。本研究中的22株耐复方新诺明嗜麦芽窄食单胞菌分型较广,不存在院内暴发流行。
[1]郝会青,胡利民.嗜麦芽窄食单胞菌医院感染分布及耐药性分析[J].实验与检验医学,2012,30(4):414-417.
[2]Hu LF,Chang X,Ye Y,et al.Stenotrophomonas maltophilia resistance to trimethoprim/sulfamethoxazole mediated by acquisition of sul and dfrA genes in a plasmid-mediated class 1 integron[J].Int J Antimicrob Agents,2011,37(3):230-234.
[3]Toleman MA,Bennett PM,Bennett DM,et al.Global emergence of trimethoprim/sulfamethoxazole resistance in Stenotrophomonas maltophilia mediated by acquisition of sul genes[J].Emerg Infect Dis,2007,13(4):559-565.
[4]Byrne-Bailey KG,Gaze WH,Kay P,et al.Prevalence of sulfonamide resistance genes in bacterial isolates from manured agricultural soils and pig slurry in the United Kingdom[J].Antimicrob A-gents Chemother,2005,53(2):696-702.
[5]Levesque C,Piche L,Larose C,et al.PCR mapping of integrons reveals several novel combinations of resistance genes[J].Antimicrobial Agents and Chemotherapy,1995,39(1):185-191.
[6]Munoz MA,Welcome FL,Schukken YH,et al.Molecular epidemiology of two Klebsiella pneumoniae mastitis outbreaks on a dairy farm in New York State[J].J Clin Microbiol,2007,45(12):3964-3971.[7]Domingues S,da Silva GJ,Nielsen KM.Integrons:Vehicles and pathways for horizontal dissemination in bacteria[J].Mob Genet Elements,2012,2(5):211-223.
[8]Huang YW,Hu RM,Lin YT,et al.The Contribution of Class 1 Integron to Antimicrobial Resistance in Stenotrophomonas maltophilia[J].Microb Drug Resist,2014,22.
[9]Gould VC,Okazaki A,Avison MB.Coordinate hyperproduction of SmeZ and SmeJK efflux pumpsextendsdrugresistance in Stenotrophomonas maltophilia[J].Antimicrob Agents Chemother,2013,57(1):655-657.
[10]Okazaki A,Avison MB.Aph(3')-IIc,an aminoglycoside resistance determinant from Stenotrophomonas maltophilia[J].Antimicrob A-gents Chemother,2007,51(1):359-360.
[11]Toleman MA,Bennett PM,Walsh TR.ISCR elements:Novel gene-capturing systems of the 21st century[J].Microbiology and Molecular Biology,Reviews,2006,70(2):296-316.
[12]Vilacoba E,Almuzara M,Gulone L,et al.Emergence and spread of plasmid-borne tet(B):ISCR2 in minocycline-resistant Acinetobacter baumannii isolates.Antimicrob Agents Chemother,2013,57(1):651-654.
[13]Liaw SJ,Lee YL,Hsueh PR.Multidrug resistance in clinical isolates of Stenotrophomonas maltophilia:roles of integrons,efflux pumps,phosphoglucomutase (SpgM),and melanin and biofilm formation[J].Int J Antimicrob Agents.2010,35(2):126-130.
[14]Gould VC,Avison MB.SmeDEF-mediated antimicrobial drug resistance in Stenotrophomonas maltophilia clinical isolates having defined phylogenetic relationships[J].J Antimicrob Chemother,2006,57(6):1070-1076.
[15]Zhang L,Li XZ,Poole K.SmeDEF multidrug efflux pump contributes to intrinsic multidrug resistance in Stenotrophomonas maltophilia[J].Antimicrob Agents Chemother.2001,45(12):3497-3503.
[16]Juhász E,Krizsán G,Lengyel G,et al.Infection and colonization by Stenotrophomonas maltophilia:antimicrobial susceptibility and clinical background of strains isolated at a tertiary care centre in Hungary[J].Ann Clin Microbiol Antimicrob,2014,13(1):333.
