秦巴山区野生桑黄rDNA ITS序列及亲缘关系分析*
2015-03-07张文隽吴亚召
张文隽,吴亚召,雷 萍,杜 芳
(陕西省微生物研究所,陕西 西安 710043)
秦巴山区野生桑黄rDNA ITS序列及亲缘关系分析*
张文隽,吴亚召,雷 萍**,杜 芳
(陕西省微生物研究所,陕西 西安 710043)
对采自秦巴山区的6株野生桑黄菌,进行rDNA ITS区段克隆测序和序列特征比较分析,并对其rDNA ITS序列进行核酸序列数据库GenBank同源性检索比对,将从GenBank检索获得的18个最相似物种的ITS序列连同6个不同桑黄菌株的ITS序列一起用于系统发育分析。结果表明,桑-W、桑-F、桑-D、桑-Y、桑-H与Inonotus linteus亲缘关系最近,相似性分别为98.23%、98.23%、98.10%、98.23%、98.23%,其序列长度均为757 bp;桑-H1与Fuscoporia qilva亲缘关系最近,相似性为95.90%,其序列长度均为796 bp。
桑黄;rDNA ITS;序列分析;亲缘关系
桑黄属于担子菌亚门(Basidiomycotina)、层菌纲(Hymenomycetes)、非褶菌目(Aphyllophorales)、锈革孔菌科 (Hymenochaeyaceae)、针层孔菌属(Phellinus)[1],属内又分为火木层孔菌(P.igniarius)、鲍氏针层孔菌(P.baumii) 和裂蹄木层孔菌(P.linteus)[2],是近几年开发的一种多年生的珍贵药用真菌,有“森林黄金”之美称。据《中药大辞典》记载,其可治血崩、血淋、带下、闭经等妇科疾病[3]。现代研究发现其抗癌、抗菌、抗纤维化、抗氧化等效果显著,广泛应用于医药、食品、日用化工、保健品等行业。
随着科学技术的不断进步,国外特别是日本和韩国权威医药机构研究发现桑黄抗肿瘤、抗菌、抗纤维化、抗氧化等效果显著,是目前公认的生物领域治疗肿瘤有效率排在第1位的高等真菌[4]。近年来,人们将药用菌桑黄作为一种重要生物资源,应用于抗肿瘤产品开发,在科技界和实业界都倾注了很高的热情。
桑黄来源于菌物的子实体,在真菌传统形态分类中,由于不同桑黄真菌间子实体形态差异较小,使得依赖于形态特征的传统鉴定较为困难,频频出现“同物异名”、“同名异物”的现象,造成了名称和应用上的混乱。根据文献记载不是每个俗名叫“桑黄”的都有药效,目前据资料报道鲍氏针层孔菌(P.baumii)、裂蹄针层孔菌(P.linteus)以及火木层孔菌(P.igniarius)等几种可入药。为此,对获得的6株桑黄菌株进行了基于rDNA ITS序列的分子鉴定,对更好的认识和利用秦巴山区野生桑黄具有重要意义。
1 材料与方法
1.1 供试菌株
供试的桑-W、桑-H、桑-F、桑-D、桑-Y、桑-H1六株菌株,为陕西省微生物研究所微生物资源中心第三研究室采集的秦巴山区野生桑黄子实体,通过组织分离获得。
对分离获得的菌种纯培养物进行取样检测。菌株编号及来源见表1。

表1 供试桑黄菌株的编号及来源Tab.1 Strain No.and fruit body collection site
1.2 方法
1.2.1 菌丝体培养
将保藏菌种取出活化后,接种于100 mL PDA液体培养基(300 mL三角瓶)中,每个菌株各接3瓶,28℃、150 r·min-1摇床培养7 d,纱布过滤,蒸馏水冲洗2次,灭菌纸吸干后,保存于-80℃备用。
1.2.2 基因组DNA提取及检测
取0.2 g~0.5 g菌丝体,用植物基因组提取DNA试剂盒(天根生化科技有限公司)提取菌丝体的基因组DNA,再用0.8%琼脂糖凝胶电泳对基因组DNA进行检测。
1.2.3 ITS序列扩增及检测
PCR扩增ITS序列,引物为ITS1:TCCGTAG GTGAACCTGCG和ITS4:TCCTCCGCTTATTGATAT GC,由上海生工生物工程股份有限公司合成。20 μL反应体系,反应条件如下:Genomic DNA(1 μL)、ITS1(0.5 μL)、ITS4(0.5 μL)、2×Mix(10 μL)、dd H2O(8 μL)。扩增程序为:95℃反应5 min;95℃反应30 s,55℃反应30 s,72℃反应60 s,35个循环;72℃反应7 min。用0.8%的琼脂糖凝胶电泳对PCR产物进行电泳检测。
1.2.4 rDNA ITS序列克隆和测序
扩增产物经0.8%琼脂糖凝胶电泳分离,以PCR纯化试剂盒回收后连接入pGM-T载体,转化大肠杆菌DH5α。每个样品均随机挑选5个阳性克隆,送至上海生工生物工程股份有限公司进行序列测定,所得序列进行BLAST同源性比对,利用分析软件MEGA4.0构建进化树。
2 结果与分析
2.1 基因组DNA提取及ITS序列扩增结果
样品基因组DNA提取电泳检测结果见图1。
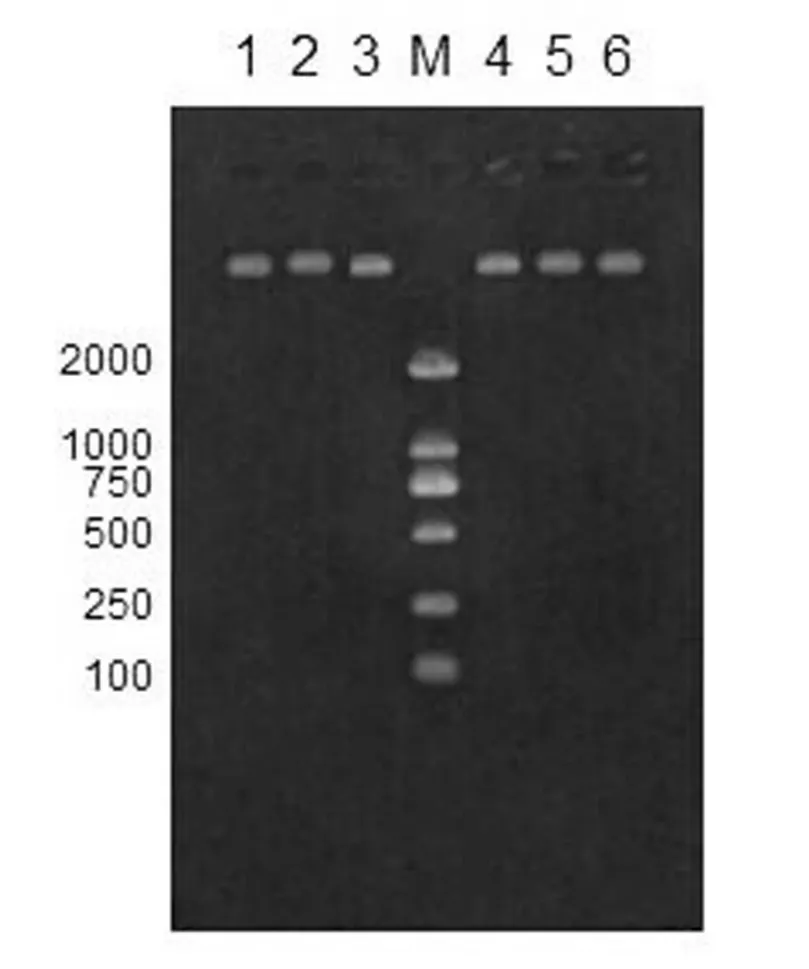
图1 样品基因组DNA提取电泳检测结果Fig.1 The test results of genomic DNA extraction electrophoresis
由图1可以看出,以桑黄摇床培养菌丝为出发材料,参照天根生化科技有限公司的植物基因组提取DNA试剂盒提取方法,所得基因组DNA经琼脂糖电泳主带清晰,无拖尾、降解现象,质量好,完全能满足后续PCR扩增对样品基因组DNA质量的要求。样品rDNA ITS序列扩增电泳检测结果见图2。
由图2可以看出,以桑黄菌丝体基因组DNA为模板,ITS1、ITS4为引物,PCR扩增出的目的片段条带单一清晰,6个测试样品扩增产物长度分布在750 bp~800 bp范围内。

图2 样品rDNA ITS序列扩增电泳检测结果Fig.2 The test results of rDNA ITS sequence amplification electrophoresis
2.2 桑黄rDNA ITS序列分析
对桑黄rDNA ITS扩增目标产物进行回收、克隆、测序,用DNA Man软件分析6个样品序列精确长度,桑-W、桑-F、桑-D、桑-Y、桑-H均为757 bp,桑-H1为796 bp。通过GenBank数据库BLAST同源性比对,桑-W、桑-F、桑-D、桑-Y、桑-H与Inonotus linteus最为接近,相似性分别为98.23%、98.23%、98.10%、98.23%、98.23%;桑-H1与Fuscoporia qilva最为接近,相似性为95.90%。
2.3 基于rDNA ITS序列的系统进化树
基于rDNA ITS序列构建系统进化树,见图3。
由图3可知,依据rDNA ITS序列信息进行系统进化树的构建,发现桑-W、桑-F、桑-D、桑-Y、桑-H与Inonotus linteus聚在一起,桑-H1与Fusco poria qilva聚在一起。系统进化树得到的结果与供试菌株rDNA ITS全序列信息分析结果表现出较高的一致性,进一步说明了系统进化树的可靠性。
3 讨论
ITS rDNA区进化速率较快,在绝大多数真核生物中表现出极为广泛的序列多态性,对高等植物和大型真菌鉴定有更好的准确性和灵敏性,适用于不同分类水平的物种鉴定及系统发育分析[5-6]。由于桑黄真菌各物种间形态差异不大,通过形态鉴定较为困难。利用rDNA ITS序列分析技术,可以直接提取蕴含于基因组内的遗传信息,检测和比较传统形态学无法区分的相似菌株种间或种内差别,具有快速、灵敏、准确等优点。
本研究应用ITS序列分析技术,对6株桑黄菌rDNA ITS区段进行克隆测序,并在GenBank数据库中对所有序列进行比对分析和构建系统发育树,结果显示,桑-W、桑-F、桑-D、桑-Y、桑-H与Inonotus linteus(裂蹄木层孔菌)最为接近,相似性分别为98.23%、98.23%、98.10%、98.23%、98.23%,其序列长度均为757 bp;桑-H1与Fuscoporia qilva最为接近,相似性为95.90%,其序列长度均为796 bp。因此,可以确定桑-W、桑-F、桑-D、桑-Y、桑-H是裂蹄木层孔菌(也称Phellinus linteus),5株菌是可以入药的桑黄菌。
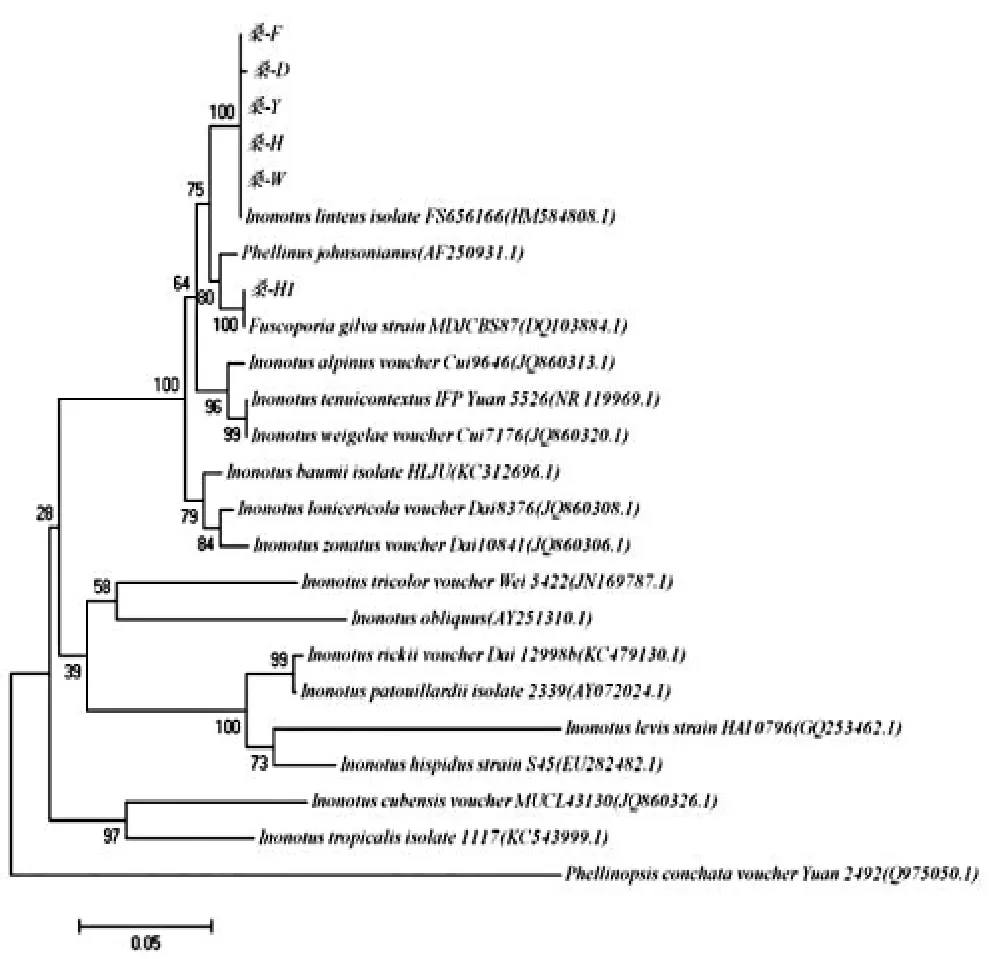
图3 基于rDNA ITS序列的系统进化树Fig.3 Phylogenetic tree based on rDNA ITS sequence
[1]张小青,戴玉成.中国真菌志 [M].北京:科学出版社,2005:117-191.
[2]刘艳芳,杨焱,贾薇,等.药用真菌桑黄总黄铜测定方法研究 [J].食用菌学报,2006,13(2):45-48.
[3]江苏新医学院.中药大辞典 [M].上海:上海科学技术出版社,1995,1976.
[4]Iikekawa T,Nakanishi M,Uehara N.Antitumor action of some basidiomycetes,especially Phellinus linteus[J].Gann(Japanese Journal of Cancer Research),1968,59(9):155-157.
[5]王建波,张文驹,陈家宽.核rDNA的ITS序列在被子植物系统与进化研究中的应用 [J].植物分类学报,1999,37(4):407-416.
[6]Agarwal M,Shrivastava N,Padh H.Advances in molecular marker techniques and their applications in plant sciences[J]. Plant Cell Reports,2008,27(4):617-631.
Analysis on Sequence and Genetic Relationship of rDNA ITS of Wild Phellinus in Qinba Mountains
ZHANG Wen-jun,WU Ya-zhao,LEI Ping,DU Fang
(Shaanxi Microbioogy Rereach Institute,Xi’an 710043,China)
The ITS sequences of six wild Phellinus strains from Qiaba mountain district were sequenced and analyzed by ITS method.Moreover,ITS sequences were followed by sequence comparison with GenBank database using the basic local alignment search tool algorithm.The retrieval sequences of 18 most similar species along with six different Phellinus strains was analyzed for system development.Results showed Inonotus linteus with Sang-W,Sang-F,Sang-D,Sang-Y and Sang-H had closest genetic relationship,the similarity was 98.23%,98.23%,98.10%,98.23%,98.23%,respectively,and the sequence length was 757 bp.Inonotus linteus with Sang-H had the most similarity with Fuscoporia qilva(95%).The sequence length was 796 bp.
Phellinus;rDNA ITS;sequence analysis;genetic relationship
S646.9
A
1003-8310(2015)01-0050-03
10.13629/j.cnki.53-1054.2015.01.014
陕西省科技厅自然科学基础研究计划项目(2013JM3021);陕西省科技厅农业攻关计划项目(2013K01-22);西安市科技局现代农业创新计划 [NC1320(1)];西安市科技局技术转移促进工程 [CXY1348(4)];陕西省科学院应用基础研究项目(2014K-14);陕西省科学院重点项目与产业化专项(2013K-03)。
张文隽(1977-)女,硕士,助理研究员,主要从事药用真菌研究与开发。
**通信作者:雷萍(1966-),女,本科,副研究员,主要从事药用真菌研究与开发。E-mail:wuleiping2529@126.com
2014-11-16
