基于线粒体控制区的中华绒螯蟹群体遗传多样性分析
2015-02-27龙宋娜王俊鹿志创高天翔
闫 龙宋 娜王 俊鹿志创高天翔
(1. 中国海洋大学海洋生物多样性与进化研究所, 青岛 266003; 2. 中国水产科学研究院黄海水产研究所, 青岛 266002; 3. 辽宁省海洋水产科学研究院, 大连 116000; 4. 浙江海洋学院水产学院, 舟山 316022)
基于线粒体控制区的中华绒螯蟹群体遗传多样性分析
闫 龙1宋 娜1王 俊2鹿志创3高天翔4
(1. 中国海洋大学海洋生物多样性与进化研究所, 青岛 266003; 2. 中国水产科学研究院黄海水产研究所, 青岛 266002; 3. 辽宁省海洋水产科学研究院, 大连 116000; 4. 浙江海洋学院水产学院, 舟山 316022)
中华绒螯蟹(Eriocheir sinensis H. Milne Edwards)隶属于十足目(Decapoda)、方蟹科(Grapsidae)、绒螯蟹属(Eriocheir), 广泛分布于我国沿海各大水系[1]。中华绒螯蟹肉味鲜美、营养价值高, 是我国重要的水产经济物种。目前, 中华绒螯蟹的增殖放流和水产养殖等生产活动的开展已造成不同水系种质资源严重混杂[2,3]。因此, 为深入了解不同水系中华绒螯蟹的遗传特征和种质差异, 有效管理利用和保护中华绒螯蟹资源, 对不同水系中华绒螯蟹的遗传多样性和群体遗传结构研究势在必行。
分子生物学技术的发展为研究生物的遗传变异提供了有效的手段。进化速度快、较小的基因组、结构简单而稳定、世代传递过程中几乎不发生重组、无组织特异性、单性母系遗传等特征使线粒体DNA(mtDNA)成为进化和群体遗传学研究的有效标记[4,5]。线粒体DNA控制区(D-loop)因缺乏编码选择压力而成为线粒体基因组中进化速率最快的基因, 比线粒体PNA其他基因片段更适合用于种间或种下水平的遗传学研究[6,7]。
目前, 国内外学者已对中华绒螯蟹的形态学和遗传学开展了大量研究。形态学方面, 有学者运用多元统计方法对中国沿海不同水域中华绒螯蟹的形态差异进行研究[8,9],唐伯平等[10]基于光镜和扫描电镜对中华绒螯蟹的触角形态特征进行了研究; 遗传学方面, Sui等[11]以及Xu和Chu[12]分别运用微卫星(SSR)和扩增片段长度多态性(AFLP)核基因标记对国内各大水系的中华绒螯蟹开展了群体遗传学分析; 另有诸多学者运用线粒体COⅠ、Cyt b等基因片段对中华绒螯蟹开展了群体遗传学研究[13-15]。迄今, 尚未见mtDNA控制区序列在中华绒螯蟹群体遗传学研究中的报道。本研究采用mtDNA控制区高变区序列对中华绒螯蟹四个野生群体的遗传多样性及群体遗传结构展开研究, 以揭示不同水系中华绒螯蟹的遗传差异,探讨其遗传多样性水平, 为中华绒鳌蟹资源的合理开发利用与保护提供理论依据。
1 材料与方法
1.1 实验材料
本研究所用中华绒螯蟹样品于1999年 7月至 2013年 10月间分别采自丹东(DD)、盘锦(PJ)、垦利(KL)、南京(NJ), 采用地点如图1所示。另外, 我们还采集了广西北海合浦绒螯蟹(Eriocheir hepuensis)19个个体作为外群来进行比较分析。样品采样方式为天然水域抓捕或地笼网捕捉, 新鲜或冷冻样品送至中国海洋大学渔业生态实验室低温保存备用。
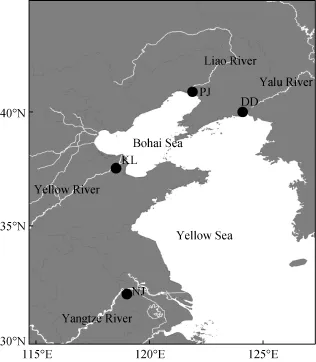
图1 中华绒螯蟹采样地点Fig. 1 Sampling sites of E. sinensis
1.2 DNA提取与PCR扩增
取中华绒螯蟹螯足肌肉组织约100 mg, 经细胞裂解液STE和蛋白酶K完全消化, 采用酚-氯仿抽提法提取基因组DNA, 100 μL超纯水溶解, 4℃保存。
根据GenBank发布的中华绒螯蟹线粒体基因组全序列[16]设计中华绒螯蟹和合浦绒螯蟹控制区高变区引物序列为YL-F: 5′-ACGTAACTGAAATGCTGTTC-3′和YL-R: 5′-ACCCGTTTCCCCTCTAGAGGA-3′。PCR反应体系为25 μL, 包括: 基因组DNA(20 ng), 1.25 U Taq DNA聚合酶(大连宝生物工程有限公司), 200 nmol/L的正反向引物, 200 μmol/L的每种dNTP, 10 mmol/L Tris pH 8.3, 50 mmol/L KCl和1.5 mmol/L MgCl2(大连宝生物工程有限公司)。PCR循环参数为: 95℃预变性5min, 95℃变性45s, 50℃退火50s, 72℃度延伸1min, 循环35次, 然后72℃后延伸10min。所有PCR均在Biometra热循环仪上完成。每组PCR均设阴性对照用来检测是否存在污染, 取2 μL PCR扩增产物进行1%琼脂糖凝胶电泳检测, 用UNIQ-10柱式DNA凝胶回收试剂盒对目的片段进行回收纯化, 然后送生物公司进行双向测序。
1.3 数据分析
运用DNAStar(DNASTAR, inc, Madison, USA)软件包对序列进行比对排序并辅以人工校正。运用MEGA4.0[17]计算序列的碱基组成、多态性、序列间的转换颠换比。利用 PAUP和 Modeltest3.7[18]软件筛选核苷酸最佳替代模型并计算不同位点间异质性的gamma分布性状参数。以合浦绒螯蟹为外群, 基于模型筛选所得参数在 MEGA4.0软件中构建 NJ系统发育树, 系统树的可靠性采用 1000次重抽样评估。利用Arlequin3.0[19]软件计算单倍型数、单倍型多样度、核苷酸多样度等遗传学参数; 计算两两群体间的遗传分化指数 FST值, 并进行分子差异分析(AMOVA)和确切P检验, 以评估中华绒螯蟹的群体遗传结构。
2 结果
2.1 序列组成与变异分析
对4个群体共84个中华绒螯蟹个体扩增后所得目的片段长度为 475 bp, 其中包括 18 bp的 tRNA序列和457 bp的控制区高变区序列。18 bp的tRNA序列内无变异位点, 457 bp的控制区高变区共检测到58个多态位点, 31个简约信息位点, 47处转换和9处颠换(转换颠换比为5.2), 同时有 8处插入/缺失。在碱基组成上, A+T含量(81.58%)明显高于G+C含量(18.42%)。58个多态位点定义了41个单倍型, 包括28个独有单倍型和13个共享单倍型。共享单倍型中6个单倍型被两个以上群体的个体所共享。Hap.16是分布最普遍的单倍型, 被三个群体的 13个个体共享, 丹东群体和南京群体之间没有出现共享单倍型(表2)。合浦绒螯蟹与中华绒螯蟹在线粒体控制区序列上存在较大差异。19个合浦绒螯蟹的目的片段长度为476 bp, 在18 bp的tRNA序列内也无变异位点, 在458 bp的控制区高变区序列共检测到55个多态位点, 定义了19个单倍型, 其中包括 44处转换和2处颠换, 转换颠换比为22, 同时有9处插入/缺失。在碱基组成上, A+T含量(80.13%)明显高于G+C含量(19.87%)。两种绒螯蟹控制区的A+T含量均高于G+C, 但中华绒螯蟹的G+C含量更低。
总体来看, 中华绒螯蟹遗传多样性处于较高水平,单倍型多样度、核苷酸多样度平均值分别为 0.958±0.010和0.016±0.009(表1)。南京群体的单倍型多样度为0.848± 0.070, 核苷酸多样度为 0.010±0.006, 略低于其他三个群体。四个群体遗传多样性水平从高到低依次为: 垦利>盘锦>丹东>南京。合浦绒螯蟹的单倍型多样度为1.000±0.017,核苷酸多样度为0.027±0.014, 均高于中华绒螯蟹。
2.2 群体遗传结构分析
两两群体间遗传分化指数 FST值显示: 丹东群体与其他群体间FST值较大, 且经Bonferroni校正后差异均显著(P<0.05); 盘锦群体与其他群体间 FST值较小, 经Bonferroni校正后差异显著(P<0.05); 垦利群体和南京群体之间FST值最小, 且差异不显著(P>0.05)(表3)。将所有群体作为一个组群进行 AMOVA分析的结果显示: 组内
群体间遗传差异极显著(ΦST=0.0681, P=0)。为进一步检测群体遗传结构, 运用确切 P检验来揭示群体间是否存在随机交配现象。在 0.05置信水平下, 除垦利群体和南京群体之间存在随机交配现象外, 其他两两群体间都不存在随机交配(P<0.05)(表3)。以上结果均表明: 丹东群体与其他三个群体间存在中度且显著的遗传分化, 盘锦群体与垦利、南京群体间存在微弱但显著的遗传分化, 垦利群体和南京群体之间遗传差异最小且不显著。

表1 中华绒螯蟹各群体的采样信息和遗传多样性参数Tab. 1 The sample information and genetic diversity parameters of different populations for E. sinensis

表2 中华绒螯蟹单倍型在不同群体间分布情况Tab. 2 Distribution of haplotype of populations of E. sinensis

表3 中华绒螯蟹群体间的遗传分化指数(下对角线)和确切P值(上对角线)Tab. 3 Pairwise FST(below diagonal) and exact P values (above diagonal) among E. sinensis populations
通过PAUP和Modeltest软件计算得到中华绒螯蟹控制区的核苷酸最佳替代模型为 TVM+I+G(I=0.54, G=0.97)。以合浦绒螯蟹为外群, 基于筛模型所得参数构建的NJ系统发育树显示: 中华绒螯蟹邻接关系树的拓扑结构比较简单, 没有检测到与采样地点相对应的分支,不存在显著的谱系结构(图 2)。单倍型网络图与 NJ系统发育树的结果一致, 也没有出现与地理分布相对应的谱系结构(图3)。
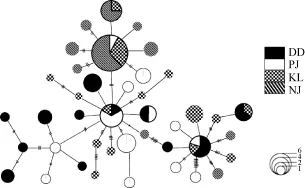
图2 中华绒螯蟹单倍型网络关系图Fig. 2 Reduced median-network showing genetic relationship in E. sinensis
3 讨论
遗传多样性的高低是评价物种对环境适应能力和物种进化潜力的重要依据。遗传多样性越丰富, 物种对环境变化的适应能力越强, 遗传多样性的贫乏会导致物种低的进化适应性, 使得物种易于遭受生态环境改变的影响[20]。种群遗传结构是指遗传多样性在种内和种间的分布, 即遗传分化。掌握种群的遗传结构可以明确物种在分布范围内是否存在随机交配, 确定物种的管理单元[21]。20世纪70年代以来, 随着河蟹养殖业的蓬勃发展, 商品蟹流通、养殖亲蟹移植等人为因素打破了各水系中华绒螯蟹天然分布格局, 导致不同水系中华绒螯蟹种质资源混杂、退化情况越来越严重[22—24]。因此, 准确了解中华绒螯蟹的遗传多样性水平和群体遗传结构可以为其资源的可持续利用与保护提供依据。

图3 基于邻接法构建的中华绒螯蟹系统发育树Fig. 3 Phylogenetic tree in E. sinensis based on neighbor joining method
本研究结果表明, 中华绒螯蟹的控制区序列的碱基组成与三疣梭子蟹[25]以及合浦绒螯蟹一致, 都表现出A+T含量明显偏高的特点。核苷酸多样度是衡量线粒体DNA遗传多样性的一个重要指标。本研究中丹东、盘锦、垦利三个群体的核苷酸多样度处于较高水平, 南京群体的核苷酸多样度明显低于其他三个群体, 但维持在中等水平。运用Cyt b基因的研究结果也表明南京中华绒螯蟹的核苷酸多样度明显低于其他水系[26]。这可能与长江水系中华绒螯蟹过度捕捞利用有关, 长期的过度捕捞和水体污染、生境破坏等造成中华绒螯蟹有效种群数量减少,可能经历了瓶颈效应[27,28], 导致南京群体核苷酸多样度低于其他群体。
有关中华绒螯蟹群体的遗传多样性研究报道较多,运用不同分子标记得到的结果有所差异。运用AFLP、RAPD等的研究表明中华绒螯蟹的遗传多样性处于较低水平[12,14,29]; 而运用微卫星和Cyt b的研究表明中华绒螯蟹的遗传多样性水平较高[11,13]。本研究结果支持中华绒螯蟹遗传多样性处于较高水平的结论。本研究4个中华绒螯蟹群体的单倍型多样度水平均较高, 南京群体的单倍型多样度虽略低于其他三个群体, 但差异不明显。中华绒螯蟹广泛分布于中国沿海各大水系且具有较强的繁殖力和环境适应能力, 大的有效群体可能是造成中华绒螯蟹遗传多样性水平较高的原因。另外, 多年来不同水系中华绒螯蟹之间频繁的基因交流在加剧了各水系中华绒螯蟹混杂的同时[23—25], 也可能在一定程度上提高了中华绒螯蟹的遗传多样性。
Sui等[11]和Chang等[30]运用SSR对中华绒螯蟹群体遗传学度研究表明: 辽河水系与黄河水系、长江水系中华绒螯蟹群体之间存在显著的遗传差异, 而黄河水系与长江水系中华绒螯蟹群体间遗传差异不明显。本研究结果与上述研究结果相近, 除垦利与南京中华绒螯蟹群体间遗传分化不显著外, 其他两两群体之间都存在不同程度的遗传差异。中华绒螯蟹有限的扩散能力和不同群体沿岸距离较远可能是造成群体间产生显著遗传分化的主要原因。虽然中华绒螯蟹蚤状幼体可随海流沿岸被动迁移, 但其蚤状幼体的浮游期短且中国沿岸海流流速缓慢[31,32], 可能尚不足以使不同水系中华绒螯蟹群体间产生基因交流[11]。生存环境的差异和地理隔离使得不同地理群体为适应各自环境而发生定向变异, 经过长期积累而最终出现遗传分化。虽然四个中华绒螯蟹群体之间存在一定的遗传结构,但不同地理群体之间还未能积累足够多的遗传变异。因此NJ系统树和单倍型网络图均未检测到与采样地点相对应的谱系结构。商品蟹流通、增殖放流和人工养殖中亲蟹及苗种的异地使用可能是导致南京群体和垦利群体间不存在遗传分化的主要原因。
综上所述, 丹东、盘锦、垦利三个中华绒螯蟹群体的遗传多样性均处于较高水平。南京群体的遗传多样性略低于其他三个群体, 需尽快采取措施对其野生资源进行保护, 以实现资源的可持续开发。不同水系中华绒螯蟹群体间存在一定程度的遗传分化, 必须建立科学有效的监督管理体系, 防止盲目的人工移植和引种导致各水系中华绒螯蟹种质资源退化和优良性状的丢失, 保证中华绒螯蟹野生资源得到有效保护和合理开发利用。
致谢:
感谢南京市农林局渔政处孙希福在采样过程中提供的帮助。感谢李宁博士和李渊在数据分析方面给予的帮助。
[1] Wang Q, Li X D, Dai W, et al.The morphological discrimination of populations of mitten crab in different river systems [J]. Fisheries Science, 2006, 25(3): 125—128 [王茜,李晓东, 戴伟, 等. 不同水系绒螯蟹群体外部形态的判别分析. 水产科学, 2006, 25(3): 125—128]
[2] Tang B P, Zhou K Y, Song D X. Biodiversity of the mitten crab Eriocheir [J]. Journal of Hebei University (Nature Science Edition), 2000, 20(3): 304—308 [唐博平, 周开亚, 宋大祥. 绒螯蟹属的生物多样性. 河北大学学报(自然科学版), 2000, 20(3): 304—308]
[3] Gu X H, Zhao F H. Resources and culturing situation of Chinese mitten crab (Eriocheir sinensis) and species character conservation [J]. Journal of Lake Sciences, 2001, 13(3): 267—271 [谷孝鸿, 赵福顺. 长江中华绒螯蟹的资源与养殖现状及其种质保护. 湖泊科学, 2001, 13(3): 267—271]
[4] Peng J L, Wang X Z, He S P. The progress and application of DNA barcoding [J]. Acta Hydrobiologica Sinica, 2008, 32(6): 916—919 [彭居俐, 王绪桢, 何舜平. DNA条形码技术的研究进展及其应用. 水生生物学报, 2008, 32(6): 916—919]
[5] Sbisa E, Tanzariello F, Reyes A, et al.Mammalian mitochondrial D-loop region structure analysis: identification of new conserved sequences and their functional and evolutionary implications [J]. Gene, 1997, 205(1-2): 125—140
[6] Xiao W H, Zhang Y P. Genetics and evolution of mitochondrial and in fish [J]. Acta Hydrobiologica Sinica, 2000, 24(4): 384—391 [肖武汉, 张亚平. 鱼类线粒体DNA的遗传与进化. 水生生物学报, 2000, 24(4): 384—391]
[7] Diniz F M, Maclean N, Ogawa M, et al. The hypervariable domain of the mitochondrial control region in Atlantic spiny lobsters and its potential as a marker for investigating phylogeographic structuring [J]. Marine Biotechnology, 2005, 7(5): 462—473
[8] He J, Xu P, Zhu J. Morphological variations and species validity of Eriocheir sinensisin the north and south drainage systems of china [J]. Transactions of Oceanology and Limnology, 2009, 3: 79—86 [何杰, 徐跑, 朱健. 南北水系中华绒螯蟹形态差异分析. 海洋湖沼通报, 2009, 3: 79—86]
[9] Li C H, Li S F. Phylogenies of populations of mitten crabs (Eriocheri sinensis, Eriocheir japonica) in six river systems of mainland China: morphology discriminant analysis [J]. Journal of Fisheries of China, 1999, 23(4): 337—342 [李晨虹, 李思发. 中国大陆沿海六水系绒螯蟹(中华绒螯蟹和日本绒螯蟹)群体亲缘关系: 形态判别分析. 水产学报, 1999, 23(4): 337—342]
[10] Tang B P, Chen L Q, Zhou K Y, et al. On the morphological characters of mitten crabs antennae and its application in the classification [J]. Acta Zootaxonomica Sinica, 2009, 34(1): 79—86 [唐伯平, 陈立侨, 周开亚, 等. 绒螯蟹触角形态特征及其在分类中的应用. 动物分类学报, 2009, 34(1): 79—86]
[11] Sui L, Zhang F, Wang X, et al. Genetic diversity and population structure of the Chinese mitten crab Eriocheir sinensis in its native range [J]. Marine Biology, 2009, 156(8): 1573—1583
[12] Xu J, Chu K H. Genome scan of the mitten crab Eriocheir sensu stricto in East Asia: Population differentiation, hybridization and adaptive speciation [J]. Molecular Phylogenetics and Evolution, 2012, 64(1): 118—129
[13] Wang C H, Li C, Li S. Mitochondrial DNA inferred population structure and demographic history of the mitten crab (Eriocheir sensu stricto) found along the coast of mainland China [J]. Molecular Ecology, 2008, 17(15): 3515—3527
[14] Ge J C, Xu Z Q, Li X H, et al. Genetic characters of Eriocheir sinensis populations from four water systems revealed by mitochondrial COI gene sequence [J]. Journal of Fishery Sciences of China, 2011, 18(1): 16—22 [葛家春, 许志强,李晓晖, 等. 利用线粒体COI序列分析4水系中华绒螯蟹群体遗传学特征. 中国水产科学, 2011, 18(1): 16—22]
[15] Zhang F Y, Ma L B. Study on partial sequences comparison of mtl2SrDNA gene of Chinese mitten crabs from the Changjiang, Liaohe and Oujiang Rivers [J]. Marine Fisheries, 2004, 26(2): 147—151 [张凤英, 马凌波. 长江、辽河、瓯江三水系中华绒螯蟹mt12SrDNA片断序列的比较. 海洋渔业, 2004, 26(2): 147—151]
[16] Sun H, Zhou K, Song D. Mitochondrial genome of the Chinese mitten crab Eriocheir japonica sinensis (Brachyura: Thoracotremata: Grapsoidea) reveals a novel gene order and two target regions of gene rearrangements [J]. Gene, 2005, 349: 207—217
[17] Tamura K, Dudley J, Nei M, et al. MEGA4: molecular evolutionary genetics analysis (MEGA) software version 4.0 [J]. Molecular Biology and Evolution, 2007, 24(8): 1596—1599
[18] Posada D, Crandall K A. Modeltest: testing the model of DNA substitution [J]. Bioinformatics, 1998, 14(9): 817—818
[19] Excoffier L, Laval G, Schneider S. Arlequin (version 3.0): an integrated software package for population genetics data analysis [J]. Evolutionary Bioinformatics, 2005, 1: 47
[20] Chen L Z. Biodiversity Status and Protection Measures of China [M]. Beijing: Science Press. 1993, 93—113 [陈灵芝.中国的生物多样性现状及其保护对策. 北京: 科学出版社. 1993, 93—113]
[21] Waples R S. Separating the wheat from the chaff: patterns of genetic differentiation in high gene flow species [J]. Journal of Heredity, 1998, 89(5): 438—450
[22] Wang Y P, Zhao N G. The culture of Chinese mitten crab and the prospect of its development [J]. Acta Hydrobiologica Sinica, 1999, 23(6): 735—740 [王怡平, 赵乃刚. 中国的河蟹养殖及其发展前景. 水生生物学报, 1999, 23(6): 735—740]
[23] Zhang X M, Liu G D, Gao T X. Advance in the study of Eriocheir germplasm resource [J]. Journal of Ocean University of Qingdao, 2002, 32(4): 533—542 [张秀梅, 柳广东,高天翔. 绒螯蟹种质资源研究进展. 青岛海洋大学学报:自然科学版, 2002, 32(4): 533—542]
[24] Zheng F, Lü X L, Sun H Y, et al. Application of ISSR markers in germplasm analyzing of mitten crab [J]. Journal of Fishery Sciences of China, 2007, 14(1): 46—51 [郑芳, 吕秀玲, 孙红英, 等. ISSR标记在河蟹种质检测中的应用. 中国水产科学, 2007, 14(1): 46—51]
[25] Yamauchi M M, Miya M U, Nishida M. Complete mitochondrial DNA sequence of the swimming crab, Portunus trituberculatus (Crustacea: Decapoda: Brachyura) [J]. Gene, 2003, 311: 129—135
[26] Xu J, Chan T Y, Tsang L M, et al. Phylogeography of the mitten crab in East Asia: Pleistocene isolation, population expansion and secondary contact [J]. Molecular Phylogenetics and Evolution, 2009, 52(1): 45—56
[27] Wang Z Q, Huang S, Mao H C, et al. Genetic differentiation analysis of the even and odd year populations of Chinese mitten crab [J]. Journal of Shanghai Ocean University, 2013, 22(5): 657—664 [王中清, 黄姝, 茅海成, 等. 中华绒螯蟹奇、偶年天然群体的遗传差异分析. 上海海洋大学学报, 2013, 22(5): 657—664]
[28] Sbordoni V, De Matthaeis E, Cobolli Sbordoni M, et al. Bottleneck effects and the depression of genetic variability in hatchery stocks of Penaeus japonicas (Crustacea, Decapoda) [J]. Aquaculture, 1986, 57(1): 239—251
[29] Gao Z Q, Zhou K Y. Genetic variation of the Chinese mitten-handed crab (Eriocheir sinensis) populations detected by RAPD analysis [J]. Biodiversity Science, 1998, 6(3): 186—190 [高志千, 周开亚. 中华绒螯蟹遗传变异的RAPD分析. 生物多样性, 1998, 6(3): 186—190]
[30] Chang Y, Liang L, Ma H, et al. Microsatellite analysis of genetic diversity and population structure of Chinese mitten crab (Eriocheir sinensis) [J]. Journal of Genetics and Genomics, 2008, 35(3): 171—176
[31] Anger K. Effects of temperature and salinity on the larval development of the Chinese mitten crab Eriocheir sinensis (Decapoda: Grapsidae) [J]. Marine Ecology Progress Series, 1991, 72(1): 103—110
[32] Wei Q S, Yu Z G, Ran X B, et al. Characteristics of the Western coastal current of the Yellow Sea and its impacts on material transportation [J]. Advance in Earth Science, 2011, 26(2): 145—156 [韦钦胜, 于志刚, 冉祥滨, 等.黄海西部沿岸流系特征分析及其对物质输运的影响.地球科学进展, 2011, 26(2): 145—156]
STUDIES ON GENETIC DIVERSITY OF CHINESE MITTEN CRAB ERIOCHEIR SINENSIS BASED ON MITOCHONDRIAL DNA CONTROL REGION
YAN Long1, SONG Na1, WANG Jun2, LU Zhi-Chuang3and GAO Tian-Xiang4
(1. Institute of Evolution & Marine Biodiversity, Ocean University of China, Qingdao 266003, China; 2. Yellow Sea Fisheries Research Institute Chinese Academy of Fishery Sciences, Qingdao 266002, China; 3. Liaoning Ocean and Fisheries Science Research Institute, Dalian 116000, China; 4. School of Fishery, Zhejiang Ocean University, Zhoushan 316022, China)
中华绒螯蟹; 线粒体控制区; 单倍型多样度; 核苷酸多样度; 遗传结构; 遗传多样性
Chinese mitten crab; mtDNA control region; Haplotype diversity; Nucleotide diversity; Genetic structure; Genetic diversity
Q349+.1
A
1000-3207(2015)03-0615-06
10.7541/2015.81
2014-05-26;
2014-08-25
国家公益性行业(农业)科研专项(201303050)资助
闫龙(1988—), 男, 甘肃高台人; 硕士; 主要从事水生生物系统发育与遗传多样性研究。E-mail: 307605811@qq.com
高天翔, E-mail: gaotianxiang0611@163.com
