草鱼CD81基因的克隆及功能分析
2015-02-27李华英黄容杜富宽廖兰杰李勇明汪亚平
李华英黄 容杜富宽廖兰杰李勇明汪亚平
(1. 中国科学院水生生物研究所, 淡水生态与生物技术国家重点实验室, 武汉 430072; 2. 中国科学院大学, 北京 100049)
草鱼CD81基因的克隆及功能分析
李华英1,2黄 容1杜富宽1廖兰杰1李勇明1汪亚平1
(1. 中国科学院水生生物研究所, 淡水生态与生物技术国家重点实验室, 武汉 430072; 2. 中国科学院大学, 北京 100049)
为研究白细胞表面分化抗原81(CD81)的功能, 对草鱼CD81进行了克隆, CD81全长共1376 bp, 其中5′非翻译区87 bp, 3′非翻译区581 bp, 开放阅读框为708 bp, 包括8个外显子, 7个内含子, 编码235个氨基酸。实验采用实时荧光定量PCR的方法检测了CD81在健康草鱼不同组织中的表达情况及草鱼出血病病毒(GCRV)攻毒前后的表达变化情况。结果显示草鱼 CD81在所有被检测组织中均有表达, 在头肾中表达量最高。在GCRV攻毒前后草鱼鳃、脾、肝、肠及头肾5个组织中的CD81表达量均有明显变化。同时, 采用绿色荧光蛋白(GFP)来示踪CD81的亚细胞表达部位, 激光共聚焦显微镜显示, 同人类一样, 草鱼CD81定位于细胞膜上。
草鱼; CD81; 四跨膜蛋白家族; 基因克隆; 组织表达; 亚细胞定位
白细胞表面分化抗原81(CD81)是四跨膜蛋白家族(TM4SF)的重要成员之一。TM4SF是一类包含四次跨膜结构域的蛋白家族, 他们主要表达在细胞膜上, 其蛋白N端与C端均在细胞质内部, 包含两个大小不同的胞外环[1]。研究表明TM4SF可以跟 MHC分子、整合素、CD4/CD8及B细胞受体复合物相互作用[2], 在细胞生长、分化、增殖、细胞间融合、黏附信号传导等过程中均发挥作用[3—10]。TM4SF并没有明显的信号基序和模块, 它们可以装配形成多蛋白复合物以便形成广泛的网络参与细胞作用, 所有的TM4SF间均存在直接的相互作用, 并可与第三者CD81相互作用[11]。
CD81在包括免疫在内的多种生理过程中发挥重要功能。研究证实CD81与T、B细胞分化有关, 缺失CD81的小鼠, 其B细胞功能异常, 但增强了T细胞的增殖[12]。也有证据显示缺失CD81的小鼠, T、B细胞数目正常, 但在抗原入侵时, 抗体生成过程推迟[13]。CD81分子在不同病毒入侵过程中也发挥着重要作用。王全楚和聂青和[14]提到CD81与曼氏血吸虫和日本血吸虫的某些抗原, 如SM23和Sj2有结构上的同源性。在人类的研究中, 也已证明CD81作为丙型肝炎病毒(HCV)的受体, 可以与HCV的E2区结合[15]。不同CD81分子表达水平的细胞系在低HCV数量感染细胞时, 表现出不同的敏感性[16]。
到目前为止, CD81已经在包括斑马鱼(Danio rerio)、斑点叉尾(Ictalurus punctatus )、鲈(Lateolabrax japonicus)、大西洋鲑(Salmo salar)、七鳃鳗(Lampetra japonica)等鱼类中得到克隆, 实验也证实同人类一样CD81也参与了七鳃鳗的免疫过程, 而斑点叉尾在受到爱德华杆菌感染后CD81出现了相应的上调表达[17—20]。草鱼在我国是一种重要的经济鱼类, 但其养殖常因出血病等各种病毒感染受到影响, 因此本研究克隆了草鱼中的CD81基因, 并对其进行了初步的功能研究, 希望为草鱼染病机制的研究提供一些线索。
本研究采用基因组搜索、ORF预测、同源扩增及cDNA末端快速扩增(RACE)等多种方法相结合克隆得到了CD81全长cDNA序列, 并对其结构特征进行分析探讨; 采用实时荧光定量的方法检测了CD81的表达, 同时采用GFP融合蛋白的方法对CD81的表达部位进行了亚细胞定位, 以期为草鱼免疫相关基因的克隆研究奠定基础。
1 材料与方法
1.1 实验鱼及草鱼CIK细胞
在本实验中所有DNA及RNA提取材料均取自健康1龄草鱼, 由中国科学院水生生物研究所官桥养殖基地提供, 剪尾鳍提取DNA, 取腮、脑、肝、脾、中肾、头肾和后肠提取总RNA。实验中所用CIK细胞购自武汉大学细胞保藏中心, 在28℃、5%CO2浓度的培养条件下培养。
1.2 病毒液制备及草鱼攻毒
挑选感染GCRV病毒症状非常典型的草鱼, 弃内脏, 按照其体重的3倍加入0.7%的生理盐水, 用搅拌机制备成组织匀浆液; 28 ℃, 140 r/min,摇床震荡30min, 让病毒尽量施放出来; 然后冷冻离心, 4000×g, 30min; 收集上清液即为病毒液。储存于–70℃。
选取健康1龄草鱼, 采用腹腔注射法进行攻毒,每100 g鱼注射2 mL病毒液, 分别于攻毒前、攻毒后2d、10d和18d各取3尾草鱼, 每条鱼取鳃、肝、脾、后肠、中肾和头肾6个组织, 每个时间点3尾鱼的相同组织混合, 加入Trizol裂解液中。
1.3 总RNA的提取及cDNA第一链的合成
将取自健康1龄草鱼的组织加入Trizol (Invitrogen, USA), 充分研磨, 按照Trizol法提取总RNA。提取的RNA经DnaseⅠ(Promega, USA)消化后用琼脂糖凝胶电泳和分光光度计(Eppendorf, Germany)方法检测其质量和浓度。使用 Revertra ace (TOYOBO, JAPAN)利用oligo dT (Invitrogen, USA)反转得到3′RACE文库, 使用SMARTer™RACE cDNA Rapid Amplification (Clontech, USA)的试剂盒由特异性引物合成5′RACE文库, –20℃保存。
1.4 基因组DNA提取
剪取健康草鱼的尾鳍, 加入抽提液(10 mmol/L Tris-Cl, pH 8.0; 1 mmol/L EDTA, pH 8.0; 0.5% SDS; 100 µg/mL蛋白酶K)匀浆后, 55℃消化2—3h, 然后采用酚-氯仿-异戊醇方法进行抽提, 用乙醇沉淀纯化, 最后将沉淀溶于ddH2O。用琼脂糖凝胶电泳和分光光度计检测其质量和浓度, –20℃保存。
1.5 基因组搜索及ORF的预测与验证
在NCBI上下载斑马鱼CD81序列, 利用putty软件在本实验室草鱼全基因组(未发表)序列中搜索包含CD81的草鱼基因组Contig, 利用http://genes.mit. edu/GENSCAN.html外显子从头预测在线软件对CD81ORF进行预测, 预测得到的CD81在NCBI上进行Blast比对验证其正确性。
1.6 引物设计及草鱼CD81 cDNA序列和基因组序列的获得和分析
在已经预测到的CD81 cDNA序列部分设计特异性引物, 未预测到的cDNA部分根据NCBI其他物种的同源性设计兼并引物, PCR扩增得到尽量长的CDS区。根据已经得到的CDS区设计特异性引物用于3′RACE库及5′RACE库的制备, 设计时保证引物扩增片段有重叠序列, 以便拼接。
以3′RACE库及5′RACE库为模板。反应条件为: 94 ℃ 2min; 94 ℃ 30s; 59 ℃30s; 72 ℃ 1min; 35个循环, 72 ℃ 10min。PCR产物经1.2%琼脂糖凝胶电泳检测, 回收后与pMD18-T(Takara, Japan)载体连接, 并转化入JM109感受态细胞中。涂平板, 挑取阳性克隆菌落送往华大基因公司测序, 将分别得到的CD81的3′端及5′端部分拼接并在NCBI中进行Blast同源性分析验证其正确性。
根据实验得到的草鱼CD81的cDNA序列和搜索到的包含草鱼 CD81的 Contig比对, 得到草鱼CD81的基因组结构。
1.7 蛋白结构预测及进化分析
使用NCBI在线软件翻译草鱼CD81所编码氨基酸序列, 运用简单模块构架搜索工具(Simple Modular Architecture Research Tool, SMART)对氨基酸序列进行蛋白质结构功能域分析, 利用http:// www.cbs.dtu.dk/services/SignalP/在线预测软件进行信号肽预测。用Mega 4.1对草鱼及其他物种CD81全长cDNA序列BLAST比对并做系统进化分析。
1.8 亚细胞定位
设计引物, 克隆CD81全长ORF, 用BamHⅠ和EcoRⅠ(Takara, Japan)对p-EGFP-N3质粒和PCR产物双酶切, T4连接酶(Takara, Japan)连接, 构建重组质粒, 同时以未重组的p-EGFP-N3空质粒做对照。将重组质粒及空质粒转入JM109感受态细胞, 阳性检测后提取阳性质粒, 送华大基因测序验证无误后, 转染CIK细胞。按照每10 μL Lipofectamine 2000 (Invitrogen, USA)加4 μg质粒的比例转染细胞, 6h后更换培养基, 72h后用DAPI细胞核染色液(碧云天,中国)染色, 置于激光共聚焦显微镜(Leica, Germany)下观察并拍照。

表1 本实验所用引物Tab. 1 Primers and their sequences
1.9 实时荧光定量PCR
运用荧光实时定量PCR的方法检测健康草鱼不同组织中CD81的表达水平及GCRV攻毒前后表达水平变化。实时定量 PCR 反应体系为 0.6 μL 10 μmol/L的引物, 1 μL模板, 10 μL 2×SYBR mix, 7.8 μL ddH2O。反应条件为: 95 , 10s; 95 , 5s, 60 ,℃℃℃20s; 40个循环。实时定量RT-PCR 的反应及信息收集均荧光定量PCR仪(BIO-RAD, USA)上进行。程序运行完成后进行溶解曲线分析以确定 PCR 产物是否专一, 使用β-actin 作内参对照, 据扩增曲线得到的 Ct值(荧光信号达设定阈值时所经历的循环数)计算出目标基因CD81和对照基因β-actin Ct值的差异ΔCt; 然后以表达量最小的样本作为参照样本, 计算出不同样品相对于参照样本基因的表达倍数2–ΔΔCt进而制作出相对定量图表。
2 结果
2.1 CD81cDNA全长序列及基因组结构
CD81 cDNA全长共1376 bp, 其中5′ UTR 87 bp, 3′UTR 581 bp, CDS区为708 bp, 编码235个氨基酸, 3′端具有典型的AATAAA 加尾信号。
CD81基因组共包括 8个外显子, 7个内含子,其中第一、二内含子很大, 第一内含子共 8422 bp,第二内含子21601 bp(图1)。
2.2 CD81蛋白结构预测及cDNA序列分析
运用SMART对CD81 ORF所编码蛋白质序列进行蛋白质结构功能域分析, 发现 CD81基因所编码蛋白具有明显的TM4SF家族特征, 含有四跨膜蛋白结构域。利用信号肽在线预测软件进行信号肽预测, 发现CD81并没有明显信号肽。利用NCBI在线软件比对草鱼 CD81氨基酸序列, 比对结果发现CD81蛋白结构在发现草鱼CD81在跨膜区保守, 但在胞外区变异性较高。将草鱼CD81cDNA序列与斑马鱼(Danio rerio CD81, NM_131518.2)、大西洋鲑(Salmo salar CD81, BT049555.1)、青斑河豚(Tetraodon nigroviridis CD81, CR686575.2)、人(Homo sapiens CD81, NM_004356.3)、小家鼠(Mus musculus CD81, NM_133655.2)、原鸡(Gallus gallus CD81, NM_ 001030339.1)、牛(Bos taurus CD81, NM_001035099.1)、野猪(Sus scrofa CD81, NM_001078679.1)、黑猩猩(Pan troglodytes CD81, NM_001009023.1)、非洲爪蟾(Xenopus (Silurana) tropicalis CD81, NM_203940.1)序列使用 MEGA4.1软件构建 NJ系统进化树。NJ系统进化树分析表明草鱼 CD81与斑马鱼及其他鱼类聚为一类, 这与分类学结果是一致的(图2)。

图1 CD81基因组结构Fig. 1 Genomic structure of CD81 gene
2.3 草鱼CD81亚细胞定位
细胞转染融合质粒72h后用DAPI细胞核染色液染色, 置于激光共聚焦显微镜下观察并拍照。图3中蓝色为细胞核, 绿色为 CD81与 GFP融合蛋白,图A为转染空质粒染色结果, 结果显示绿色荧光较为均匀的分布于细胞质和细胞膜上, 而图B为转染融合质粒结果, 结果显示点状绿色荧光颗粒分布于细胞周缘, 而胞质内仅有散在的零星发光为典型的细胞膜分布[21,22]。
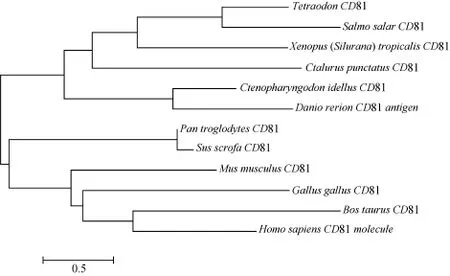
图2 基于草鱼与其他物种的NJ系统进化树Fig. 2 Neighbor-Joining phylogenetic tree analysis of grass carp CD81 cDNA with other vertebrates
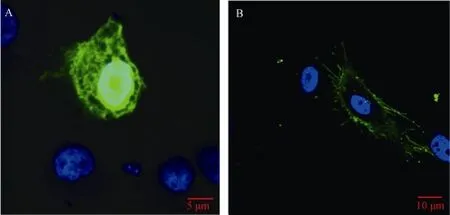
图3 草鱼肾细胞系CIK细胞中CD81的亚细胞定位Fig. 3 Distributing of CD81 in kidney cell line in grass carp
2.4 草鱼CD81的组织表达分析及GCRV攻毒前后表达变化
采用实时荧光定量PCR检测草鱼CD81在组织表达分析及 GCRV攻毒前后表达变化, 结果显示:草鱼 CD81基因在所有被检测组织中均有表达, 由低到高依次为:鳃<肠道<肝<中肾<脑<脾脏<头肾(图4A)。此外, 被检测草鱼的6个组织CD81基因在攻毒前后表达量均发生变化, 攻毒第 2天在鳃、头肾即出现表达变化, 在鳃中第 2天表达明显上调, 随攻毒时间延长又出现表达下调情况; 而头肾则在攻毒第 2天下调表达, 至攻毒中期又出现上调反应,然后在攻毒后期表达量又开始下降; 在肝、脾和肠中均到攻毒中期才出现明显上调, 至攻毒后期表达量又开始下降。
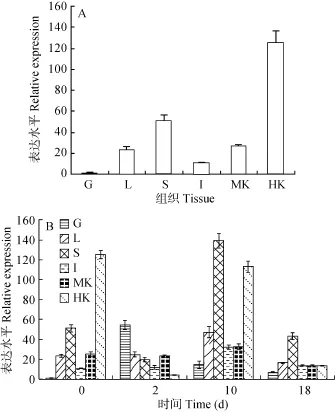
图4 CD81在不同组织表达情况及在GCRV攻毒前后表达变化Fig. 4 Expression of CD81 mRNA in grass carp in different tissues and in different periods after GCRV injection
3 讨论
CD81在人类免疫过程中的作用研究已较为深入, 而在鱼类研究中, CD81虽然在多个鱼类物种中得到克隆, 并有提示 CD81可能与鱼类的免疫过程相关, 但尚未有详细研究。基于对草鱼出血病进程表达谱的分析, 我们发现 CD81发生了较为显著的变化(未发表), 加之其在人类免疫过程中发挥的重要作用及其在各物种间的保守性, 我们希望探究CD81是否在草鱼免疫过程中也发挥作用。
本实验克隆了草鱼CD81的全长cDNA, 并根据实验室草鱼基因组序列得到了其基因组结构。CD81比对分析表明草鱼CD81是一个保守性很高的基因,与鱼类相似度均达到 70%以上, 与斑马鱼相似度达88%, 与斑点叉尾响相似度达 87%, 与大西洋鲑相似度达83%, 与青鳉(Oryzias sinensis)相似度达78%, 与非洲爪蟾(Xenopus (Silurana) tropicalis)的相似度也达到了 78%, 这提示草鱼 CD81很可能作为一种基础分子在草鱼的生物过程中发挥着重要作用。更值得关注的是 CD81分子的保守区域主要集中在跨膜区, 而胞外区高度可变, 正是这个高度可变的胞外区在人类中可以与HCV结合, 介导HCV的入侵[23], 这提示具有种属特异性的 CD81的大胞外区很有可能与不同物种的不同免疫过程相关。
在实验中发现草鱼CD81的基因组内包含了2个很大的内含子, 其中一个甚至达21601 bp。搜索其他物种CD81, 我们发现在人(Homo sapiens)、牛(Bos Taurus)和小耳大婴猴(Otolemur garnettii)中也发现了类似现象, 如此大的内含子到底在基因的功能或者基因的进化中扮演了什么角色, 这或许是一个有意思的问题。
王铁辉等[24,25]在研究感染出血病的稀有鲫的细胞病理中发现, 鳃是GCRV 侵袭的主要组织。此外, 研究者在染病草鱼肝、脾、肾、肠中均检测到过病毒颗粒。本实验结果表明草鱼CD81基因在GCRV攻毒前后在鳃、肝、脾、肾、肠中表达量均发生了变化, 鳃中CD81在第2天即出现了上调表达,而肝、脾、肾、肠中则到攻毒中期才出现上调表达,这与鳃作为GCRV的感染器官, 而肝、脾、肾、肠作为效应器官的推测是吻合的。值得一提的是, 本研究采用了腹腔注射的方法攻毒, 因此可以认为GCRV在相同时间以相同的病毒量到达各个组织,可以排除由于不同组织接触病毒的时间及由于病毒量差异而造成的影响。
细胞膜的迁移是病毒进入细胞的一种重要方式,而病毒进入细胞后其感染能引起细胞融合, 进而导致病毒的释放与扩散[26—28]。在人类中的研究表明, CD81分子可与包含TM4SF在内的其他蛋白形成复合体, 在细胞膜迁移和细胞融合中起作用[12], 本实验已证实, 草鱼中CD81在鱼类细胞中也定位在细胞膜上, 因此我们猜测CD81是否有可能在GCRV的入侵中也起到重要作用。
基于以上研究结果, 为我们所提出的 CD81可能参与了草鱼GCRV进程的免疫应答过程的推测提供了部分线索, 但相关研究仍需继续深入。
[1] Stipp C S, T V Kolesnikova, M E Hemler. EWI-2 is a majorCD9 and CD81 partner and member of a novel Ig protein subfamily [J]. Journal of Biological Chemistry, 2001, 276(44): 40545—40554
[2] Lee H, Bae S, Jang J, et al. CD53, a suppressor of inflammatory cytokine production, is associated with population asthma risk via the functional promoter polymorphism -1560 C>T [J]. Biochimica et Biophysica Acta, 2013, 1830(4): 3011—3018
[3] Boucheix C, E Rubinstein. Tetraspanins [J]. Cellular and Molecular Life Sciences, 2001, 58(9): 1189—1205
[4] Hemler M E. Tetraspanin functions and associated microdomains [J]. Nature Reviews Molecular Cell Biology, 2005, 6(10): 801—811
[5] Horejsi V, C Vlcek. Novel structurally distinct family of leucocyte surface glycoproteins including CD9, CD37, CD53 and CD63 [J]. FEBS Letters, 1991, 288(1—2): 1—4
[6] Maecker H T, S C Todd, S Levy. The tetraspanin superfamily: molecular facilitators [J]. FASEB Journal, 1997, 11(6): 428—442
[7] Richardson M M, Jennings L K, Zhang X A. Tetraspanins and tumor progression [J]. Clinical & Experimental Metastasis, 2011, 28(3): 261—270
[8] Wright M D, M G Tomlinson. The ins and outs of the transmembrane 4 superfamily [J]. Immunology Today, 1994, 15(12): 588—594
[9] Zhang F, Kotha J, Jennings L K, et al. Tetraspanins and vascular functions [J]. Cardiovasc Research, 2009, 83(1): 7—15
[10] Berditchevski F, E Odintsova. Tetraspanins as regulators of protein trafficking [J]. Traffic, 2007, 8(2): 89—96
[11] Claas C, Wahl J, Orlicky D J, et al. The tetraspanin D6.1A and its molecular partners on rat carcinoma cells [J]. Biochemistry Journal, 2005, 389(Pt 1): 99—110
[12] Hemler M E. Specific tetraspanin functions [J]. Journal of Biochemistry & Cell Biology, 2001, 155(7): 1103—1107
[13] Maecker H T, S Levy. Normal lymphocyte development but delayed humoral immune response in CD81-null mice [J]. Journal of Experimental Medicine, 1997, 185(8): 1505—1510
[14] Wang Q C, Nie Q H. CD81 molecule and its role in infectious diseases [J]. Epidemiology Lemology Foreign Medical Sciences, 1999, 26(6): 244—247 [王全楚, 聂青和. CD81分子及其在感染性疾病中的作用. 国外医学流行病学传染病学, 1999, 26(6): 244—247]
[15] Koutsoudakis G, Ruprecht N, B nsch C, et al. Characterization of the early steps of hepatitis C virus infection by using luciferase reporter viruses [J]. Journal of Virology, 2006, 80(11): 5308—5320
[16] Koutsoudakis G, Herrmann E, Kallis S, et al. The level of CD81 cell surface expression is a key determinant for productive entry of hepatitis C virus into host cells [J]. Journal of Virology, 2007, 81(2): 588—598
[17] Yeh H Y, Klesius P H. Channel catfish, Ictalurus punctatus Rafinesque 1818, tetraspanin membrane protein family: characterization and expression analysis of CD81 cDNA [J]. Veterinary Immunology and Immunopathology, 2009, 128(4): 431—436
[18] Kuhl H, Tine M, Hecht J, et al. Analysis of single nucleotide polymorphisms in three chromosomes of European sea bass Dicentrarchus labrax [J]. Comparative Biochemistry Physiology-Part D: Genomics & Proteomics, 2011, 6(1): 70—75
[19] Leong J S, Jantzen S G, von Schalburg K R, et al. Salmo salar and Esox lucius full-length cDNA sequences reveal changes in evolutionary pressures on a post-tetraploidization genome [J]. BMC Genomics, 2010, 11: 279
[20] Uinuk-Ool T, Mayer W E, Sato A, et al. Lamprey lymphocyte-like cells express homologs of genes involved in immunologically relevant activities of mammalian lymphocytes [J]. Proceedings of the National Academy of Sciences of the United States of America, 2002, 99(22): 14356—14361
[21] Yang D Y, Li C H, Lan F, et al. Expression and localization of strong epitope hAFP542—550 on eukaryotic cytoplasm icmembrane [J]. China Journal Cell Molecular Immunology, 2009, 25(7): 592—599 [羊东晔,李彩虹, 蓝方, 等. 强抗原决定簇 hAFP542—550在真核细胞膜定位表达研究. 细胞与分子免疫学杂志, 2009, 25(7): 592—599]
[22] Yu Y G, Xu R X, Jiang X D, et al. Subcellular localization of NMDA receptor protein on neuronmembrane by confocal laser Scanning microscopy [J]. China Journal Neuromedicine, 2004, 3(6): 408—410 [郁毅刚, 徐如祥, 姜晓丹, 等.神经细胞膜NMDA受体蛋白激光共聚焦显微镜亚光共聚焦显微镜亚细胞定位研究. 中华神经医学杂志, 2004, 3(6): 408—410]
[23] Higginbottom A, Quinn E R, Kuo C C, et al. Identification of amino acid residues in CD81 critical for interaction with hepatitis C virus envelope glycoprotein E2 [J]. Journal of Virology, 2000, 74(8): 3642—3649
[24] Wang T H, Chen H X, Liu P L, et al. Lobservations on the ultra-thin sections of the main organs and tissues of hemorrhagic Gobiocypris rarus artificially infectied by grass carp hemorrhagic virus (GCHV) [J]. Acta Hydrobiologica Sinica, 1993, 17(4): 343—346 [王铁辉, 陈宏溪, 刘沛霖,等. 草鱼出血病病毒人工感染稀有鲫出血病鱼主要器官组织的超薄切片观察. 水生生物学报, 1993, 17(4): 343—346]
[25] Li J, Wang T H, Lu R H, et al. Advances in research of hemorrhagic virus of grass carp [J]. Oceanologia et Limnologcia Sinica, 1999, 30(4): 445—453 [李军, 王铁辉,陆仁后, 等. 草鱼出血病病毒的研究进展. 海洋与湖沼, 1999, 30(4): 445—453]
[26] Eckert D M, Kim P S. Mechanisms of viral membrane fusion and its inhibition [J]. Annual Review of Biochemistry, 2001,70: 777—810
[27] Grove J, Marsh M. The cell biology of receptor-mediated virus entry [J]. Journal Cell Biology, 2011, 195(7): 1071—1082
[28] Benavente J, Martinez-Costas J. Avian reovirus: structure and biology [J]. Virus Research, 2007, 123(2): 105—119
MOLECULAR CLONING AND PRIMARY FUNCTIONAL ANALYSIS OF CD81 IN GRASS CARP
LI Hua-Ying1,2, HUANG Rong1, DU Fu-Kuan1, LIAO Lan-Jie1, LI Yong-Ming1and WANG Ya-Ping1
(1. State Key Laboratory of Freshwater Ecology and Biotechnology, Institute of Hydrobiology, Chinese Academy of Sciences, Wuhan 430072, China; 2. University of Chinese Academy of Sciences, Beijing 100049, China)
Cluster of Differentiation 81 (CD81) is a transmembrane protein also known as tetraspanins that plays an important role in cell growth, differentiation, signal conduction and adhesion. In this study, we cloned the CD81 gene of grass carp. The length of the cDNA was 1376 bp containing an 87 bp 5′ untranslated region (UTR), a 581 bp 3′ untranslated region (UTR), and a 708 bp open reading flame (ORF). The ORF had 8 exons and 7 introns, and encoded 235 amino acid residues. We applied real-time RT-PCR analysis to determine the pattern of CD81 gene transcription in different tissues of grass carp, and to measure the change in the transcript level after GCRV infection. The results showed that CD81 was expressed in all the tested tissues and was highest in head kidney. The transcript level was altered in gill, spleen, liver, intestines and head kidney after GCRV infection. Moreover, we used green fluorescent protein (GFP) to trace the subcellular expression of CD81, and the laser confocal electron microscopy revealed that grass carp CD81 was located on the cell membrane, which was the same in human.
Grass carp; CD81; TM4SF; Gene cloning; Tissue expression; Subcellular localization
Q144+.2
A
1000-3207(2015)03-0468-07
10.7541/2015.62
2014-05-08;
2014-06-12
国家863项目(2011AA100403); 国家自然科学基金(31130055)资助
李华英(1988—), 女, 山东东平人; 硕士; 研究方向为鱼类功能基因组学。E-mail: lihuaying_1988@126.com
汪亚平(1963—), 男, 博士; 研究方向为鱼类功能基因组学。E-mail: wangyp@ihb.ac.cn
