食管癌组织中RASSF1A的表达变化及意义
2014-12-02李秀梅秦炳照
李秀梅,秦炳照,刘 波
(湖北医药学院附属襄阳医院,湖北襄阳441000)
食管癌是人类最常见的恶性肿瘤之一,据统计,全球每年大约有30万食管癌新增病例[1,2]。食管癌的发生、发展是多基因、多因素共同参与的过程。Ras相关区域家族1A(RASSF1A)是2000年发现的新型候选肿瘤抑制基因(TSG),其在调节细胞周期和细胞凋亡方面具有重要作用,在肺、乳腺、肾、消化系统等部位的癌组织中存在不同程度的表达缺失[3~5]。本研究通过检测RASSF1A在食管癌组织中的表达,进一步探讨其在食管癌发生、发展中的作用。
1 资料与方法
1.1 临床资料 收集2013年1~12月湖北医药学院附属襄阳医院住院并接受开放手术治疗的120例食管癌患者的癌组织及其相应的癌旁正常组织(距肿瘤边缘8 cm以上,并经病理检查排除肿瘤累及),术前均未行任何抗癌药物治疗或放疗。其中,男94例,女26例;年龄 43~83岁,中位年龄为 63岁;TNM分期(UICC-AJCC,2003)Ⅰ~Ⅱ期53例,Ⅲ~Ⅳ期67例;分化程度分为高、中、低分化各40例;均经组织病理学检查确诊。
1.2 方法
1.2.1 RASSFIA检测方法 所有标本常规用甲醛固定,石蜡包埋,4 μm厚连续切片;HE染色,光镜观察分级和分期。采用免疫组化 SP法,羊抗人RASSF1A多克隆抗体购于北京中山生物技术公司,按说明书操作。2名医师双盲法观察切片,细胞质呈棕黄色染色为RASSF1A阳性细胞。阳性细胞所占百分比计分:<5%为0分,5%~25%为1分,26%~50%为2分,51%~75%为3分,>75%为4分。染色强弱计分:无色0分,淡黄色1分,黄色2分,棕褐色3分。阳性细胞百分比与染色强度计分的乘积0~2为阴性,3~8分为阳性,9~12分为强阳性。阳性、强阳性均判为阳性表达。
1.2.2 统计学方法 采用SPSS11.5统计软件。计数资料比较采用χ2检验。P≤0.05为差异有统计学意义。
2 结果
2.1 RASSF1A在食管癌及其癌旁正常组织中的表达 RASSF1A在食管癌及其癌旁正常组织中的阳性表达率分别为 50.08%(61/120)、91.60%(110/120),P <0.01。
2.2 RASSF1A表达与食管癌患者临床病理参数的关系 RASSF1A阳性表达与食管癌的TNM分期、浸润深度、淋巴结转移相关(P均<0.05),见表1。
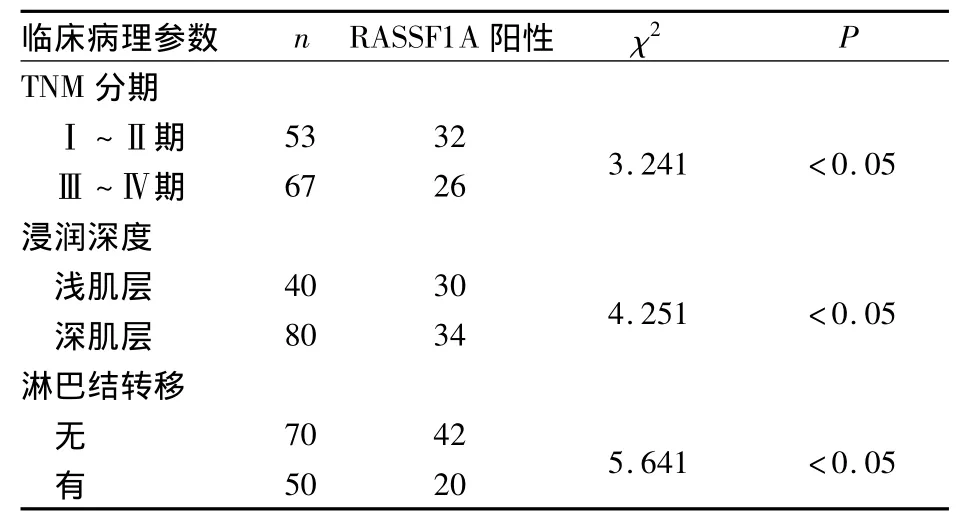
表1 RASSF1A表达与食管癌患者临床病理参数的关系(例)
3 讨论
食管癌的发生、发展是多基因改变、多阶段变异累积的复杂病理过程,患者就诊时多属中、晚期。因此,要改善食管癌患者的预后,早期诊断是相当重要的。RASSF1A是2000年报道从3号染色体短臂克隆出来的肺癌新型候选抑癌基因,其作为抑癌基因的具体机制尚不清楚。目前已证实[6~9],Ras-GTPase是调节增殖与凋亡的分子开关超家族一员,它的不同功能取决于细胞外的信号分子;由此,活化的Ras可能因与一系列不同的下游效应分子相互作用而发挥两种截然不同的功能,既可以促进细胞生长和分化,又可通过诱发细胞休眠,诱导终末分化和凋亡来抑制细胞生长。Lee等[10]用甲基化特定PCR法检测正常结肠组织、结肠腺瘤、结直肠癌,发现RASSF1A CpG岛甲基化在结直肠癌灶很频繁,远比癌灶旁及癌远隔部位的甲基化概率高。有研究[11~15]发现,食管癌细胞、食管癌组织、胃癌细胞、原发性胃癌组织及膀胱癌细胞中RASSF1A明显甲基化,且甲基化程度与临床分期相关,进一步表明RASSF1A的表达失活部分是由RASSF1A启动子区CpG岛的高甲基化所引起的。本研究显示,RASSF1A蛋白表达与食管癌TNM分期、浸润深度、淋巴结转移相关(P均<0.05)。因此,该基因有望在多种肿瘤的早期诊断和筛选中发挥重大作用,为消化道肿瘤早期诊断、预后判断和干预治疗提供新思路。
[1]Jenkins TD,Friedman LS.Adenocarcinoma of the esophagogastric junction[J].Dig Dis,1999,17(3):153-162.
[2]Parkin DM.Global cancer statistics in the year 2000[J].Lancet Oncol,2001,2(9):533-543.
[3]Dammann R,Schagdarsurengin U,Seidel C,et al.The tumor suppressor RASSF1A in hunman carcinogenesis:an update[J].Histol Histopathol,2005,20(2):645-663.
[4]Dammann R,Li C,Yoon JH,et al.Epigenetic inactivation of a RAS association domain family protein from the lung tumor suppressor locus 3p21.3[J].Nat Genet,2000,25(3):315-319.
[5]Hamilton DW,Lusher ME,Lindsey JC,et al.Epigenetic inativation of the RASSF1A tumour suppressor gene in ependymoma[J].Cancer Lett,2005,227(1):75-81.
[6]Dammann R,Schagdarsurengin U,Strunnikova M,et al.Epigenetic inactivation of the Ras-association domain family 1(RASSF1A)gene and its function in human carcinogenesis[J].Histol Histopathol,2003,18(2):665-677.
[7]Liu L,Tommasi S,Lee DH,et al.Control of microtubule stability by the RASSF1A tumor suppressor[J].Oncogene,2003,22(50):8125-8136.
[8]Strunnikova M,Schagdarsurengin U,Kehlen A,et al.Chromatin inactivation precedes de novo DNA methylation during the progressive epigenetic silencing of the RASSF1A promoter[J].Mol Cell Biol,2005,25(10):3923-3933.
[9]Chow LS,Lo KW,Kwong J,et al.RASSF1A is a target tumor suppressor from 3p21.3 in nasopharyngeal carcinoma[J].Int J Cancer,2004,109(6):839-847.
[10]Lee S,Hwang KS,Lee HJ,et al.Aberrant CpG island hypermethylation of multiple genes in colorectal neoplasia[J].Lab Invest,2004,87(4):884-893.
[11]Kuroki T,Trapasso F,Yendamuri S,et al.Allele loss and promoter hypermethylation of VHL,RAR-beta,RASSF1A,and FHIT tumor suppressor genes on chromosome 3p in esophageal squamous cell carcinoma[J].Cancer Res,2003,63(13):3724-3728.
[12]Kuroki T,Trapasso F,Yendamuri S,et al.Promoter hypermethylation of RASSF1A in esophageal squamous cell carcinoma[J].Clin Cancer Res,2003,9(4):1441-1445.
[13]Byun DS,Lee MG,Chae KS,et al.Frequent epigenetic inactivation of RASSF1A by aberrant promoter hypermethylation in human gastric adenocarcinoma[J]. Cancer Res,2001,61(19):7034-7038.
[14]Lee MG,Kim HY,Byun DS,et al.Frequent epigenetic inactivation of RASSF1A in human bladder carcinoma[J].Cancer Res,2001,61(18):6688-6692.
[15]Hoque MO,Begum S,Topaloglu O,et al.Quantitative detection of promoter hypermethylation of multiple genes in the tumor,urine,and serum DNA of patientswith renal cancer[J].Cancer Res,2004,64(15):5511-5517.
