1株西沙群岛野生诺尼种子内生细菌CICC 10599的分离与鉴定*
2014-11-20刘洋曹艳花姚粟翟磊张明娟张欣程池
刘洋,曹艳花,姚粟,翟磊,张明娟,张欣,程池
(中国食品发酵工业研究院中国工业微生物菌种保藏管理中心,北京,100015)
诺尼(Morinda citrifolia L.),又称为海巴戟天、诺丽、萝梨、四季果或印桑椹等,是一种生长于热带、亚热带的茜草科植物,原产于亚洲、澳大利亚及一些太平洋岛屿,富含160余种营养成分,具有非常独特的保健功效与药用价值[1]。国内外已有众多关于诺尼植物对多种病症具有显著疗效的科学研究报道[2],诺尼保健食品已被人们用来辅助治疗多种疾病。
植物内生细菌是能够在健康植物组织内栖居而对植物不造成实质性危害而与植物建立了和谐联合关系的微生物[3],其中不乏能够促进植物生长健康及功效性成分产生的有益微生物种类[4]。本研究以自西沙群岛野生诺尼种子中分离筛选得到的内生细菌菌株CICC 10599为研究对象,对其进行多相分类学鉴定与分析,旨在为进一步了解其生物功能及其相关机制奠定微生物资源基础。
1 材料和方法
1.1 实验材料
1.1.1 供试菌株
供试菌株分离自我国西沙群岛野生诺尼种子(采集自中国海南省三沙市永兴岛,具体位置为东经112.34°,北纬16.83°)中分离获得,并保藏于中国工业微生物菌种保藏管理中心(CICC),保藏编号为CICC 10599。
1.1.2 培养基
LB培养基(蛋白胨 10.0 g,酵母粉 5.0 g,NaCl 10.0 g,琼脂 15.0 g,蒸馏水 1.0L,pH7.0)。
1.1.3 主要试剂
Taq DNA聚合酶、dNTP、D2000 Marker购自天根生化科技有限公司;GoldView购自北京塞百盛基因技术有限公司;溶菌酶购自Sigma公司、蛋白酶K购自Merk公司;API 50 CHB和API 20E试剂条购自法国梅里埃公司;其他化学药品均为进口分析纯产品。
1.2 实验方法
1.2.1 菌株分离
在限菌条件下称取10 g野生诺尼种子,放入盛有90 mL无菌水带玻璃珠的三角瓶中,充分振荡20 min,之后静置5 min。采用梯度稀释法制备10-2到10-6的系列稀释液,分别涂布到LB培养基中置于30℃下培养48h。挑取单菌落转接到新鲜的LB斜面上于4℃保藏。
1.2.2 形态学观察
将供试菌株接种于LB固体培养基,置于30℃培养48h后,观察其在LB培养基上的培养特征;并利用光学显微镜及电子显微镜进行菌体特征观察[5]。
1.2.3 生理生化特征API检测
采用API 20E及API 50CH试剂条对菌株CICC 10599的酶活性、碳源利用及其产酸情况等生理生化特征进行检测,具体操作方法按试剂条说明书进行。
1.2.4 16 S rRNA基因序列分析
利用细菌基因组DNA提取试剂盒(Tiangen公司)提取菌株CICC 10599基因组DNA,具体步骤参见试剂盒说明书,提取的基因组DNA用0.8%的琼脂糖进行检测。
以基因组DNA为模板,利用通用引物对16S rRNA基因进行扩增。引物序列为:正向引物27f(5'-AGAGTTTGATCCTGGCTCAG-3'),反向引物 1492r(5'-GGTTACCTTGTTACGACTT-3')[5]。50 μL PCR 反应体系:50 ng DNA模板,1×Taq reaction buffer,引物各20 pmol,20 μmol dNTP,1.5 单位 Taq 酶(Ferments)。反应程序为:94℃预变性5 min,94℃变性1 min,52℃复性1 min,72℃延伸1 min 30 s,30个循环后72℃延伸10 min。PCR扩增产物用1%的琼脂糖进行检测。纯化后的PCR产物用ABI 3700基因测序仪测序。测序由北京诺赛基因组研究中心有限公司完成。测序结果用Chromas软件参照正反序列图谱人工校对。将测序得到的结果在EzTaxon server 2.1数据库中进行比对[6],确定与已知近缘种序列的相似性。采用CLUSTAL W对CICC10599及其若干近缘种16S rRNA基因序列进行多序列比对[7],并利用N-J法通过MEGA 5软件进行系统发育及分子进化分析[8]。
1.2.5 DNA促旋酶B亚基基因(gyrB gene)序列分析
以提取获得的菌株CICC 10599基因组DNA为模板,利用特异性引物DNA促旋酶B亚基基因(gyrB gene)进行扩增,上游引物(BC-gyrB-f)为 5'-GAAGTCATCATGACCGTTCTGCAYGCNGGNGGNAARTTYGA-3',下游引物(BC-gyrB-r)为 5'-AGCAGGATACGGATGTGCGAGCCRTCNACRTCNGCRTCN GTCAT-3'[9]。50 μL PCR反应体系:50 ng DNA模板,1× Taq reaction buffer,引物各 20 pmol,20 μmol dNTP,1.5单位Taq酶(Ferments)。反应程序为:94℃预变性5 min,94 ℃变性30 s,50℃复性30 s,72 ℃延伸1 min 30 s,30个循环后72℃延伸10 min。PCR扩增产物用1%的琼脂糖进行检测。纯化后的PCR产物用ABI 3700基因测序仪测序。引物合成及测序均由北京诺赛基因组研究中心有限公司完成。测序得到的结果在GenBank数据库中进行比对。
2 结果
2.1 形态学观察
菌株CICC 10599在LB培养基上菌落呈黄色,圆形,边缘不整齐,表面光滑并凸起(见图1a);利用光学显微镜观察显示菌体细胞杆状,单个排列,大小为0.4 μm ×(0.9~1.5)μm,革兰氏阴性(见图1b)。菌体电子显微镜成像效果见图1c和1d。
2.2 生理生化特征鉴定
经API 20E及API 50CH试剂条对菌株CICC 10599的酶活性、碳源利用及其产酸情况等生理生化特征进行检测,结果显示CICC 10599能够水解明胶和利用柠檬酸盐;具有β-半乳糖苷酶活性;并能够利用葡萄糖、果糖、半乳糖、甘露醇、肌醇、鼠李糖、麦芽糖、蔗糖、蜜二糖、阿拉伯糖等多种碳源物质,结果见表1。
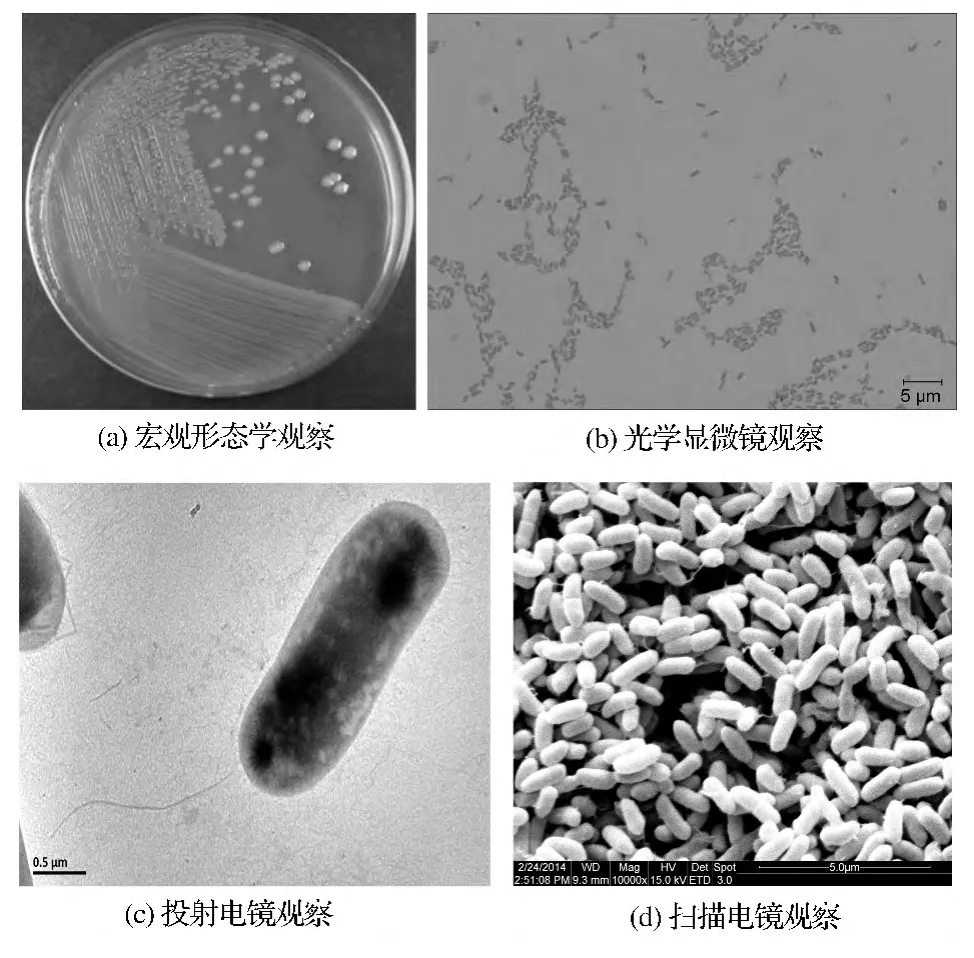
图1 菌株CICC 10599形态学观察结果Fig.1 Morphological observation of strain CICC 10599
2.3 16S rRNA基因序列分析
菌株CICC 10599的基因组DNA提取结果见图2。所提取的DNA片段约为23 kb,利用引物27f和1492r扩增得到16S rRNA基因全长序列,结果显示,PCR产物仅有一条带,大小约为1500 bp(见图3)。扩增产物经测序及分析处理后,将序列信息提交至Gen-Bank,登录号为 KJ562862。以 Escherichia coli KCTC 2441T(EU014689)为外群,构建CICC 10599与相关近缘模式菌株的系统发育树(图4)。由系统发育分析可断,菌株CICC 10599归属为泛菌属(Pantoea sp.)。

图2 CICC 10599基因组DNAFig.2 CICC 10599 Genomic DNA
2.4 DNA促旋酶B亚基基因的克隆与序列分析
DNA促旋酶B亚基基因(gyrB gene)可作为泛菌属内各菌种间分子生物学鉴定的重要判据之一[10]。以菌株CICC 10599的基因组DNA为模版,利用特异性引物(BC-gyrB-f/BC-gyrB-r)对DNA促旋酶B亚基基因进行扩增,结果如图5所示,扩增片段大小约为1300 bp左右,产物经测序及分析处理后,将序列信息提交至GenBank,登录号为KJ562866。将DNA促旋酶B亚基基因序列在GenBank中进行Blast比对,与分散泛菌(Pantoea dispersa)DNA促旋酶B亚基基因序列的相似度为99%,而与泛菌属中的其他近缘种的相似度均不高于90%。

图3 CICC 10599 16S rRNA基因扩增产物Fig.3 CICC 10599 16S rRNA gene PCR amplifi cation

图4 CICC 10599 16S rRNA基因系统发育分析Fig.4 Phylogenetic analysis of CICC 10599 16S rRNA gene
2.5 菌株CICC 10599分类鉴定结论

图5 CICC 10599DNA促旋酶B亚基基因PCR扩增产物Fig.5 CICC 10599 gyr B gene PCR amplification
通过形态学观察,生理生化特征鉴定,16S rRNA基因序列系统发育分析及持家基因——DNA促旋酶B亚基基因的比对分析,并结合相关文献报道[11],西沙群岛野生诺尼种子内生细菌菌株CICC 10599被鉴定为分散泛菌(Pantoea dispersa)。
3 讨论
本文采用多相分类(polyphasic taxonomy)鉴定技术对我国西沙群岛野生诺尼种子内生细菌菌株CICC 10599进行“种”水平分类地位的鉴定。多相分类的概念最早是由Colwell[12]提出的,综合利用微生物多种不同信息,包括表型、基因型和系统发育分析,对微生物进行分类的方法,已广泛应用于微生物分类学研究领域。
本研究利用细菌通用引物27f和1492r对菌株CICC 10599的16S rRNA基因序列扩增和序列测定,并通过GenBank数据库进行分析比较和利用MEGA 5进行系统进化分析,CICC 10599被鉴定为泛菌属(Pantoea sp.)。以基因系统发育分析为基础,结合形态及培养特征观察、生理生化试验及持家基因——DNA促旋酶B亚基基因序列分析结果,并参考相关文献报道[11],最终确定 CICC 10599为分散泛菌(Pantoea dispersa)。
分散泛菌(P.dispersa)由 Gavini等[11]于1989年首次分离得到并命名和描述;洪永聪等[13]自稻谷上分离获得的分散泛菌对水稻白叶枯病菌(Xanthomonas campestris pv.oryzae)具有良好的拮抗作用,但自诺尼植物中被发现并分离得到本研究尚属首次,鉴于该菌作为分离自野生诺尼种子内生细菌群落中的优势菌,可以推测该菌在诺尼植物生长发育及功效性成分物质产生方面具有不可忽视的作用。对其进行全面、准确的多相分类学鉴定是研究其生物学功能的必要前提。此外,本研究还针对CICC 10599进行碳源物质利用情况及相关酶的活性进行研究,确定其具有β-半乳糖苷酶的活性,并能够利用葡萄糖、果糖、半乳糖、鼠李糖、麦芽糖、蔗糖及阿拉伯糖等近30种碳源物质,为进一步发挥该菌的生物学功能和实现其生产利用价值奠定重要的基础。
[1]褚大为.来自伊甸园的礼物——热带野果萝荔的故事[M].上海:上海科学普及出版社,2002:1-2.
[2]WANG MY,West BJ,Jensen CJ,et al.Morinda citrifolia(Noni):A literature review and recent advances in Noni research[J].Acta Pharmacol Sin,2002,23(12):1 127-1 141.
[3]Di Fiore S,Del Gallo M.Endophytic bacteria:there possible role in the host plant.In:Fendrik I,et al.(eds),Azospirillum VI and related microorganisms[J].NATO ASI Series,1995,37:169-187.
[4]刘洋,刘琳,邹媛媛,等.与植物联合的细菌生物膜及其形成机制的研究进展[J].自然科学进展,2009,19(9):896-905.
[5]LIU Y,LIU L,QIU FB,et al.Paenibacillus hunanensis sp.nov.,isolated from rice seeds[J].International Journal of Systematic and Evolutionary Microbiology,2010,60:1 266-1 270.
[6]Chun J,Lee JH,Jung Y,et al.EzTaxon:a web-based tool for the identification of prokaryotes based on 16S ribosomal RNA gene sequences[J].Int J Syst Evol Micr,2007,57(10):2 259-2 261.
[7]Thompson JD,Higgins DG,GibsonTJ.CLUSTAL W:improving the sensitivity of progressive multiple sequence alignment through sequence weighting,position-specific gap penalties and weight matrix choice[J].Nucleic Acids Res,1994,22(22):4 673 -4 680.
[8]Tamura K,Dudley J,Nei M,et al.MEGA4:Molecular Evolutionary Genetics Analysis(MEGA)software version 4.0[J].Molecular Biology and Evolution,2007,24:1 596-1 599.
[9]Yamamoto S,Harayama S.PCR amplification and direct sequencing of gyrB genes with universal primers and their application to the detection and taxonomic analysis of Pseudomonas putida strains[J].Applied and Environmental Microbiology,1995,61(3):1 104-1 109.
[10]Rezzonico F,Smits TH,Montesinos E,et al.Genotypic comparison of Pantoea agglomerans plant and clinical strains[J].BMC Microbiology,2009,9:204 -221.
[11]Gavini F,Mergaert J,Beji A,et al.Transfer of Enterobacter-Agglomerans(Beijerinck 1888)Ewing and Fife 1972 to Pantoea Gen-Nov[J].as Pantoea-Agglomerans Comb Nov and Description of Pantoea-Dispersa Sp-Nov.International Journal of Systematic Bacteriology,1989,39(3):337-345.
[12]Cowell RP.Polyphsic taxonomy of the genus Vibiro:numberical taxonomy of Vibiro cholera,Vibiro parahaemolyticus,and related Vibro species[J].Journal of Bacteriology,1970,104(1):410-433.
[13]洪永聪,胡方平,陈庆河,等.从稻谷上分离、筛选用于拮抗水稻白叶枯病的泛菌[J].福建农业科技,2011(4):1-3.
