鹿科与牛科动物IGF-1基因序列比较及分子进化分析
2014-09-02宋兴超巴恒星王桂武杨福合
宋兴超,徐 超,王 雷,巴恒星,王桂武,杨福合
(中国农业科学院特产研究所 吉林省特种经济动物分子生物学重点实验室,吉林 长春 130112)
鹿科与牛科动物IGF-1基因序列比较及分子进化分析
宋兴超,徐 超,王 雷,巴恒星,王桂武,杨福合*
(中国农业科学院特产研究所 吉林省特种经济动物分子生物学重点实验室,吉林 长春 130112)
以GenBank中已登录的梅花鹿(Cervusnippon)、马鹿(Cervuselaphus)、普通牛(Bostaurus)、水牛(Bubalusbubalis)、山羊(Caprahircus)和绵羊(Ovisaries)胰岛素样生长因子-1(IGF-1)基因mRNA序列为研究材料,通过DNAStar 7.0等生物信息学软件对6个物种的IGF-1基因进行序列比较及分子进化分析。结果表明:鹿科与牛科动物IGF-1基因编码区均为465 bp,并且碱基组成表现为G>C>A>T,且G+C含量高于A+T;6个物种IGF-1基因密码子第1位富含A,密码子第2位4种碱基的分布相对均匀,密码子的第3位碱基C含量较高;编码区碱基序列中共检测到20个变异位点,包括10个单一变异和10个简约变异,鹿科和牛科动物IGF-1基因核苷酸和氨基酸序列的相似性均较高;(4) NJ和UPGMA两种方法构建的分子进化树均把6个物种聚为2个大类:鹿科和牛科,其中牛科又分支出牛亚科和羊亚科两类。
鹿科;牛科;胰岛素样生长因子-1基因;序列比较;密码子
胰岛素样生长因子(Insulin-like growth factors,IGFs)是一类广泛存在于动物体组织内且能够促进细胞分化、蛋白质沉积、骨骼增长以及个体生长发育的多功能细胞增殖调控因子[1]。IGFs系统包括I型胰岛素样生长因子(IGF-1)、II型胰岛素样生长因子(IGF-2)、胰岛素样生长因子受体(IGF-1 R和IGF-2 R)、胰岛素样生长因子结合蛋白(IGFBP)、IGFBP相关蛋白及IGFBP酶等[2]。其中,IGF-1基因被认为是影响动物生长性状的重要候选基因,随着分子生物学技术在畜禽育种研究中的逐渐应用,IGF-1基因也作为一种有效的遗传标记在猪[3]、牛[4]、羊[5]和鸡[6]等畜禽上进行了较为深入的研究,特别是近年来,有关鹿科动物IGF-1基因分离、鉴定及其与鹿茸生长关系的研究资料相对丰富。杜智恒等[7]首次在国内对梅花鹿IGF-1基因进行了克隆,获得DNA核苷酸序列长度为764 bp且与猪、牛和羊该基因同源性均为90%以上。胡薇等[8]通过RT-PCR方法分离得到梅花鹿IGF-1基因465 bp mRNA序列,相对荧光定量PCR差异分析发现该基因在鹿茸顶端的前软骨和软骨组织的表达水平高于真皮和间充质组织。郝林琳等[9]采用生物信息学方法预测马鹿该基因5′调控区含有CpG岛和253个潜在的转录因子结合位点并且与牛、羊存在差异。李兆志等[10]利用PCR-SSCP技术检测到左家地区梅花鹿群体IGF-1基因启动子区包括多个突变位点。到目前为止,IGF-1基因被认为是刺激鹿茸细胞分裂增殖最强的生长因子,并且NCBI的GenBank数据库中已经能够检索到多个鹿科动物IGF-1基因序列,同时,牛科与鹿科动物同属偶蹄目反刍动物,而生长发育对于这两种动物都是非常重要的经济性状。因此,本研究利用生物信息学方法对鹿科和牛科动物IGF-1基因mRNA序列进行比较,进一步构建6个物种的分子进化树,旨在探讨鹿科和牛科动物该基因序列变异特点及物种进化关系,为进一步寻找IGF-1基因功能分化位点提供生物学基础资料。
1 材料与方法
1.1 序列来源
从GenBank的核苷酸序列数据库中下载梅花鹿、马鹿、普通牛、水牛、山羊和绵羊的胰岛素样生长因子-1(IGF-1)基因全长cDNA序列,各物种序列信息见表1。
1.2 分析方法
鹿科和牛科动物IGF-1基因核苷酸序列碱基组成由BioEdit 7.0和DNAStar 7.0软件完成;采用DNAStar 7.0软件中MegAlign程序对6个物种IGF-1基因编码区核苷酸和氨基酸序列进行差异分析及相似性比较;利用Clustal X 1.83对IGF-1基因氨基酸序列进行完全比对,进一步基于MEGA 5.05软件的NJ(neighbor-joining,邻近法)和UPGMA(unweighed pair group method with arithmetic mean,非对组算数平均法)2种方法分别进行鹿科与牛科动物IGF-1基因分子进化分析。

表1 从NCBI中获取的鹿科和牛科动物 IGF-1基因核苷酸和氨基酸序列Table 1 Nucleotide and amino acid sequences of IGF-1 gene of Cervidae and Bovidae species
2 结果与分析
2.1 cDNA序列碱基组成
由表2可知,鹿科与牛科动物该基因编码序列碱基组成基本一致,均为G>C>A>T,且G+C含量高于A+T,梅花鹿和马鹿的T碱基百分含量最低。从4种碱基在密码子的分布情况可知,鹿科和牛科动物密码子中碱基分布存在相似的规律,密码子第1位富含A,密码子第2位4种碱基的分布相对均匀,密码子的第3位C碱基含量较高,A碱基含量最低,其中梅花鹿C碱基含量达39.4%,而水牛的A碱基含量最低,为11.0%。
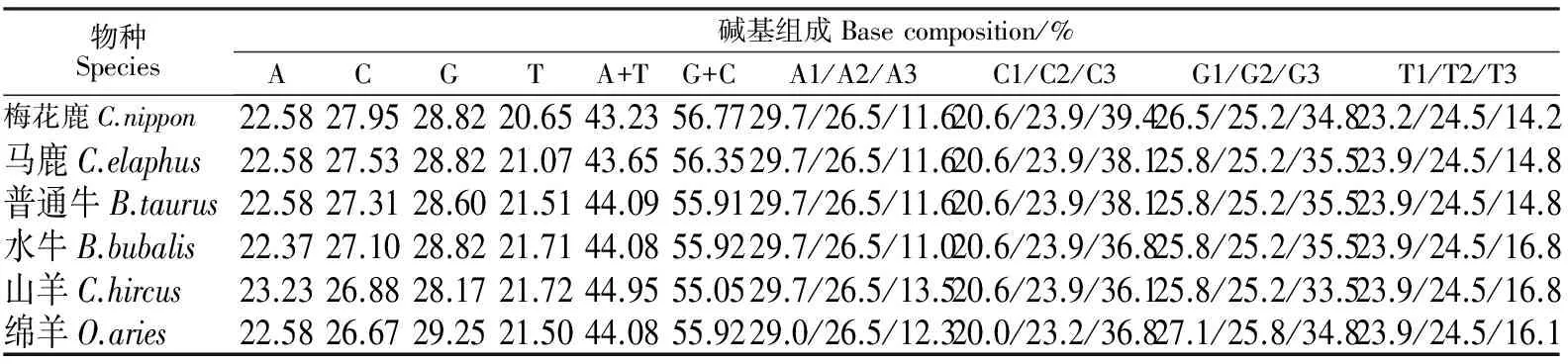
表2 鹿科和牛科动物IGF-1基因cDNA序列碱基组成Table 2 Base composition of IGF-1 gene of Cervidae and Bovidae species
注:表中数字1,2,3分别代表密码子的第1、2、3位。
Note:No.1,2 and 3 represent the 1st,2ndand 3rdcodon position,respectively.
2.2 核苷酸与氨基酸序列变异
GenBank数据库中公布的鹿科和牛科动物IGF-1基因mRNA序列长度不一致,其中鹿科中的梅花鹿和马鹿分别为465 bp和809 bp,牛科动物中普通牛、水牛、山羊和绵羊分别为874 bp、538 bp、978 bp和595 bp,但鹿科与牛科动物该基因完整编码区序列均为465 bp,这6个物种编码序列碱基变异情况如图1所示,以梅花鹿IGF-1基因序列作为标准,共检测到20个变异位点,其中,10个单一变异位点(Singleton variable sites)为:g.84A>G,g.111C>T,g.131C>G,g.201C>T,g.240C>T,g.258G>A,g.343C>G,g.354G>A,g.435C>G,g.439G>T,10个简约变异位点(Parsimony informative sites)为:g.78C>A,g.79G>A,g.105C>T,g.153 C>T,g.270 G>A,g.276A>C,g.330C>T,g.346A>G,g.352G>T,g.429C>T。在梅花鹿与马鹿及普通牛与水牛IGF-1基因序列中,仅存在3个变异位点,而山羊与绵羊间却产生了8个碱基变异。另外,在变异的碱基中,发生在第3密码子位置上有14处,占70%,密码子第1位存在5处碱基变异,只有1处变异发生在密码子第2位,表现出密码子的第2位非常保守,密码子碱基第3位的变异率明显高第1、2位,符合密码子摆动性的特点。序列相似性比对表明梅花鹿与马鹿、普通牛、水牛、山羊和绵羊IGF-1基因编码区核苷酸序列相似性分别为99.4%、98.1%、97.6%、97.6%和97.6%。

图1 鹿科与牛科动物IGF-1基因编码区碱基变异位点(·表示相同核苷酸)Fig.1 Nucleotide variable sites of IGF-1 gene between Cervidae and Bovidae species(·Indicates the identity of nucleotides)
从氨基酸序列比对结果来看(图2),鹿科与牛科动物IGF-1基因均编码154个氨基酸,鹿科动物中梅花鹿与马鹿IGF-1基因编码氨基酸序列相似性为99.4%,牛科动物中普通牛与水牛、山羊和绵羊的相似性分别为100%、98.7%和98.1%,普通牛和水牛的氨基酸序列完全一致。

图2 鹿科与牛科动物IGF-1基因编码区氨基酸变异位点(·表示相同氨基酸)Fig.2 Amino acid variable sites of IGF-1 gene in Cervidae and Bovidae species (·indicates the identity of amino acid)
2.3 分子进化树构建
鹿科和牛科动物的IGF-1基因编码区核苷酸序列通过MEGA 5.0软件的NJ法和UPGMA法分别构建6个物种分子进化树(图3和4),Bootstrap值是基于1000次模拟产生的,各个分支的置信度较高,结果表明,两种方法构建的进化树均把6个物种聚为2个大类:鹿科和牛科,其中牛科又分支出牛亚科和羊亚科两类,这种聚类与NCBI中动物系统分类结果基本相符。
3 讨 论
3.1 鹿科与牛科动物IGF-1基因序列差异
本研究通过生物信息学方法分析了鹿科与牛科动物IGF-1基因的碱基组成特点,同时参考了前人的研究结果,最终发现,该研究中所涉及到的6种动物的A、T、C、G的含量与胡薇等[8]研究结果基本一致,均为G+C含量明显高于A+T。王宁等[11]研究表明,G+C含量与核苷酸替代速率之间呈反比,由于IGF-1基因在哺乳动物进化过程中起主要调控作用,该基因的表达量决定了物种的生长速度,其编码区核苷酸替代速率必然低于非功能基因,因此导致该基因G+C含量较高。根据本研究结果,鹿科和牛科动物IGF-1基因核苷酸序列中共检测到20个变异位点,其中梅花鹿与马鹿间变异位点较少,相似性达到99.4%,胡薇等[8]对梅花鹿IGF-1基因克隆测序表明,梅花鹿与马鹿IGF-1基因核苷酸序列同源性达到99.78%,与本研究结果相似;而山羊和绵羊之间存在8个变异位点,这些变异位点与IGF-1基因的表达产物以及与鹿科和牛科物种表型差异的关系将是我们今后的工作目标。此外,从IGF-1基因编码区密码子碱基的分布情况可以看出,鹿科和牛科动物该基因存在相同的变化规律并且密码子第3位的变异率明显高于第1和第2位,符合摇摆假说(wobble hypothesis),即密码子第3位碱基允许有一定程度的摆动[12]。这与李鹏飞等[13]分析鱼类线粒体DNA Cyt b基因序列密码子组成特点的研究结果一致。
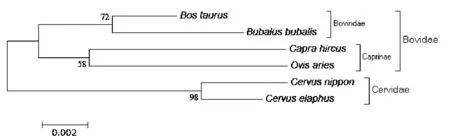
图3 NJ法构建的分子系统进化树Fig.3 Molecular phylogenetic tree by NJ method
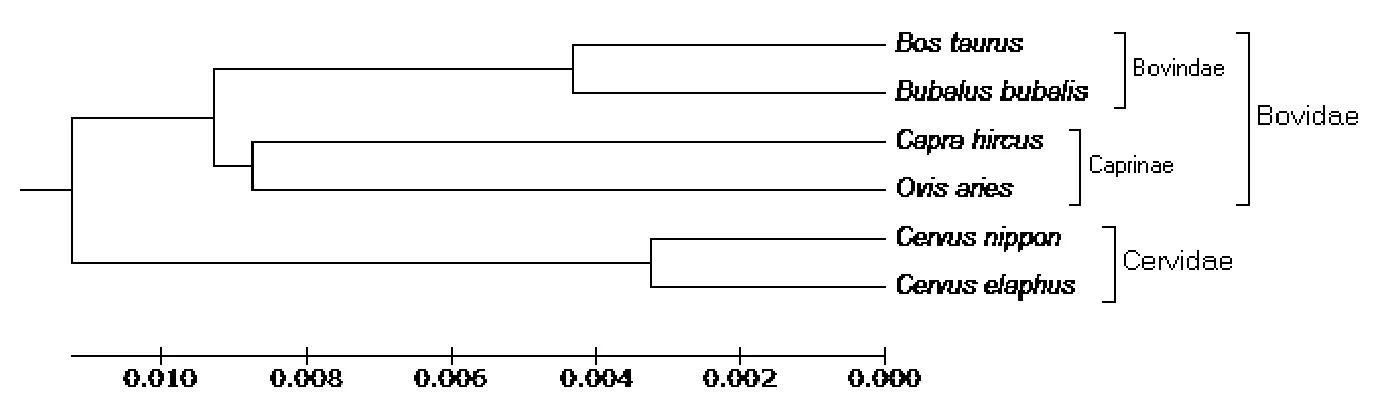
图4 UPGMA法构建的分子系统进化树Fig.4 Molecular phylogenetic tree by UPGMA method
3.2 鹿科与牛科动物的分子进化分析
从动物分类学角度分析,本研究中梅花鹿和马鹿属于鹿科物种[14],普通牛、水牛、山羊和绵羊同属牛科物种,其中普通牛和水牛属牛科中的牛亚科,而山羊和绵羊则属于牛科中的羊亚科[15]。众多研究者以不同基因为研究对象,采用不同方法对鹿科与牛科动物的遗传分化进行了分析。Wada等[16]分别通过NJ和MP方法,构建了包括鹿科和牛科在内的12个物种线粒体基因组核苷酸及其编码氨基酸序列系统进化树,发现鹿科、牛亚科及羊亚科在不同的进化枝上。Klungland等[17]利用核基因黑素皮质激素受体-1(MC1R)基因分析了鹿科与牛科动物的遗传分进化关系,表明鹿科和牛科动物的MC1R基因核苷酸歧异度为5.3%~6.8%,构建的有根进化树与当前动物分类学一致。侯佩兴等[18]研究表明,偶蹄目动物的性别决定(Sex-determining Region of Y-chromosome,SRY)基因按照其所处的科聚成了2类,即鹿科和牛科。本研究以GenBank中公布的2种鹿科动物和4种牛科动物IGF-1基因编码氨基酸序列为研究对象,分别通过NJ和UPGMA两种方法构建系统进化树,分析鹿科和牛科动物的分子进化关系,表明梅花鹿与马鹿间的遗传距离最短且普通牛、水牛与山羊、绵羊分属两个不同的亚科。本研究结果与前人研究基本一致,进一步解释了物种间的分化程度与碱基序列差异之间存在相关性,同时也证明了通过核内基因来分析鹿科和牛科动物系统进化的可行性。
[1]Tang S Q,Sun D X,Ou J T,et al.Evaluation of the IGFs(IGF1 and IGF2)genes as candidates for growth,body measurement,carcass,and reproduction traits in Beijing you and silkie chickens[J].Animal Biotechnology,2010,21: 104-113.
[2]Duan C M,Ren H X,Gao S.Insulin-like growth factors(IGFs),IGF receptors,and IGF-binding proteins: Roles in skeletal muscle growth and differentiation[J].General and Comparative Endocrinology,2010,167: 344-351.
[3]Niu P X,Kim S W,Choi B H,et al.Porcine insulin-like growth factor 1(IGF1)gene polymorphisms are associated with body size variation[J].Genes Genom,2013,35: 523-528.
[4]Maskur,Arman C,Sumantri C,et al.A novel single nucleotide polymorphism in exon 4 of insulin-like growth factor-1 associated with production traits in bali cattle[J].Media Peternakan,2012,8: 96-101.
[5]Bahrami A,Behzadi S,Miraei-Ashtiani S R,Roh S G,et al.Genetic polymorphisms and protein structures in growth hormone,growth hormone receptor,ghrelin,insulin-like growth factor 1 and leptin in mehraban sheep[J].Gene,2013,527: 397-404.
[6]Paswan C,Bhattacharya T K,Nagaraja C S,et al.Nucleotide variability in partial promoter of IGF-1 gene and its association with body weight in fast growing chicken[J].Animal Research,2013,3(1): 31-36.
[7]杜智恒,王宇祥,白秀娟.梅花鹿胰岛素样生长因子I(IGF1)基因的克隆及序列分析[J].黑龙江畜牧兽医,2007,5:98-99.
[8]胡 薇,孟星宇,田玉华,等.梅花鹿IGF1全长cDNA克隆及在鹿茸组织的表达[J].东北林业大学学报,2011,39(11): 71-75.
[9]郝林琳,刘松财,赵志辉,等.马鹿茸IGF-1基因端克隆及生物信息学分析[J].畜牧与兽医,2007,39(12): 47-49.
[10]李赵志,严昌国,张立春,等.吉林梅花鹿IGF-1基因启动子区单核苷酸多态性分析[J].安徽农业科学,2010,38(13):6 729-6 730.
[11]王 宁,陈润生.基于内含子和外显子的系统发育分析的比较[J].科学通报,1999,44(19):2 095-2 102.
[12]邹思湘.动物生物化学[M].4版.北京:中国农业出版社,2005: 287-306.
[13]李鹏飞,周永东,徐汉祥.大黄鱼、鮸鱼及美国红鱼线粒体DNA的Cyt b基因序列比较[J].南方水产,2008,4(3):43-47.
[14]赵世臻.中国养鹿大成[M].北京: 中国农业出版社,2001: 257-265.
[15]杨秋丽,哈福,李大林,等.牛科物种FSH 基因外显子3编码区遗传多态性及其生物信息学分析[J].华北农学报,2013,28(2):52-59.
[16]Wada K,Nishibori M,Yokohama M.The complete nucleotide of mitochondrial genome in the Japanese Sika deer (Cervus nippon),and a phylogenetic analysis between Cervidae and Bovidae[J].Small Ruminant Research,2007,69: 46-54.
[17]Klungland H,RФED K H,NESBФ C,et al.The melanocyte-stimulating hormone receptor (MC1-R) gene as a tool in evolutionary studies of Artiodactyles[J].Hereditas,1999,131: 39-46.
[18]侯佩兴,孙立彬,赵洪波.偶蹄目牛科和鹿科SRY基因编码区进化研究[J].上海交通大学学报:农业科学版,2009,27(4): 350-352.
SequenceComparisonandMolecularEvolutionAnalysisofIGF-1GeneofCervidaeandBovidaeSpecies
SONG Xing-chao,XU Chao,WANG Lei,Ba Heng-xing,WANG Gui-wu,YANG Fu-he*
(StateKeyLaboratoryofSpecialEconomicAnimalMolecularBiology,InstituteofSpecialAnimalandPlantScienceofCAAS,Changchun,Jilin130112,China)
Based on the IGF-1 gene nucleotide and amino acid sequences ofCervusnppon,Cervuselaphus,Bostaurus,Bubalusbubalis,Caprahircusand Ovis aries registered in GenBank,sequence variation and molecular evolution of IGF-1 gene were analyzed by means of bioinformatics software.The results showed that the lengths of IGF-1 gene coding sequences of Cervidae and Bovidae species were both 465 bp with the base composition being G>C>A>T and the content of G+C higher than that of A+T.Tthe first position of IGF-1 gene codon of the 6 species were rich in A,and the four bases in the second position were relatively even in distribution.A higher content of C in the third position.A total of 20 polymorphic sites including 10 singleton sites and 10 parsimony informative sites were detected among the 6 species,and a greater similarity of nucleotide and amino acid sequence in Cervidae and Bovidae species was observed.According to the molecular evolutionary tree constructed by NJ and UPGMA method,the 6 species fell into two broad categories: Cervidae and Bovidae with Bovidae including Bovidae and Capinae.
cervidae;bovidae;IGF-1 gene;sequence comparison;codon
2013-09-26,
2013-12-09
国家重点基础研究发展计划(2012CB722907);国家科技支撑计划项目(2011BAI03B02)
宋兴超(1982-),男,河北保定人,在读博士,助理研究员,研究方向:特种经济动物遗传育种。E-mail:tcssxc@126.com
*[通讯作者]杨福合(1956-),男,河北人,研究员,博士生导师,研究方向:特种经济动物种质资源收集、评价及遗传育种。E-mail:yangfh@126.com
S811.6
A
1005-5228(2014)03-0014-05
