改进型K-Means算法在肠癌病理图像分割中的应用
2014-08-25,,
,,
(浙江工业大学 计算机科学与技术学院,浙江 杭州 310023)
大肠癌的病发率呈逐年上升趋势,大肠癌患者的平均五年存活率仅为四成左右[1].因此,大肠癌的识别对临床诊断和治疗至关重要.目前,大肠癌临床诊断最可靠的方法是通过对患者的大肠病理切片图像进行病理性诊断[2].在现阶段,往往依靠病理专家的主观经验进行诊断,这种方法受病理专家的水平影响,且经验丰富的病理专家集中在少数大型医院,使许多中小医院无法开展该项工作.随着模式识别、人工智能和数字图像分析技术的发展,计算机辅助技术已经越来越受到关注,利用该技术进行病变细胞识别分类也取得了一定研究成果,如胃癌[3]、肺癌[4]和肝癌[5]的识别分类等.大肠病理切片图像分割技术是基于计算机辅助技术的大肠癌识别分类的关键技术,为识别难度较高的肠癌诊断奠定了一定的技术基础.
K-Means算法是比较成熟的一种聚类分析的方法,已被广泛应用到数据挖掘、机器学习和遥感图像等领域.研究证明[6-7]:初始聚类中心的选取是进行聚类算法非常重要的一部分,它直接影响聚类的结果.根据大肠癌细胞判别的病理学诊断指标,对使用聚类分析法进行大肠病理切片图像分割进行了探索,针对传统聚类分析中确定初始中心的难点,提出了一种改进型K-Means大肠癌病理切片图像分割算法,对后续的大肠病理切片图像特征提取以及相似癌症识别分类技术具有至关重要的意义.
1 K-Means算法
设有由n个m维数据构成的数据集X={xi|xi∈Rm,i=1,2,…,n},将X划分为由K个聚类集构成的集合C={ck|k=1,2,…,K},且每个聚类集之间的数据具有较高相似性,不同聚类集之间的数据彼此相差较大.常用的聚类方法有多种,K-Means算法是应用广泛的基于划分的一种聚类方法,这种方法的K值确定,适合处理数据量较大的数据集,且更易发现球形类.
K-Means算法的核心思想是:选定K个数据作为数据集的初始中心;对于其余每个数据,根据该数据到初始中心的距离,选定距离该数据最近的类作为它的归属类;重新计算K个类的初始中心,重新确定每个数据的归属类,不断重复这个过程,直至初始中心不再变更.
常用的计算距离主要有Minkowski distance,Euclidean distance,CityBlock distance三种方法,Minkowski distance方法的计算公式为
(1)
式中λ取任意值.Euclidean distance方法的公式对应式(1)λ取2,其计算公式为
(2)
CityBlock distance方法的公式对应式(1)λ取1,其计算公式为
(3)
使用式(1—3)进行聚类时,数据集分别以星形、圆形和菱形方式逼近中心.研究对象是以接近圆形的腺体为主,因此选用Euclidean distance方法式(2)计算距离.
使用K-Means算法时要确定类别数,研究的大肠病理切片图像是大肠组织样本经过苏木精—伊红染色法(Hematoxylin-eosin staining,HE染色法)染色之后得到的大肠病理切片图.正常的大肠病理切片图像中,有意义的物质主要有三类,分别是腺腔和上皮细胞、细胞核、间质.因此,聚类分析的类别数K=3.
传统K-Means算法确定初始中心主要使用随机方法或平均方法,随机方法随机选择某一个或某几个数据点作为初始中心,平均方法计算数据集的平均数据点作为初始中心.传统计算初始中心方法确定的初始中心并不能较好地代表数据集,具有随机性和不确定性.而聚类初始中心对聚类过程有十分重要的影响,使用传统方法确定的初始中心因其较差的代表性,使得聚类收敛过程缓慢,聚类后的结果往往不理想.
2 主成分分析
主成分分析(PCA)是常用的分析、挖掘和简化数据技术,它的实质是对数据集进行线性变换到另一空间,在新空间下借助主成分或者少数的几个成分概括关系复杂的数据信息量.PCA有效简化了数据集[7],便于分析,被广泛地应用到了金融统计和神经科学等领域[8].
图像分割是主成分分析重要的应用方向之一,主成分分析通常将一幅图像经过线性变换到另一个空间,在变换后的空间提取特征值.对数据集进行主成分分析时,协方差矩阵和相关系数矩阵是常用的计算工具.协方差矩阵适用于单个数据指标方差大的一类数据集上,而相关系数矩阵剥离了单个数据指标的方差,适用于相关数据指标多、数据指标之间的相关程度高数据集上[8].使用主成分分析时,若数据集经过了标准化,则得到的矩阵便是协方差矩阵.使用主成分分析目的是选取具有代表性的聚类初始中心,应避免单个数据指标方差对分析产生的影响,所以使用相关系数矩阵进行主成分分析.
在一个m维空间中,设定它的中心为
(4)
将空间中的数据标准化,即将原始数据样本处理成样本均值为0和样本方差为1的标准化数据.现将位于该空间中一点投影变换到一个向量上,设定ω为m维投影向量,且满足ωωΤ=1,得到投影变换后的方差为
(5)

ωCωΤ-λ(1-ωωΤ)
(6)
将式(6)对ω求导并使之为0,可得方差最大化的条件为
CωΤ=λωΤ
(7)
式(7)取得最大值时,λ为C的最大特征值,ω为最大特征值对应的特征向量.
上述过程实现了数据集的降维和简化,以有效信息丢失最少的原则下,通过线性变换保留对应特征值最大的部分,舍弃其他无效或仅包含少量有效信息部分得到变换后的主成分.主成分分析方法得到的最大特征值对应的成分为第一主成分,第一主成分在数据空间集上的方差最大,负载的信息量最大,延伸空间最长.根据大肠病理切片图像特点,将第一主成分平均分成三个区间,分别代表腺腔和上皮细胞、细胞核、间质三类物质,取三个区间的中间点作为聚类的初始中心.使用此方法确定的初始中心较传统方法确定的初始中心更具有代表性,且在后阶段聚类中提高了收敛速度.
3 改进型K-Means大肠病理切片图像分割算法
对由大肠病理切片图像使用PCA求取聚类初始中心,之后使用K-Means算法进行聚类,最终实现大肠病理切片图像分割,假设该矩阵为
(8)
X中的每一个行向量代表一个样本数据,但RGB空间维度相关性比较大,难以选出具有代表性的三类初始中心,同时考虑到该数据集数据量庞大,难以实现特征提取,根据PCA理论将其变换到另一个更适合的维度,产生新的矩阵为Y=ωX.
将式(8)中X中每个元素减去对应每列均值得到标准化后矩阵Z,得到相关系数矩阵为
(9)
计算得到Cx特征值,并将特征值按递减排序,对应特征向量矩阵为υ∈R3×3.变换后的新矩阵为Y=υZ∈RN×3,第一列称为主维度,第一列向量为主向量.Vmax和Vmin分别表示主向量中最大与最小值,将主向量分为K段,每段长度为
(10)
第i段的范围区间为
[d=Vmax+(i-1)d,Vmin+id) 1≤i≤K
(11)
对于Y中的每一个样本数据,根据其主维度值归类到对应列.对于每个类,求出其均值中心,得到由K个类的中心构成的矩阵为M.
将M变换至原坐标得到K个初始聚类中心构成的矩阵为
(12)
确定初始中心之后,将样本数据进行K-Means聚类分析,其中计算距离时使用式(2)Euclidean distance公式.具体的算法步骤为:
1) 根据上述算法确定初始中心为Z1(0),Z2(0),…,ZK(0),括号中的值表示迭代次数.
2) 对于第T次迭代,对于除聚类中心之外的数据,根据该点到初始中心的距离确定该点属于的类.
3) 利用均值的方法确定第T+1次迭代的初始中心为Z1(T+1),Z2(T+1),…,ZK(T+1).
4) 如果聚类中心与原聚类中心重合,即Zj(T)=Zj(T+1),且在一定次数内收敛,则完成聚类,停止;否则,跳至第(2)步重复.
4 实验结果与分析
为充分验证提出算法的有效性,选取一张具有代表性的大肠病理切片图像进行分割处理,同时将算法与Lab聚类算法处理结果进行对比.
图1为一张正常的大肠病理切片图.本实验的图像样本是将大肠组织经过苏木精—伊红染色法染色获取的图像,图中形状近似圆形、轮廓清晰且排列有序的是大肠腺体,腺腔和上皮细胞分布于其中,间质分布于其间,而细胞核分布于腺体边缘和其间.正常的大肠病理切片图中细胞分布均匀、排列有序且细胞核与上皮细胞明显.而癌变的大肠病理切片图中细胞分布散乱、排列无序且细胞核与上皮细胞均消失.

图1 大肠病理切片原图
使用聚类算法处理图1所示大肠病理切片原图,得到聚类结果.聚类图中蓝色代表腺腔和上皮细胞,红色代表细胞核,绿色代表间质.图2(a)为使用提出的改进型K-Means聚类算法对原图进行处理得到的实验结果.Lab聚类算法是一种常用的聚类分割算法,该算法将彩色图像变换至Lab色彩空间,并去除代表亮度的L空间信息,再对变换后的结果进行K-Means聚类算法处理.作为对比,使用Lab聚类算法对图1所示原图进行聚类,得到实验结果如图2(b)所示.
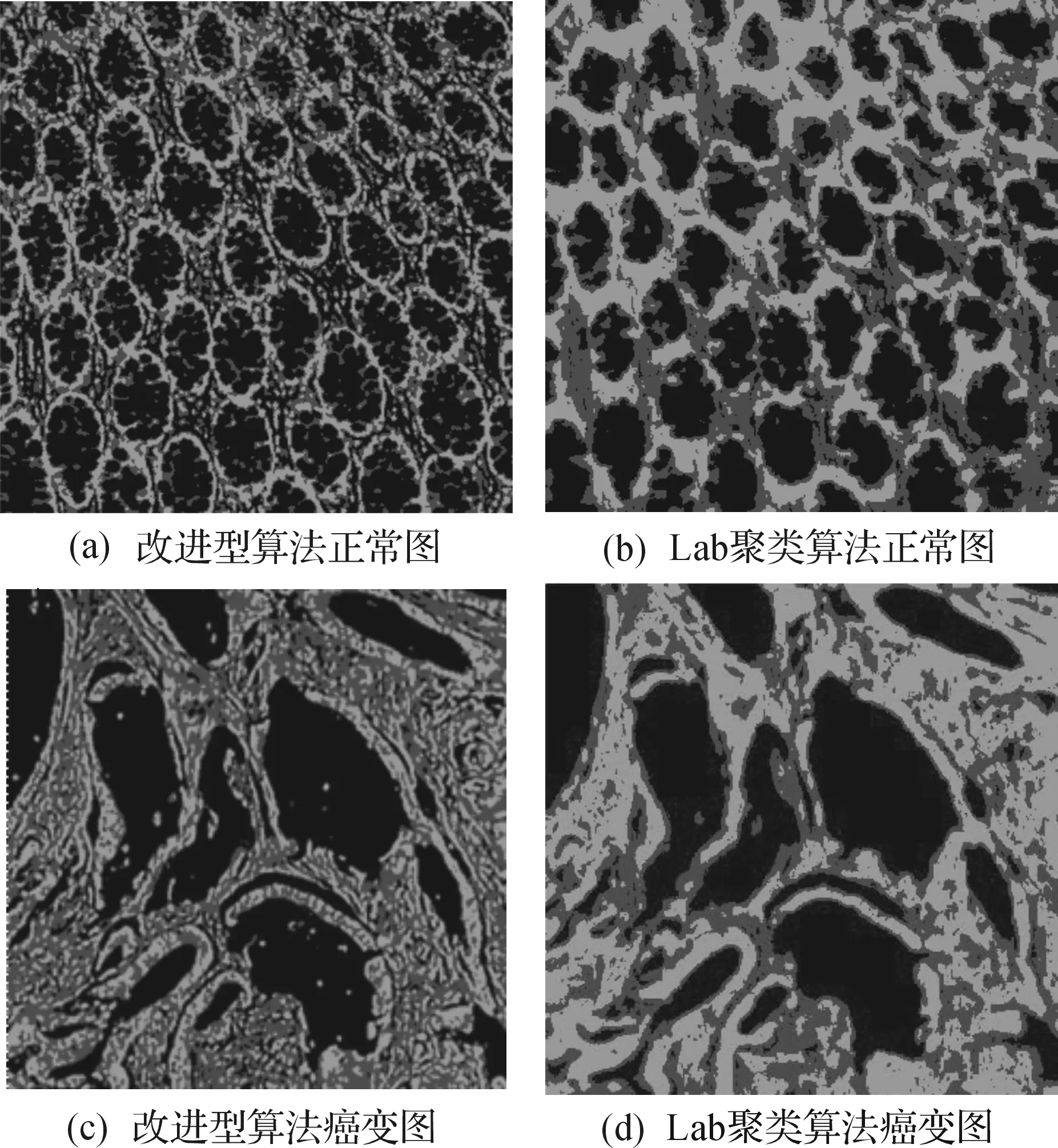
图2 聚类处理后的结果图
由实验对比结果可看出:Lab聚类算法处理结果丢失了大肠病理切片图中存在于大肠腺体中的腺腔和上皮细胞、间质的信息,这是大肠癌病理识别诊断中最关键的特征之一.同时,Lab聚类算法处理破坏了大肠病理切片图中大肠腺体的形状特征,出现了腺体重叠、轮廓不清晰等问题,因此Lab聚类算法造成了过分割的结果.改进型K-Means算法处理结果保留了正常大肠病理切片图中细胞分布均匀、排列有序且细胞核与上皮细胞明显的特征,同时腺体轮廓清晰,没有出现腺体重叠的现象.
改进型K-Means算法在具备有效性的前提下,由于使用PCA方法确定初始中心,相比于使用RANDOM方法确定初始中心的传统K-Means算法,具有收敛速度快的优势.传统K-Means选定初始中心使用随机选取的方法,确定的初始中心不能很好代表数据集,并有可能对收敛过程造成不良影响,这体现在使用RANDOM方法的传统聚类算法比使用PCA方法的算法的收敛时间更长.
为比较收敛时间,选取20,50,100幅大小为351×351的彩色大肠病理切片图像,分别使用提出的PCA方法与传统RANDOM方法进行收敛时间对比.
本实验在Intel(R) Core(TM) i7-4650U 1.70 ~2.30 GHz的PC机上实现,实验结果如表1所示.

表1 PCA方法与RANDOM方法收敛时间对比结果
综上所述,使用提出的改进型K-Means大肠病理图像分割算法有效地提取并突出了大肠病理切片中的腺腔和上皮细胞、细胞核、间质的颜色特征,同时对比于传统RANDOM方法具有收敛速度快的特点.
5 结 论
基于PCA和K-Means算法的大肠病理切片图像分割算法,将大肠病理切片图像中的腺腔和上皮细胞、细胞核、间质进行分类.通过使用基于相关系数矩阵的主成分分析法降低了数据复杂度,确定了具有代表性的聚类初始中心,再使用K-Means算法,将病理切片图像中的数据聚分成三类.与Lab聚类算法相比,改进型K-Means算法更准确地将大肠病理切片图像分割,而且使用PCA方法确定初始中心在收敛速度上优于RANDOM方法.改进型K-Means算法实现高效简单,为后续的特征提取奠定了良好的基础.
参考文献:
[1] SIEGEL R, NAISHADHAM D, JEMAL A. Cancer statistics[J]. A Cancer Journal for Clinicians,2013,63(1):11-30.
[2] SADANANDAM A, LYSSIOTIS C A, HOMICSKO K, et al. A colorectal cancer classification system that associates cellular phenotype and responses to therapy[J]. Nature Medicine,2013,19(5):619-625.
[3] CHANG K H, MILLER N, KHEIRELSEID E A H, et al. MicroRNA signature analysis in colorectal cancer:identification of expression profiles in stage II tumors associated with aggressive disease[J]. International Journal of Colorectal Disease,2011,26(11):1415-1422.
[4] SUN S, BAUER C, BEICHEL R. Automated 3-D segmentation of lungs with lung cancer in CT data using a novel robust active shape model approach[J]. Medical Imaging, IEEE TransActions on,2012,31(2):449-460.
[5] 凌华强,龙胜春,项鹏远.主动形体模型法在肝脏CT图像分割中的应用[J].浙江工业大学学报,2012,40(4):450-453.
[6] ALRABEA A, SENTHILKUMAR A V, AL-SHALABI H, et al. Enhancing k-means algorithm with initial cluster centers derived from data partitioning along the data axis with PCA[J]. Journal of Advances in Computer Networks,2013,1(2):137-142.
[7] 古辉,王益义.一种基于模板匹配的船铭牌字符分割方法[J].浙江工业大学学报,2010,38(1):33-35.
[8] 朱永忠,姚烨,张艳.基于主成分分析和Logistic回归的上市公司财务困境预警模型的研究[J].浙江工业大学学报,2012,40(6):692-694.
[9] CAMPBELL M C, MARKHAM J, FLORES H, et al. Principal component analysis of PiB distribution in parkinson and alzheimer diseases[J]. Neurology,2013,81(6):520-527.
[10] NUMPACHAROEN K, ATSAWARUNGRUANGKIT A. Generating correlation matrices based on the boundaries of their coefficients[J]. PloS One,2012,7(11):e48902.
