泛耐药鲍氏不动杆菌与耐药相关基因检测及亲缘性分析
2014-04-13杨越明金法祥钟建平
杨越明 金法祥 钟建平
泛耐药鲍氏不动杆菌与耐药相关基因检测及亲缘性分析
杨越明 金法祥 钟建平
目的了解泛耐药鲍氏不动杆菌(PDR-ABA)的菌株亲缘性。方法 对20株PDR-ABA进行3种耐药相关看家基因(carO、gyrA、parC)和54种水平转移获得的与β-内酰胺类、氨基糖苷类和喹诺酮类药物耐药相关基因以及12种接合性质粒、转座子、插入序列、整合子等可移动遗传元件的遗传标记检测。结果 20株PDR-ABA中检出4种β-内酰胺类获得性耐药基因,5种氨基糖苷类获得性耐药基因,2种获得性抗菌制剂外排泵基因和5种可移动遗传元件。耐药相关看家基因carO、gyrA、parC均存在有义突变。 结论 PDR-ABA存在5个克隆传播,其中1-3-4-5-9-15-16-18-20号菌株呈流行趋势。
鲍氏不动杆菌 看家基因 水平转移基因 菌株亲缘性分析
鲍氏不动杆菌(Acinetobacter baumannii,ABA)临床分离株的耐药性不断上升,从多重耐药到泛耐药,直至极度耐药ABA均已有报道[1-2]。近年来有学者已从ABA中检出新型β-内酰胺酶,如超广谱AmpC酶ADC-56型[3],新型金属β-内酰胺酶NDM-2型[4],新型碳青霉烯酶OXA-225型。本研究对一组(20株)泛耐药的ABA(pandrug-resistant ABA,PDR-ABA)进行了3种与耐药相关的看家基因(carO、gyrA、parC)和54种水平转移获得与β-内酰胺类、氨基糖苷类和喹诺酮类药物耐药相关基因以及12种接合性质粒、转座子、插入序列、整合子等可移动遗传元件的遗传标记检测,并对结果作了样本聚类分析,拟探讨该组菌株间的亲缘关系,现报道如下。
1 材料和方法
1.1 菌株来源 20株PDR-ABA均分离自2009-01— 2010-03本院ICU患者的痰液标本。由于PDR-ABA的定义报道者各不相同,本研究PDR-ABA的判别标准参照肖永红[5]的报道,即β-内酰胺类药物(包括碳青霉烯类药物)、氨基糖苷类药物和喹诺酮类药物(包括左氧氟沙星)3者均耐药才纳入本研究。
1.2 细菌鉴定及药敏试验 所有菌株均经法国生物梅里埃公司ATB微生物鉴定仪及配套鉴定条鉴定为ABA,并行gyrA和parC基因扩增与测序,经GenBank比对进一步确认为ABA。药敏试验采用纸片扩散法,M-H琼脂与药敏纸片为英国Oxoid公司产品。根据美国临床实验室标准化协会(CLSI)2009年版标准进行抗菌药物敏感性判断。20株菌对哌拉西林、头孢噻肟、头孢他啶、头孢吡肟、亚胺培南、美罗培南、氨苄西林/舒巴坦、哌拉西林/他唑巴坦、阿米卡星、庆大霉素、环丙沙星、左氧氟沙星、四环素、复方磺胺甲恶唑均耐药,仅对多粘菌素敏感。
1.3 细菌DNA提取 挑取培养菌落置入0.5ml离心管内(内预置200ng/ml蛋白酶K溶液400μl),56℃水浴2h后改95℃水浴10min,即为基因检测的模板液,-20℃冰箱保存备用。
1.4 基因检测 3种与耐药相关的看家基因(carO、gyrA、parC)和54种水平转移获得与β-内酰胺类、氨基糖苷类、喹诺酮类耐药相关基因以及12种接合性质粒、转座子、插入序列、整合子等可移动遗传元件遗传标记检测均采用PCR法。靶基因引物识别序列、目的序列引物由和基因检测试剂盒由无锡市克隆遗传技术研究所提供。PCR扩增体系均为:每反应体系P1引物1μl(1.0μmol/L)、P2引物 1μl(1.0μmol/L)、dNTPs 2μl(2 mmol/L)、10×缓冲液2μl(含KCl 10mmol/L、(NH4)2SO48mmol/L、MgCl22mmol/L、pH 9.0 Tris-HCl 10mmol/L、NP 40 0.5%、BSA 0.02%),Taq DNA pol 1U(不计体积),超纯水9μl,模板液5μl,总反应体积20μl。PCR扩增产物>500bp时PCR参数均为:93℃预变性2min,93℃60s,55℃60s,72℃60s,共35个循环,最后72℃延长5min。其余则为:93℃预变性2min,93℃30s,55℃30s,72℃60s,共35个循环,最后72℃延长5min。扩增产物经2%琼脂糖凝胶电泳,出现与阳性对照相应的目的条带为阳性。
1.5 基因测序 基因测序委托上海博尚生物技术有限公司,于美国ABI公司3730型毛细管全自动测序仪上完成。读序使用Chromas软件,测序结果直接作BLAST Search序列比对。
1.6 聚类分析 对3种与耐药相关的看家基因和54种水平转移获得与β-内酰胺类、氨基糖苷类、喹诺酮类耐药相关基因以及12种接合性质粒、转座子、插入序列、整合子等可移动遗传元件遗传标记检测结果作样本聚类分析(Neighbour-Joining法)。
2 结果
20株PDR-ABA中3种与耐药相关的看家基因和54种水平转移获得与β-内酰胺类、氨基糖苷类和喹诺酮类药物耐药相关基因以及12种接合性质粒、转座子、插入序列、整合子等可移动遗传元件遗传标记检测结果见表1。其中看家基因carO、gyrA、parC均存在有义突变,parC基因除第80位密码子为有义突变外,另有3个同义突变。parC(第80位)突变经与美国GenBank基因库比对为新的亚型,该基因序列已登录于美国Gen-Bank基因库(登录号:JQ003194)。parC(第80位)突变部位测序图见图1。
20株PDR-ABA检测结果样本聚类分析提示,其中1-3-4-5-9-15-16-18-20号菌株,7-10号菌株,8-11号菌株,12-14号菌株,17-19号菌株分别为同一克隆。其中7-10号菌株均携带了TEM+、PER+、ADC+、OXA-23+、aac(3)-Ⅰ+、aac(6′)-Ⅰb+、ant(3″)-Ⅰ+、adeB+、qacE△1+、tnpU+、tnp513+、IS26+、ISaba1+和intⅠ1+等14种水平转移基因;12-14号均携带了TEM+、PER+、ADC+、OXA-23+、aac(6′)-Ⅰb+、ant(3″)-Ⅰ+、adeB+、qacE△1+、tnpU+、tnp513+、IS26+、ISaba1+和intⅠ1+等13种水平转移基因。

表1 20株PDR-ABA基因检测结果
3 讨论
ABA已成为ICU患者中分离的首位细菌,多数菌株对β-内酰胺类(包括碳青霉烯类)、氨基糖苷类、喹诺酮类(包括左氧氟沙星)等抗生素耐药[6]。医院内感染是耐药ABA迅速播散的主要原因,对分离的菌株进行亲缘性分析是判别医院内感染的有效方法。菌株亲缘性分析的本质为菌株的系统发育分析,即对所观察的菌株的形成和发展过程进行分析,亦即分析该均住院的进化历史。系统发育分析需要收集多种特征包括指标和变量,而与耐药相关的看家基因、水平转移基因符合这一要求,且检出的基因型与药物敏感性表型可作直观分析。姜如金等[7]基于看家基因与水平转移基因的菌株亲缘性分析提示,如果不考虑水平转移基因,菌株分析中将有15种多态性被遗漏。
对所研究的一组细菌中的每一株进行全基因组测序,并对结果进行样本聚类分析目前是菌株亲缘性分析最为可靠的方法。近年来国际著名学术刊物上相继刊登了全基因组测序后并对所发现的单核苷酸多态性(single nucleotide polymorphism,SNPs)作聚类分析,从而解析菌株亲缘性的文献报道[8-10]。不久前德国暴发致患者腹泻、肠出血、肾功能衰竭的大肠埃希菌O104:H4感染,其分离的菌株也用此法进行亲缘性分析[11]。但对所研究的每一株细菌进行全基因组测序由于价格昂贵,不适用于临床实践。因此,在所研究的菌株基因组中选择一些有代表性的看家基因和水平转移基因进行抽样检测,再作样本聚类分析成为合理的选择[7]。
本研究中以3种与耐药相关的看家基因和54种水平转移获得与β-内酰胺类、氨基糖苷类和喹诺酮类药物耐药相关基因以及12种接合性质粒、转座子、插入序列、整合子等可移动遗传元件遗传标记测得结果作样本聚类分析,结果显示20株PDR-ABA中1-3-4-5-9-15-16-18-20号、7-10号、8-11号、12-14号及17-19号菌株均为克隆传播,而且其中1-3-4-5-9-15-16-18-20号菌株呈流行趋势。其中7-10号菌株均携带了14种水平转移基因,12-14号均携带了13种水平转移基因,如不考虑水平转移基因该4个菌株则无法鉴别。本研究中20株PDR-ABA经看家基因和水平转移基因检测分析提示,3种看家基因均存在有义突变,并检出多种β-内酰胺酶基因、多种氨基糖苷类修饰酶基因和抗菌药物外排泵基因adeB、qacE△1,同时检出5种可移动遗传元件。可见本组PDR-ABA泛耐药的表型除了看家基因carO、gyrA、parC的有义突变,还借助可移动遗传元件获得了多种抗菌药物灭活酶基因和外排泵基因。
综上所述,与全基因组测序后并对SNPs作聚类分析解析菌株亲缘性相比,看家基因和水平转移基因样本聚类分析实用性强,能够用于对医院内感染的实时监测。
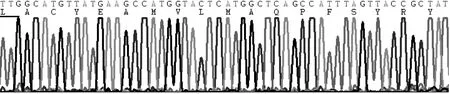
图1 parC(第80位)突变部位测序图(敏感株第80位密码子为TCA)
[1]Munoz-Price L S,Weinstein R A.Acinetobacter Infection[J].N Engl J Med,2008,358(12):1271-1281.
[2]Park Y K,Peck K R,Cheong H S,et al.Extreme drug resistance in Acinetobacter baumannii infections in intensive care units,South Korea[J].Emerg Infect Dis,2009,15(8):1325-1326.
[3]Tian G B,Adams-Haduch J M,Taracila M,et al.Extended-spectrum AmpC cephalosporinase in Acinetobacterbaumannii: ADC-56 confers resistance to cefepime[J].Antimicrob Agent Chemother,2011,55(10):4922-4925.
[4]Kaase M,Nordmann P,Wichelhaus T A,et al.NDM-2 carbapenemase in Acinetobacter baumannii from Egypt[J].J Antimicrob Chemother,2011,66(6):1260-1262.
[5]Kuo L C,Teng L J,Yu C J,et al.Dissemination of a clone of unusual phenotype of pandrug-resistant Acinetobacter baumannii at auniversity hospital in Taiwan[J].J Clin Microbiol,2004,42(4): 1759-1763.
[5]肖永红.Mohnarin 2008年度ICU细菌耐药性监测[J].中华医院感染学杂志,2010,20(16):2384-2388.
[6]秦楠,栗东芳,杨瑞馥.高通量测序技术及其在微生物学研究中的应用[J].微生物学报,2011,51(4):445-457.
[7]姜如金,朱健铭,吴康乐.基于管家基因与水平转移基因的菌株亲缘性分析[J].中华医院感染学杂志,2011,21(18):3765-3769.
[8]Alam M T,Merlo M E,Takano E,et al.Genome-based phylogenetic analysis of Streptomyces and its relatives[J].Mol Phylogenet Evol, 2010,54(3):763-772.
[9]Harris S R,Feil E J,Holden M T,et al.Evolution of MRSA during hospital transmission and intercontinental spread[J].Science,2010, 327(5964):469-474.
[10]Morelli G,Song Y,Mazzoni C J,et al.Yersinia pestis genome sequencing identifies patterns of global phylogenetic diversity[J].Nat Genet,2010,42(12):1140-1143.
[11]Rasko D A,Webster D R,Sahl J W,et al.Origins of the E.coli strain causing an outbreak of hemolytic-uremic syndrome in Germany [J].N Engl J Med,2011,365(8):709-717.
Phylogenetic analysis of drug resistance-related genes in pandrug-resistant Acinetobacter baumannii(PDR-ABA)
Objective To conduct phylogenetic analysis in 20 strains of pandrug-resistant Acinetobacter baumannii (PDR-ABA).Methods The analysis was completed in three drug resistance-related housekeeping genes(carO,gyrA,parC) and 54 species level shift obtained with β-lactams,aminoglycosides,quinolones resistance-related genes as well as 12 kinds of bonding nature of the particles,transposons,insertion sequences,integrons and other mobile genetic elements of genetic markers in 20 pan-resistant Acinetobacter baumannii.Results In 20 strains of PDR-ABA,4 kinds of β-lactam-acquired drug resistance genes,5 kinds of aminoglycoside acquired drug resistance genes,2 kinds of acquired drug efflux genes,5 kinds of removable genetic markers genetic components were detected.There were sense mutations in housekeeping genes carO,gyrA, parC. Conclusion The cluster analysis showed that five clones spread exist in sample group of strains,and the strains 1-3-4-5-9-15-16-18-20 have epidemic trend.
Acinetobacter baumanniiHousekeeping gene Gene acquired by horizontal transfer Phylogenetic analysis

2013-12-30)
(本文编辑:胥昀)
312000 绍兴市第六人民医院感染控制科
