脱羧酶基因ARO10的过量表达及其 对Saccharomyces cerevisiae的β-苯乙醇合成代谢影响
2014-03-22,,,*,,,,*,
, ,,*, ,,,*,
(1.北京工商大学北京市食品添加剂工程技术研究中心,北京 100048;2.北京市食品风味化学重点实验室,北京 100048;3.河南中大生物工程有限公司,郑州 451162)
β-苯乙醇,又称为2-苯乙醇,是一种具有持久淡雅玫瑰香气的芳香醇类物质,广泛应用于食品、化妆品和烟草行业[1-2]。天然β-苯乙醇主要存在于显花植物的花果及其提取的精油中,但因其浓度低,提取困难,目前β-苯乙醇主要还是通过化学方法进行合成。虽然化学合成的成本低、产量高,但存在着环境污染严重,产生有毒副产物等弊端,不符合现代消费者崇尚“天然、安全和环保”的绿色消费理念[3-4]。由微生物转化制备的香料,被称为天然等同香料,符合“绿色生产”的安全标准,而且其构型、手性单一,有助于香气品质的提高,是近年来天然香料制备的重要发展方向[5-6]。
国外已有部分学者通过研究酿酒酵母中氨基酸代谢来探索β-苯乙醇的微生物制备方法[7-8],而国内还鲜有报道。在酿酒酵母中,以L-苯丙氨酸为底物,经艾利希途径合成β-苯乙醇,是β-苯乙醇生物转化的快捷途径,受到转氨酶、脱羧酶和醇脱氢酶的调节[6,9]。Vuralhan等以苯丙氨酸、蛋氨酸和亮氨酸为唯一氮源进行恒化器培养,对培养得到的酵母细胞提取物进行分析,发现存在一种具有广泛底物活性的脱羧酶Aro10p(EC 4.1.1.72)参与催化反应。ARO10是编码这种脱羧酶的基因,其转录情况与α-酮酸脱羧酶在恒化器中培养时的表达情况密切相关;将除ARO10基因外的其他α-酮酸脱羧酶基因全部敲除,研究发现此突变株仍然表现出了脱羧酶活性[10-12]。因此认为增强ARO10基因拷贝与表达,有利于增强目的产物β-苯乙醇的产量。本文以S.cerevisiaeS288C为出发菌株,将脱羧酶基因ARO10进行过量表达,研究其对β-苯乙醇合成途径的影响,为β-苯乙醇工程菌构建与改造提供指导。
1 材料与方法
1.1 实验材料
EscherichiacoliTOP10感受态细胞 购自北京天根公司;SaccharomycescerevisiaeS288C(ATCC 26108TM)、质粒PYES2-G418(G418抗性) 由本实验室制备保藏;质粒小提试剂盒、胶回收试剂盒 购于OMEGA公司;酵母质粒提取试剂盒 购于北京天根生化科技有限公司;pMD19-T Simple Vector、DNA marker、Ex Taq、限制性内切酶、T4 DNA 连接酶 购于TaKaRa公司;酵母提取物、胰蛋白胨 购于北京拜尔迪生物公司;甲醇(色谱纯) 购于Fisher公司;β-苯乙醇 为本院香精香料实验室制备;其余试剂均为国产分析纯。
1.2 培养基
LB培养基[13]、YPD种子培养基[13]。
发酵培养基(g/L):无氨基酸氮源YNB 1.8,蔗糖40,L-苯丙氨酸7,Na2HPO40.5。
1.3 方法
1.3.1ARO10基因的克隆与表达载体构建 根据Genbank和相关文献[14]中公布的S.cerevisiae3-磷酸甘油激酶基因启动子片段Ppgk和S.cerevisiaeAR010基因编码序列,利用Primer 5.0软件设计引物。扩增Ppgk所用引物:PGK-F:5′-TATCAGACCGGTTATTTTAGATTCCTGACTT-3′,下划线部分为酶切位点AgeI,PGK-R:5′-CGCTCGCAGCTGTGTTTTTATATTTGTTGT-3′,下划线部分为酶切位点PvuII。扩增ARO10的引物ARO10F:5′-CGAGCTCATGGCACCTGTTACAATT-3′,下划线部分为酶切位点SacI;ARO10R:5′-CTCTAGACTATTTTTTATTTCTTTTAAGTGCC-3′,下划线部分为酶切位点XbaI。酵母转化子检测所用引物:
T7F:5′-CAGCTGTAATACGACTCACTATAGGG-3′,
CYC1R:5′-AAATAAATAGGGACCTAGACTTC AGG-3′。
以S.cerevisiaeS288C 基因组为模板,Ex Taq DNA Polymerase进行PCR反应。25μL反应体系如下:Ex Taq聚合酶0.3μL,基因组<1μg,上下游引物各1μL(10μmol/L),dNTP混合物4μL(2.5mmol/L),10×Ex Taq Buffer 5μL,ddH2O 至25μL;
扩增Ppgk和ARO10基因的反应条件分别为:94℃ 5min;94℃ 30s,55℃ 30s,72℃ 40s,30 个循环;72℃ 10min。
94℃ 5min;94℃ 30s,58℃ 30s,72℃ 2min,30个循环;72℃ 10min。
分别将扩增的Ppgk片段和ARO10片段连接到pMD19-T Simple质粒,进行测序;对可能带有Ppgk片段和ARO10基因的质粒pMD19-T-Ppgk、pMD19-T-ARO10进行酶切回收。
重组质粒pYES2-Ppgk-ARO10构建见图1。先用AgeI和PvuII分别对质粒pMD19-T-Ppgk和pYES2-G418于37℃酶切5h。将酶切产物经1%琼脂糖凝胶电泳分离,用DNA琼脂糖凝胶回收试剂盒回收目的片段,经 T4 DNA Ligase于4℃连接过夜,连接产物转化于E.coliTOP10感受态细胞,选取阳性菌落扩繁并进行重组质粒提取,得到质粒pYES2-Ppgk。再用SacI和XbaI分别对质粒pMD19-T-ARO10和pYES2-Ppgk进行酶切,其余方法同上,最终得到重组质粒pYES2-Ppgk-ARO10。
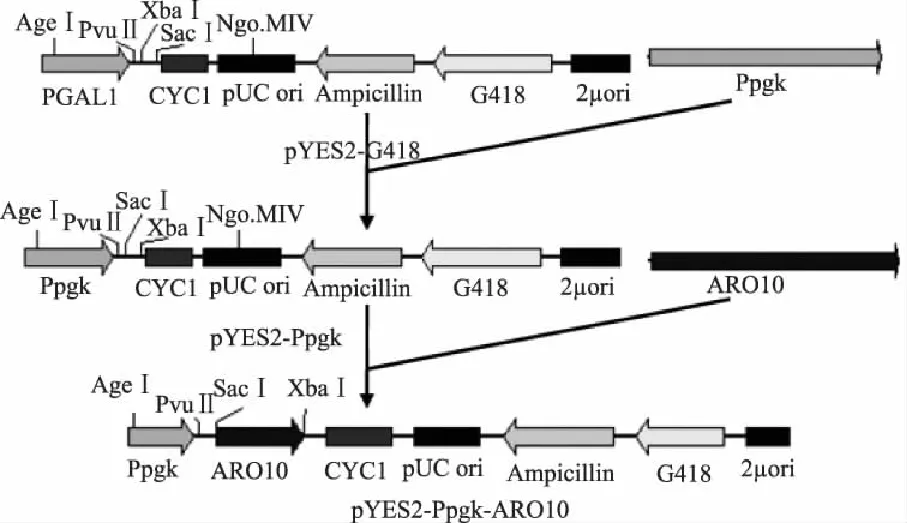
图1 重组质粒pYES2-Ppgk-ARO10的构建策略 Fig.1 The construction strategy of recombinant plasmid of pYES2-Ppgk-ARO10
1.3.2 重组质粒在酿酒酵母中的表达 利用PEG/LiAc高效酵母转化方法将空载pYES2-Ppgk和重组表达载体pYES2-Ppgk-ARO10分别导入S.cerevisiaeS288C感受态细胞中,获得的酵母转化子经PCR验证。将酵母阳性克隆接种到YPD液体培养基中,于30℃,200r/min,过夜振荡培养。取适量菌液接种于发酵培养基中,最终调至OD600=0.25,30℃继续培养,每隔一定时间收集发酵液。高效液相色谱(HPLC)检测发酵液中的β-苯乙醇。
1.3.3 β-苯乙醇的HPLC检测与数据处理 准确吸取0.100g β-苯乙醇并加水定容于10mL容量瓶中,制成10g/L的贮备液,再分别吸取贮备液10、30、50、70、80、100和150μL加水混匀定容于7个10mL容量瓶中制成0.01、0.03、0.05、0.07、0.08、0.10和0.15g/L的标准溶液,经0.45μm微孔滤膜过滤装入色谱分析瓶,HPLC检测其峰面积,以标准溶液浓度为横轴,测得的峰面积为纵轴绘制其标准曲线。
将获得的酿酒酵母转化子SP10发酵培养,收集得到的发酵液8000r/min离心,取适量上清液稀释10倍用于β-苯乙醇的HPLC检测。检测β-苯乙醇采用LC-20A岛津液相色谱仪,检测条件[15]为:色谱柱 RP-C18 色谱柱(4.6mm×150mm,5μm),V(甲醇)∶V(水)=50∶50作为流动相,检测波长215nm,进样量10μL,流速1.0mL/min。由此测出发酵液中β-苯乙醇特征峰的峰面积,代入上述标准曲线,即可得出发酵液中的β-苯乙醇浓度,再利用Excel 2003对其浓度和发酵时间作图。
2 结果与分析
2.1 ARO10基因的克隆与表达载体构建
以S.cerevisiaeS288C基因组为模板,分别用上下游引物PGK-F,PGK-R;ARO10F,ARO10R,扩增目的片段Ppgk和ARO10,PCR产物电泳检测。由图2和图3可知,扩增得到的单一DNA片段分别为780bp和1900bp左右,与预期大小相符。
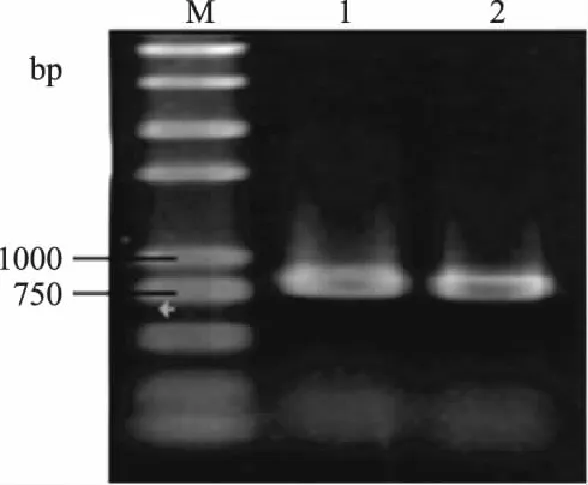
图2 基因Ppgk的扩增 Fig.2 Amplification of Ppgk gene by PCR
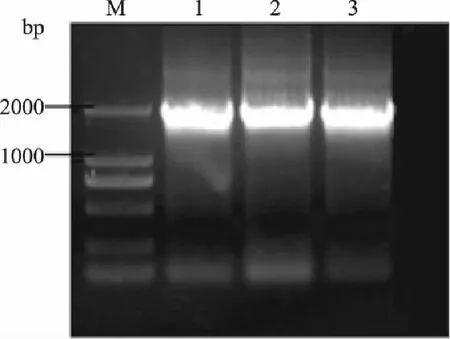
图3 基因ARO10的扩增 Fig.3 Amplification of ARO10 gene by PCR
DNA琼脂糖凝胶回收试剂盒回收目的片段,连接到pMD19-T Simple Vector,连接产物转化大肠杆菌E.coliTOP10感受态细胞,选取阳性菌落扩繁并进行酶切验证,1%琼脂糖凝胶电泳检测。筛选得到的转化子均切下大小正确的条带,且测序结果与Genbank 所公布基因序列一致。
按照1.3.1的方法构建重组质粒pYES2-Ppgk和pYES2-Ppgk-ARO10,将经测序正确的阳性转化子过夜培养,用质粒小提试剂盒提取质粒pYES2-Ppgk和pYES2-Ppgk-ARO10。分别对两种质粒进行酶切验证,酶切鉴定见图4及图5。

图4 质粒pYES2-Ppgk的酶切验证 Fig.4 Restriction analysis of plasmid pYES2-Ppgk
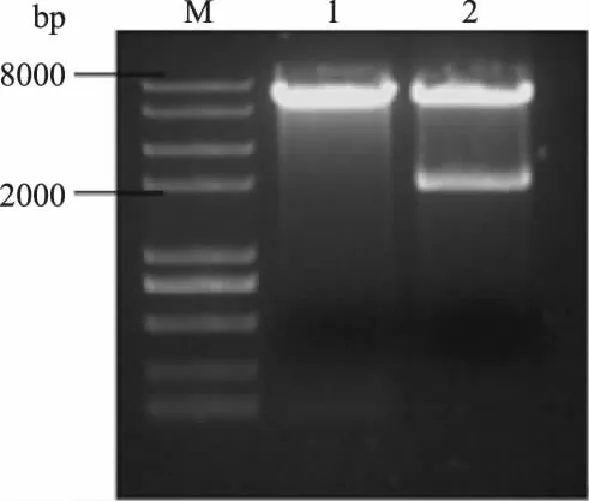
图5 质粒pYES2-Ppgk-ARO10的酶切验证 Fig.5 Restriction analysis of plasmid pYES2-Ppgk-ARO10
2.2 重组酿酒酵母的筛选与鉴定
重组质粒pYES2-Ppgk和pYES2-Ppgk-ARO10转入酿酒酵母后,在含G418的YPD平板上进行筛选,在G418浓度达到200μg/mL的平板上未见酿酒酵母S288C菌株生长。
分别从转入质粒pYES2-Ppgk和pYES2-Ppgk-ARO10的筛选平板上挑取几个单菌落作为重组菌株,提取转化子的质粒,为避免假阳性结果的出现,分别选择引物PGK-F/CYC1R和引物T7F/CYC1R对两种质粒进行PCR扩增,扩增的片段大小约为1000bp和2000bp。凝胶电泳检测结果如图6、图7显示,筛选到的转化子中均扩增出目的基因特异性条带,表明构建的两种重组质粒均已转化进入酿酒酵母菌株中;阴性对照为已转化质粒pYES2-G418的酿酒酵母S288C菌株,无目的条带。

图6 pYES2-Ppgk转化子PCR验证 Fig.6 The detection of pYES2-Ppgk transformants by PCR
图7 pYES2-Ppgk-ARO10转化子PCR验证 Fig.7 The detection of pYES2-Ppgk-ARO10 transformants by PCR
2.3 摇瓶发酵实验和β-苯乙醇检测
β-苯乙醇的标准溶液经高效液相色谱(HPLC)分析,β-苯乙醇特征峰的出峰时间约在9min. 按照1.3.3中β-苯乙醇标准曲线绘制的方法,处理和进样测定,以β-苯乙醇质量浓度为横坐标,峰面积为纵坐标,绘出标准曲线(图8)。β-苯乙醇在0.01~0.15g/L 范围内呈现良好线性关系,线性回归方程为y=2×107x-10005(R2=0.9976)。

图8 β-苯乙醇标准曲线 Fig.8 Standard Curve of β-phenethyl alcoho
按照1.3.3中的检测条件,对发酵液进行液相色谱分析,能够较好的分离β-苯乙醇,并通过其峰面积与标准曲线,计算其在发酵液中的浓度。由图9可知,β-苯乙醇生成量随着发酵时间的延长逐渐积累,发酵60h时其生成量达到最高,导入空质粒pYES2-Ppgk的重组菌株SP和S.cerevisiaeS288C野生菌,β-苯乙醇的生成量均为0.86g/L,转化子SP10的β-苯乙醇的生成量为1.0g/L,相较前两者均提高16.3%;继续发酵到96h,由于β-苯乙醇的积累对于酵母细胞有一定的毒性作用,抑制其生长,且在摇瓶过程中有一些挥发损失,所以β-苯乙醇产量没有再增加,而是均有小幅度的下调。结果表明,S.cerevisiaeS288C中脱羧酶是β-苯乙醇生物合成途径的关键酶,增强脱羧酶基因ARO10的克隆及基因表达,有利于提高β-苯乙醇的产量,为将来β-苯乙醇的酿酒酵母工程菌构建提供了方向。
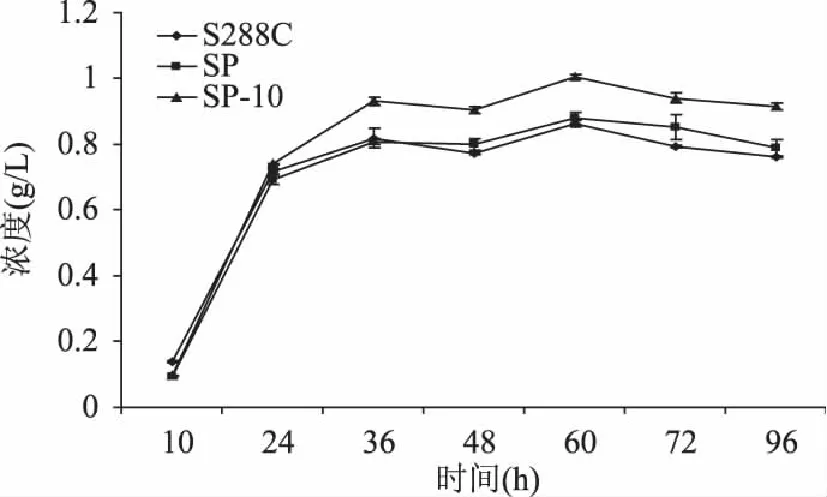
图9 发酵时间对S. cerevisiae的β-苯乙醇合成的影响 Fig.9 The β-phenethyl alcohol content in different time
3 结论与讨论
3.1 结论
将PGK1启动子片段克隆并替换原pYES2-G418穿梭质粒中的诱导型启动子,构建新的表达载体pYES2-Ppgk;再将脱羧酶基因ARO10克隆与表达载体pYES2-Ppgk连接,构建了酿酒酵母表达质粒pYES2-Ppgk-ARO10。经测序验证后,LiAc/SSDNA/PEG方法转化至S.cerevisiaeS288C中进行表达,通过基因表达、发酵产物测定等不同水平检测,结果表明,构建的酿酒酵母转化子SP10 β-苯乙醇产量有所提高,发酵60h时,其生成量达到1.0g/L,较野生型菌株提高了16.3%。因此,PGK1启动子可用于穿梭质粒的启动表达,且新构建的表达载体可以使脱羧酶基因ARO10在酿酒酵母中过量表达;S.cerevisiaeS288C中脱羧酶Aro10p是β-苯乙醇生物合成途径的关键酶,增强脱羧酶基因ARO10的克隆及基因表达,有利于增强β-苯乙醇的合成代谢及目的产物产量的提高
3.2 讨论
启动子是表达载体上不可或缺的核心元件之一,其活性强弱直接影响目的基因转录效率的高低,进而影响基因表达水平。本研究使用的酿酒酵母3-磷酸甘油酸激酶PGK1基因的启动子为组成型强启动子,转录活性高,已广泛应用于外源基因在酿酒酵母中的表达;此外,采用组成型表达的策略无需加入诱导剂,避免了对诱导物浓度、诱导时间和温度等条件进行优化的繁琐工作,使目的基因的表达更高效方便,同时消除了诱导物对目的产物代谢途径的影响并有利于后续实验开展重组菌株的代谢流量分析。
脱羧酶基因ARO10在酿酒酵母中的过量表达有利于增强β-苯乙醇的合成代谢及目的产物产量的提高。但其产量并没有达到最理想水平,可能有几方面的原因:一是实验所用培养基与培养条件没有进行优化;二是只进行了小规模的摇瓶发酵,还未进行发酵罐的发酵实验,由于在发酵过程中,无法进行补料和一些重要发酵参数的控制,所以在一定程度上限制了β-苯乙醇的产量;三是β-苯乙醇的合成代谢通路中有多种酶进行调控,改变其中一些基因的表达,对于目的产物的代谢流量的影响有限,本实验室也正在构建氨基转移酶ARO8基因与脱羧酶ARO10基因耦合过量表达以及副反应途径基因敲除的工程菌,以增强主反应途径的相关酶基因的表达及代谢流量,减弱或阻断副产物支路代谢流量,为后续的研究提供了思路和技术数据的支持。
[1]孙宝国.食品添加剂[M]. 北京:化学工业出版社,2008:59-85.
[2]Zhu Y J,Zhou H T,Hu Y H,etal. Antityrosinase and antimicrobial activities of 2-phenylethanol,2-phenylacetaldehyde and 2-phenylacetic acid[J]. Food Chemistry,2011,124(1):298-302.
[3]王成涛,苏伟,陈刚.天然食品配料--生产与应用[M]. 北京:化学工业出版社,2010:1-87.
[4]Etschmann M M W,Sell D,Schrader J. Production of 2-phenylethanol and 2-phenylethylacetate from L-phenylalanine by coupling whole‐cell biocatalysis with organophilic pervaporation[J]. Biotechnology and bioengineering,2005,92(5):624-634.
[5]Perpète P,Duthoit O,De Maeyer S,etal. Methionine catabolism in Saccharomyces cerevisiae[J]. FEMS yeast research,2006,6(1):48-56.
[6]Hazelwood L A,Daran J M,Antonius JA,etal. The Ehrlich pathway for fusel alcohol production:a century of research on
Saccharomyces cerevisiae metabolism[J]. Applied and environmental microbiology,2008,74(8):2259-2266.
[7]Dickinson J R,Salgado L E J,Hewlins M J E. The catabolism of amino acids to long chain and complex alcohols in Saccharomyces cerevisiae[J]. Journal of Biological Chemistry,2003,278(10):8028-8034.
[8]Kneen M M,Stan R,Yep A,etal. Characterization of a thiamin diphosphate-dependent phenylpyruvate decarboxylase from Saccharomyces cerevisiae[J]. FEBS Journal,2011,278(11):1842-1853.
[9]Eshkol N,Sendovski M,Bahalul M,etal. Production of 2-phenylethanol from L-phenylalanine by a stress tolerant Saccharomyces cerevisiae strain[J]. Journal of Applied Microbiology,2009,106(2):534-542.
[10]Vuralhan Z,Luttik M A H,Tai S L,etal. Physiological characterization of the ARO10-dependent,broad-substrate-specificity 2-oxo acid decarboxylase activity of Saccharomyces cerevisiae[J]. Applied and environmental microbiology,2005,71(6):3276-3284.
[11]Vuralhan Z,Morais M A,Tai S L,etal. Identification and characterization of phenylpyruvate decarboxylase genes in Saccharomyces cerevisiae[J]. Applied and environmental microbiology,2003,69(8):4534-4541.
[12]Romagnoli G,Luttik M A H,Kötter P,etal. Substrate specificity of thiamine pyrophosphate-dependent 2-oxo-acid decarboxylases in Saccharomyces cerevisiae[J]. Applied and environmental microbiology,2012,78(21):7538-7548.
[13]奥斯伯F M,布伦特R,金斯顿R E,等. 分子生物学实验指南[M]. 北京:科学出版社,2005.
[14]阳辛凤,郭安平,孔华,等. 工业酿酒酵母PGK启动子的克隆与功能分析[J].中国农学通报,2012,28(6):178-182.
[15]诸葛庆,李博斌,郑云峰,等. 反相高效液相色谱法测定黄酒中的β-苯乙醇[J].食品科学,2009,30(14):175-177.
