一种快速聚合酶链式反应方法的探究和法医学应用
2014-03-22董会李彩霞王晶贾竟刘超
董会,李彩霞,王晶,贾竟,刘超
(1.南方医科大学基础医学院法医学系,广州510515;2.公安部物证鉴定中心,北京市现场物证检验工程技术研究中心,法医遗传学公安部重点实验室,北京100038;3.郑州大学基础医学院遗传学实验室,郑州450000)
·技术方法·
一种快速聚合酶链式反应方法的探究和法医学应用
董会1,李彩霞2,王晶2,贾竟3,刘超1
(1.南方医科大学基础医学院法医学系,广州510515;2.公安部物证鉴定中心,北京市现场物证检验工程技术研究中心,法医遗传学公安部重点实验室,北京100038;3.郑州大学基础医学院遗传学实验室,郑州450000)
目的优化和建立一套快速聚合酶链式反应(PCR)扩增体系和程序,缩短扩增时间,提高短串联重复序列分型的速率和办案效率。方法运用AmpFLSTR®Identifiler®Plus试剂盒和7种快速扩增酶进行筛选和优化,最后对筛选出的酶优化其扩增体系和扩增程序,并对几种常规检材中提取的DNA进行扩增和检测,然后对其分型结果进行验证和讨论。结果最终优化的扩增程序由常规的3 h左右缩短至24 min,DNA检验结果与常规方法一致。结论应用优化后的快速PCR扩增体系可以成功准确扩增DNA样本,显著提高DNA分型速率。
法医学;快速聚合酶链式反应;复合短串联重复序列
生物物证的DNA检验在公安办案实践中发挥着举足轻重的作用,案(事)件尤其是一些大要案往往要求在极短时间甚至在现场就提供检验结果,为案(事)件的快速处置、侦查诉讼等提供及时的线索和证据。目前,生物物证常规检测步骤包括DNA提取、定量、复合短串联重复序列(short tandem repeat,STR)扩增、电泳检测等,最终获得常染色体或Y染色体STR分型,整个过程大致需要10~12 h。而聚合酶链式反应(polymerase chain reaction,PCR)一般需要3 h左右,是生物物证检验的一个重要限速过程[1]。如果能将此过程进一步缩短,将会显著提升物证检验的速度,提高法医检验的效率,满足案件侦查和诉讼对于时效性的要求,提升生物物证的证据价值和证明力,为公安机关进行PCR扩增提供便利。
快速PCR就是基于普通PCR的工作原理,在保证PCR反应特异性、灵敏度和保真度的前提下,在更短时间内完成对核酸分子的扩增[2]。近年来,快速PCR主要从3个方面分别进行了研发:聚合酶的改进、添加剂的研发以及热循环仪的革新创造[3]。快速扩增DNA聚合酶以及快速升/降温速率的PCR扩增仪[4]的出现,使快速PCR技术在法医学中大规模应用成为可能。快速PCR技术在核基因组DNA和线粒体DNA的相关研究[5]中均占据重要位置。本文以当前法医DNA实验室常用的STR复合扩增试剂[6]和快速扩增酶为主要研究对象,建立了一套快速扩增体系和程序,整个扩增程序只需24 min即可完成,显著缩短了物证检验时间,为公安机关及科研院所进行PCR扩增和检验提供了便利。
1 材料与方法
1.1 样本
50份无关个体静脉血由中山大学第一附属医院提供,84份案例检材(血布片10份、血棉签10份、唾液布片10份、唾液棉签10份、接触类检材10份、骨头7份、牙齿7份、精斑5份、口腔拭子10份、头发毛囊5份)由公安部物证鉴定中心提供。
1.2 主要试剂和仪器
SpeedSTAR®HS DNA酶(日本TaKaRa公司),KAPA2GTM快速PCR试剂盒(美国Kapa Biosystems公司),QIAGEN快速PCR试剂盒和M48 QIAGEN®试剂盒(美国QIAGEN公司),PyroStartTMFast PCR Master Mix(2×)(加拿大Fermentas公司),FastStart Taq DNA酶,dNTPack(德国Roche Applied Science公司),PfuUltraⅡFusion HS DNA酶(美国Agilent公司),Fast HiFidelity PCR试剂盒(TIANGEN公司),AmpFLSTR®Identifiler®Plus试剂盒和ABI 3130XL遗传分析仪(美国Applied Biosystems公司),蛋白酶K(德国Merck公司),Mastercycler pro Eppendorf®热循环仪(德国Eppendorf公司),SpeedCycler2热循环仪(德国Analytik Jena公司),NANODROP 2000C分光光度计(美国Thermo Scientific公司)。
1.3 方法
1.3.1 模板DNA的提取:采用磁珠法[7]提取全部的实验样本,具体操作参照M48 QIAGEN®试剂盒使用说明。
1.3.2 对模板DNA的定量:使用NANODROP 2000C分光光度计进行DNA定量,操作参照仪器试剂使用说明。
1.4 快速扩增酶的筛选和优化
1.4.1 扩增时间为1 h左右的快速酶的筛选和优化:对以上7种酶和1种常规扩增试剂盒进行扩增,扩增时间在1 h左右,扩增体系为每种酶的标准扩增体系。最终筛选出TaKaRa、Roche、Fermentas这3家公司的3种酶,位点扩出较多,可以进一步优化和筛选[8,9]。7种酶的推荐扩增体系如下:(1)TaKaRa 10 μL PCR扩增体系:Fast BufferⅠ(10×,含有30 mmol/L MgCl2)1 μ L,Identifiler primer mix 2 μ L,SpeedSTAR®HS DNA酶(5 U/μL)0.1 μL,dNTP Mixture(2.5 mmol/L each)0.8 μL,灭菌水5.1 μL,模板DNA 1 μL。(2)QIAGEN 10 μL PCR扩增体系:QIAGEN Fast Cycling PCR Master Mix 5 μL,Identifiler primer mix 2 μL,灭菌水2 μL,模板DNA 1 μL。(3)Roche 10 μL PCR扩增体系:PCR reaction buffer(10×,含有20 mmol/L MgCl2)1 μL,Identifiler primer mix 2 μL,FastStart Taq DNA酶(5 U/μL)0.2 μL,MgCl2(25 mmol/L)0.4 μL,PCR Grade Nucleotide Mix 0.2 μL,灭菌水5.2 μL,模板DNA 1 μL。(4)KAPA 10 μL PCR扩增体系:5×KAPA2G Buffer A(含有20 mmol/L MgCl2)2 μL,dNTP Mix(10 mmol/L each)0.2 μL,Identifiler primer mix 2 μL,KAPA2G Fast DNA酶(5 U/μL)0.2 μL,灭菌水4.6 μL,模板DNA 1 μL。(5)Aglient 10 μL PCR扩增体系:10×PfuUltraⅡreaction buffer 1 μL,dNTP mix(25 mmol/L each)0.2 μ L,Identifiler primer mix 2 μL,PfuUltraⅡfusion HS DNA酶0.2 μL,灭菌水5.6 μL,模板DNA 1 μL。(6)TIANGEN 10 μL PCR扩增体系:5×Fast HiFidelity PCR Buffer 2 μL,Identifiler primer mix 2 μL,Fast HiFidelity酶0.4 μL,灭菌水4.6 μL,模板DNA 1 μL。(7)Fermentas 10 μL PCR扩增体系:PyroStartTMFast PCR Master Mix(2×)5 μL,Identifiler primer mix 2 μL,灭菌水2 μL,模板DNA 1 μL。
2组筛选扩增程序(此程序是综合扩增程序时间在1 h左右以及根据每种酶推荐程序推导得出)如下:扩增分别在Mastercycler pro Eppendorf®热循环仪(扩增仪的升降温速率分别为6℃/s和4.5℃/s)和SpeedCycler2热循环仪(扩增仪的升降温速率分别为12℃/s和8℃/s)上进行,热循环参数1为:95℃2 min,95℃20 s,59℃20 s,72℃20 s,30个循环;72℃延伸3 min;25℃保存[10]。扩增时间分别为63 min和43 min。热循环参数2为:95℃4 min,95℃30 s,59℃30 s,72℃30 s,30个循环;72℃延伸7 min;25℃保存。扩增时间分别为78 min和58 min。
1.4.2 扩增时间为30 min左右的快速酶的筛选和优化:用Roche、TaKaRa、Fermentas 3种酶分别扩增AmpFlSTR®Identifiler®Plus Primer Mix。在Mastercycler pro Eppendorf®热循环仪和SpeedCycler2热循环仪上进行优化和调整。从以下10个方面对热循环程序进行优化调整:(1)酶的用量:0.05、0.10、0.20、0.40 μL;(2)引物的用量:2、3、4 μL;(3)镁离子终浓度调整:2、3、4、5 mmol/L;(4)预变性时间调整:1、2、3、4 min;(5)变性时间调整:5、10、15、20、30 s;(6)退火时间调整:5、10、15、20、30 s;(7)延伸时间调整:5、10、15、20、30 s;(8)终延伸时间调整:1、3、5、7、10、20 min;(9)退火温度调整:56、57、58、59、60、61、62℃;(10)循环数调整:25、28、30、33、35个循环[11,12]。
最终优化出扩增时间最短、扩增效力最好的Ta-KaRa公司的SpeedSTAR酶的10 μL PCR扩增体系:Fast BufferⅠ(10×,含有30 mmol/L MgCl2)1 μL,Identifiler primer mix 2 μL,SpeedSTAR®HS DNA酶(5 U/μL)0.05 μL,dNTP Mixture(2.5 mmol/L each)0.8 μL,灭菌水5.15 μL,模板DNA 1 μL。扩增在SpeedCycler2热循环仪上进行,热循环参数为:95℃1 min,95℃10 s,59℃10 s,72℃10 s,30个循环;72℃延伸1 min;25℃保存。扩增时间为24 min。
1.4.3 对优化的扩增体系和扩增程序进行一致性和样品适应性验证:应用优化的热循环条件对收集的134份样本进行复合扩增,将1 μL扩增产物和9 μL甲酰胺内标混合物混合变性后,应用ABI 3130XL型遗传分析仪进行电泳检测,用GeneMapper ID V3.3软件自动分析数据。每组均设置阳性对照和空白试剂阴性对照。甲酰胺内标混合物配比参照AmpFLSTR®Identifiler®Plus试剂盒使用说明。
1.4.4 对优化的扩增体系和扩增程序进行灵敏度测试:将9947A(阳性标准品)和样本1、2、3、4、5五份DNA分别稀释为1、0.750、0.500、0.250、0.125、0.063、0.032 ng/μL[13],用优化的热循环条件进行扩增,产物用ABI 3130XL型遗传分析仪进行电泳检测。每组均设置空白试剂阴性对照。
1.4.5 对优化的扩增体系和扩增程序进行稳定性检测:运用优化后的快速PCR扩增程序和扩增体系对同一份样品在同一批次进行5次重复实验[14],扩增后用ABI 3130XL型分析仪进行电泳检测。每组均设置阳性对照和空白试剂阴性对照。
1.5 统计学分析
所得DNA检测图谱均与已知分型结果比对,以各等位基因峰高大于50相对荧光单位(relative fluorescence unit,RFU)者为有效分型结果[15,16]。检出率(%)=检测到的样本数/总样本数。
2 结果
2.1 快速扩增酶的筛选和优化结果
2.1.1 扩增时间为1 h左右的快速酶的筛选和优化结果:图1为7种酶的商品化试剂盒推荐扩增体系和Identifiler plus引物在同一扩增程序1下,在Eppendorf热循环仪上进行扩增,然后进行3130电泳检测的分型图,通过综合分析DNA分型图谱的准确性、均衡性等初次筛选出3个公司的3种酶:Roche、TaKaRa、Fermentas。
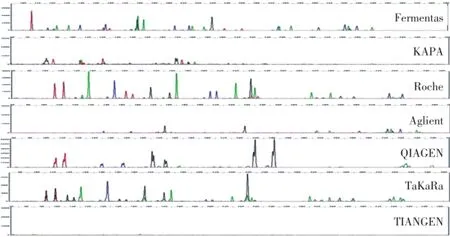
图1 7种快速酶的初次筛选电泳分型图Fig.1 Classification of the seven fast PCR enzyme for the initial selection
2.1.2 扩增时间为30 min左右的快速酶的筛选和优化结果:通过系统研究和优化,以时间最短和扩增效力最好为优化选择条件,最终从Fermentas、 Roche和TaKaRa 3个公司的3种酶中筛选出TaKaRa公司的酶,扩增程序在SpeedCycler2热循环仪上进行,扩增时间仅为24 min,见图2。

图2 3种快速酶的最终优化筛选电泳分型图Fig.2 Classification of the three fast PCR enzyme for the final selection
2.2 一致性与样品适应性验证实验结果
应用优化的热循环条件对50份静脉血DNA和84份案件检材DNA进行扩增和电泳,在扩增不同类型的检材时均获得完整分型结果,且所得分型结果均与常规扩增方法分型结果一致,见表1和图3。

表1 快速PCR分型与常规分型一致性的实验结果Tab.1 Classification results of the fast PCR and the standard procedure
2.3 灵敏度测试
实验结果显示当样品DNA浓度达到或超过0.25 ng/μL可以获得完整基因座分型,且无非特异扩增现象,每个浓度6份样品平均的峰高即RFU值以散点图的形式呈现。见表2和图4。
2.4 体系稳定性
运用快速PCR程序和体系用AmpFLSTR®Identifiler®Plus试剂盒引物对同一份样品在同一批次进行5次重复实验,PCR分型结果一致,峰面积无较大差异。见图5和图6。
3 讨论
本文从缩短PCR扩增的时间和快速热循环仪参数的优化出发,针对AmpFLSTR®Identifiler®Plus试剂盒引物和7种快速PCR扩增酶,进行快速酶的筛选以及扩增体系和扩增程序的建立和优化,构建适合法医学检验的快速扩增体系和程序。本研究对快速扩增酶的筛选、快速PCR扩增体系的构建和优化进行了较完整和系统的实验研究和汇报,为快速PCR实验提供了可供参考的实验条件和理论依据。对快速PCR的实验条件进行优化后,扩增时间明显缩短,平均耗时仅为24 min,比标准的耗时3 h左右的热循环程序明显缩短,与Tsukada[13]和Wang[2]的文献报道的2 h左右的快速扩增时间相比也明显缩短。在实验初筛中,Fermentas和Roche的快速酶在扩增时间为1 h左右时,扩增效力较好,明显优于TaKaRa公司的快速酶,但在扩增时间缩短至30 min内时,Fermentas和Roche的快速酶出现丢点现象,峰高均衡性较差,而TaKaRa公司的快速酶在扩增时间分别为1 h与30 min内时相对保持稳定,未出现丢点现象,峰高相对均衡。因此实验最终针对TaKaRa公司的快速酶进行了深度优化,最终获得了扩增效力较好、扩增时间仅为24 min的扩增体系和程序。

图3 不同类型的检材用最终优化的TaKaRa酶的快速扩增体系扩增所得的电泳分型结果Fig.3 Classification results of the final fast PCR with DNA extracted from different types of samples using AmpFLSTR Identifiler plus PCR amplification kits and the fast PCR enzyme of TaKaRa

表2 灵敏度测试的实验结果Tab.2 Experimental results of sensitivity tests
实验通过134份包含不同种类的检材,进行一致性和样品适应性验证,均获得良好分型图谱,从图3可以得出,此扩增体系和扩增程序能够广泛适用于各种法医常规检材。对随机一份样品同一条件下重复实验5次,PCR分型结果一致稳定,峰高和峰面积无较大差异,从图5和图6可以看出,峰高分布集中,5次重复实验无较大差异。表明此扩增体系和扩增程序不是随机性,而是具有良好的稳定性且具有峰高均衡性。Vallone等[6]的一些实验研究中出现大量非特异扩增现象和双尖情况,而使用本研究建立的快速扩增体系和扩增程序进行扩增时则无此现象,体现了此快速扩增体系和扩增程序的优势。

图4 不同浓度时9947A的各基因座的平均峰高散点图Fig.4 The scatter diagram of the average peak height of different concentration in all loci of 9947A
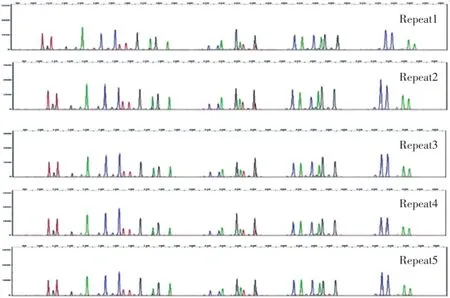
图5 样品5次重复的电泳分型图Fig.5 Classification results of the sample for five repeat

图6 样品5次重复的各基因座的平均峰高散点图Fig.6 The scatter diagram of the average peak height of different concentration in all loci of the sample for five repeat
针对随机5份样品和9947A进行灵敏度验证,结果显示只要模板DNA浓度达到或超过0.250 ng/μL就能保证等位基因不丢失,表明此扩增体系和程序对样品量要求较低。从图4可以得出,样本在不同浓度扩增时峰高较均衡集中,体现了此扩增体系和扩增程序的均衡性较好,也从侧面反映了此体系和程序的稳定性。常规扩增时AmpFLSTR®Identifiler®Plus试剂盒的灵敏度为0.125 ng/μL,在Vallone等[6]的一些实验研究中,其快速检测体系和程序的灵敏度也均在0.125 ng/μL左右。与常规扩增相比,本研究建立的快速扩增体系和扩增程序显著提升了检测速率,但在一定程度上降低了检测的灵敏度,其灵敏度为0.250 ng/μL,但是仍能满足各种常见法医检材检验的需求,且与目前国际上关于快速PCR检测的各项研究中的灵敏度相一致。
在Vallone[6]和Laurin[3]的研究中,其用酶量均消耗较大,本文建立的扩增体系和扩增程序显著提高了扩增效率,10 μL扩增体系中酶的用量仅为0.25 U且适用于各种常规检材,这为公安机关以及科研院所进行PCR扩增提供了便利。随着新的快速酶的不断产生、热循环仪的不断优化,PCR扩增时间和扩增效力将有望进行进一步缩短和提高。
综上所述,本研究建立的快速PCR扩增体系和扩增程序,扩增时间仅为24 min,比标准的耗时3 h左右的热循环程序明显缩短,为公安机构和科研院所进行快速PCR反应提供了便捷方法。
[1]Verheij S,Harteveld J,Sijen T.A protocol for direct and rapid multiplex PCR amplification on forensically relevant samples[J].Forensic Sci Int Genet,2012,6(2):167-175.
[2]Wang DY,Chang CW,Hennessy LK.Rapid STR analysis of single source DNA samples in 2 h[J].Forensic Sci Int Genet Suppl Ser,2009,2(1):115-116.
[3]Laurin N,Fregeau C.Optimization and validation of a fast amplification protocol for AmpFLSTR profiler plus for rapid forensic human identification[J].Forensic Sci Int Genet,2012,6(1):47-57.
[4]杨文超,张晓东.快速PCR研究进展[J].中国生物工程杂志,2007,27(4):99-103.
[5]张明阳,单海燕,徐冬冬,等.PCR-SSCP技术分析线粒体DNA多态性及其法医学应用[J].中国医科大学学报,2011,40(4):334-336.
[6]Vallone PM,Hill CR,Podini D,et al.Rapid amplification of commercial STR typing kits[J].Forensic Sci Int Genet Suppl Ser,2009,2(1):111-112.
[7]Kishore R,Reef WH,Anderson VJ,et al.Optimization of DNA extraction from low-yield and degraded samples using the BioRobot EZ1 and BioRobot M48[J].J Forensic Sci,2006,51(5):1055-1061.
[8]Pavlov AR,Pavlova NV,Kozyavkin SA,et al.Recent developments in the optimization of thermostable DNA polymerases for efficient applications[J].Trends Biotechnol,2004,22(5):253-260.
[9]Collins PJ,Hennessy LK,Leibelt CS,et al.Developmental validation of a single-tube amplification of the 13 CODIS STR loci,D2S1338,D19S433 and amelogenin:the AmpFLSTR Identifiler PCR Amplification Kit[J].J Forensic Sci,2004,49(6):1265-1277.
[10]Vallone PM,Hill CR,Butler JM.Demonstration of rapid multiplex PCR amplification involving 16 genetic loci[J].Forensic Sci Int Genet,2009,3(1):42-45.
[11]Bienvenue JM,Legendre LA,Ferrance JP,et al.An integrated microfluidic device for DNA purification and PCR amplification of STR fragments[J].Forensic Sci Int Genet,2010,4(3):178-186.
[12]Wang Y,Prosen DE,LI M,et al.A novel strategy to engineer DNA polymerases for enhanced processivity and improved performance in vitro[J].Nucleic Acids Res,2004,32(3):1197-1207.
[13]Tsukada K,Harayama Y,Kurasawa Y,et al.Fast PCR amplification AmpFLSTR Identifiler:second report[J].Forensic Sci Int Genet Suppl Ser,2009,2(1):108-110.
[14]Choung CM,Lee DS,Park KW,et al.Test of the rapid PCR method using AmpFLSTR Identifiler Kit[J].Forensic Sci Int Genet Suppl Ser,2011,3(1):475-476.
[15]Butler JM.Forensic DNA typing[M].2nd ed.New York:Elsevier,2005:410-420.
[16]Butler JM.Genetics and genomics of core STR loci used in human identity testing[J].J Forensic Sci,2006,51(2):253-265.
[17]韩俊萍,李彩霞,严红,等.低体积PCR扩增用于单细胞分离和检验[J].中国法医学杂志,2012,28(2):123-125.
(编辑 陈姜)
An Exploration ofthe Rapid Polymerase Chain Reaction Method and Its Application in Forensic Science
DONGHui1,LICai-xia2,WANGJing2,JIAJing3,LIUChao1
(1.DepartmentofForensic Science,Basic Medicine College,Southern MedicalUniversity,Guangzhou 510515,China;2.Institute ofForensic Science,Key Laboratory ofForensic Genetics,Beijing Engineering Research CenterofCrime Scene Evidence Examination,Ministry ofPublic Security,Beijing 100038,China;3.Institute ofGenetics,Basic Medicine College,Zhengzhou University,Zhengzhou 450000,China)
ObjectiveTo establish a rapid polymerase chain reaction(PCR)cycling protocol,so as to help shorten amplification time and improve generating short tandem repeats profiles and working efficiency.MethodsDNA extracted from the common samples were amplified with the optimized PCR method and cycling protocol using AmpFLSTR®Identifiler®Plus PCR Amplification Kits and the fast PCR enzyme filtrated from seven fastPCRenzyme.The genotyping results were then validated and discussed.ResultsThe developed PCRmethod,using filtrated fastPCRenzyme,shortened the time from 3 h of standard procedure to 24 min,and the genotyping results were in accord with the results generated by standard procedure.ConclusionWe successfully amplified the STR loci of several routine forensic specimens using the established rapid cycling protocol.Rapid thermalcycling conditions can significantly improve the rate ofDNA profiling.Moreover,unbalanced amplification ofthe loci,which may occurusing commercialkitsassingle primerconcentration cannotbe adjusted,now can be overcame with the established system.
forensic medicine;rapid polymerase chain reaction;multiplex short tandem repeat
R89
A
0258-4646(2014)10-0931-07
国家自然科学基金(81202384);中央公益类基本科研业务费(2012JB012)
董会(1990-),女,硕士研究生.
刘超,E-mail:liuchaogaj@21cn.com
2014-07-05
网络出版时间:
