针对ALK4 基因的TALEN 质粒构建与活性鉴定
2014-01-14曾凡才顾洪王轲周红
曾凡才 顾洪 王轲 周红
(1.电子科技大学生命科学与技术学院,成都 610054;2.泸州医学院生物化学与分子生物学实验室,泸州 646000)
TGF-β 超家族包括40 个结构相关成员,它们参与细胞的增殖、分化、粘附、迁移和凋亡等生物学效应[1,2]。这些成员通过与具有丝氨酸/苏氨酸激酶活性的两种膜受体(Ⅰ型和II 型)结合和装配
发挥作用,一般是配体分子先与II 型受体结合后招募和激活Ⅰ型受体,激活的Ⅰ型受体促使下游信号分子Smads 磷酸化,磷酸化的Smads 从胞质穿梭到核并调节靶基因的转录[3,4]。至今,已发现5 种哺乳动物II 型受体(TβR-II、ActR-IIA、ActR-IIB、BMPR-II 和AMHR-II)和7 种I 型受体(Activin receptor-like kinase 1-7,ALK1-7)[5],其中Activin 的II 型受体(ActRIIA 和ActRIIB)和Ⅰ型受体(ALK4)除与Activins 形成复合物外,还结合GDF1、GDF3、GDF8(myostatin)和Nodal 等家族成员,同时这些配体也激活ALK4 以外的其它受体信号[6]。因此,为了更好地研究这些配体和受体的功能及其相互关系,敲除该信号通路中起关键作用的Ⅰ型受体ALK4,对阐明这些复杂的信号通路具有重要意义。
类转录激活因子效应物核酸酶(Transcription activator-like effector nuclease,TALEN)是一种设计简便、特异性好和毒性低的基因敲除技术。自2010年底应用于基因敲除[7],目前已在大鼠、小鼠、斑马鱼、酵母、植物和人类细胞等多种生物体中成功应用[8]。TALEN 的设计理念主要来自类转录激活因子效应物(Transcription activator-like effector,TALE)和另一基因敲除技术锌指核酸酶(Zinc-finger nuclease,ZFN)的研究成果。TALE 是主要存在于植物病原体黄单胞菌的一类高度保守的蛋白家族[9]。TALE 的结构主要由N 末端的转移结构域(TS)、中间DNA 结合结构域、C 末端的核定位信号(NLS)和转录激活结构域(AD)组成。TALE 的不同主要在于DNA 结合结构域中重复片段的数量和重复可变的双残基(Repeat variable di-residue,RVD)[10]。RVD 是指每个重复单位中+12 和+13 位的氨基酸残基,它们是实现靶向特异识别DNA 碱基的关键位点,随靶点核苷酸序列的不同而异。2009 年,两个研究小组分别阐明了TALE 蛋白中RVD 与靶序列碱基的对应关系,最常见的4 种RVD 与碱基的识别关系分别是NI(Asn Ile)-A、NG(Asn Gly)-T、HD(His Asp)-C 和NN(Asn Asn)-G[11,12]。通过TALE 蛋白的氨基酸识别碱基规则,结合锌指核酸酶的研究成果将TALE 中的转录激活结构域(AD)替换成核酸内切酶(Fok Ⅰ)的切割结构域,即构建为TALEN。人们只要将TALEN 的DNA 结合结构域替换为设计的靶点识别域,即可对基因组的特定靶点进行切割,从而实现基因打靶。因此,TALEN 技术的关键步骤是TALEN 靶点识别域的构建。本试验采用改进后的质粒文库快速直接构建法,只需一步即可完成构建反应。鉴于ALK4 在TGF-β 超家族信号通路中的重要性和信号通路本身的复杂性,本研究详细介绍用于敲除ALK4 基因的TALEN 质粒构建过程,并进一步鉴定其活性,为建立不同的ALK4 基因敲除细胞系,研究ALK4 基因的功能及其与不同配体之间的关系做准备。
1 材料与方法
1.1 材料
1.1.1 细胞 HEK293T 细胞购自中国科学院上海细胞库。
1.1.2 试剂和引物 嘌呤霉素购自Sigma 公司;Taq DNA 聚合酶、基因组DNA 提取试剂盒、高纯度中抽质粒提取试剂盒、核酸分子量标准购自天根生物有限公司;BamH I、EcoR I 和Hha I 核酸内切酶、小抽质粒提取试剂盒购自宝生物(大连)公司;PCR 产物纯化试剂盒购自罗氏公司;卡那霉素购自上海生工;LipofectamineTM2000、DMEM 高糖培养基、Opti-MEM 液体培养基购自Invitrogen 公司;pEGFPN1 质粒由本实验室保存;TALEN 试剂盒购自上海斯丹赛生物技术有限公司;测序和引物合成由上海英骏生物技术有限公司完成。本研究所用引物序列如表1 所示。

表1 测序及PCR 扩增所用引物序列
1.2 方法
1.2.1 确定ALK4 基因的靶位点和识别序列 从美国国家生物技术信息中心(NCBI)搜索到ALK4 基因序列。利用TALEN 设计专业网站(https://tale-nt.cac.cornell.edu/),根据天然TALEs 识别序列的特征和TALEN 靶点的选择原则[13],并综合考虑ALK4的不同剪接异构体序列,最终确定一个作用靶点和TALEN 左右臂识别序列。
1.2.2 根据识别序列构建质粒 采用上海斯丹赛生物技术有限公司的TALEN 试剂盒构建TALEN 质粒。这种方法基于含有TALEN 的DNA 结合重复序列的172 个质粒文库模块(图1),将左、右臂识别序列的最后一位T 去除,以1 个或2 个碱基组合为一个模块,首个标记为1,最后一个标记为9,依次类推进行标记。每次必须选择9 个模块,最后两个模块必须为双模块。左右臂标记情况见图1,其中以右臂为例,模块选择情况见图中粗体加下划线显示,左臂按照相同方法构建。
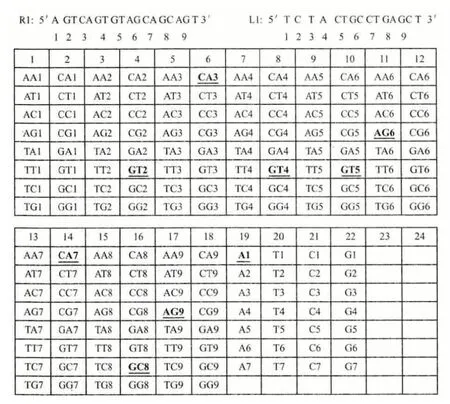
图1 TALEN 左右臂的模块选择
每 个 模 块 取1.5 μL,9 个 模 块 共13.5 μL。连接反应体系如下:9 个模块 13.5 μL,左臂或右臂TALEN 骨架载体 1.5 μL,溶液3 2.0 μL,溶液1 1.0 μL,溶液2 1.0 μL,ddH2O 1.0 μL。总体积为20 μL。混匀后在PCR 仪中连接,反应程序如下:37℃ 5 min,16℃ 10 min,15 个 循 环;80℃ 10 min;12℃ 1 min。取出PCR 管并加入1 μL 溶液4 和0.5 μL 溶液5,混匀,37℃孵育1 h,转化后涂板于含卡那霉素的LB 琼脂平板,37℃培养过夜。其中,溶液1-5为TALEN 试济盒中所配。
1.2.3 酶切鉴定 从平板中挑取15 个单克隆菌落,在含卡那霉素的LB 琼脂平板上划线培养,同时接种于5 mL 含抗卡那霉素的LB 培养液中,37℃ 250 r/min 培养16 h。用常规质粒小抽试剂盒提取质粒进行酶切:ddH2O 8 μL,10×K Buffer 2 μL,Plasmid 8 μL,BamH I 1 μL,EcoR I 1 μL;总体积20 μL。置于37℃水浴中孵育1 h。在1%琼脂糖中电泳鉴定,根据酶切后片段大小,选择符合条件的单克隆测序。
1.2.4 BLAST 比 对 在 网 址http://blast.ncbi.nlm.nih.gov/Blast.cgi 中选择tblastx(使用氨基酸比对),勾选Align two or more sequences(两序列或更多序列比对),将拼接好的标准核苷酸序列和测序序列分别输入到上、下文本框中,在BLAST 比对结果中查找比对正确的结果。比对正确的质粒进行反向测序,其中右臂TALEN 表达质粒由于插入片段较长,除正反方向测序外,还在中间设计引物以便测序完整(测序引物见表1)。经BLAST 比对完全正确的单克隆,用中抽质粒提取试剂盒提取质粒。
1.2.5 最低嘌呤霉素浓度的确定 TALEN 质粒带有抗嘌呤霉素基因,可利用嘌呤霉素筛选转染阳性细胞,其最低浓度由以下试验确定:取对数生长期HEK293T 细胞,生长于24 孔板(1×105个/孔),24 h 后分别加入含0、0.5、1、2、3 和4 μg/mL 嘌呤霉素的新鲜筛选培养基,每天更换筛选培养基且监测存活细胞数量,最终确定3-4 d 能杀死所有细胞的最低嘌呤霉素浓度。
1.2.6 转染试验 取对数生长期HEK293T 细胞,以1×106个/孔的密度接种于6 孔板中,24 h 后转染。转染体系如下:左臂TALEN 质粒 2 μg,右臂TALEN 质 粒 2 μg,pEGFP-N1 质 粒 0.5 μg,Opti-MEM 2×250 μL,LipofectamineTM2000 13.5 μL; 对照组细胞不转染。24 h 后观察GFP 表达并替换成含2 μg/mL 嘌呤霉素的筛选培养基。每天更换筛选培养基,观察到对照细胞全部死亡后,收集试验组细胞,一半细胞继续培养,一半细胞用于提取基因组DNA。对照组不进行细胞转染和不加筛选培养基,直接提取基因组DNA。
1.2.7 PCR 扩增和酶切鉴定 提取HEK293T 细胞基因组DNA,定量后进行PCR 扩增,反应体系为:基因组DNA 2 μL、10 mmol/L dNTPs 1 μL、2.5 mmol/L Mg2+4 μL、10 pmol/L ALK4-TALEN-F 和ALK4-TALEN-R 各1 μL、Taq DNA 聚合酶1 μL、10×PCR Buffer 5 μL 和ddH2O 35 μL。扩 增 程 序 为:95℃ 5 min;94℃ 30 s,60℃ 30 min,72℃ 30 s,共30 个循环;72℃ 10 min。取5 μL PCR 产物电泳鉴定,剩余PCR 产物纯化后Hha Ⅰ内切酶处理:Hha Ⅰ 1 μL,10×M Buffer 2 μL,PCR 产 物12 μL,ddH2O 5 μL;总体积20 μL。37℃水浴中孵育3 h,3%琼脂糖凝胶电泳30 min,凝胶成像系统拍照和灰度分析。
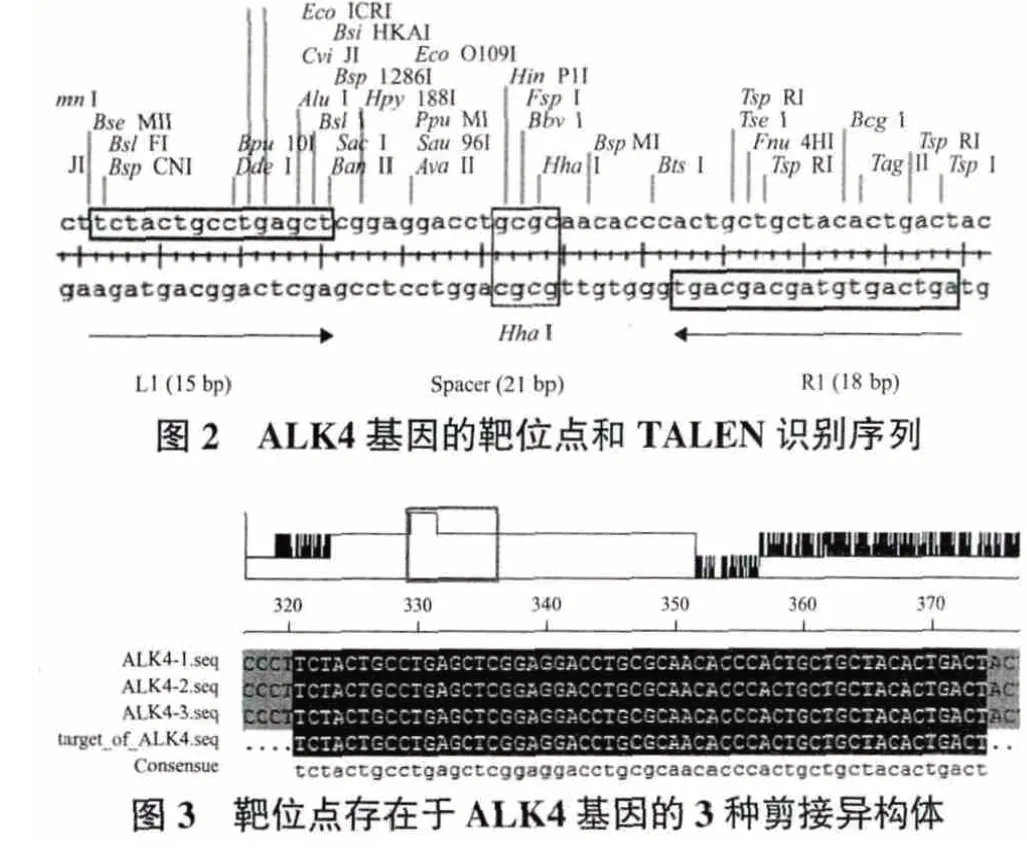
2 结果
2.1 ALK4基因的靶位点及TALEN识别序列的确定
根据Bogdanove 和Voytas 描述的TALEN 靶位点选择原则,在ALK4 基因的TALEN 靶位点左右臂识别序列5'端的前一位(第0 位)碱基都为胸腺嘧啶(T),且靶序列的最后一个碱基(3'端碱基)也都是胸腺嘧啶(T)(对应于最后的0.5 个TALE 重复单元)。左臂识别序列的长度为15 bp,右臂识别序列的长度为18 bp,中间间隔序列的长度为21 bp,间隔序列中有Hha Ⅰ酶切位点,可用于判断TALEN质粒的活性(图2)。90%人类基因都有剪切异构体,目前发现至少有3 种可编码蛋白的ALK4 剪接异构体,本试验设计的TALEN 质粒可作用于ALK4 基因的3 种剪接异构体的第二外显子(图3)。为了避免SNP 影响TALEN 的识别效果,PCR 扩增HEK293T细胞基因组中的靶序列并测序,发现靶序列与设计识别序列完全一致(图4)。
2.2 TALEN质粒构建及酶切鉴定
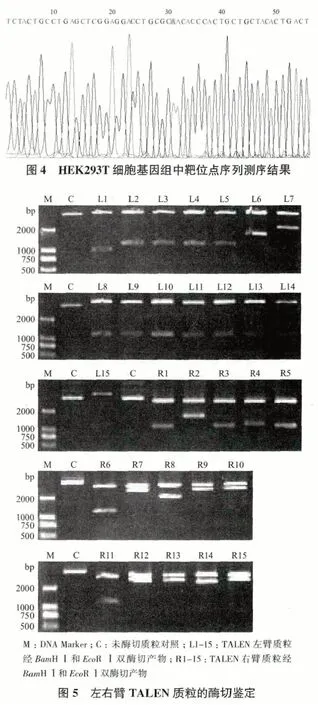
构建的左右臂TALEN 质粒均挑取15 个单克隆菌落,抽提质粒进行酶切鉴定(图5)。TALEN 左臂骨架载体为5 057 bp,其中BamH Ⅰ和EcoR Ⅰ两酶切位点之间的片段为803 bp。构建敲除ALK4 基因的TALEN 左臂质粒在BamH Ⅰ和EcoR Ⅰ酶切位点之间插入识别重复单位,左臂质粒插入15 bp 的识别序列,除最后一个碱基胸腺嘧啶(T)设计在载体骨架上外,还需插入14 个碱基的识别序列,每个碱基的识别序列由102 bp 核苷酸组成,因此需在骨架载体中插入1 428 bp 的核酸片段。如果构建正确,TALEN 左臂表达质粒经上述双酶切和电泳分离后应观察到4 254 bp 和2 231 bp 的两条片段,据此分析判断L7 最符合此条件(图5),用于测序。右臂骨架质粒缺少抗嘌呤霉素基因,只有3 792 bp 大小,双酶切位点之间的片段也为803 bp,右臂应插入18 个碱基的识别序列,除最后一位T 设计在载体骨架上外,还需插入1 734 bp 的核酸片段。如果构建成功,TALEN 质粒经双酶切和电泳分离后应观察到2 989 bp 和2 537 bp 的两条片段。结果发现R7、R9、R10、R12、R13、R14 和R15 编号质粒均可能构建正确(图5),将它们测序。
2.3 BLAST比对分析
由于识别4 种碱基的重复单位仅在+12 和+13位的氨基酸残基不一致,因而不论是拼接的标准序列还是测序序列都是一些相似度很高的序列重复,它们的BLAST 比对结果会有很多,需要从中找到正确的比对结果。根据左臂插入的标准核苷酸序列长度为1 428 bp 和L7 质粒的测序结果,正向测序比对正确的结果应选择标准核苷酸序列从第1 位,测序序列从第64 位碱基开始进行比对,且两序列一致性为100%的结果(图6-A);反向测序比对正确的结果应选择标准核苷酸序列是以第1 428 位碱基比对结束,且对应的反向测序序列应在第28 位碱基,两序列的一致性为100%的结果(图6-B)。L7 质粒正反向测序比对正确的结果中有327 个核苷酸所编码的氨基酸重叠且序列完全一致(图6),说明TALEN左臂质粒L7 构建成功。其它质粒以相同方法分析,结果与L7 质粒类似(图略)。右臂质粒正向测序结果表明R13 存在一个位点的突变,其它质粒序列正确。选择其中R9 和R12 进行完整测序,BLAST 比对确认右臂R9 和R12 质粒构建成功。

图6 L7 质粒序列BLAST 比对结果
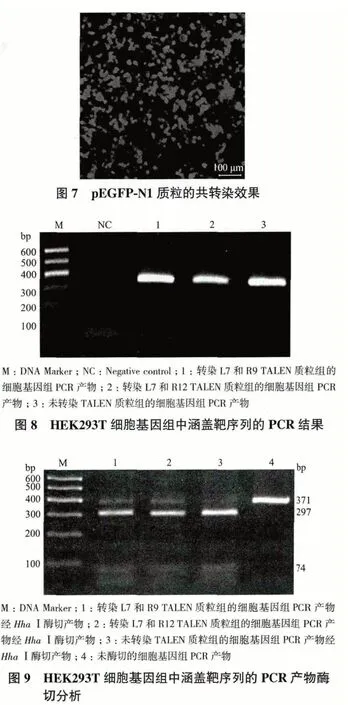
2.4 TALEN质粒的活性鉴定
通过共转染24 h 的pGFP-N1 质粒,荧光显微镜观察到至少60%的转染效率(图7)。同时利用嘌呤霉素进行筛选,以最低嘌呤霉素浓度试验确定的2 μg/mL 嘌呤霉素在加药3 d 时可致未转染细胞全部死亡,而转染TALEN 质粒的HEK293T 细胞还剩余至少50%,一定程度反映了转染效率。用ALK4 基因靶序列两端设计的PCR 引物扩增HEK293T 细胞基因组DNA,电泳结果表明无论是否转染TALEN 质粒都可获得与预期371 bp 一致的特异带(图8)。此PCR 产物纯化后经Hha Ⅰ内切酶酶切,未转染组的PCR 产物完全被切开,生成与预期297 bp 和74 bp一致的两条带,而转染TALEN 质粒的一部分PCR产物未被切开,灰度分析大约占总条带的10%,说明其Hha Ⅰ内切酶位点已发生突变,部分HEK293T细胞的ALK4 基因被敲除(图9)。此外,L7+R9 和L7+R12 两个质粒组合因为识别序列一样,作用于靶点的活性也基本相同(图9)。
3 讨论
本研究综合考虑靶点选择原则和ALK4 剪接异构体的共同序列,最终将靶点确定在ALK4 基因的第二外显子,确保如果ALK4 基因被敲除后,ALK4功能不会被其剪接异构体所补偿。靶序列中间有一Hha Ⅰ酶切位点以方便判断质粒活性[14]。如果识别序列中只要有一个碱基出现错配就有可能较大幅度地降低TALEN 的活性。因此,通过PCR 扩增HEK293T 细胞基因组中的靶序列并测序,比对后发现该段序列在HEK293T 细胞基因组中不存在SNP现象。通过一步法构建TALEN 质粒,用酶切鉴定和测序,最后通过BLAST 序列比对成功获得敲除ALK4 基因的TALEN 质粒,并用酶切鉴定的方法证明在HEK293T 细胞中有一定活性。
与其它基因敲除技术相比,TALEN 简便实用,理论上可作用于基因组中的任何位点[15]。目前TALEN 的构建方法包括DNA 序列直接合成、Golden Gate 克隆、PCR 与Golden Gate 克隆结合、Toolbox组装、基于同尾酶的单元组装、FLASH 组装和LIC组装等,它们各有特点[14,16-18]。例如,北京大学张博[19]课题组开发的基于同尾酶的单元组装法已在斑马鱼的基因敲除中取得很好的效果,该方法费用较低,但步骤较多。如果构建TALEN 较多,积累了较多不同重复单元的模块后,效率也比较高。本研究采用改进后的质粒文库快速直接构建,可通过一步连接反应构建识别序列为12-19 bp 之间任意长度的TALEN 质粒。一般挑选15 个克隆即可筛选到构建正确的质粒,而且在TALEN 载体质粒上设计有嘌呤霉素抗性基因,可用嘌呤霉素提高筛选效率。
TGF-β 超家族信号通路中存在一个配体作用于多个受体,一个受体也可接受多个配体信号这一复杂现象。为揭示这种复杂的关系,敲除其中的重要基因是一个不错的选择。ALK4 作为多种TGF-β 超家族成员的受体分子,在这一复杂的关系中处于关键位置,但早期研究表明敲除ALK4 基因导致胚胎死亡[20],因此限制了对ALK4 基因功能的研究。最近发展起来的TALEN 基因敲除技术方便快捷,希望能利用该技术建立敲除ALK4 基因的不同细胞系,以研究ALK4 的功能。
本研究成功构建了敲除ALK4 基因的TALEN 质粒,并证明在HEK293T 细胞中有一定的活性。虽然通过观察共转染的GFP 表达以及嘌呤霉素的筛选都证明TALEN 质粒的转染效率较高,但TALEN的切割效率可能并不高,导致发生基因组突变的HEK293T 细胞并不多。这说明TALEN 技术虽在多个物种已取得成功,但不能保证每个位点都有高活性。这是因为TALEN 这一新兴技术还没有完全被认识,但其有效性是肯定的。我们下一步将通过大量筛选细胞得到敲除ALK4 基因的细胞系或者在同一位点设计多条左右臂TALEN 质粒,组合使用这些质粒以筛选到更高活性的TALEN 质粒。
4 结论
本研究成功构建了敲除ALK4 基因的TALEN质粒,并采用脂质体法导入HEK293T 细胞,且TALEN 质粒有一定的活性。
[1] Pardali K, Moustakas A. Actions of TGF-beta as tumor suppressor and pro-metastatic factor in human cancer[J]. Biochim Biophys Acta, 2007, 1775(1):21-62.
[2] Massague J, Gomis RR. The logic of TGFbeta signaling[J]. FEBS Lett, 2006, 580(12):2811-2820.
[3] Shi Y, Massague J. Mechanisms of TGF-beta signaling from cell membrane to the nucleus[J]. Cell, 2003, 113(6):685-700.
[4] Schmierer B, Hill CS. TGFbeta-SMAD signal transduction :molecular specificity and functional flexibility[J]. Nat Rev Mol Cell Biol, 2007, 8(12):970-982.
[5] Piek E, Heldin CH, Ten Dijke P. Specificity, diversity, and regulation in TGF-beta superfamily signaling[J]. FASEB J, 1999, 13(15):2105-2124.
[6] Harrison CA, Gray PC, Vale WW, et al. Antagonists of activin signaling:mechanisms and potential biological applications[J]. Trends Endocrinol Metab, 2005, 16(2):73-78.
[7] Christian M, Cermak T, Doyle EL, et al. Targeting DNA doublestrand breaks with TAL effector nucleases[J]. Genetics, 2010, 186(2):757-761.
[8] Shen Y, Xiao A, Huang P, et al. TALE nuclease engineering and targeted genome modification[J]. Yi Chuan, 2013, 35(4):395-409.
[9] Bogdanove AJ, Schornack S, Lahaye T. TAL effectors:finding plant genes for disease and defense[J]. Curr Opin Plant Biol, 2010, 13(4):394-401.
[10] Yang B, Sugio A, White FF. Avoidance of host recognition by alterations in the repetitive and C-terminal regions of AvrXa7, a type III effector of Xanthomonas oryzae pv. oryzae[J]. Mol Plant Microbe Interact, 2005, 18(2):142-149.
[11] Boch J, Scholze H, Schornack S, et al. Breaking the code of DNA binding specificity of TAL-type III effectors[J]. Science, 2009, 326(5959):1509-1512.
[12] Moscou MJ, Bogdanove AJ. A simple cipher governs DNA recognition by TAL effectors[J]. Science, 2009, 326(5959):1501.
[13] Cermak T, Doyle EL, Christian M, et al. Efficient design and assembly of custom TALEN and other TAL effector-based constructs for DNA targeting[J]. Nucleic Acids Res, 2011, 39(12):e82.
[14] Huang P, Xiao A, Zhou M, et al. Heritable gene targeting in zebrafish using customized TALENs[J]. Nat Biotechnol, 2011, 29(8):699-700.
[15] 梁龙, 皮文辉. TAL 效应子应用研究进展[J].生物技术通报, 2012(11):54-59.
[16] Weber E, Gruetzner R, Werner S, et al. Assembly of designer TAL effectors by Golden Gate cloning[J]. PLoS One, 2011, 6(5):e19722.
[17] Li T, Huang S, Zhao X, et al. Modularly assembled designer TAL effector nucleases for targeted gene knockout and gene replacement in eukaryotes[J]. Nucleic Acids Res, 2011, 39(14):6315-6325.
[18] Zhang F, Cong L, Lodato S, et al. Efficient construction of sequence-specific TAL effectors for modulating mammalian transcription[J]. Nat Biotechnol, 2011, 29(2):149-153.
[19] Shen Y, Huang P, Zhang B. A protocol for TALEN construction and gene targeting in zebrafish[J]. Yi Chuan, 2013, 35(4):533-544.
[20] Gu Z, Nomura M, Simpson BB, et al. The type I activin receptor ActRIB is required for egg cylinder organization and gastrulation in the mouse[J]. Genes Dev, 1998, 12(6):844-857.
