烟酸转磷酸核糖激酶和丙酮酸羧化酶共表达对大肠杆菌BA002产丁二酸的影响
2013-10-31曹伟佳苟冬梅梁丽亚刘嵘明陈可泉马江锋姜岷
曹伟佳,苟冬梅,梁丽亚,刘嵘明,陈可泉,马江锋,2,姜岷
1 南京工业大学生物与制药工程学院 材料化学工程与国家重点实验室,江苏 南京 211816
2 中国石化扬子石油化工有限公司南京研究院,江苏 南京 210048
大肠杆菌 NZN111由于缺乏了乳酸脱氢酶的编码基因 (ldhA)和丙酮酸-甲酸裂解酶的编码基因 (pflB)而使副产物乳酸和甲酸的生产途径阻断,具有潜在生产丁二酸的能力。但同时由于NADH依赖的乳酸脱氢酶 (LDH)无法合成而限制了糖酵解过程中 NADH再生为 NAD+的过程,造成辅酶不平衡,导致该菌株厌氧条件下不能利用葡萄糖进行生长[1-3]。烟酸转磷酸核糖激酶(NAPRTase)是NAD(H)合成系统中的限速酶[4-6]。Liang等[7]通过在 NZN111中过量表达 NAPRTase基因 pncB,NAD+的浓度提高了 6.2倍,NADH/NAD+的比例大大降低,从而细胞生长和丁二酸产量有了大幅度提高。然而,发酵过程中伴随着大量的副产物丙酮酸生成,这是因为葡萄糖磷酸转移酶系统 (简称PTS)摄入胞外葡萄糖时,需要消耗磷酸烯醇式丙酮酸 (PEP)生成副产物丙酮酸,直接影响了丁二酸生产得率[8-9]。丙酮酸羧化酶(PYC)可催化丙酮酸到丁二酸前体草酰乙酸的反应,在重组大肠杆菌中表达有利于减少丙酮酸生成,提高丁二酸产量[10-12]。
本研究通过在E. coli BA002 (E. coli K12 △ldhA、△pflB)共表达烟酸转磷酸核糖激酶和外源丙酮酸羧化酶,构建了重组菌 BA016(BA002/pTrc99a-pncB-pyc),增加NAD(H)的总量,降低NADH/NAD+比例,同时减少了副产物丙酮酸的积累并促进了丁二酸的合成。
1 材料与方法
1.1 材料
BA002、BA014、BA015 (BA002/pTrc99a-pyc)和BA016,由南京工业大学生物过程与系统工程实验室自主构建;质粒pTrc99a,由南京师范大学邵蔚蓝教授惠赠。所有菌株用 20%甘油保藏在–80 ℃。发酵培养基:LB培养基:酵母粉5 g/L,蛋白胨10 g/L,氯化钠5 g/L,pH 7.0,氨苄青霉素的终浓度为100 µg/mL。血清瓶发酵为添加 20 g/L葡萄糖和16 g/L碱式碳酸镁;发酵罐发酵为添加35 g/L葡萄糖和 28 g/L 碱式碳酸镁。加入终浓度分别为0.5 mmol/L烟酸和0.3 mmol/L诱导剂IPTG (异丙基-β-D-硫代半乳糖苷)以诱导表达NAPRTase和PYC。
1.2 方法
1.2.1 培养方法
种子培养:将保存于–80 ℃的菌种在加有氨青霉素的平板上活化,挑单菌落于5 mL种子培养基中,37 ℃、200 r/min培养12 h。
发酵培养:接1 mL种子培养液加入到150 mL LB,有氧培养6 h。将种子液以15%的接种量接入装有1.5 L发酵培养基的3 L发酵罐中,温度和搅拌速率分别维持在30 ℃和170 r/min。以0.2 L/min的速率持续通入CO2。
1.2.2 分析方法
细胞生长用紫外可见分光光度计于波长 600 nm处测定吸光度值,有机酸、葡萄糖用高效液相色谱法 (HPLC)检测。
测定蛋白浓度用 Bradford法[13],以牛血清白蛋白 (BSA)为标准。NAPRTase酶活测定参照文献[14]。PYC酶活测定参照文献[15]。辅酶NADH与NAD+的浓度根据酶循环机理[16]测定。
2 结果与分析
2.1 对照菌与重组菌在血清瓶中纯厌氧发酵
由表1可知:72 h纯厌氧发酵后,BA002细胞干重仅为 0.52 g/L,合成了 1.57 g/L丁二酸,1.90 g/L丙酮酸。重组菌株BA014恢复了生长能力和发酵产酸能力,细胞干重是对照菌的 3.33倍,合成丁二酸5.04 g/L,但积累了4.71 g/L的丙酮酸。而BA015细胞干重为0.93 g/L,虽然丙酮酸减少至0.29 g/L,但合成的丁二酸只有3.61 g/L。重组菌BA016合成 15.68 g/L的丁二酸,比对照菌增加了7.97倍,而丙酮酸只有0.15 g/L。表2可知72 h厌氧条件下诱导后,BA016中 PYC的酶活增加至1.87 U/mg。BA014和BA016中NAPRTase的酶活明显高于BA002中的NAPRTase酶活。表2可以看出,重组菌株 BA016中 NADH/NAD+的比例从BA002中的0.60下降至BA014中的0.16和BA016中的0.05,该比例不断趋于辅酶平衡。
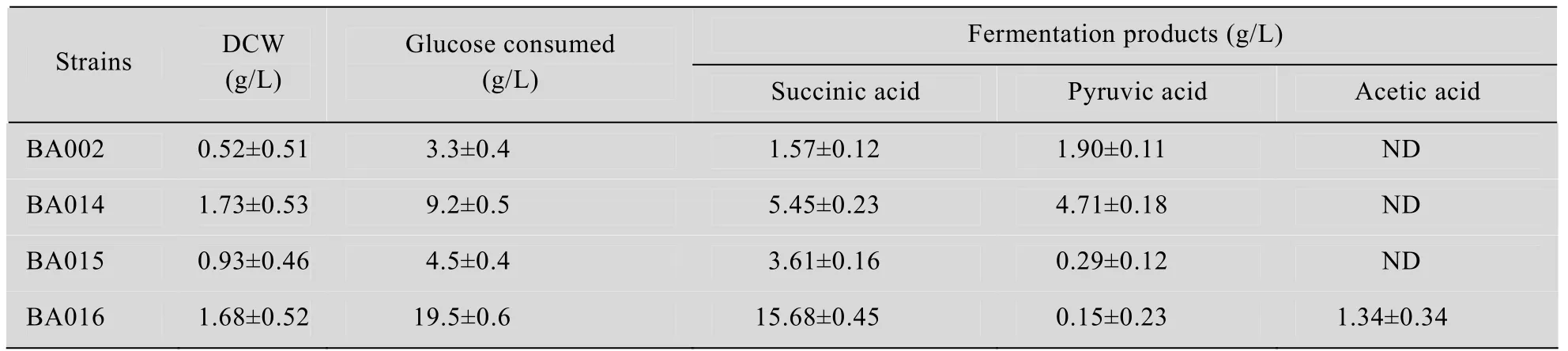
表1 不同菌株在LB培养基中72 h纯厌氧发酵结果Table 1 Results of exclusively anaerobic fermentations of the strains after 72 h in LB media
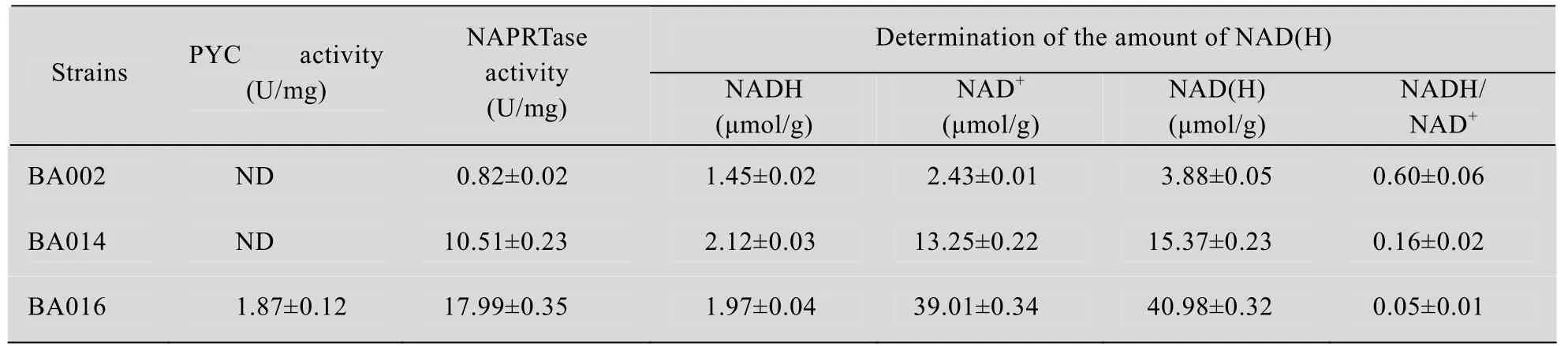
表2 不同菌株发酵72 h后PYC酶活、NAPRTase酶活和NAD(H)的测定Table 2 Enzyme assays and determination of the amount of NAD(H)in anaerobic fermentations of the strains after 72 h
2.2 BA016在3 L发酵罐中的纯厌氧发酵
由图1可以看出,发酵112 h后,共消耗了35 g/L的葡萄糖,菌体 OD600为 4.64,产生了 25.09 g/L丁二酸。41 h时,丙酮酸的浓度积累到3.23 g/L,随后开始下降,52 h时为0.09 g/L,而乙酸增加至2.35 g/L。表3可以看出41 h相比于0 h的PYC酶活显著增加至0.88 U/mg,此时代谢流从丙酮酸转向丁二酸的前体物草酰乙酸,这很可能是丙酮酸下降的原因。此外,41 h到64 h,NADH/NAD+下降了 4倍,丁二酸的生产效率达到最高,为0.4 g/(L·h)。说明下降的 NADH/NAD+对菌株生长和合成丁二酸具有促进作用。64 h后虽然葡萄糖消耗速率仍然很高,但是丁二酸生产效率下降,此时碳通量流向部分副产物的支路。酶活PYC的下降导致丙酮酸的积累,菌株在后期厌氧发酵时,NADH的不足导致苹果酸及富马酸的积累。
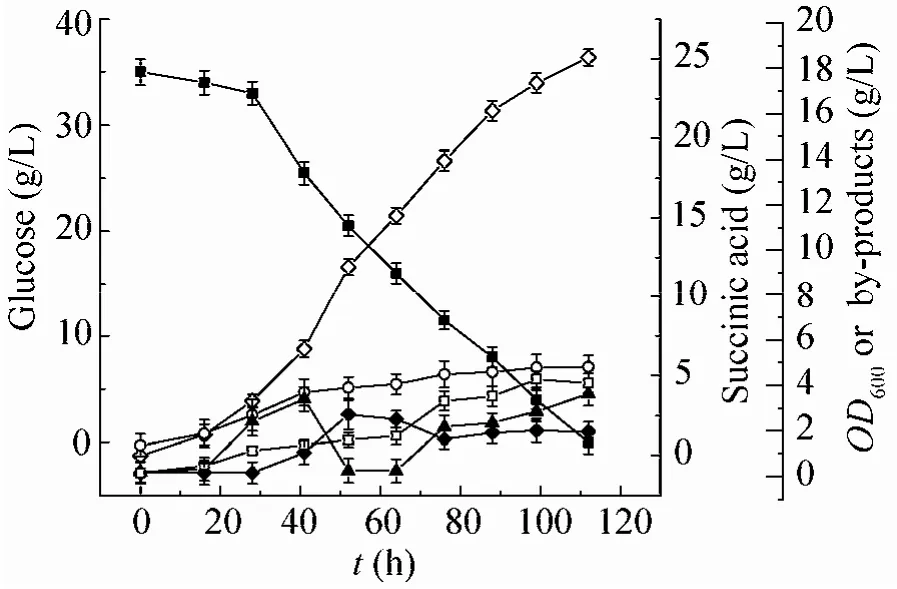
图1 BA016在 3 L发酵罐中厌氧发酵的细胞生长(OD600)、葡萄糖消耗及有机酸的生成Fig. 1 Anaerobic fermentation in 3 L fermentor by BA016.OD600 (open circle), glucose (filled square), succinic acid(open diamond), acetic acid (filled diamond), pyruvic acid(filled triangle), malic acid (open square).
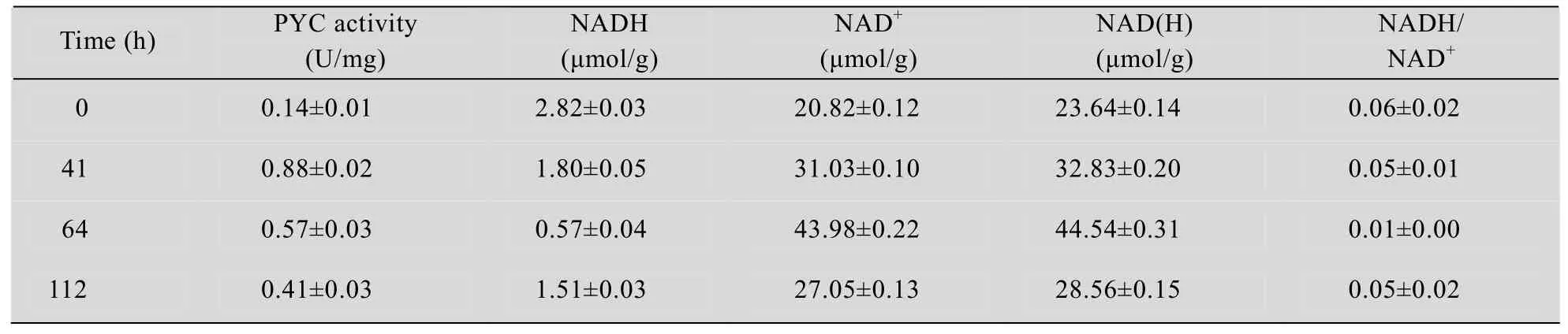
表3 3 L发酵罐中不同时间段PYC的酶活和NADH/NAD+的比例Table 3 Determination of specific enzyme activity of PYC and NAD(H)at different time in 3 L fermentor
[1]Bunch PK, Mat-Jan F, Lee N, et al. The ldhA gene encoding the fermentative lactic acid dehydrogenase of Escherichia coli. Microbiology, 1997, 143: 187–195.
[2]Singh A, Lynch MD, Gill RT, et al. Genes restoring redox balance in fermentation-deficient E. coli NZN111.Metab Eng, 2009, 11: 347–354.
[3]Mat-Jan F, Alam KY, Clark DP, et al. Mutants of Escherichia coli deficient in the fermentative lactate dehydrogenase. J Bacteriol, 1989, 171: 342–348.
[4]Gokarn RR, Eiteman MA, Altman E, et al. Metabolic analysis of Escherichia coli in the presence and absence of the carboxylating enzymes phosphoenolpyruvate carboxylase and pyruvate carboxylase. Appl Environ Microbiol, 2000, 66(5): 1844–1850.
[5]Heuser F, Schroer K, ltüz S, et al. Enhancement of the NAD(P)(H)pool in Escherichia coli for biotransformation. Eng Life Sci 2007, 7(4): 343–353.
[6]San KY, Bennett GN, Berríos-Rivera SJ, et al.Metabolic engineering through cofactor manipulation and its effects on metabolic flux redistribution in Escherichia coli. Metab Eng, 2002, 4: 182–192.
[7]Liang LY, Liu RM, Wang GM, et al. Regulation of NAD(H)pool and NADH/NAD+ratio by overexpression of nicotinic acid phosphoribosyltransferase for succinic acid production in Escherichia coli NZN111. Enzyme Microbiol Technol, 2012, 51: 286–293.
[8]Gosset G. Improvement of Escherichia coli production strains by modif i cation of the phosphoenolpyruvate:sugar phosphotransferase system. Microb Cell Factories,2005, 4: 14.
[9]Clark DP. The fermentation pathways of Escherichia coli. FEMS Microbiol Lett, 1989, 63(3): 223−234.
[10]Gokarn RR, Evans JD, WaLker JR, et al. The physioLogical effects and metabolic alterations caused by the expression of Rhizobium etli pyruvate carboxylase in Escherichia coli. Appl Microbiol Biotechnol, 2001, 56:188–195.
[11]Vemuri GN, Eiteman MA, Altman E, et al. Effects of growth mode and pyruvate carboxylase on succinic acid production by metabolically engineered strains of Escherichia coli. Appl Environ Microbiol, 2002a, 68(4):1715–1727.
[12]Vemuri GN, Eiteman MA, Altman E, et al. Succinate production indual-phase Escherichia coli fermentations depends on the time of transition from aerobic to anaerobic conditions. J Ind Microbiol Biot, 2002b, 28:325–332.
[13]Bradford MM. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principles of protein-dye binding. Anal Biochem,1976, 72: 248–254.
[14]Vinitsky A, Grubmeyer C. A new paradigm for biochemical energy coupling: Salmonella typhimurium nicotinate phosphoribosytransferase. J Biol Chem, 1993,268: 26004–26010.
[15]Payne J, Morris JG. Pyruvate carboxylase in Rhodopseudomonas spheroides. J Gen Microbiol, 1969,59: 97–101.
[16]Li J, Chen KQ, Huang XM, et al. Establishment of the determination of NAD+and NADH in the anaerobic fermentation of organic acids. Food Sci Technol, 2008,33(12): 254−257 (in Chinese).李建, 陈可泉, 黄秀梅, 等. 厌氧发酵有机酸体系中NAD+和 NADH测定方法的建立. 食品科技, 2008,33(12): 254−257.
