南极冰藻Chlamydomonas sp.ICE-L RNA解旋酶CiDDX5的基因特征及其表达分析*
2013-09-20宓妍妍黄晓航刘晨临
宓妍妍,王 燕,黄晓航,刘晨临*
(1.山东轻工业学院 食品与生物工程学院,山东 济南250353;2.国家海洋局 第一海洋研究所,山东 青岛266061)
DEAD-box RNA解旋酶能介导NTP依赖的双链RNA解旋,利用ATP依赖的RNA酶催化RNA二级结构的构象变化,在许多RNA参与的代谢活动中发挥关键作用。DEAD-box RNA解旋酶参与RNA转录、前体mRNA剪切、核糖体发生、核质运输、蛋白质翻译和RNA降解等重要的生命活动,广泛存在于从病毒到人类几乎所有已知的生命形式中[1]。
越来越多的研究表明,DEAD-box RNA解旋酶参与植物细胞中的各种代谢过程,具有广泛的生理意义,并且在对逆境的适应中具有重要作用[2]。2000年,Chamot等[3]在蓝藻中发现了一些冷诱导的基因,RNA解旋酶基因就是其中之一[4-7]。除温度以外,RNA解旋酶基因还受光照、氧气和渗透压等条件变化的影响[8]。2005年,在拟南芥中发现1个DEAD-box RNA解旋酶基因发生突变,这个DEAD-box RNA解旋酶与m RNA输出、植物发育和逆境反应有关系[9]。集胞藻PCC6803只有1个RNA解旋酶基因,即crh R(sir0083),它的表达也受低温诱导,还在一定程度上受组氨酸激酶基因hik33的影响[2,7]。研究表明,crh R突变使groES,groEL1和groEL2分子伴侣基因的转录不受低温诱导上调表达[8]。RNA解旋酶可能直接或间接地影响某些酶或蛋白质的合成,因而在生物的逆境适应过程中发挥作用。
本研究从南极分离到一株严格嗜冷的海冰衣藻Chlamydomonas sp.ICE-L,严酷的极地生境造就了生活在海冰环境中的真核光合微藻(冰藻)独特的生物学特性。在南极海冰的形成、生长和融化的季节性变化中,冰藻也经受了温度、光照等生态条件的剧烈变化。温度和盐度是影响海冰中冰藻种类生存的主要因素,Chlamy domonas sp.ICE-L对低温、高盐等逆境具有很强的适应能力。实验室培养发现,受正常海水3倍的盐度变化,其最适生长温度为4~10℃。目前对DEAD-box RNA解旋酶基因的研究主要集中在人类,以及玉米,水稻,蓝藻和集胞藻等植物方面,对于极地浮游植物的研究还很少。
本研究通过对筛选到的1条南极冰藻DEAD-box RNA解旋酶CiDDX5基因进行生物信息学分析,并研究其在低温、高盐环境中表达量的变化,探寻DEAD-box RNA解旋酶基因的功能,为揭示南极冰藻在低温、高盐环境中的适应机制提供参考和依据。
1 材料与方法
1.1 实验材料
南极冰藻Chlamydomonas sp.ICE-L分离自由中国极地研究所(上海)提供的南极中山站(69°S,77°E)附近的浮冰中。采用Provasoli培养基培养,培养条件为:温度7℃,光照强度1 300~1 600 lx,光照周期12 h/12 h L/D。南极冰藻Chlamydomonas sp.ICE-L的RNA解旋酶基因序列来自于本实验室构建的南极衣藻Chlamydomonas sp.ICE-L EST文库,测序由上海桑尼生物技术有限公司完成。
1.2 DEAD-box RNA解旋酶基因的生物信息学分析
DEAD-box RNA解旋酶CiDDX5基因的开放阅读框(open reading frame,ORF)分析通过NCBI网站(http://www.ncbi.nlm.nih.gov)进行,用DNAStar软件包中的EditSeq软件预测蛋白的理论分子量和等电点。通过 SWISS-MODEL[10-12](http://swissmodel.expasy.org/)网站预测 RNA 解旋酶基因的三级结构。
将测定的CiDDX5基因序列用BLAST软件与GenBank(http://www.ncbi.nlm.nih.gov)中已知的其他RNA解旋酶基因序列进行比对。选取相似性较高的序列[13]用BioEdit软件进行对位排列,人工校正和合并序列。用MEGA 4.0[14]软件分析中的紧邻法(neighbor-joining method,NJ)推测系统关系,构建系统发育树,通过自展检测法(bootstrap test)估计所构建的系统发育树的可靠性,重复次数为1 000次。
运用DNAStar软件将Chlamydomonas sp.ICE-L的CiDDX5基因和其它3个遗传距离较近的绿藻DEAD-box RNA解旋酶基因进行氨基酸序列同源性分析。
1.3 CiDDX5基因定量表达分析
1.3.1 样品处理
低温胁迫处理,在-20℃避光条件下,将7℃培养的对数期南极冰藻Chlamy domonas sp.ICE-L样品分别处理0.5,1,3,6和12 h。以7℃避光处理0 h和12 h的混合样品作为对照。
高盐胁迫处理,将正常海水盐度(33)培养的对数期南极冰藻Chlamydomonas sp.ICE-L样品浓缩后置于3倍海水盐度(99)的培养基中,正常光照条件下分别处理0.5,1,3,6和12 h,以正常盐度海水培养0 h和12 h的混合样品作为对照。
样品离心收集,液氮迅速冷冻后放入-80℃冰箱保存。分别用液氮研磨后立即进行RNA提取。
1.3.2 RNA提取
采用CTAB法[15]提取南极冰藻ICE-L的总RNA:1)将研磨好的藻样加到600μL CTAB裂解液(2 g CTAB,1 g PVP,0.585 g EDTA,8.182 g NaCl,100 m L DEPC处理水)中,60℃水浴10 min;2)加入等体积的氯仿-异戊醇(24:1,V/V),抽提两次,4℃,13 000 rpm离心10 min;3)加入1/4体积10 mol/L的LiCl溶液,-20℃沉淀2~3 h;4)用75%乙醇洗涤沉淀两次,加入30μL DEPC处理水溶解沉淀;5)用琼脂糖凝胶电泳检测所提RNA的质量并用紫外分光光度计检测所提RNA的浓度和OD260/OD280。
提取出的RNA用全式金反转录试剂盒(北京全式金生物科技有限公司)进行反转录,用于实时定量PCR。
1.3.3 实时定量PCR
根据南极冰藻ICE-L的CiDDX5基因序列,利用Primer 5.0软件设计所需特异性引物,并由北京市理化分析测试中心代为合成。选择磷酸甘油醛脱氢酶GAPDH和核糖体蛋白RLP19这两对基因作为本次实验的参比基因,引物序列见表1。
在荧光定量PCR仪(Mx3000,美国Stratagene公司)上进行扩增和数据分析。反应体系为10μL,反应程序:95℃5 min;95℃10 s,63℃20 s,72℃20 s,40个循环;循环后升温至95℃,再降至63℃,以0.5℃递增至95℃。
将实验重复3次。数值取3次重复的平均±SE,通过2-ΔΔCt方法[16]计算低温和高盐条件下不同诱导时间对ICE-L的CiDDX5基因表达的影响。

表1 实时定量PCR引物Table 1 The primers of Real-Time PCR
2 结果与分析
2.1 CiDDX5基因的序列分析
测序分析表明,CiDDX5基因全长为2 077 bp,含有1个完整的开放阅读框,编码513个氨基酸。预测该基因编码的蛋白的理论分子量为74.5 k Da,等电点为9.14。
将预测的CiDDX5氨基酸序列进行Protein Blast分析发现其含有DEAD-box解旋酶的保守结构域和解旋酶超家族C端的保守结构域(图1)。
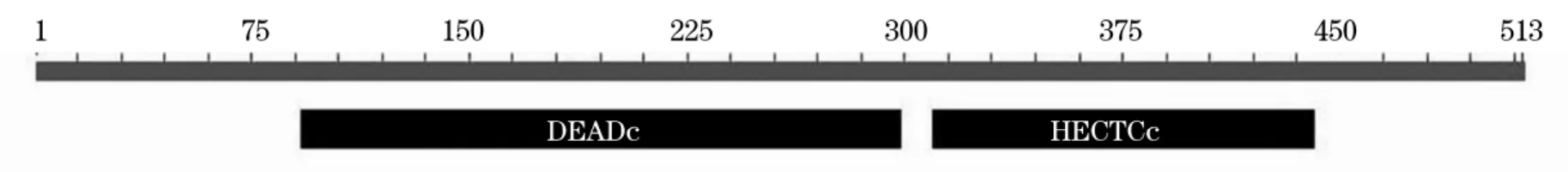
图1 Blast分析CiDDX5基因可能含有的功能域DEADc:DEAD-box解旋酶的保守结构域;HELICc:解旋酶超家族C端的保守结构域Fig.1 Potential functional domains of the CiDDX5 gene by Blast analysis DEADc:DEAD-box helicase domain;HELICc:Helicase superfamily C-terminal domain
2.2 CiDDX5基因的蛋白质三级结构预测分析
将ICE-L的CiDDX5的氨基酸序列提交到SWISS-MODEL预测分析,发现其与数据库中,来自人类的2种解旋酶Ddx5(P68)和Ddx3x的功能蛋白相似度最高。分别达到49%和41%,具有同源模建的意义。表2中a和b两个肽段的功能分别为镁辅因子和ATP结合位点。

表2 预测CiDDX5所含两个肽段的信息Table 2 The information of two predicted peptides of the CiDDX5
2.3 CiDDX5氨基酸序列的系统发育分析
从构建的CiDDX5序列的系统发育树可以看出,绿藻、高等植物和真菌分别构成了3个独立的分支,CiDDX5蛋白序列与莱茵衣藻,团藻和胶球藻等绿藻的相关序列聚类在一起,说明CiDDX5的蛋白结构与其所属物种的分类地位是相关的(图2)。

图2 CiDDX5序列的系统发育树Fig.2 Phylogenetic tree of CiDDX5 sequence constructed by the neighbor joining method
2.4 CiDDX5氨基酸序列同源性分析
运用DNAStar软件将CiDDX5氨基酸序列和3种绿藻的氨基酸序列进行同源性比对,从分析结果可看出ICE-L与莱茵衣藻,团藻和胶球藻的同源性较高,分别为66%,65%和64%。将ICE-L的CiDDX5的氨基酸序列与其它3种单细胞绿藻的RNA解旋酶蛋白序列进行比对,发现其编码DEAD-box RNA解旋酶特有的9个保守的氨基酸基序(图3),说明CiDDX5属于DEAD-box RNA解旋酶。但是,CiDDX5蛋白中的保守氨基酸基序与其它绿藻的DEAD-box RNA解旋酶的保守序列不完全一致。在所有9个保守基序中Motif Q的保守性最差,4种不同绿藻的Motif Q均有不同组成的氨基酸。CiDDX5的保守基序MotifⅧ是YIHRLGRTGR,其它绿藻的均为YVHRIGR。
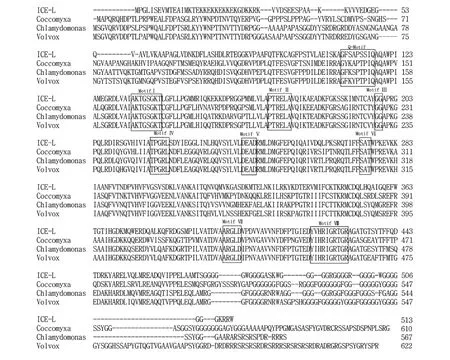
图3 ICE-L CiDDX5和3种不同DEAD-box RNA解旋酶氨基酸序列同源性分析Fig.3 Amino acid sequence comparison ICE-L CiDDX5 and other representative DEAD-box RNA helicases.
2.5 CiDDX5基因定量表达分析
在-20℃冰冻胁迫条件下,南极冰藻经不同时间的处理过程后,CiDDX5基因的表达量发生了明显的变化(图4)。随着处理时间的延续,该基因的表达量逐渐升高,处理3 h时,CiDDX5基因的相对表达量达到最大值,此时表达量为未经低温处理组的1.8倍,之后开始降低;在处理12 h后,CiDDX5基因的相对表达量降为对照组的一半。

图4 -20℃ 低温胁迫下CiDDX5基因的定量表达Fig.4 The quantitative expression of the CiDDX5 gene under low temperature stress of-20℃
检测经过3倍盐度培养处理的冰藻CiDDX5基因表达量的变化,其实时定量PCR的结果见图5。在高盐胁迫下CiDDX5基因的表达量持续升高,在6 h处理后表达量达到最大值,为对照组的2.9倍,在12 h略有降低。在所选取的处理时间段内,处理组基因的表达量都是高于对照组。表明在高盐胁迫下,CiDDX5基因的表达量较低温处理条件下发生了更为明显和持久的变化,提示该基因在冰藻应对高盐胁迫方面可能发挥更重要的作用。
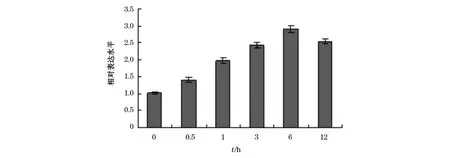
图5 3倍盐度胁迫下ICE-L DEAD-box RNA解旋酶基因的定量表达Fig.5 The quantitative expression of the CiDDX5 gene under salinity stress of 3 times
3 讨 论
越来越多的研究表明,植物中RNA解旋酶的表达受到各种生物和非生物胁迫的影响[2,8,15-16]。在本研究中,克隆得到了南极冰藻Chlamydomnas sp.ICE-L的RNA解旋酶CiDDX5基因,进行了生物信息学分析,并研究了冰藻的该基因在低温和高盐胁迫下表达量的变化。
解旋酶是存在于原核生物和真核生物中的一个庞大蛋白类群,分为5个(超)家族,即SF1~5。DEAD-box RNA解旋酶属于SF2家族成员[2],根据保守的DEAD模体的序列差异分为DEAD,DEAH,DECH和DEVH类群,这些类群的蛋白中均包含特征性的保守基序结构[2]。CiDDX5蛋白中包含了所有9个保守性模体结构,因而可以推定它是DEAD-box RNA解旋酶蛋白。DEAD-box RNA解旋酶蛋白中的保守基序与其生化功能有密切关系,其中Motif V和MotifⅧ共同参与ATP的水解,而模体Ⅵ则与RNA的解链有关。CiDDX5中的模体V为DEAD,而模体Ⅵ为SAT,这与DEAD类群的DEAD-box RNA解旋酶蛋白中的模体序列相一致。
在植物中DEAD-box RNA解旋酶的表达受到生物以及非生物条件的影响。在低温胁迫下Av DH1和STRS1的表达可能在早期调节机制中发挥作用;而在高盐胁迫下上述两基因的表达可能起到长期调节的作用[17-18]。从本研究中可以看出,低温胁迫下CiDDX5基因的表达量在处理3 h时达到了最大值;而在高盐胁迫下,在处理12 h时基因仍具有很高的表达量。以上研究结果与Liu等[19]在高等植物中所做的研究结果相一致。
Gong等[20]发现在冷胁迫的条件下,DEAD-box RNA酶缺陷型的植物中,mRNA在从细胞核输出到细胞质的过程中受损。Zhu等[21]研究结果表明,RNA代谢过程的调节对于植物的低温适应是非常重要的,DEAD-box RNA解旋酶在生理调节方面的重要作用。蓝藻中DEAD-box RNA解旋酶的有关研究表明,crh R编码的RNA解旋酶可能通过改变mRNA的构象,影响其在低温下的稳定性,从而调节某些基因的表达水平;这种对二级构象的调节可能是某些mRNA在低温下指导蛋白合成所需要的。对拟南芥的相关研究表明,低温胁迫下At RH25和At RH9两种RNA解旋酶的表达都会发生上调。本研究初步揭示了CiDDX5在南极冰藻低温、高盐环境胁迫下表达量的变化,分析了其可能的分子作用机制。在今后的研究里,还需要进一步对其是否是ATP依赖型、其受到胁迫的信号调节分子是什么,以及其靶向作用的m RNA的类型等方面进行更为深入的研究,以期加深对南极冰藻RNA解旋酶的了解,进一步揭示南极冰藻在极端环境条件下的适应机制。
:
[1] HALL M C,MATSON S W.Heliease motifs:the engine that powers DNA unwinding[J].Molecular Microbiology,1999,24(5):867-877.
[2] VASHISHT A A,TUTEJA N.Stress responsive DEAD-box helieases:a new pathway to ngineer plant stress tolerance[J].Journal of Photochemistry and Photobiology.B,Biology,2006,84:150-160.
[3] SUZUKI I,KANESAKI M K,MIKAMI K,et al.Cold-regulated genes under control of the cold sensor Hik33 in Synechocystis[J].Molecular Microbiology,2001,40(1):235-244.
[4] CHAMOT D,MAGEE W C,YU E,et al.A cold shock induced cyanobacterial RNA helicase[J].Journal of Bacteriology,1999,181:1728-1732.
[5] CHAMOT D,OWTTRIM G W.Regulation of cold shock-induced RNA helicase gene expression in the cyanobacterium Anabaena sp.strain PCC 7120[J].Journal of Bacteriology,2000,182(2):1251-1256.
[6] VINNEMEIER J,HAGEMANN M.Identification of salt-regulated genes in the genome of the cyanobacterium Synechocystis sp.strain PCC 6803 by subtractive RNA hybridization[J].Atchives of Microbiology,1999,172(6):377-386.
[7] MIKAMI K,KANESAKI Y,SUZUKI I,et al.The histidine kinase Hik33 perceives osmotic stress and cold stress in Synechocystis sp.PCC 6803[J].Molecular Microbiology,2002,46(4):905-915.
[8] OWTTRIM G W.RNA helicases and abioti stress[J].Nucleic Acids Research,2006,34(11):3220-3230.
[9] CHAMOT D,COLVIN K R,KUJAT-CHOY S L,et al.RNA structural rearrangement via unwinding and annealing by the cyanobacterial RNA helicase,CrhR[J].The Journal of Biological Chemistry,2005,280:2036-2044.
[10] ARNOLD K,BORDOLI L,KOPP J,et al.The SWISS MODEL Work space:A web based environment for protein structure homology modeling[J].Bioinformatics,2006,22(2),195-201.
[11] SCHWEDE T,KOPP J,GVEX N,et al.SWISS-MODEL:an automated protein homology-modeling server[J].Nucleic Acids Research,2003,31(13):3381-3385.
[12] GUEX N,PEITSCH M C.SWISS-MODEL and the Swiss-Pdb Viewer:an environment for comparative protein modelling[J].Electrophoresis,1997,18(15):2714-2723.
[13] SONG B,WARD B B.Molecular characterzation of the assimilatory nitrate reductase gene and its express in the marine green alga Dunaliella tertiolecta(Chlorophyceae)[J].Journal of Phycology,2004,40(4),721-731.
[14] KUMAR S,NEI M,DVDLEY J,et al.A biologistcentric software for evolutionary analysis of DNA and protein sequences[J].Briefings in Bioinformatics,2008,9(4):299-306.
[15] EICKEN H,BOCK C,WITTIG R,et al.Magnetic resonance imagine of sea-ice pore fluids:methods and thermal evolution of pore microstructure[J].Cold Regions Science and Technology,2000,31(3):207-225.
[16] LIVAK K J,SCHMITTIGEN T D.Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C[J])method[J].Methods,2001,25(4):402-408.
[17] LIU H H,LIU J,FAN S L,et al.Molecular cloning and characterization of a salinity stress-induced gene encoding DEAD-box helicase from the halophyte Apocynum venetum [J].Journal of Experimental Botany,2008,59(3):633-644.
[18] CHUNG E,CHO C H,YUN B M,et al.Molecular cloning and characterization of the soybean DEAD-box RNA helicase gene induced by low temperature and high salinity stress[J].Gene,2009,443(1-2):91-99.
[19] LI D,LIU H,ZHANG H,et al.OsBIRH1 a DEAD-box RNA helicase with functions in modulating defence responses against pathogen infection and oxidative stress[J].Journal of Experimental Botany,2008,59(8):2133-2146.
[20] GONG Z,DONG C H,LEE H,et al.A DEAD-box RNA helicase is essential for mRNA export and important for development and stress responses in Arabidopsis[J].The Plant Cell,2005,17(1):256-267.
[21] ZHU J,DONG C H,ZHU J K.Interplay between cold-responsive gene regulation,metabolism and RNA processing during plant cold acclimation.Curr.Opin[J].Current Opinion in Plant Biology,2007,10(3):290-295.
