波纹唇鱼(Cheilinus undulatus)不同地理种群遗传多样性的微卫星分析*
2013-09-20侯新远尹绍武贾一何胡亚丽
胡 静,侯新远,尹绍武,祝 斐,贾一何,胡亚丽
(1.南京师范大学生命科学学院,江苏 南京210023;2.中国水产科学研究院南海水产研究所 热带水产研究开发中心,广东 广州510300)
波纹唇鱼(Cheilinus undulatus)俗名苏眉,在分类学上隶属鲈形目(Perciformes)、隆头鱼科(Labridae)、唇鱼属(Cheilinus),是一种暖水性鱼类,同时也是体型最大的珊瑚礁鱼类之一。分布于非洲东岸、红海以及印度洋至太平洋中心,在我国主要分布于南海与东海的南部海域以及台湾海域[1-3]。波纹唇鱼为名贵的海水经济鱼类,其肉质细嫩味鲜,营养价值高,深受亚洲人喜爱[4]。因过度捕捞及珊瑚礁栖息地破坏等因素,导致自然海区的波纹唇鱼数量越来越少,目前已濒临灭绝。1996-2005年间波纹唇鱼曾4度被世界自然保护联盟(IUCN)、世界自然基金会(WWF)等组织和《濒危野生动植物种国际贸易公约》(CITES)列入濒危物种名单。因此,应加大对其野生资源的保护力度,同时加强和加快对其种质资源的研究,以期对其进行更加合理的开发和保护。
目前,国内外有关波纹唇鱼的研究报道较少,多集中在该鱼生物学研究方面[5-8],而国内仅见对其消化系统组织学研究[9]、不同组织同工酶表达差异研究[10]、核型分析[11]、营养成分分析[12]以及鳃丝结构观察[13]等方面报道,未见关于该鱼种质资源方面的研究。近年来,微卫星分子标记技术被广泛应用于物种进化、生物群体内的遗传变异、遗传多样性分析、遗传图谱构建以及种间关系甚至生物个体鉴别等领域[14]。本研究采用微卫星分子标记技术对波纹唇鱼4个野生群体进行遗传多样性分析,旨在为波纹唇鱼这一濒危物种的保护生物学和资源开发提供更多基础资料。
1 材料与方法
1.1 实验材料
野生波纹唇鱼共101尾分别取自海南陵水、马来西亚、西沙以及南沙群岛附近海域,采样信息见表1。分别取尾鳍固定在95%乙醇中带回实验室,更换两次95%乙醇后置于-40℃保存备用。

表1 波纹唇鱼样品的基本信息Table 1 Basic information of Cheilinus undulatus samples
1.2 引物合成
从本实验室自主开发的波纹唇鱼微卫星引物中选取15对,并在5′端按照扩增目的片段大小分别标记了3种荧光染料:HEX、FAM及TAMRA,引物由上海捷瑞生物工程有限公司合成,序列信息和退火温度等见表2。

表2 15对微卫星引物信息Table 2 The information of fifteen microsatellite primers
1.3 基因组DNA的提取
对常规“酚-氯仿”抽提法[15]稍加改动,提取波纹唇鱼尾鳍基因组DNA,用1.0%琼脂糖凝胶电泳检测DNA质量,并利用紫外分光光度计检测其浓度和纯度,调整浓度至50 ng/μL,4℃保存备用。
1.4 PCR扩增与产物检测
用合成的15对荧光引物对波纹唇鱼4个群体进行PCR扩增,反应体系为:10×PCR Buffer 2μL,1.5 μL 25 m M MgCl2,2μL 25 m M d NTP,正向引物F和反向引物R各1μL,taq酶0.2μL,DNA 模板为1μL,双蒸水11.3μL。反应程序为:94℃5 min后,94℃30s,退火(温度见表2)30s,72℃30 s,30个循环;72℃5 min,4℃保温。以8%聚丙烯酰胺变性凝胶电泳结合银染法检测PCR产物,如位点T462的部分PCR扩增产物见图1,将扩增目的条带清晰的PCR产物在ABI 3500XL基因分析仪上进行毛细管电泳,检测产物片段大小。

图1 位点T462的PCR扩增结果Fig.1 PCR amplification profile of the primer T462
1.5 数据统计与分析
用软件Gene Mapper 4.1分析读取毛细血管电泳片段长度后,利用群体遗传多样性分析软件POPGEN32分析计算每个群体及整体水平上的有效等位基因数(Ne)、平均观测杂合度(Ho)、平均期望杂合度(He)及Nei's遗传相似指数[16]和遗传距离,并采用UPGMA法构建群体间的系统树;PIC-CALC 0.6计算多态信息含量(PIC);最后由遗传分化分析软件Areliquin3.1[17]计算出遗传分化参数并对群体的遗传变异进行AMOVA分析。
2 结 果
2.1 SSR扩增结果
本研究所用15对微卫星引物在4个波纹唇鱼群体中均能稳定扩增,等位基因数为3~16个,平均每对引物产生9个等位基因。其中A3位点在马来西亚群体中获得的等位基因数最少(3个),位点B30(2)在南沙群体中获得的等位基因数最多(16个),扩增片段在110~289 bp之间(表3)。

表3 15个微卫星位点扩增结果Table 3 The results of amplification of fifteen microsatellite loci
2.2 群体的遗传多样性分析
15对微卫星引物在4个波纹唇鱼群体中共检测到246个等位基因,其中陵水群体(LP)131个,马来西亚群体(MP)140个,西沙群体(XP)114个,南沙群体(NP)160个。分析4个野生波纹唇鱼群体的等位基因数、期望杂合度(He)、观测杂合度(Ho)、多态信息含量(PIC)和Shannon's信息指数结果(表4)表明,马来西亚群体的各遗传多样性参数最高,而平均等位基因数(Na)、平均有效等位基因数(Ne)、平均多态信息含量(PIC)和平均Shannon's信息指数(I)均以西沙群体最低,分别为7.467,3.876,0.669和1.551,平均观测杂合度(Ho)和平均期望杂合度(He)则以南沙群体最低(0.443和0.712)。总体分析发现,4个波纹唇鱼群体的各遗传变异参数均较高,PIC均>0.5,说明这些海域的野生波纹唇鱼仍具有较高的遗传多样性水平。

表4 4个波纹唇鱼群体的遗传多样性Table 4 Genetic diversity of four C.undulatus groups
2.3 群体间的遗传距离和聚类分析
基于Nei's遗传距离构建4个波纹唇鱼群体的UPGMA系统树(图2),结果显示陵水群体与马来西亚群体的亲缘关系最近,首先聚为一支,然后与西沙群体聚在一起,最后再与亲缘关系最远的南沙群体聚在一起。Nei无偏倚遗传距离范围为0.145 7~0.226 1,遗传相似度范围0.766 7~0.864 4(表5),其中陵水群体与马来西亚群体的遗传距离最小(0.145 7),遗传距离最大的为南沙群体和马来西亚群体(0.226 1)。
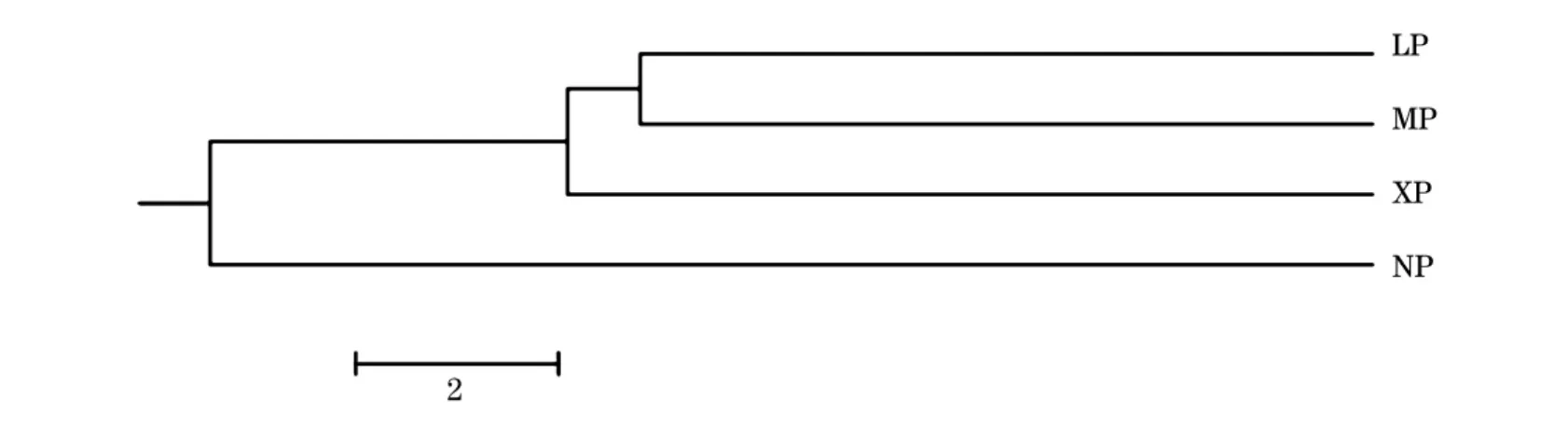
图2 4个波纹唇鱼群体的UPGMA系统树Fig.2 UPGMA tree of four C.undulatus groups

表5 4个波纹唇鱼野生群体的遗传相似指数与遗传距离Table 5 Nei's genetic identity and genetic distance of four C.undulatus groups
2.4 遗传变异和基因流
用Arlequin3.1软件对4个波纹唇鱼群体进行AMOVA遗传变异方差分析(表6),结果表明,波纹唇鱼60.90%的遗传变异来自于个体间,35.75%的遗传变异来自于群体内个体间,仅有4.15%的遗传变异来自于群体间,说明波纹唇鱼的遗传变异主要来自于群体内部,且变异水平较高。由Arlequin3.1软件生成的群体间的遗传分化系数(Fst=0.373 0)[18]及其计算所得基因流数值(Nm=0.840 5)来看,波纹唇鱼群体间存在显著地基因交流障碍并且群体可能由于遗传漂变而发生了分化[19]。

表6 基于微卫星分子标记的波纹唇鱼4个群体AMOVA分析Table 6 The AMOVA analysis of the four C.undulatus populations in microsatellite loci
3 讨 论
3.1 波纹唇鱼的遗传多样性
群体的遗传多样性高低是判断一个生物物种适应能力、生存能力和进化潜力的重要依据。丰富的遗传多样性是一个物种具有较强的环境适应能力和较大的生物进化潜力以及丰富的育种和遗传改良能力的重要特征,并对种质鉴定和亲本选择具有重要的意义[20-21]。有效等位基因数是基因纯合度的倒数,能反映基因座等位基因间的相互作用[22]。平均杂合度、PIC、Shannon's信息指数也是目前衡量遗传多样性的重要指标。本研究中,陵水群体、马来西亚群体、西沙群体和南沙群体的平均有效等位基因分别为4.114,4.887,3.876,4.243;平均观测杂合度(平均期望杂合度)分别为0.520(0.727),0.531(0.736),0.457(0.724),0.443(0.712);平均多态信息含量分别为0.687,0.696,0.669,0.673,均>0.5;平均Shannon's信息指数分别为1.620,1.705,1.551,1.675。从上述4个指标可以看出,4个不同地理海域的野生波纹唇鱼群体均具有丰富的遗传多态性,蕴藏着较大的进化潜力,且以马来西亚群体最高。因此,通过对野生波纹唇鱼遗传多样性的分析来评价自然灾害、环境污染、人为因素等对其种质资源所带来的遗传学影响,对寻找科学合理的开发保护途径具有重要的意义。
3.2 波纹唇鱼群体间的亲缘关系和遗传分化
遗传距离是研究物种遗传多态性的基础,常用来描述品种(系)的遗传结构、品种间的差异与遗传关系[23]。本研究通过4个波纹唇鱼群体的遗传距离研究发现,海南陵水群体与马来西亚群体的遗传距离最小(0.145 7),先聚为一支,然后与西沙群体聚在一起,南沙群体与其它3个群体的遗传距离较远,独立一支。这种导致遗传距离远近与地理距离不一致现象发生的原因可能是:1)与南沙海域样品采集地点偏远有关;2)与本研究所采用的分子标记技术有关。因此,要阐明遗传距离与地理距离的确切关系,还需结合其它分子标记对更多地点的样本做进一步分析。
Fst作为分化系数可用于分析群体遗传结构,Shaklee[24]指出若固定指数Fst在0~0.05之间,说明群体间的遗传差异很小;若Fst在0.05~0.15之间,说明群体间存在中等程度的遗传差异;若Fst在0.15~0.25之间,说明群体间的遗传差异比较大;若Fst>0.25,则说明群体间的遗传差异很大。通过对波纹唇鱼群体的遗传结构分析(表6)表明,本研究中的4个波纹唇鱼群体并不是一个无分化的大群体族群,而是各群体遗传差异很大(Fst=0.373 0)且遗传物质非均匀分布的族群。其中,60.90%的遗传变异来自于个体间,35.75%的遗传变异来自于群体内个体间,仅有4.15%的遗传变异来自于群体间,也充分验证了这一点。
根据群体遗传学理论,基因流越顺畅,遗传分化的程度将越低[25]。Slatkin[26]和 Hamrick等[27]认为,若每代迁入个体数Nm>1,基因流就足以抵制遗传漂变的作用,也同时防止了群体分化的发生;若Nm<1,基因流不足以抵制群体内因漂变引起的遗传分化,即有限的基因流可以促使群体发生遗传分化。本研究得出波纹唇鱼4个野生群体Nm=0.840 5,说明遗传漂变对波纹唇鱼的遗传结构有重要影响。遗传漂变会造成一些等位基因的丢失和另一些等位基因的固定,从而引起同一物种对不同环境的适应,再通过生存空间的扩展,形成物种的微进化[28]。因此,遗传漂变对本研究中波纹唇鱼群体遗传分化的影响不容忽视。当然,波纹唇鱼这种分化格局的出现还可能与其种群数量急剧减少、活动范围相对狭窄、气候环境剧变、地质结构变化等多种因素有关,有待进一步考证。
:
[1] SHEN S J.Fishes of Taiwan[M].Taipei:Department of animal science at National Taiwan University,1993.沈世杰.台湾鱼类志[M].台北:台湾大学动物学系印行,1993.
[2] CHENG Q T,ZHENG B S.Search Chinese fish system[M].Beijing:Science Press,1987.成庆泰,郑葆珊.中国鱼类系统检索[M].北京:科学出版社,1987.
[3] DONALDSON T J,SADOVY Y.Threatened fishes of the world:Cheilinus undulatus Rüppell,1835(Labfidae)[J].Environmental Biology of Fishes,2001,62(4):428.
[4] WANG Y B,CHEN G H,LUO J,et al.Morphology and anatomy on digestive system of Cheilinus undulatus Rüppell with scanning electron microscopy of intestinal tract[J].Marine Science Bulletin,2010,29(2):199-205.王永波,陈国华,骆剑,等.波纹唇鱼消化系统的形态解剖与肠道上皮的扫描电镜观察[J].海洋通报,2010,29(2):199-205.
[5] CHOAT J H,DAVIS C R,ACKERMAN J L,et al.Age structure and growth in a large teleost,Cheilinus undulatus,with a review of size distribution in labrid fishes[J].Marine Ecology Progress Series,2006,318:237-246.
[6] OLIVIER C,LAURENT W.Site fidelity and activity patterns of a humphead wrasse,Cheilinus undulatus(Labridae),as determined by acoustic telemetry[J].Environmental Biology of Fishes,2006,80(4):187-196.
[7] RANDALL J E,HEAD S M,SANDERS A P L,et al.Food habits of the giant humphead wrasse,Cheilinus undulatus(Labridae)[J].Environmental Biology of Fishes,1978,3:235-238.
[8] SLUKA R D.Humphead wrasse(Cheilinus undulatus)abundance and size structure among coral reefhabitats in maldies[J].Atoll Ites.Bull,2005,538:189-198.
[9] CHEN G H,WANG Y B,WANG J,et al.Histology of the digestive system in cheilinus undulatus rüppell[J].Acta Hydrobiologica Sinica,2010,34(4):685-693.陈国华,王永波,王珺,等.波纹唇鱼消化系统的组织学[J].水生生物学报,2010,34(4):685-693.
[10] QU Y J,QI X D,LI J E.Five kinds of isozyme in different tissues of Cheilinus undulatus[J].South China Fisheries Science,2009,5(2):51-55.区又君,齐旭东,李加儿.波纹唇鱼不同组织5种同工酶表达的差异[J].南方水产,2009,5(2):51-55.
[11] HUO R,ZHANG B,CHEN G H,et al.The karyotype of Cheilinus undulatus[J].Marine Sciences,2009,33(4):94-97.霍蕊,张本,陈国华,等.波纹唇鱼染色体核型分析[J].海洋科学,2009,33(4):94-97.
[12] OU Y J,LI J E.Analysis and evaluation of nutrition composition of double-headed parrotfish Cheilinus undulatus[J].Journal of Tropical Oceanography,2010,29(3):97-102.区又君,李加儿.野生波纹唇鱼Cheilinus undulatus营养成分分析与评价[J].热带海洋学报,2010,29(3):97-102.
[13] LIAO G Y,OU Y J,LI J E.Light,scanning and transmission electron microscopical observationof gill filaments of Cheilinus undulatus[J].Chinese Journal of Zoology,2011,46(1):7-15.廖光勇,区又君,李加儿.波纹唇鱼鳃丝的光镜、扫描和透射电镜观察[J].动物学杂志,2011,46(1):7-15.
[14] YAN H C,SI Z S,LI G L.Microsatellites and its application to the genetic diversity in fish[J].Biotechnology,2007,17(3):83-85.闫华超,司振书,李桂兰.微卫星DNA标记及其在鱼类遗传多样性研究中的应用[J].生物技术,2007,17(3):83-85.
[15] SAMBROOK J,FRITSCH E F,MANIATIS T.Molecular cloning:a laboratory manual[M].2th ed.Beijing:Science Press,1995.萨姆布鲁克J,弗里奇E F,曼尼阿蒂斯T.分子克隆实验指南第2版[M].金冬雁,黎孟枫,译.北京:科学出版社,1995.
[16] NEI M.Estimation of average heterozygosity and genetic distance from a small number of individuals[J].Genetics,1978,89:583-590.
[17] EXCOFFIER L,SMOUSE P E,QUATTRO J M.Analysis of molecular variance inferred from metric distances among DNA haplotypes:Application to human mitochondrial DNA restriction data[J].Genetics,1992,131(2):479-491.
[18] WEIR B S,COCKERHAM C C.Estimating F-statistics for the analysis of population structure[J].Evolution,1984,38(6):1358-1370.
[19] MILLAR C L,LIBBY W J.Strategies for conserving clinal,ecotypic,and disjunct population diversity in widespread species[M]∥Fald D A,Holsinger K E.Genetics and Conservation of Rare Plants.New York:Oxford University Press,1991:149-170.
[20] CHEN L Z.Chinese biodiversity:status and protection countermeasures[M].Beijing:Science Press,1993,99-113.陈灵芝.中国的生物多样性:现状及其保护对策[M].北京:科学出版社,1993,99-113.
[21] ZHANG Z M,HAN Z Q,MA A J,et al.Genetic diversities in the tongue sole(Cynoglossus semilaevis)as revealed by isozyme analysis[J].Marine Fisheries Research,2006,27(2):10-16.庄志猛,韩志强,马爱军,等.黄、渤海半滑舌鳎种群遗传结构的同工酶分析[J].海洋水产研究,2006,27(2):10-16.
[22] YAN B,LI S F,CAI W.Microsatellite analysis of Oreochromis niloticus,Sarotherodon melanotheron and their reciprocal hybrids[J].Journal of Fisheries of China,2007,31(3):412-415.颜标,李思发,蔡完其.尼罗罗非鱼与萨罗罗非鱼及其正反杂交后代的微卫星分析[J].水产学报,2007,31(3):412-415.
[23] CRAWFORD A M,LITTLEPOHN R P.The use of DNA marker in deciding conservation priorities in sheep and other livestock[J].Animal Genetic Resources Information,1998,23(1):21-26.
[24] SHAKLEE J B,TAMARU C S,WAPLES R S.Speciation and evolution of marine fishes studied by the electrophoresis analysis of proteins[J].Pacific Sci,1982,36(2):141-157.
[25] GUO B Y,ZHOU C,LV Z M,et al.Genetic diversity of different geographical populations in Octopus variabilis revealed by ISSR analysis[J].Oceanologia &Limnologia Sinica,2011,42(6):868-873.郭宝英,周超,吕振明,等.长蛸(Octopus variabilis)不同地理群体遗传多样性的ISSR 分析[J].海洋与湖沼,2011,42(6):868-873.
[26] SLATKIN M.Gene flow in natural populations[J].Annual Review of Ecology and Systematics,1985,16:393-430.
[27] HAMRICK J L,GODT M J W,SHERMAN-BROYLES L S.Gene flow among plant populations:evidence from genetic markers[M]∥HOCH P C,STEPHNON A G.Experimental and Molecular Approaches to Plant Biosystematics.Saint Louis:Missouri Botanical Garden,1995:215-232.
[28] LIU Z L,ZHAO G F.Population genetics and its implications for conservation ofrare and endangered plants[J].Chinese Biodiversity,1999,7(4):340-346.刘占林,赵桂仿.居群遗传学原理及其在珍稀濒危植物保护中的应用[J].生物多样性,1999,7(4):340-346.
