斑玉蕈菌株的IGS2序列和RAPD分析
2013-05-15冯志勇陈明杰张津京
潘 越,陈 辉,冯志勇,*,陈明杰,汪 虹,张津京
(1.南京农业大学生命科学学院,南京210095;2.国家食用菌工程技术研究中心,农业部南方食用菌资源利用重点开放实验室,上海市农业遗传育种重点开发实验室,上海市农业科学院食用菌研究所,上海201403)
斑玉蕈(Hypsizygus marmoreus)又名真姬菇、蟹味菇、鸿喜菇等,隶属白蘑科玉蕈属,是重要的工厂化食用菌之一[1]。斑玉蕈味道鲜美,是一种珍稀食用菌品种,在国内外都受到食用菌生产行业的重视,其在日本、韩国和国内上海等地已经实现了工厂化生产模式,且效益良好。近年来,随着食用菌市场竞争的日趋激烈,假劣菌种充斥着市场,极大地影响了整个产业的健康发展。为保护我国食用菌物种的遗传资源,迫切需要简便、可靠的方法研究斑玉蕈菌株种质资源的遗传多样性。
在对真菌rDNA的研究中,通常认为IGS2序列是高变区,该序列变异性大,进化较快,可用于真菌种间和种内遗传多样性研究。Saito等[2]对16个香菇商业栽培菌株的IGS2序列进行过研究,表明香菇种内菌株间IGS2序列有着显著的差异。在国内,黄晨阳等[3]对8个刺芹侧耳菌株的IGS2序列进行研究,研究显示刺芹侧耳不同菌株的IGS2序列有丰富的多样性。
RAPD技术是1990年由Williams首先报道的一种分子标记技术[4],如今已经成为生物种内变种和种群差异性研究的通用手段和标记。它具有操作简单、多态性高、DNA用量少、信息量大等优点。这一方法已经广泛应用于草菇[5]、松口蘑[6]、双孢蘑菇[7]、木耳[8]、杏鲍菇[9]等主要食用菌的菌种资源研究中。在斑玉蕈中,许占伍等[10]使用RAPD技术成功分析了斑玉蕈单核菌株的遗传多态性,为斑玉蕈育种材料的选择提供了一定的分子水平依据。
本文拟利用IGS2和RAPD分子标记技术,以20个不同的斑玉蕈菌株为实验材料,探讨各菌株间的亲缘关系,旨在为斑玉蕈菌株的快速准确鉴别提供技术依据,为斑玉蕈种质资源的知识产权保护奠定基础。
1 材料与方法
1.1 材料与仪器
本实验中斑玉蕈菌株共20份 具体来源见表1;感受态细胞 Tiangen公司;PCR试剂、pGEM-T载体TaKaRa公司;Axy Prep DNA试剂盒 爱思进生物技术有限公司;IGS2引物 由上海捷瑞生物工程有限公司合成;RAPD随机引物 由上海生工生物工程有限公司合成。

表1 供试菌株Table 1 Tested strains of Hypsizygus marmoreus
5810R型高速冷冻离心机 Eppendorf;PCR扩增仪,Power PAC 3000水平电泳仪,VDS凝胶成像系统等。
1.2 实验方法
1.2.1 DNA提取 将低温保藏的斑玉蕈菌种活化后接种于PDA平板上,25℃暗室培养15~18d,收集菌丝。采用CTAB法[11]提取基因组DNA,1.5%琼脂糖凝胶电泳检测DNA质量,使用Nano Drop测定DNA浓度,将基因组DNA样品浓度稀释到50ng/μL,-20℃保存备用。
1.2.2 IGS2分析
1.2.2.1 IGS2片段的扩增 使用真菌通用引物5SRNAR/InvSR1R(表2)对供试菌株进行扩增。25μL反应体系:10×PCR buffer 2.5μL(含Mg2+20mmol/L),dNTPs(2.5mmol/L)2μL,引物(10umol/L)0.5μL,模板DNA(50ng/L)0.5μL,Taq DNA聚合酶(5U/μL)0.25μL,ddH2O补平至25μL。PCR反应条件:94℃3min;94℃ 30s,55℃ 30s,72℃ 1.5min,30个循环;72℃7min。4℃保存。
1.2.2.2 扩增片段的回收与克隆 PCR扩增产物经1.5%琼脂糖凝胶电泳后,利用Axy Prep DNA试剂盒进行回收,按照PGEM-T Vector试剂盒说明书进行连接,转化于大肠杆菌E.coli DH5α感受态细胞中,将菌液涂布于含氨苄青霉素的LB平板(含X-gal和IPTG),于37℃倒置培养12~16h,挑取阳性转化子送往上海生工生物工程有限公司测序。
1.2.2.3 数据分析 对所得序列中计算机误读的个别碱基进行人工校对,校对后的DNA序列用NCBI BLAST进行序列搜索,确定为IGS2序列后,使用DNAStar软件进行序列相似度比对。
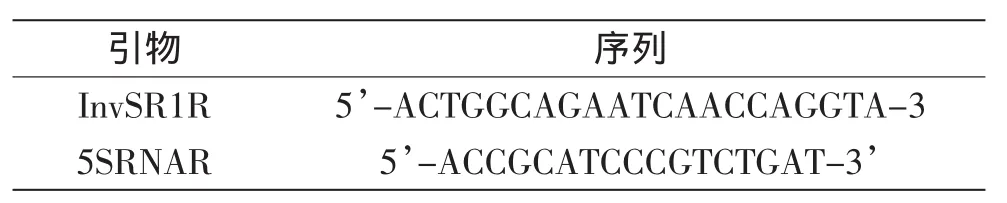
表2 IGS2引物Table 2 Primer of IGS2
1.2.3 RAPD分析
1.2.3.1 引物筛选 从随机引物中筛选出对所有20个菌株扩增效果较好、多态性高、稳定性较强的引物对样品进行扩增。
1.2.3.2 反应体系及参数 25μL反应体系:10×PCR buffer 2.5μL(含Mg2+20mmol/L),dNTPs(2.5mmol/L)2μL,引物(10umol/L)0.5μL,模板DNA(50ng/L)2μL,Taq DNA聚合酶(5U/μL)0.25μL,ddH2O补平至25μL。PCR反应程序为:94℃ 3min;94℃ 1min,38℃ 1min,72℃ 2min,30个循环;72℃ 7min。4℃保存。PCR产物电泳检测:PCR产物用1.5%琼脂糖凝胶电泳检测,EB染色后,在VDS摄影系统下观察结果并拍照。
1.2.3.3 数据处理 根据片段的迁移率及有无(有带记为1,无带记为0),统计得到二元数据,转换为数字矩阵。使用NTSYSpc分析软件对数据进行处理,采用UPGMA法构建遗传相关聚类图谱。
2 结果与分析
2.1 20株斑玉蕈形态学特征
选取六株具有代表性的菌株,其子实体形态图见图1。20株斑玉蕈的形态学特征见表3。
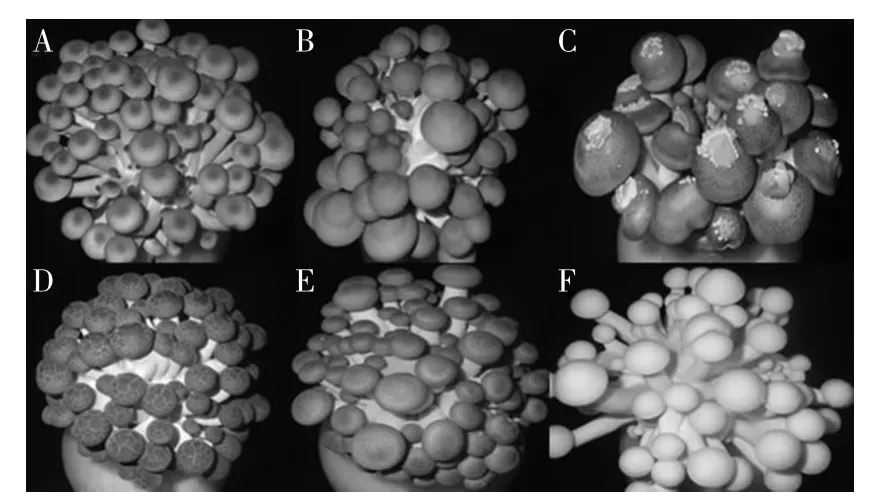
图1 部分菌株的子实体形态图Fig.1 The morphology picture of part strains
2.2 IGS2分析
2.2.1 IGS2-PCR 对20个供试菌株的扩增结果显示,所有菌株均可扩增出目的片段,不同菌株间IGS2序列大小没有显著差异,均在1200bp左右(图2)。
2.2.2 IGS2-测序 测序结果表明,不同斑玉蕈菌株的IGS2序列大小存在差异,在1150~1203bp之间,将所有菌株的序列提交到GenBank,登录号见表4。使用DNAStar对序列进行比对和分析并计算序列相似性系数。20个斑玉蕈菌株的IGS2片段总的变异位点数为139个,占总碱基数的11.6%。序列间相似性系数约在95%以上(表5),除了19号菌株与20号菌株序列完全一致以外,其他菌株序列均存在差异。
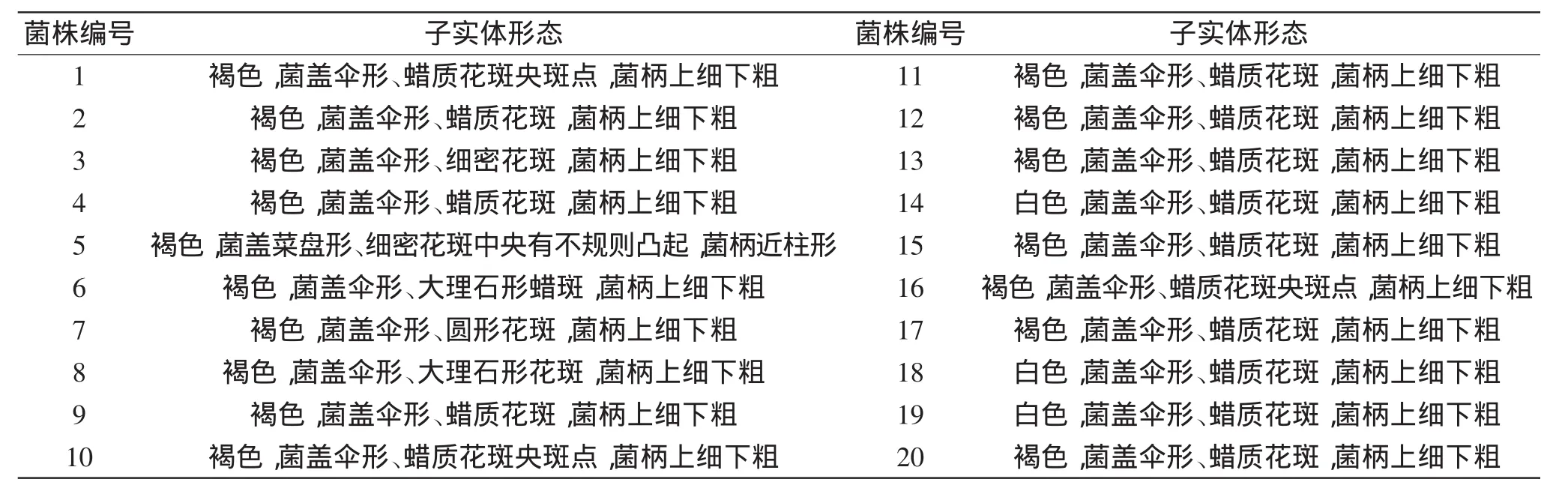
表3 20株斑玉蕈形态学特征Table 3 Morphology character of 20 Hypsizygus marmoreus

图2 20个斑玉蕈菌株的IGS2扩增电泳图Fig.2 IGS2-PCR profile of 20 tested strains
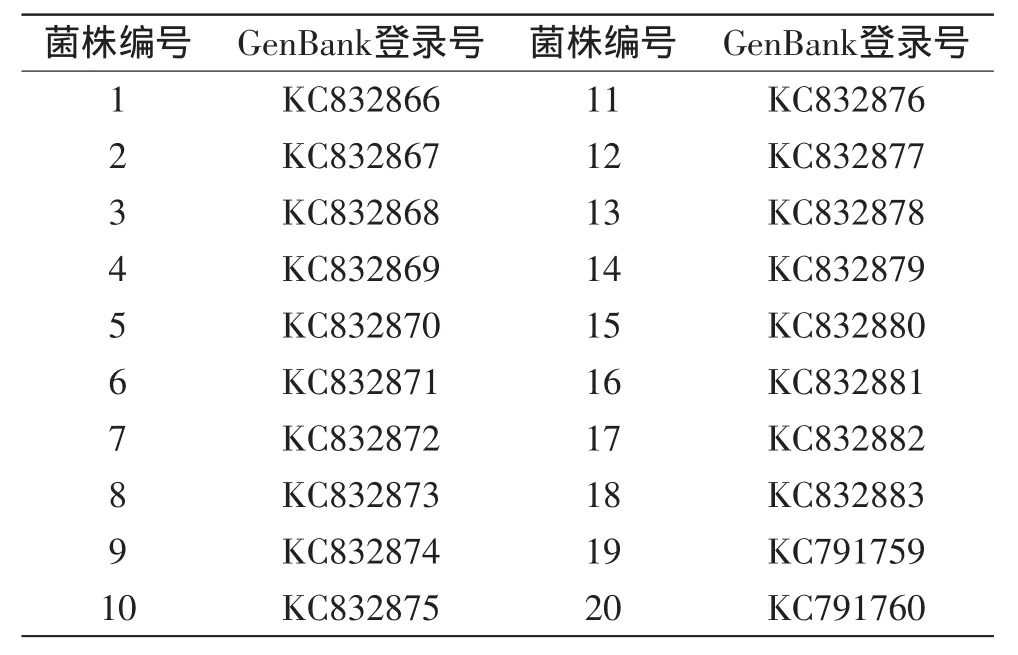
表4 斑玉蕈菌株IGS2序列的登录号Table 4 GenBank accession numbers of IGS2 sequences of 20 strains

表5 20个斑玉蕈菌株IGS2序列相似度Table 5 Table of similarity between sequence
2.3 RAPD分析
2.3.1 RAPD扩增图谱分析 从供试的100个随机引物中筛选出条带清晰稳定、多态性高、重复性好的11个引物(表6)。11个引物共扩增出142条清晰可用的DNA条带,其中特异性条带134条,多态性94.4%。不同引物扩增出的DNA片段数不尽相同,在8~21条之间,其分子量绝大多数在0.5~4ku之间,不同菌株的DNA指纹图谱均包含丰富的多态信息(图3)。

表6 RAPD-PCR扩增反应的随机引物Table 6 Primers of RAPD-PCR

图3 部分引物对20个斑玉蕈菌株的扩增结果Fig.3 DNA fragments amplified by part random primers
2.3.2 RAPD聚类分析 根据所得图谱资料,综合11个引物对20个菌株的DNA扩增结果,构建遗传距离矩阵。应用NTSYSpc分析软件计算菌株间的相似性系数,用UPGMA法进行聚类分析,生成供试菌株的亲缘关系树状图(图4)。

图4 RAPD聚类分析图Fig.4 Dendrogram of 20 strains based on RAPD cluster analysis
根据图3显示的结果,20个斑玉蕈菌株的相似系数主要在0.57~0.92之间。在大约0.68的相似性水平上,所有的菌株可以分为5个组,第一组以0.77水平值又分成A、B两个亚组。A组共6个菌株,分别为1、2、4、7、10、11号菌株,B组共5个菌株,分别为3、6、8、15、16号菌株。13和17号菌株归为第二组,9和1号菌株归为第三组,14、18、19、20号菌株归为第四组,其中白色突变菌株(19号)与原野生型菌株(20号)聚为一类,两个白色野生型菌株(14号和18号)聚为一类。5号菌株与其他所有菌株分开,单独列为一组。
3 结论与讨论
本研究首先对20个供试菌株进行了IGS2序列测序分析,虽然有研究报道IGS2序列为低保守基因区域,在种内不同菌株间存在明显差异。但本研究对斑玉蕈不同菌株的测序结果显示,斑玉蕈的不同菌株间IGS2序列差异不明显,仅存在个别碱基的差异,大部分菌株的IGS2序列相似度在95%以上,在IGS2片段的琼脂糖凝胶电泳图上也不能将不同菌株区分开来,这与香菇[2]、刺芹侧耳[3]、双色蜡蘑[12]等食用菌IGS2序列的研究结果不一致。在其他部分真菌的IGS研究中,也观察到与本研究类似的现象,即一个菌株的IGS2序列仅为单一条带,不存在大小有显著差异的多态性条带。如梁宏等[13]对腥黑粉菌11个菌株的IGS2区域进行了研究,研究结果表明各菌株的IGS2区域均为单一条带,IGS2片段的保守性较强,没有显著的差异。在食用菌中,李黎等[14]在研究木耳不同菌株的IGS2序列时发现,木耳不同菌株的IGS2序列相似度很高,仅存在少数碱基的差异。因此,不同真菌的IGS2序列存在着明显的差异,还有待进一步的深入研究。
RAPD是一种能快速、简便且准确地检测基因组DNA多态性的分子标记,本研究通过优化RAPDPCR反应体系,使用筛选出的11个引物扩增出了142条DNA条带,其中多态性条带134条,多态性比例很高,说明RAPD技术可以用于斑玉蕈不同菌株亲缘关系的鉴定。根据RAPD对供试菌株的扩增结果,不同菌株间的扩增图谱有明显的差异,因此所选引物可用于斑玉蕈不同菌株的快速鉴定,但由于RAPD技术仍存在不足之处,还需将其进一步转化为SCAR标记才可以更加准确地对菌株进行鉴定[15]。
通过对遗传距离及RAPD图谱的分析,两个白色野生型菌株单独聚为一类,表明RAPD技术可以有效地将不同颜色的斑玉蕈菌株区分。5号菌株与其他所有菌株遗传距离较远,单独聚为一类,这与董岩等的研究结果一致[16]。推测其原因可能是5号菌株从国外引进,与其他来源于国内的斑玉蕈菌株的遗传背景存在差异,且5号菌株的子实体形态与其他菌株也存在较大的差异(表2)。白色突变菌株(19号)与原褐色野生型菌株(20号)在RAPD图谱中聚为一类,且相似系数为100%,在IGS2测序分析中,两个菌株的序列相似度同样为100%,这一结果表明19号菌株确实由20号菌株突变而来。
本研究表明,IGS2分析与RPAD技术相结合的方法准确可靠,其中RAPD技术较IGS2分析具有更加丰富的多态性,能够在DNA水平上鉴别不同菌株是否存在差异,为科学、准确地判断特定菌株的特异性奠定了基础。
[1]孙培龙,魏红福,杨开,等.真姬菇研究进展[J].食品科技,2005(9):54-56.
[2]Saito T,Tanaka N,Shinozawa T.Characterization of subrepeat regions with rDNA intergenic spacers of the edible basidiomycete Lentinula edodes[J].Bioscience Biotechnology and Biochemistry,2002,66(10):2125-2133.
[3]黄晨阳,张金霞,郑素月,等.刺芹侧耳rDNA的IGS2多样性分析[J].农业生物技术学报,2005,13(5):592-595.
[4]Williams J G K.DNA polymorphisms amplified by arbitrary primers are genetic markers[J].Nucleic Acids Research,1990,18(22):6531-6535.
[5]徐学锋,姬宏超,李巧玲,等.草菇有性世代间rDNA序列变异分析及RAPD分析[J].食品工业科技,2012,33(10):198-202.
[6]马银鹏,陈强,赵梦然,等.中国三省松口蘑遗传多样性分析[J].食用菌学报,2012,19(1):17-21.
[7]Moore A J,Challen M P,Warner P J,et al.RAPD discrimination of Agaricus bisporus mushroom cultivars,Appl Microbiol Biotechnol[J].2001,55:742-749.
[8]Yan P S,Luo X C,Zhou Q.RAPD molecular differentiation of the cultivated strains of the jelly mushrooms,Auricularia auricula and A polytricha[J].World Journal of Microbiology&Biotechnology,2004,20:795-799.
[9]许峰,刘宇,尹永刚,等.北京地区杏鲍菇菌株遗传多样性的RAPD分析[J].中国食用菌,2012,31(6):35-37.
[10]许占伍,陈辉,冯志勇,等.真姬菇SIEF3136菌株单核体RAPD多态性分析[J].安徽农业科学,2010,38(6):2820-2823.
[11]杨华,李联泰.食用菌DNA提取方法研究[J].中国食用菌,2003,22(1):19-211.
[12]Francis M,Marc-Andre S,Francois L T.The nuclear rDNA intergenic spacer of the ectomycorrhizal basidiomycete Laccaria bicdor:structural analysis and allelic polymorphism[J].Microbiology,1999,145:1605-1611.
[13]梁宏,彭友良,张国珍,等.腥黑粉菌属3种检疫性真菌rDNA-IGS区的扩增及其序列分析[J].植物病理学报,2006,36(5):407-412.
[14]李黎.中国木耳栽培种质资源的遗传多样性研究[D].武汉:华中农业大学,2011.
[15]Lahgue F,This P,Bouquet A.Identification of a codominant scar marker linked to the seedlessness character in grapevine.Theoretical and Applied Genetics[J].1998,97(5-6):950-959.
[16]董岩,陈辉,冯志勇,等.真姬菇栽培菌株的ITS和SSR分析[J].上海农业学报,2009,25(3):59-64.
