苹果过敏原Mald4蛋白抗原表位预测及交叉反应分析①
2012-07-30夏宏林邹泽红陶爱林广州医学院第二附属医院呼吸疾病国家重点实验室变态反应研究室过敏反应与临床免疫重点实验室广州510260
夏宏林 何 颖 邹泽红 陶爱林 (广州医学院第二附属医院/呼吸疾病国家重点实验室变态反应研究室/过敏反应与临床免疫重点实验室,广州510260)
苹果属于蔷薇科植物,因其丰富的营养物质以及愉悦的风味而被人们推荐为健康食品,虽然是一种健康食品,但在部分人群中会引起过敏反应。据统计,欧洲大约有200万人口对蔷薇科水果过敏[1,2]。苹果过敏主要临床症状表现为口腔过敏综合征、皮肤瘙痒、哮喘、荨麻疹及过敏性鼻炎等,严重时可导致过敏性休克,甚至危及生命安全[2,3]。
苹果过敏原主要有4种,Mald4蛋白是苹果主要过敏原之一,在烘烤或经胃肠道消化等条件下,理化性质不稳定。该过敏原蛋白与桦树花粉过敏原Bet v 2有很高的同源性,存在交叉反应,10% ~20%苹果过敏患者血清的前纤维蛋白能与桦树过敏患者血清中的IgE结合,在免疫交叉反应中前纤维蛋白扮演非常重要的角色[4]。引起免疫交叉反应的物质基础是抗原表位(Antigenic epitope),分为线性表位和构象表位,其中T细胞仅能识别抗原递呈细胞加工处理的线性表位,B细胞则可识别线性或构象表位[5]。
目前对蛋白抗原表位预测主要集中在B细胞单参数预测,有一定的局限性,为此我们引入多参数综合预测分析B/T细胞抗原表位,以便提高预测的准确性[6,7]。通过对苹果 Mald4蛋白 B/T细胞抗原表位预测及同源序列空间构象对比,寻找含有相同/相似优势抗原表位区域位点,预测目的蛋白与其同源蛋白之间的交叉反应性,为我们以后利用基因突变技术改造苹果Mald4蛋白及其同源蛋白的过敏原性提供了理论依据。
1 材料与方法
1.1 材料 苹果Mald4蛋白(GenBank登录号Q9XF41.1),其氨基酸序列为:MSWQAYVDDHLMCEIEGNHLSAAAIIGHNGSVWAQSATFPQLKPEEVTGIMNDFNEPGSLAPTGLYLGGTKYMVIQGEPGVVIRGKKGPGGVTVKKSTMALLIGIYDEPMTPGQCNMVVERLGDYLIEQGL。
1.2 方法
1.2.1 Mald4蛋白二级结构预测及特性分析 利用在线分析软件HNN对苹果Mald4蛋白结构进行预测。运用SignaIP 3.0和TMHMM在线软件分析该蛋白氨基酸序列信号肽和跨膜区域(表1)。
1.2.2 Mald4蛋白B/T细胞抗原表位预测 运用Bcepred在线软件预测B细胞抗原表位,选取亲水性好、抗原性强、可塑性好、分子可及性高的片段作为候选表位。采用DNAStar Protean软件对目的蛋白抗原表位进行预测,根据Kyte-Doolittle方案、Emini方案、Karplus-Schulz方案和 Jameson-Wolf方案,分别预测其蛋白的亲水性、可及性、可塑性和抗原指数。在预测4种单参数中,取至少三个以上参数重叠区作为B细胞抗原表位候选区域。对上述二种软件预测结果综合分析,并兼顾二级结构各参数,避免α-螺旋区,确定Mald4蛋白B细胞抗原表位区域;综合运用网络服务器NetMHCⅡ、NetMHCⅡpan、Propred、Syfpeithi预测能与MHCⅡ类分子结合的外源性抗原肽,选取9个滑动抗原肽,预测T细胞抗原表位亲和力[8-10]。

表1 生物信息学在线软件Tab.1 Bioinformatics online software
以上述预测Mald4蛋白B细胞抗原表位区域参数为基础,结合T细胞抗原表位预测结果,获得B/T细胞共同抗原表位优势区域[11]。
1.2.3 Mald4蛋白抗原表位免疫交叉反应预测利用NCBI数据库的Blastp工具,搜索与苹果Mald4蛋白同源性高的前纤维蛋白序列,运用Clustal X1.83软件对其同源蛋白氨基酸序列进行比对分析,并采用Mega 3.1软件中的N-J法构建进化树。利用Swiss-Model在线软件,对Mald4蛋白与其同源蛋白氨基酸序列空间结构进行对比,预测它们之间可能发生交叉反应的空间位点。
2 结果
2.1 Mald4蛋白二级结构预测及信号肽、跨膜区分析 运用HNN在线软件预测Mald4蛋白,结果显示以无规则卷曲为主,分布在11个区域,高达53.44%;α-螺旋集中在 4 个区域,占 19.08%;伸展链主要分布在7个区域,占27.48%(表2)。利用在线软件SignaIP 3.0分析Mald4蛋白的氨基酸序列,结果显示该蛋白无信号肽。采用TMHMM软件对Mald4蛋白跨膜区进行预测,发现具有131个氨基酸残基的Mald4蛋白无跨膜区。
2.2 Mald4蛋白的B细胞抗原表位预测 通过Bcepred在线软件对Mald4蛋白相关参数进行预测,结果显示该蛋白B细胞抗原表位区域为40~46、53~61、85~95、107 ~113(表 3)。使用 DNAStar Protean软件对Mald4蛋白相关参数进行预测,结果表明B细胞抗原表位的区域为16~18、40~48、52~61、85~95、107~114(表4)。综合上述2种方法预测B细胞抗原表位区域参数,并兼顾二级结构各参数预测结果,确定Mald4蛋白B细胞优势抗原表位区域为40~46、53~61、85~95、107~113。

表2 HNN预测Mald4二级结构结果Tab.2 The result of Mald4 secondary structure predicted by HNN
2.3 T细胞抗原表位的预测 利用网络服务器Propred和Syfpeithi对Mald4蛋白T细胞抗原表位亲和力进行预测,结果显示亲和力高的评分区域为2~5、49~93(图1);运用 NetMHCⅡ、NetMHCⅡpan软件对Mald4蛋白T细胞抗原表位进行预测,结果发现40~43、51~62、76~93、105~112为 T细胞抗原表位优势区域(图2)。综合上述T细胞表位预测结果,确定其T细胞共同抗原表位优势区域为51~62和76~93。
2.4 B细胞和T细胞抗原表位综合分析 结合上述Mald4蛋白B细胞与T细胞抗原表位预测结果,对二者的重叠区域进行拟合分析,确定Mald4蛋白B/T细胞共同表位优势区域为53~61和85~93。
2.5 Mald4蛋白抗原表位对比交叉反应预测分析 以苹果Mald4蛋白氨基酸序列为基础,经NCBIBlastP分析,并运用Cluster X1.83软件对其同源序列进行比对,结果显示苹果Mald4蛋白与桃Pru p 4(Q8GT39.1)、甜樱桃 Pru du 4(ACE80972)、草莓Fra a 4(POCOY3.1)、芒果 Man i3(ABD62998)的氨基酸序列同源性分别为95%、95%、91%、88%,都属于前纤维蛋白家族(Profilin family),而苹果Mald4蛋白的抗原表位优势区域53~61和85~93,在这几者之间是高度保守的(图3),因此它们之间发生交叉反应的概率很高。采用Mega 3.1软件构建系统进化树,发现有二个明显的分支,桃Pru p 4和甜樱桃Pru du 4前纤维蛋白首先聚在一组,然后与苹果Mald4、芒果Man i3、草莓Fra a 4前纤维蛋白聚在一起(图4)。进化比对发现,苹果Mald4处于核心地位,与桃Pru p 4和甜樱桃Pru du 4中前纤维蛋白亲缘关系较近,具有较高保守遗传稳定性和演化趋同性,表明它们之间可能存在相似的功能。

表3 Bcepred预测Mald4蛋白B细胞表位Tab.3 Prediction results of the Mald4 B cell epitopes with Bcepred

表4 DNAStar预测Mald4蛋白B细胞表位Tab.4 Prediction results of the Mald4 B cell epitopes with DNAStar

图1 Propred和Syfpeithi预测Mald4蛋白T细胞表位分析结果Fig.1 The results of the Mald4 protein T cell antigenic epitopes predicted by Propred and Syfpeithi
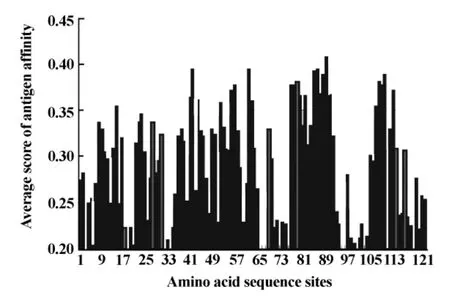
图2 NetMHCⅡ和NetMHCⅡpan预测Mald4蛋白T细胞表位分析结果Fig.2 The results of the Mald4 protein T cell antigenic epitopes predicted by NetMHCⅡ and NetMHCⅡpan

图3 不同物种前纤维蛋白优势抗原表位(方框表示区)的对比分析Fig.3 Comparisons of dominant epitopes(Box said area)among different species profilins

图4 苹果Mald4蛋白与其它物种前纤维蛋白的系统进化树(N-J法)Fig.4 Phylogenetic tree of apple Mald4 protein and profilins from other species(N-J method)

图5 不同物种蛋白空间构象表位对比图Fig.5 Conformational epitope comparison among different species proteins
利用Swiss-Model在线服务器,分别预测上述同源蛋白空间构象表位所在区域,并用PyMOL 1.0软件绘图。结果表明苹果Mald4蛋白与桃、甜樱桃、草莓、芒果的前纤维蛋白空间构象都含有4个α-螺旋,11个无规则卷曲,其中苹果、桃和芒果的前纤维蛋白含有7个伸展链,甜樱桃和草莓的前纤维蛋白含有6个伸展链(图5),虽然它们空间结构略有差异,但是具有很高的相似性。这提示前纤维蛋白家族成员之间相似的空间构象和功能在决定过敏原性特征方面可能起到重要作用。
综合上述预测结果可知,苹果Mald4蛋白与桃、甜樱桃、草莓、芒果的前纤维蛋白同源性高,空间结构相似,有较近的亲缘关系,发生交叉反应可能区域为53~61和85~93。
3 讨论
近年来,生物信息学软件在预测蛋白结构、功能及生物学特性等方面起到了非常重要的作用,已广泛运用于蛋白质抗原表位的预测[12]。理想的抗原表位是兼有B细胞表位和T细胞表位的功能[13],既可以通过刺激B细胞来产生抗原的特异性抗体,也可以与MHCⅡ类分子结合,通过抗原递呈细胞把抗原信息递呈给T淋巴细胞,引发T细胞的活化,产生特异性免疫应答,为B细胞产生抗体提供支持。因此,引入两种表位共预测的设想可提高抗原表位的预测准确率。
目前对B细胞抗原表位预测主要是以线性表位为主,代表软件有DNAStar、Bcepred等。由于不同预测软件算法不同,预测的结果存在一些差异,所以综合运用不同软件对多肽表位进行多参数预测非常必要,对提高预测结果准确性将起到一定作用[14]。
在预测B细胞抗原表位的同时,对蛋白质二级结构预测也非常重要[15]。对B细胞抗原表位进行预测时,应选择无规则卷曲的区域,避免 α-螺旋。因螺旋区的结构较为规则,化学键键能比较高,结构稳定,多位于蛋白质内部形成构象表位,不易与配体结合;而无规卷曲的区域多位于蛋白质分子的表面,易形成线性表位,有利于与配体结合,容易嵌合抗体,成为抗原表位的可能性较大[5]。随着生物信息学发展及网络数据库的扩充,应用多参数综合预测,已成为当前抗原表位预测的主流[16]。基于人工神经网络软件对T细胞表位进行预测,本文选择其中敏感性和特异性较高的软件NetMHCⅡ、NetMHCⅡpan、Syfpeithi和Propred进行综合预测。
本研究以苹果过敏原Mald4蛋白为基础,为排除信号肽或跨膜区对抗原表位筛选的干扰,首先采用SignaIP 3.0和TMHMM在线软件分析Mald4蛋白氨基酸序列的信号肽及跨膜区,结果表明该蛋白无信号肽和跨膜区。在B细胞表位的选择上,综合运用Bcepred、DNAStar两种软件预测结果,选取亲水性高、表面可及性强、抗原指数高以及柔韧性好的区域,兼顾该蛋白二级结构,选取无规则卷曲区,避免 α-螺旋区,并结合 NetMHCⅡ、NetMHCⅡpan、Propred及Syfpeithi在线软件预测T细胞抗原表位结果,最后得出优势抗原表位区域为53~61,85~93。此外,苹果Mald4蛋白与桃、甜樱桃、草莓、芒果的前纤维蛋白之间具有高度的同源性,并有非常相似的空间构象,表明它们之间发生交叉反应的概率很高。目前多数学者认为,若超过80个氨基酸的蛋白质,其序列的同源性大于35%,或有6个连续相同的氨基酸序列,则蛋白之间发生交叉反应可能性就很高[17]。然而,Silvanovich 等[18]通过概率学研究表明,以8个或更少连续相同氨基酸序列来判定蛋白的交叉过敏反应,只是一个随机产生的结果,对致敏性评估不具备任何意义。由于序列相似性分析仅仅局限于氨基酸顺序的比较,不能包括构象表位分析,因此对同源蛋白保守区空间构象进行相似性分析,预测目的蛋白与其它同源蛋白之间是否会存在交叉反应是很有意义的。
总之,本研究利用生物信息学方法,综合多种相关网络软件预测结果获得苹果Mald4蛋白T/B细胞的优势抗原表位。同时对苹果Mald4蛋白及其同源蛋白进行氨基酸序列比对和空间构象预测,获得它们之间可能发生交叉反应的优势表位区域。这为我们以后利用基因突变技术改造苹果Mald4蛋白及其同源蛋白的过敏原性,指导并深入开展无过敏性或低过敏性苹果制品以及表位疫苗的相关研究奠定了理论基础。
1 Marzban G,Puehringer H,Dey R et al.Localisation and distribution of the major allergens in apple fruits[J].Plant Science,2005;169(2):387-394.
2 Fernandez-Rivas M,Bolhaar S,Gonzalez-Mancebo E et al.Apple allergy across Europe:how allergen sensitization profiles determine the clinical expression of allergies to plant foods[J].JAllergy Clin Immunol,2006;118(2):481-488.
3 Ausukua M,Dublin I,Echebarria M A et al.Oral Allergy Syndrome(OAS).General and stomatological aspects[J].Med Oral Patol Oral Cir Bucal,2009;14(11):e568-572.
4 Ma Y,Zuidmeer L,Bohle B et al.Characterization of recombinant Mald4 and its application for component resolved diagnosis of apple allergy[J].Clinical& Experimental Allergy,2006;36(8):1087-1096.
5 Barlow D J,Edwards M S,Thornton J M.Continuous and discontinuous protein antigenic determinants[J].Nature,1986;322(6081):747-748.
6 Anandarao R,Swaminathan S,Khanna N.The identification of immunodominant linear epitopes of dengue type 2 virus capsid and NS4a proteins using pin-bound peptides[J].Virus Res,2005;112(1-2):60-68.
7 Saha S,Raghava G P.Prediction of continuous B-cell epitopes in an antigen using recurrent neural network[J].Proteins,2006;65(1):40-48.
8 Nielsen M,Lundegaard C,Lund O.Prediction of MHC classⅡ binding affinity using SMM-align,a novel stabilization matrix alignment method[J].BMC Bioinfomatics,2007;8:238-249.
9 Singh H,Raghava G P.ProPred:prediction of HLA-DR binding sites[J].Bioinformatics,2001;17(12):1236-1237.
10 Vordermeier M,Whelan A O,Hewinson R G.Recognition of mycobacterial epitopes by T cells across mammalian species and use of a program that predicts human HLA-DR binding peptides to predict bovine epitopes[J].Infect Immun,2003;71(4):1980-1987.
11 南文龙,金宁一,鲁会军et al.H5N1亚型禽流感病毒血凝素 Th和B细胞表位预测及抗原性分析[J].中国免疫学杂志,2009;25(7):630-633.
12 Korber B,LaBute M,Yusim K.Immunoinformatics comes of age[J].PLoSComput Biol,2006;2(6):484-492.
13 Parida R,Shaila M,Mukherjee S et al.Computational analysis of proteome of H5N1 avian influenza virus to define T cell epitopes with vaccine potential[J].Vaccine,2007;25(43):7530-7539.
14 Saha S,Raghava G P.Prediction of continuous B-cell epitopes in an antigen using recurrent neural network[J].Proteins,2006;65(1):40-48.
15 Jameson B A,Wolf H.The antigenic index:a novel algorithm for predicting antigenic determinants[J].Comput Appl Biosci,1988;4(1):181-186.
16 Florea L,Halldorsson B,Kohlbacher O et al.Epitope prediction algorithms for peptide-based vaccine design[J].Proc IEEE Comput Soc Bioinform Conf,2003;2:17-26.
17 Goodman R E.Practical and predictive bioinformatics methods for the identification of potentially cross-reactive protein matches[J].Mol Nutr Food Res,2006;50(7):655-660.
18 Silvanovich A,Nemeth M A,Song P et al.The value of short amino acid sequence matches for prediction of protein allergenicity[J].Toxicol Sci,2006;90(1):252-258.
