人免疫球蛋白重链可变区基因引物设计方法的改良
2012-01-12姜晶孙颖刘红艳牟玲罗恩杰
姜晶,孙颖,刘红艳,牟玲,罗恩杰*
(1.中国医科大学病原生物学教研室,辽宁沈阳110001,2.沈阳市传染病院肾综合征出血热研究所,辽宁沈阳110006)
基因工程抗体具有分子量小、组织穿透力强、体内清除快、免疫原性低、易于原核表达大量生产和进行基因工程改进等优点[1]。基因工程抗体的构建关键是抗体可变区(V)基因的获得[2]。自Orlandi等[3]最早进行抗体V基因扩增以来,20多年来V基因扩增仍然是一个热点课题,倍受科研工作者的重视。这是由于从杂交瘤细胞或外周血单核细胞中扩增抗体可变区,是构建ScFv[4]、克隆单克隆抗体和研究抗原与抗体相互作用的关键步骤。而抗体可变区的高度变异性使扩增相对困难,至今仍是一个难点。其中,PCR引物的设计对于能否顺利扩增抗体基因起关键作用。在引物设计时应尽可能保持亲本抗体可变区序列的完整性和真实性,尽管抗体分子可变区有着数量巨大的多样性,但其骨架区、前导区和恒定区相对保守。目前,包含抗体胚系(germ line)基因序列的数据库主要有Vbase2、IMGT和IgBLAST数据库。随着人类抗体胚系基因序列的测序完成,人们对抗体结构的认识更深入了一步,使得对人抗体基因的操作更加简单易行,使PCR扩增人全部可变区基因成为可能。本研究通过一个开放的简并引物设计平台iCODEHOP[5],针对可变区基因框架区FR1和FR4自主设计一套新的重链可变区引物,为获得新型基因工程抗体及构建噬菌体抗体库奠定基础。
1 材料与方法
1.1 材料
1.1.1 数据库、生物信息学软件人免疫球蛋白对应的V、J胚系基因序列来自国际免疫遗传学信息数据库(IMGT)(http://www.imgt.org/)和IgBLAST数据库(http://www.ncbi.nlm.nih.gov/ig blast)。开放的简并引物设计平台iCODEHOP(https://icodehop.cphi.washington.edu/i-codehop-context/Welcome)。
1.1.2 质粒、菌株、工具酶及试剂PMD18-T载体购自TAKARA公司,大肠埃希菌E.coli TG1由本实验室保存;DNA Marker DL2000、RNA提取试剂盒、RT-PCR试剂盒(RNA PCR Kit(AMV)Version 3.0)等均购自TAKARA公司;小量质粒提取试剂盒、凝胶回收试剂盒、Taq酶购自天根生化科技有限公司;淋巴细胞分离液购自鼎国生物制品有限公司;PAGE级引物由大连宝生物公司合成。
1.2 方法
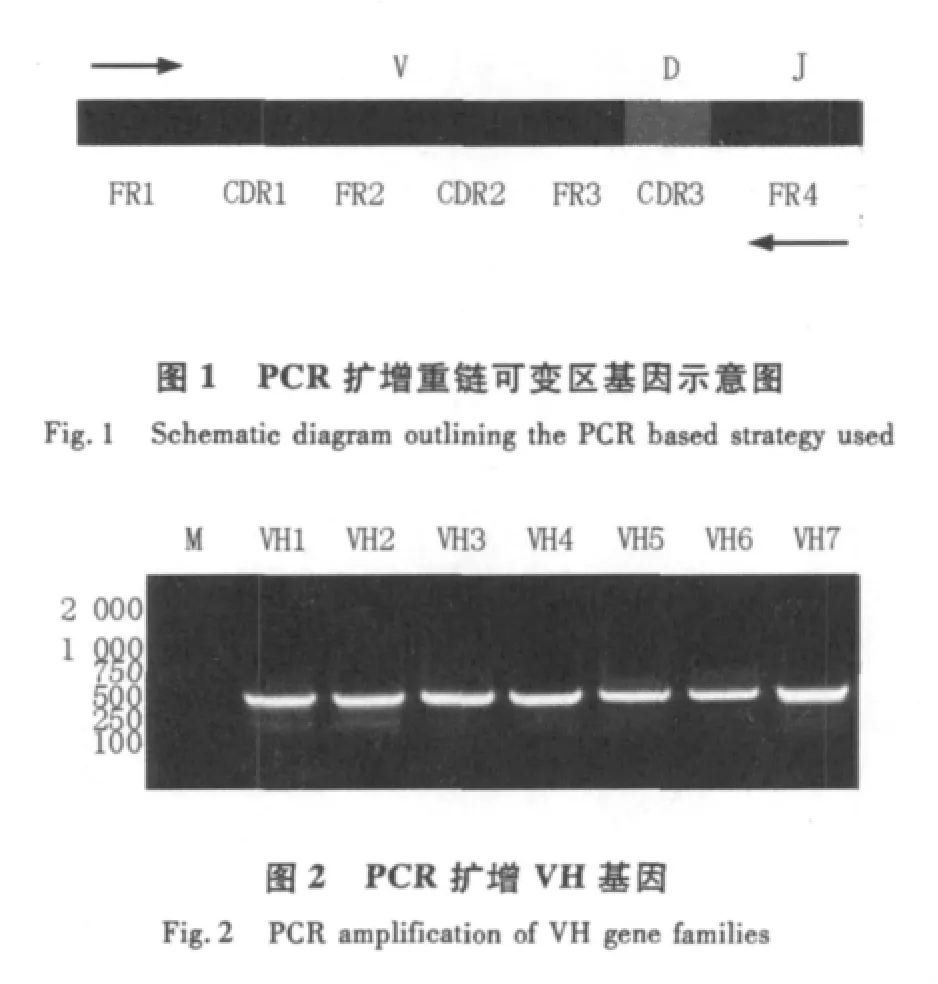
1.2.1 生物信息学分析首先,从IMGT和Ig-BLAST数据库获得了全部胚系基因序列,其中包含功能性基因及假基因。由于假基因序列的不完整性和复杂性,在引物设计时必须去除假基因,只覆盖所有的功能性基因。根据编码VH基因FR1的胚系基因V片段基因家族的不同分成7组。然后通过BlockMaker服务器将同一家族内的序列进行比对,找出高度保守区域。编码VH基因FR4的胚系基因J片段共有13个核苷酸序列放在一组中自行找出保守区。
1.2.2 CODEHOP引物设计CODEHOP引物是由简并的核心区和非简并的夹板区组成。核心区位于引物的3'端,是通过多序列比对获得的高度保守区的3~4个氨基酸对应的所有可能的密码子集合。夹板区位于引物的5'端,是由紧邻高度保守区的氨基酸序列决定的,主要根据人胚系基因的序列比对后的常用密码子设计,这个区域的长度通常是15~20个碱基,可以根据用户需要自行调整。在通过BlockMaker服务器获得的高度保守区域中使用iCODEHOP系统软件的默认参数设计可变区引物。反向引物是通过13个J片段核苷酸序列手动设计。
1.2.3 VH基因的扩增①cDNA合成:取未经免疫的健康人外周血5 mL,用淋巴细胞分离液分离淋巴细胞。参照RNA提取试剂盒使用说明,提取总RNA。按照TAKARA RNA PCR Kit(AMV)Version 3.0说明进行第一链反转录合成得到cDNA;②PCR扩增VH基因:以cDNA为模板应用自主设计的CODEHOP引物扩增抗体重链可变区基因。反应体系50 μL;反应条件:94℃5 min;94℃30 s、55℃30 s、72℃1 min;共进行35个循环,72℃延伸10 min。取5 μL PCR产物于1%凝胶中电泳,电压80 V,30 min后用凝胶成像系统进行扫描;③VH基因的鉴定:PCR产物纯化后连入PMD18-T载体中,对多个单克隆进行测序鉴定。
2 结果与分析
2.1 引物设计
通过iCODEHOP设计简并引物共获得5'端引物7条。根据13条胚系基因J区的核苷酸序列手动设计获得3'引物1条,分别与7条5'端引物配对使用(表1)。这套引物可以扩增人免疫球蛋白重链可变区框架区1(FR1)至框架区4(FR4)(图1)。
2.2 VH基因的扩增
应用自主设计的引物,经RT-PCR扩增获得VH基因。如图所示,每对引物扩增的基因大小都与预期一致,大小约为350 bp(图2)。

表1 扩增人免疫球蛋白重链可变区基因的引物Table 1 Primers for PCR amplification of Ig VH genes
2.3 VH基因多样性分析
为了鉴定PCR产物的特异性和多样性,将PCR产物纯化,克隆入PMD18-T载体中,转化TG1菌,多个单克隆提取后测序,与GenBank和IMGT/V-QUEST数据库比对表明,序列分析均为人Ig重链可变区基因,包含4个FR区和3个CDR区,V、D、J 3个基因经重排后相互连接在一起,形成V-D-J复合体,完成重链重排。对编码VH基因序列的胚系基因来源进行了分析,显示了其多样性(表2)。
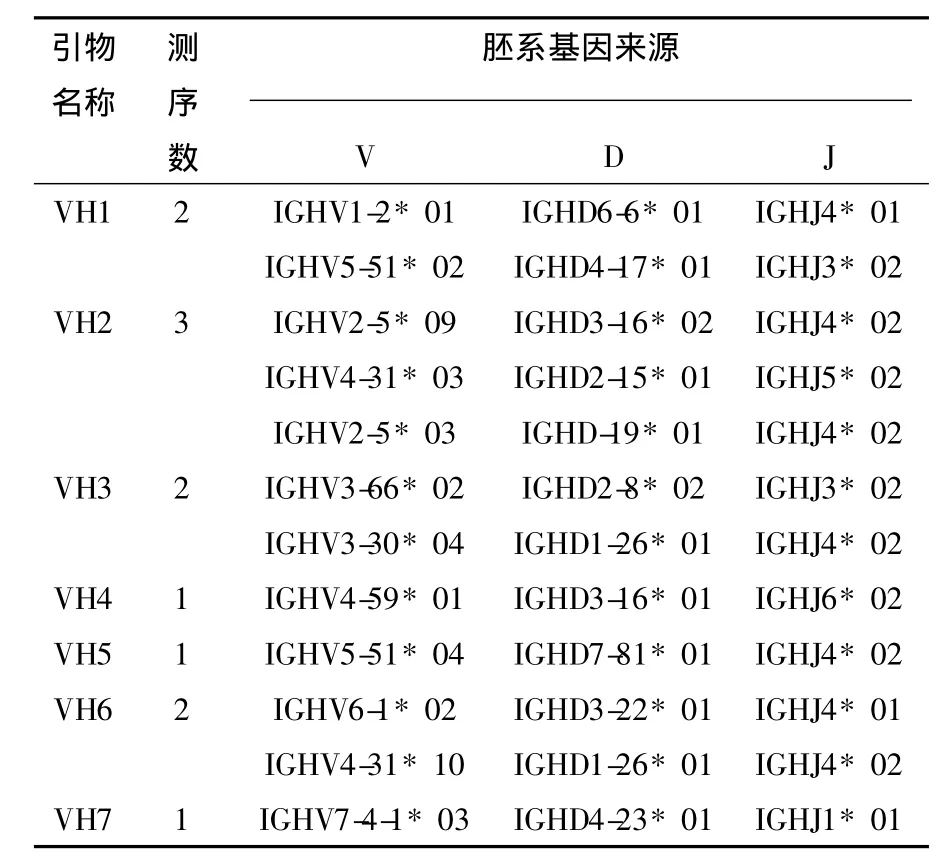
表2 重链可变区基因的多样性分析Table 2 Ig VH gene diversity analysis
3 讨论
随着人类抗体胚系基因序列的测序完成,编码重链可变区基因的V、D、J基因片段可从IMGT数据库中获得,使PCR扩增人全部可变区基因成为了可能。为了有效地扩增人Ig可变区基因,首先设计引物时应考虑的原则就是不影响产物多样性的前提下减少引物的数量。本研究通过选择不同胚系基因家族内的高度保守区来达到这一目的。
为了获取抗体基因,通常有3种设计思路:①在框架区FRl和FR4设计引物,可直接获得V基因;②设计套式PCR引物,先在信号肽和C区设计引物,再在FR1和FR4区设计引物,可提高引物的特异性并获得完全真实的V区序列,但信号肽序列变化较大,不易设计;③5'-RACE或3'-RACE,设计的引物可针对C区或polyA区,用此方法也可获得完全真实的V区序列,而且不易漏过可用的抗体基因,可用于通用引物无法扩增的杂交瘤细胞,但操作比较繁琐[6]。本研究在框架区FRl和FR4设计引物,直接获得了Ig重链可变区基因,设计方法简便有效。
目前有许多公共的开放工具都是根据核苷酸序列为基础设计简并引物,然而对于数量较大的基因片段,从氨基酸序列中寻找高度保守区可行性更高。GeneFisher[7]和iCODEHOP都是以氨基酸序列为基础设计引物,用GeneFisher所设计的引物在任何位点都可以有简并碱基,在PCR过程每一循环中都不能与模板完全匹配。而用iCODEHOP所设计的引物包括两部分,一部分是长度为11~12个碱基的3'简并核心区,一部分是根据保守氨基酸和密码子偏好性原则设计的5'非简并夹板区。用iCODEHOP引物扩增时,在第1个循环只有1种组合的特异性引物的3'核心区与目的基因完全匹配,但从第2个循环开始,每一个引物的5'夹板区都与第1轮扩增的模版完全匹配,所以就增加了扩增的特异性、准确性和成功率。
iCODEHOP过去常被用于对未知基因的引物设计,如:不同物种的同源基因、相同基因组的种内同源基因或者不同宿主中的新病原体[8-9]。本研究首次将iCODEHOP用于Ig可变区基因的引物设计。为了验证引物的特异性,对一些重组的单克隆进行了测序分析。尽管测序的数量不大、每一对引物也不是特异性扩增所属家族内的序列,但序列分析仍然显示了基因的多样性。也即表明了iCODEHOP所设计引物的有效性,为扩增其他物种可变区基因或T细胞受体(TCR)提供了新的设计思路。
[1] 马颖.单链抗体及其在生物医学中的应用[J].免疫学杂志,2006,22(3):1-3.
[2] Li J,Wang Y,Wang Z,et al.Influences of amino acid sequences in FR1 region on binding activity of the scFv and Fab of an antibody to human gastric cancer cells[J].Immunol Lett,2000,71(3):157-165.
[3] Orlandi R,Gussow PT,Jones.Cloning immunoglobulin variable domains for expression by the polymerase chain reaction[J].Proc Natl Acad Sci USA,1989,86(10):3833-3837.
[4] 邵长利,史利军,章金刚,等.抗人红细胞H抗原单链抗体基因克隆和表达[J].免疫学杂志,2008,24(2):238-242.
[5] Boyce R,Chilana P,Rose TM.iCODEHOP:a new interactive program for designing COnsensus-DEgenerate Hybrid Oligonucleotide Primers from multiply aligned protein sequences[J].Nucleic Acids Res,2009,37(Web Server issue):W222-W228.
[6] Ozawa T,Kishi H,Muraguchi A.Amplification and analysis of cDNA generated from a single cell by 5'-RACE:application to isolation of antibody heavy and light chain variable gene sequences from single B cells[J].BioTechniques,2006,40(4):469-478.
[7] Lamprecht AL,Margaria T,Giegerich R,et al.GeneFisher-P:variations of Gene-Fisher as processes in Bio-jETI[J].BMC Bioinformatics,2008,9(Suppl.4):S13.
[8] Staheli JP,Boyce R,Rose TM,et al.CODEHOP PCR and CODEHOP PCR primer design[J].Methods Mol Biol,2011,687:57-73.
[9] Staheli JP,Ryan JT,Rose TM.Consensus-degenerate hybrid oligonucleotide primers(CODEHOPs)for the detection of novel viruses in non-human primates[J].Methods,2009,49(1):32-41.
