氯化钠促进溶菌酶聚集机制的分子动力学研究
2011-12-26邹志远何剑为宋有涛
关 婧,李 辉,邹志远,何剑为,宋有涛
(辽宁大学生命科学学院,辽宁 沈阳 110036)
氯化钠促进溶菌酶聚集机制的分子动力学研究
关 婧,李 辉,邹志远,何剑为,宋有涛
(辽宁大学生命科学学院,辽宁 沈阳 110036)
氯化钠能够在一定的条件下促进溶菌酶发生聚集,但其具体的作用机制目前还尚不明确.采用分子动力学模拟的方法研究了鸡溶菌酶单体在含500mmol/L NaCl和不含NaCl两种体系中的结构变化.模拟的结果表明:NaCl能够使溶菌酶聚集关键区域(40~110位残基)内二硫键cys76-cys94的结合强度减弱,同时破坏了该区域内的氢键网络,从而导致了蛋白结构的不稳定性;另一方面,NaCl也促使疏水核心发生了一定的扩张,使疏水核心内氨基酸残基的结合变得更加松散,从而促进了溶菌酶分子间的相互作用.
溶菌酶;氯化钠;淀粉样聚集;分子动力学模拟
近年来,许多蛋白质错误折叠导致的疾病,如阿尔茨海默病、疯牛病、帕金森氏病等,已经越来越被关注,它们的发生均与蛋白质的淀粉样纤维化沉积有关,但蛋白质淀粉样纤维化的机制目前尚不清楚[1-2].已有研究结果表明,这些蛋白的成纤维倾向除了与其序列和结构有一定的关系外,更重要的是与蛋白质所处的环境条件密切相关.生物体体液中的各种盐离子可通过与蛋白质的相互作用,使蛋白质分子能够保持正确的结构.一旦体液中的离子强度发生变化,蛋白质就可能产生错误折叠而生成淀粉样聚集体[3].
溶菌酶广泛存在于鸟类的蛋清和哺乳动物的泪液、唾液、血浆、乳液、胎盘以及体液、组织细胞内,以蛋清中含量最丰富(约0.3%).迄今在世界上发现的两种由溶菌酶突变导致的家族遗传性淀粉样变性病,患者主要在内脏中都产生了大量淀粉样蛋白的沉淀物,其致死原因是肾损伤或肝出血[4].溶菌酶蛋白家族具有高度的序列和结构保守性.作为该超家族的成员,鸡卵清溶菌酶蛋白同样具有淀粉样纤维化倾向[5].以鸡卵清溶菌酶作为模型,近年来美国的Clyston等通过酶交联的方法比较分析了酸性条件下NaCl对溶菌酶聚集的影响,结果表明,该蛋白从3% (500mmol/L)的NaCl梯度开始出现明显的聚集体并随着离子强度的增加而增多[6].另外,李文涓等采用荧光光谱法发现,在长时间热胁迫的情况下,NaCl可促进溶菌酶分子的聚集而纤维化[3].戴国亮等通过动态光散射法研究不同浓度NaCl对溶菌酶生长晶体的影响时发现,较高浓度的NaCl可以诱导溶菌酶在溶液中形成较大的聚集体[7].这暗示着NaCl可能通过某种机制促进溶菌酶的构象改变,进而诱导溶菌酶发生了分子间聚集.但通过常规实验的方法对相关聚集的分子机制是很难探明的.
较传统实验方法而言,基于测定的蛋白质三级结构和牛顿力学、量子力学的分子动力学模拟(molecular dynamics simulation)能够利用计算机强大的计算能力模拟再现蛋白质纳秒数量级的结构变化过程,并在原子水平上提供有关蛋白结构变化的详细信息,可用于辅助并指导传统实验的研究.目前,这种技术已被逐渐应用于蛋白质错误折叠疾病分子机制的研究中[8-9].本研究通过模拟分析酸性条件下500mmol/L NaCl对鸡卵清溶菌酶单体稳定性的影响,从结构动力学的角度深入探讨了NaCl促进溶菌酶聚集的分子机制,对于探索蛋白质错误折叠疾病的机制,理解蛋白质在生命过程中的行为具有重要意义.
1 模拟方法和条件
本文采用Gromacs 4.0[10]软件将鸡卵清溶菌酶模型在含500mmol/L NaCl和不含NaCl两种体系中分别进行10ns分子动力学模拟,使用的力场为GROMDS9643a1,模拟条件为温度25℃,pH=1.0.初始结构文件来自于RCSB数据库射线衍射三维坐标文件(PDB编号1GXV)[11].确定模拟体系的温度耦合采用“Berendsen-thermostat”法,耦合常数为0.1ps;压力耦合采用“Parrinello-Rahman”方法,耦合常数为0.5ps,参照压力为1.0×105Pa[12].采用标准立方盒包裹模型及其他分子,晶体置于盒子中心,选择溶质原子到盒子壁的距离为1nm以保证蛋白质有足够的构象变化空间.水溶液环境下采用SPC模型(含29 174个水分子),加入18个Cl—分子使体系电荷量达到平衡.然后在其中一个体系内加入500mmol/L的NaCl.同时,为了减少结构文件转换过程中氢原子被自动加上后,由于粒子与粒子距离过紧而引起不合理的碰撞,将体系以最陡下降法(steep)优化1 000步的能量.模拟体系中的远程范德华力作用距离取1.4nm,经典相互作用使用球型截断半径“cut-off”方法计算.体系在所有方向上采用周期性边界条件,时间步长为2fs,模拟结果用VMD(visual molecular dynamics)[13]软件进行了系统可视化分析.
2 结果
2.1 模拟过程中溶菌酶RMSD值的比较分析
RMSD(root mean square deviation,均方根偏差)可用来测量两个进行比对的蛋白质相同位点上的氨基酸残基碳原子之间的距离,是测定蛋白质骨架变化指标衡量模拟过程中蛋白是否达到稳定结构的重要参数[14].RMSD值的大小直接反映了蛋白质结构的稳定性.如图1所示,溶菌酶单体在500mmol/L的NaCl模拟体系中整体结构的RMSD值在5ns左右达到平衡(约0.30nm).与不含NaCl的模拟体系相比,其平均值整体上要高0.05nm左右.这说明了NaCl使得溶菌酶单体的稳定性变差,与先前其他研究者在体外生化实验体系中观察到的现象是一致的.
2.2 模拟后溶菌酶的结构比对分析
RMSF(root mean square fluctuation,均方根涨落)是蛋白体系最终原子位置同参考位置的比较,它可以通过蛋白中各个氨基酸相对于平均结构的位置偏差来计算蛋白质二级结构变化的剧烈程度[13].如图2(A)所示,500mmol/L的NaCl模拟体系中溶菌酶的氨基酸残基整体变化幅度均大于不含NaCl的模拟体系,其中变化比较显著的区域是第40~110位残基.这与通过分子生物学实验得到的结论,即溶菌酶上触发蛋白聚集的关键区域为57~107位残基基本上是一致的[15].因此,我们选取了多肽片段40~110位残基进行了结构比对(见图2B).结果显示,在添加NaCl的模拟体系中溶菌酶的α3片段发生了部分解旋,其螺旋残基从13个降为8个,而该区域的环状(loop)结构和β片层结构却分别增加了4.3%和7.1%,表明了片段40~110区域结构在NaCl存在的情况下具有较高的柔性,另外这些二级结构的变化也是典型的蛋白质淀粉样聚集时的特征.
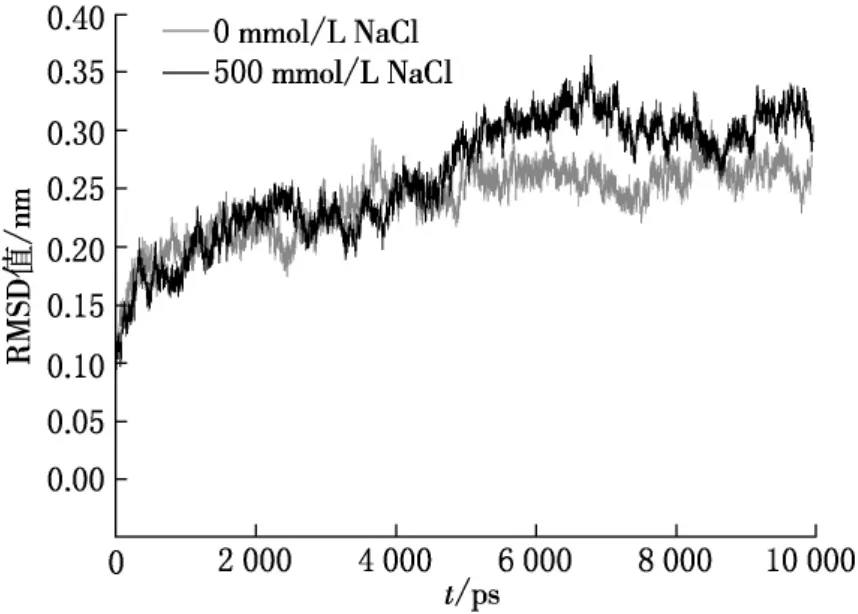
图1 RMSD值随模拟时间的变化

图2 (A)RMSF值随模拟时间变化及(B)模拟后结构比对
2.3 关键区域中二硫键的分析
溶菌酶结构中主要存在4个二硫键.前期的分子生物学实验结果表明,两个位于关键区域内的二硫键cys64-cys80和cys76-cys94在蛋白成纤维过程中起了重要作用,而其他两个二硫键并没有参与纤维的形成[15].因此,我们定量分析了溶菌酶二硫键cys64-cys80和cys76-cys94在含盐和不含盐两种模拟体系中的变化情况(见图3).在分子动力学模拟中,一般认为两个半胱氨酸之间的距离小于0.4nm为二硫键的形成状态,而大于0.4nm的为二硫键断裂状态[16].模拟的结果显示,二硫键cys64-cys80在两种模拟体系中均未发生断裂(见图3A).值得注意的是:NaCl使得二硫键cys76-cys94从4ns开始表现出明显的断裂趋势,这种趋势一直延续到模拟结束,而不含NaCl的模拟体系中溶菌酶的二硫键cys76-cys94并未发生断裂(见图3B).由于本模拟体系中的二硫键没有被保护,所以并不能确定推导出生化实验中溶菌酶的二硫键cys76-cys94是断裂的,但是这个结果暗示着溶液中NaCl可能通过削弱二硫键cys76-cys94的强度,使得溶菌酶的构象容易发生转换,从而降低其单体稳定性,最后形成分子间聚集.

图3 二硫键距离随时间的变化
2.4 关键区域中氢键存活时间的分析
氢键作为一种非共价结合作用在维持蛋白的结构稳定性方面起着重要的作用.近年来许多研究发现了蛋白质的淀粉样聚集与氢键网络破坏密切相关[17].因此,我们分析了多肽片段40~110位残基中的氢键数量及存活时间(见图4).结果表明,虽然添加的NaCl模拟体系中溶菌酶氢键的数量略高于不含NaCl体系中的氢键数量(234∶212),但是从整个模拟过程中大于95%的氢键存活时间来看,不含NaCl的模拟体系中的氢键数为21个,而NaCl模拟体系中的氢键数仅为14个.由于氢键的存活时间相对于单纯的数量对于蛋白结构的稳定性更有直接意义,这个结果说明了NaCl的加入可能会导致溶菌酶关键区域内氢键网络的破坏,从而降低其单体的稳定性,趋向于分子间的聚集.

图4 氢键随模拟时间的变化
2.5 溶菌酶疏水核心的比较分析
蛋白质的疏水核心是维持蛋白结构稳定性的重要组成.此前关于另一种淀粉样蛋白胱抑素(cystatin)的分子动力学研究表明,其疏水核心的扩张是形成胱抑素分子间聚集的重要原因[9,18].因此,本研究分析了NaCl对溶菌酶疏水核心稳定性的影响.鸡溶菌酶疏水核心结构主要是由15个疏水残基组成,包括β区域的Ile-55,Leu-56,Ile-58和α区域的 Leu-8,Met-12,Trp-28,Ala-31,Ala-32,Phe-34,Phe-38,Ala-95,Ile-98,Ala-107,Trp-108,Ala-110.通过比较疏水核心的溶剂可及表面积(SASA)不仅可以说明疏水核心内氨基酸残基结合的紧密程度,而且还可确定蛋白质整体结构的稳定性[19].如图5(A)所示,溶菌酶疏水核心在NaCl模拟体系中的溶剂可及表面积整体要高于其在不含NaCl模拟体系的数值,这暗示着NaCl促使溶菌酶的疏水核心氨基酸残基有向外扩张的趋势.模拟过程中疏水核心内具体每个氨基酸残基的位移叠加结果显示,Trp28,Phe38,Ala107,Trp108,Ala110在NaCl模拟体系中均向外发生了显著地扩张(见图5C),而这些氨基酸残基在不含NaCl的模拟体系中并未发生明显地偏移(见图5B).

图5 疏水核心SASA值随模拟时间的变化(A)及疏水核心位移叠加图
3 结论
综上所述,我们通过对鸡卵清溶菌酶蛋白质单体结构模型的分子动力学模拟的研究,提出了NaCl促进溶菌酶的聚集可能的分子机制:一方面NaCl使得溶菌酶聚集关键区域(40~110位残基)内二硫键cys76-cys94的结合强度减弱,同时破坏了该区域内的氢键网络,从而导致了蛋白结构的不稳定性;另一方面,NaCl促使疏水核心发生扩张,使得疏水核心内氨基酸残基的结合变得更加松散,从而促进了溶菌酶分子间的相互作用.这对于深入研究溶菌酶引发的蛋白质错误折叠疾病的发病机制有重要的促进意义,虽然本分子动力学模拟的结论还有待更多的分子生物学和生物化学实验的证据支持.此外,本研究中所采用的研究方法,对有关这一类由于淀粉样纤维化沉积引起的蛋白质错误折叠疾病的研究及治疗也提供了一个新的思路.
[1] ROSS C A,POIRIER M A.Protein aggregation and neurodegenerative disease[J].Nature Med,2004,10:10-17.
[2] STEFANI M.Protein misfolding and aggregation:new examples in medicine and biology of the dark side of the protein world[J].Biochim Biophys Acta,2004,1739(1):5-25.
[3] 李文涓,李芳,唐燕红,等.溶液中金属盐对溶茵酶高级结构的影响[J].化学应用与研究,2009,21(3):423-426.
[4] GILLMORE J D,BOOTH D R,MADHOO S,et al.Hereditary renal amyloidosis associated with variant lysoayzme in a large English family[J].Nephrol Dial Transplant,1999,14(11):2639-2644.
[5] PEPYS M B,HAWKINS P N,BOOTH D R,et al.Human lysozyme gene mutations cause hereditary systemic amyloidosis[J].Nature,1993,362(6420):553-557.
[6] CLAYTON L H,JOHN R C,AMANDA M B,et al.Supersaturated lysozyme solution structure studied by chemical crosslinking[J].Acta Cryst,2005,61(6):813-818.
[7] 戴国亮,胡文瑞.NaCl对液-液扩散法生长溶菌酶晶体的影响[J].化学学报,2003,61(4):520-545.
[8] HANSSON T,OOSTENBRINK C,VAN GUNSTEREN W.Molecular dynamics simulations[J].Curr Opin Struct Biol,2002,12(2):190-196.
[9] YU Y,WANG Y,HE J,et al.Structural and dynamic properties of a new amyloidogenic chicken cystatin mutant I108T[J].J Biomol Struct Dyn,2010,27(5):0739-1102.
[10] HESS B,KUTZNER C,LINDAHL E.GROMACS 4:algorithms for highly efficient,load-balanced,and scalable molecular simulation[J].J Chem Theory Comput,2008,4(3):435-447.
[11] REFAEE M,TEZUKA T,AKASAKA K.Pressure-dependent changes in the solution structure of hen egg-white lysozyme[J].Mol Biol,2003,327(4):857-65.
[12] PARRINELL O M,RAHMAN A.Polymorphic transitions in single crystals:a new molecular dynamics method[J].J Appl Phys,1981,52(12):7182-7190.
[13] HUMPHREY W,DALKE A,SCHULTEN K.VMD:visual molecular dynamics[J].J Mol Graph,1996,14(1):27-38.
[14] EASTWOOD M P,HARDIN C,LUTHEY-SCHULTEN Z.Evaluating protein structure-prediction schemes using energy landscape theory[J].IBM J Res Dev,2001,45:475-497.
[15] FRARE E,POLVERINO DE LAURETO P,ZURDO J,et al.A highly amyloidogenic region of hen lysozyme[J].J Mol Biol,2004,340(5):1153-1165.
[16] KOHLER P,KECK-GASSNMEIER B,WIESER H,et al.Molecular modeling of the N-terminal regions of high molecular weight glutenin subunits 7and 5in relation to intramolecular disulfide bond formation[J].Cereal Chem,1997,74(2):154-158.
[17] BERNARD G,GUILBERT C,DAVID P.Unfolding of hen egg lysozyme by molecular dynamics simulations at 300K:insight into the role of the interdomain interface[J].Proteins,2000,41(1):58-74.
[18] JANOWSKI R,KOZAK M,JANKOWSKA E,et al.Human cystatin C,an amyloidogenic protein,dimerizes through threedimensional domain swapping[J].Nat Struct Biol,2001,8(4):316-320.
[19] MILLER S,JANIN J,LESK A M,et al.Interior and surface monomeric proteins[J].J Mol Biol,1987,196(3):641-656.
Mechanism study of sodium chloride on lysozyme aggregation investigated by molecular dynamics simulation
GUAN Jing,LI Hui,ZOU Zhi-yuan,HE Jian-wei,SONG You-tao
(College of Life Sciences,Liaoning University,Shenyang 110036,China)
Sodium chloride(NaCl)could induce lysozyme aggregation under some conditions,but the detail mechanism has been poorly understood.In this study,performed 10ns molecular dynamic simulations to investigate the structure changes of chicken lysozyme monomer when treated with 500 mmol/L NaCl.The simulation results indicated that NaCl could weaken the disulfide bond cys76-cys94 as well as the hydrogen bonding network in the aggregation critical region,which might lead instability of the lysozyme structure.In addition,results also showed that the hydrophobic core had an apparent expanding tendency when treated with NaCl,which might promote the intermolecular interactions between lysozyme monomers.
lysozyme;sodium chloride;amyloid aggregation;molecular dynamics simulation
Q 71
180·37
A
1000-1832(2011)03-0117-05
2011-04-29
国家自然科学基金资助项目(30970152);辽宁省教育厅优秀人才基金资助项目(2009R26).
关婧(1985—),女,硕士研究生;通讯作者:宋有涛(1973—),男,博士,教授,辽宁省动物资源与疫病防治重点实验室主任,主要从事分子生物学研究
方 林)
