2007~2010年江苏地区猪繁殖与呼吸综合征病毒RT-PCR检测及部分Nsp2基因序列分析
2011-08-21顾丙泉王明珍金文杰秦爱建
钱 琨,顾丙泉,王明珍,金文杰,秦爱建
(1.扬州大学兽医学院 禽类预防医学省部共建教育部重点实验室 江苏省动物预防医学重点实验室,扬州 225009;2.江苏省南通市动植物进出境检疫局,南通 226000)
猪繁殖与呼吸综合征(Porcine reproductive and respiratory syndrome,PRRS)是由猪繁殖与呼吸综合征病毒(Porcine reproductive and respiratory syndrome virus,PRRSV)引起的一种严重危害养猪业的病毒性传染病,其主要的临床表现为妊娠母猪的繁殖障碍以及各年龄猪的呼吸系统疾病[1]。PRRS于1987年在美国的北卡罗纳、衣阿华等州首先报道,不久即传遍到了中西部,并在全美迅速蔓延。随后加拿大[2]、德国、荷兰[3]等一些国家先后暴发了该病。自1991年,亚洲地区的日本、韩国、菲律宾、中国台湾等都相继报道了该病[4,5]。中国大陆于1996年首次报道并分离到病毒[6]。现在PRRS已经成为世界性疾病,遍及世界各主要养猪国家与地区,并己成为危害养猪业最严重的传染病之一。2006年以来,中国暴发了一种以高热、高发病率、高死亡率为特征的“猪高热综合症”。通过国内学者的大量研究,证实高致病性PRRSV变异株是“猪高热综合症”的主要病因,其病毒基因与典型PRRSV相比在多处发生了特异性的变异,因此高致病性PRRSV有时也称之为非典型性PRRSV[7-9]。江苏省猪场PRRSV病毒感染情况虽然有报道,但是仍然缺乏比较系统的流行病学调查数据,因此对江苏地区高致病性PRRSV的诊断及病毒基因的遗传变异分析就显得非常必要。
PRRSV的基因组包含相互重叠的9个开放阅读框(open reading frames,ORFs),分别为ORF1a、ORF1b、ORF2-ORF7。ORF1a编码的聚合蛋白经裂解后产生6个非结构蛋白(nonstructural protein,Nsp):Nsp1α、Nsp1β和Nsp2-5。其中Nsp2是PRRSV毒株间差异较大的的一个编码区,具有种特异性,与PRRSV对细胞或组织的嗜性有关,也可能与毒株的致病性有关,在遗传关系很近的毒株间也可能有较大差异。因此,Nsp2常常作为分子流行病学研究,病毒基因多样性和进化研究的重要的靶基因之一[9]。
本研究建立并优化了一种能够区分经典型和非经典型PRRSV的RT-PCR检测方法,用此方法对江苏地区病料进行了检测,同时对Nsp2基因进行了比对分析,希望为江苏地方猪繁殖与呼吸综合征的临床诊断和防治提供参考依据。
1 材料和方法
1.1 病毒、细胞与临床病料 Marc-145细胞由中国农科院上海兽医研究所袁世山研究室惠赠;PRRSV阳性毒株JXwm06由中国农业大学杨汉春教授惠赠;高致病性 PRRSV JXA1、SY0608、WUH1、HuN4等毒株登录号等信息见参考文献[9]。
临床病料来自规模猪场患病猪的新鲜淋巴结和肺脏组织,以及2007~2010年采集的江苏省不同地区发病猪的肺、脾、肾、淋巴结等组织病料。
1.2 主要试剂与仪器 高糖型DMEM购自Sigma公司;新生牛血清(NBS)购自兰州民海生物公司;Rnasin Inhibitor 、dNTP 、1kb DNA Marker、TaqDNA聚合酶购自Fermentas;反转录酶(M-MLV)、T4 DNA Ligase、pGEM T easy购自promega公司;AxyPrep Multisource Total RNA Miniprep Kit 为美国AxyGEN公司产品;DL2000 DNA Marker 购自TaKaRa 公司;紫外分光光度仪(ND-1000)购自美国Nano Drop公司;BIO-RAD PTC-100 PCR仪购自BIO-RAD公司;高速冷冻离心机Centrifuge 5804R购自Eppendorf公司;凝胶成像系统购自美国UVP公司。
1.3 病料处理及总RNA的提取 取病猪的肺脏、脾脏和淋巴结等组织,剪碎研磨,制成匀浆,反复冻融3次,以3200×g离心5 min,取上清液,-20℃保存。
取上述上清液(或病猪血清) 200 μ L,置于DEPC水处理过的1.5 mL灭菌离心管中,然后按照RNA提取试剂盒AxyPrep Multisource Total RNA Miniprep Kit的操作说明提取病毒总RNA。
1.4 引物设计 参考GenBank登陆的基因序列,在Nsp2基因缺失区的两端保守区设计了一对引物,缺失毒株(高致病性PRRSV变异株)和非缺失毒株(经典PRRSV株),预期扩增产物长度分别约644 bp和734 bp。上游引物NSP2-F: 5'- ATC CCA GCC GCT CTG GCC GAA-3';下游引物NSP2-R:5'- GAC AGG AGC TGC TTG ATG ACAC-3'。
1.5 RT-PCR扩增 RNA样品采用25 μ L反应体系进行反转录,将检测PRRSVNsp2基因片段的下游引物(25 pmol/L)1 μ L与15 μL RNA 样品混合,70℃水浴5 min,然后迅速冰浴5 min,再加入 dNTP 2 μL(10 mmol/L),RNasin 1 μL(40 U/μL),M-MLV reverse transcriptase(10 U/μL)1μL,混匀,于42℃反转录80 min后,95℃ 5 min,立即使用或-20℃冻存备用。
取1 μL cDNA为模板,依次加入10× buffer 5 μL、Mg2+4 μL 、dNTP(10 mmol/L) 2 μL、用于检测PRRSVNsp2基因片段的上下游引物 (25 pmol/L)各 1 μL、TaqDNA 聚合酶(5 U/μL)1 μL,加超纯水至总体积为50 μL。反应条件为:95℃预变性5min;94℃变性1 min,59℃退火1 min,72℃延伸1 min,共30个循环;72℃延伸10 min。取PCR产物用1%琼脂糖凝胶电泳鉴定。
1.6 RT-PCR灵敏性试验 采用NanoDrop ND-1000型分光光度计测定纯化的PRRSV Nsp2质粒 DNA浓 度(1065 ng/μL), 将 标 准 质 粒DNA以10倍比稀释后进行浓度测定,确认稀释后标准品的浓度基本符合倍比关系,证明标准品稀释的准确性,进而得到1 μL标准品的拷贝 数 为:1065×10-6×6.023×1023/[(3015+644)×330×2]=2.7×1011copy/μL。
1.7 特异性试验 利用设计的Nsp2引物以相同的反应条件对猪圆环病毒2型(Porcine circovirus type 2 ,PCV2)、猪细小病毒(Porcine parvovirus,PPV)、 猪 流 感 病 毒(Swine influenza virus,SIV)、猪瘟病毒(Hog cholera virus,HCV)、非典型PRRSV变异株进行RT-PCR检测。
1.8 临床样品检测 取不同来源的45份PRRS疑似病料处理后按照上述的方法提取总RNA,利用NSP2-F/NSP2-R引物进行RT-PCR扩增,并将PCR产物克隆,送上海生工进行序列测定。
1.9 序列测定与分析 利用DNAStar软件,分析PRRSV各株Nsp2编码基因部分序列,按照软件中的MegAlign的Clustal W方法比较所测毒株基因序列的同源性及与参考毒株的同源性,并构建系统发生进化树。
2 结果
2.1 RT-PCR反应条件的优化 建立的RT-PCR方法在退火温度为50℃时出现隐约片段且非特异多,然后在其他条件不变的情况下提高退火温度,分别应用52℃、55℃、57℃、59℃的退火温度进行PCR,结果在退火温度为59℃时,出现明显清晰的目的条带,结果见图1。
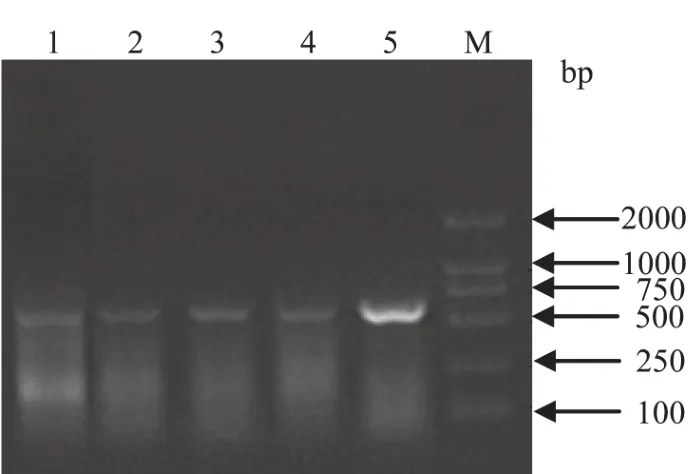
图1 不同退火温度PCR结果Fig.1 PCR in different anneal temperature
2.2 灵敏性实验 将Nsp2质粒DNA进行浓度测定,进而通过公式计算得到1μL标准品的拷贝 数 为:1065×10-6×6.023×1023/[(3015+644)×330×2]=2.7×1011copy/μL。以10倍比稀释后,采用设计的Nsp2-F/Nsp2-R引物进行PCR扩增,比较检测最低拷贝数为27copy/μL,结果见图2。

图2 不同质粒DNA浓度PCRFig.2 PCR in different plasmid concentration
2.3 特异性实验 利用设计的Nsp2引物以相同的反应条件对PCV2、PPV、SIV、HCV、PRRSV变异株进行RT-PCR检查,结果仅PRRSV变异株能扩增出644 bp的特异性条带,其他4种病毒均没有扩增出条带(见图3),证实所建立的RT-PCR检测方法特异性良好。

图3 RT-PCR检测方法的特异性鉴定Fig.3 Specif i city of RT-PCR for PRRSV
2.4 RT-PCR 方法检测临床样品的电泳结果 采用建立的RT-PCR方法对临床采集的45病例进行检测,其中共检测到阳性样品18份,阳性率为40%,部分阳性结果PT-PCR结果见图4。

图4 部分临床样品的RT-PCR扩增结果Fig.4 The fragments amplif i ed by RT-PCR from some clinical samples
2.5Nsp2基因的序列分析 利用DNAStar软件将18株PRRSV的部分Nsp2氨基酸序列与国内外已发表的毒株进行同源性的比较,结果表明:所检测的18株PRRSV之间高度同源,Nsp2的核苷酸同源性在95%~99.7%,氨基酸同源性在89.7%~99.5%之间。这些阳性毒株与美洲型代表毒株VR-2332的氨基酸同源性在57.5%~68.7%,与中国高致病性PRRSV代表毒株JXA1、WUH1的同源性分别在82.2%~97.6%、80.4%~95.3%。
Nsp2部分氨基酸比对结果见图5,这些序列与美洲型代表毒株VR-2332相比最显著的序列特征是:测定的18个分离株的Nsp2蛋白出现30个氨基酸的不连续缺失,即Nsp2蛋白的481aa处有一个氨基酸的缺失,在533-561aa处有29个氨基酸的连续缺失,与国内同时期所分离得到的高致病性PRRSV在Nsp2蛋白中有相同的序列特征[9-12]。
2.6 分离株Nsp2蛋白部分氨基酸序列的系统进化分析 基于Nsp2蛋白部分氨基酸序列绘制的遗传进化树(图6)。结果表明:所有PRRSV可分为美洲型和欧洲型,而本研究所分离的18株PRRSV,均属于美洲型毒株,与国内分离的高致病性PRRSV JXA1、SY0608、WUH1、HUN4等处于同一亚群,表明它们之间具有非常接近的亲缘关系。
3 讨论
用于PRRSV诊断的方法主要有病毒的分离法、血清学方法(IFA,ELISA,IPMA,SN)和RT-PCR等方法[13],前两种方法耗时长并且不能够区分高致病性PRRSV和经典PRRSV。

图5 部分Nsp2基因编码氨基酸序列对比分析Fig.5 Alignment of deduced amino acids sequences of partial Nsp2 gene

图6 PRRSV Nsp2部分氨基酸序列的系统发生进化树Fig.6 Phylogenetic analysis based on the partial amino acids sequences of Nsp2
相比而言,RT-PCR方法更利于临床样品快速特异的检测。本研究参考GenBank发表的PRRSV经典病毒株和高致病性毒株的Nsp2基因序列,在高致病性毒株缺失区外侧两端的保守区设计并合成了一对引物NSP2-F/NSP2-R,对临床病料进行RTPCR扩增。其中高致病性毒株、经典毒株扩增片段分别为644 bp和734 bp,从而通过PCR产物大小将二者区分开来。通过特异性实验和敏感性实验证明所建立的方法对PRRSV特异性、敏感性强且能够一步法区分高致性PRRSV和经典PRRSV毒株,适合临床以及各进出口岸的快速检测,有一定的应用推广价值。用优化后的RT-PCR方法对45份2007~2010年采集于江苏地区的临床病料进行RTPCR检测,结果显示检测到18份阳性样品。值得注意的是,这18份阳性PCR产物都为644 bp,表现为高致病性毒株的特征,这一结果与PCR产物测序的结果完全吻合。RT-PCR的结果说明,近年来高致病性PRRSV在江苏省内部分地区仍然存在,分析原因这些阳性病料往往来自于饲养管理条件较差的小规模养殖场或者直接来自于农户,可能与PRRSV疫苗免疫失败有关。但是这些阳性病毒是否具有相同的致病性和毒力还需要进一步的研究来证实。
PRRSV的Nsp2具有种特异性,与细胞或组织的嗜性有关,也可能与毒株的致病性有关,在遗传关系很近的毒株间也可能有较大差异[8]。因此,Nsp2常常作为分子流行病学研究的重要基因之一。本研究Nsp2部分序列比对分析结果表明,18株PRRSV的Nsp2的氨基酸与高致病性PRRSV代表毒株JXA1、WUH1同源性高。同时,测定的18个分离株的Nsp2蛋白出现30个氨基酸的不连续缺失,与国内同时期所分离得到的高致病性PRRSV在Nsp2蛋白中有相同的序列缺失特征,证明了这些毒株为高致病性PRRSV,而且基因进化树分析表明这些毒株与国内流行的高致病性PRRSV JXA1处于同一分支,亲缘关系非常近,推测可能来源于同一祖先。这些推测需要进一步的全基因组分析证实。另外,在45份采集的样本中没有检测到一株经典PRRSV毒株,是否说明在江苏地区经典的PRRSV已经不存在,这一结论还需要更多的样本数,以及更为全面的流行病学调查来证实。
综合以上实验结果,本研究通过RT-PCR检测以及Nsp2部分基因的变异分析对江苏地区近几年PRRSV的流行状况有了初步的了解,为地方猪繁殖与呼吸综合征的临床诊断和防治提供确实可靠的依据。
[1] Nielsen H S, Liu G, Nielsen J,et al.Generation of an infectious clone of VR-2332, a highly virulent north american-type Isolate of porcine reproductive and respiratory syndrome virus [J].J Virol, 2003,77(6):3702-3711.
[2] Bilodeau R, Dea S, Sauvageau R A,et al.Porcine reproductive and respiratory syndrome' in Quebec [J].Vet Rec, 1991, 129(5): 102-103.
[3] Wensvoort G, Terpstra C, Pol J M,et al.Mystery swine disease in the Netherlands: the isolation of lelystad virus [J].Vet Q, 1991,13(3): 121-130.
[4] Albina E, Piriou L, Hutet E,et al.Immune responses in pigs infected with porcine reproductive and respiratory syndrome virus (PRRSV) [J].Vet Immunol Immunopathol,1998, 61(1): 49-66.
[5] Blaha T.The "colorful" epidemiology of PRRS[J].Vet Res, 2000, 31(1): 77-83.
[6] 郭宝清, 陈章水, 刘文兴, 等.从疑似PRRS 流产胎儿分离PRRSV的研究[J].中国畜禽传染病, 1996.18(2): 1-4.
[7] 张建武, 庄金山, 袁世山.中国部分地区高致病性猪繁殖与呼吸综合症病毒的分子流行病学研究[J].中国农业科学, 2008, 41(6): 1822-1831.
[8] 王兴龙,李玉峰,周业飞,等.我国华东地区猪繁殖与呼吸综合征病毒流行株NSP2基因的遗传变异分析[J].中国预防兽医学报, 2011, 33(4): 319-322.
[9] Zhou L, Chen S, Zhang J,et al.Molecular variation analysis of porcine reproductive and respiratory syndrome virus in China [J].Virus Res, 2009.145: 97-105.
[10]高志强,郭鑫,杨汉春,等.猪繁殖与呼吸综合征病毒缺失变异株的基因组特征[J].畜牧兽医学报, 2005, 36 (6):578-584.
[11]童光志, 仇华吉, 周彦君, 等.猪生殖-呼吸综合征病毒CH-1a结构蛋白基因的克隆和序列分析[J].自然科学进展, 2000, 10(2): 147-153.
[12]王文成,边少国, 舒秀伟,等.猪繁殖与呼吸综合症病毒国内分离经典株与变异株的全基因组序列分析[J].畜牧与兽医, 2007, 40(7): 34-38.
[13]胡建华,高骏, 孙风萍,等.猪繁殖与呼吸综合征诊断技术研究进展[J].上海农业学报, 2004, 20(2): 106-108.
