猪脑心肌炎病毒NJ08株基因组序列测定与分析
2011-05-21蒋康富李玉峰王先炜
白 娟,蒋康富,李玉峰,王先炜,姜 平
(南京农业大学农业部动物疫病诊断与免疫重点开放实验室,江苏 南京 210095)
脑心肌炎病毒(Encephalomyocarditis virus,EMCV)是单股正链RNA病毒,属于微RNA病毒科心病毒属的成员。EMCV可感染多种哺乳动物、昆虫和人类等,啮齿类动物是EMCV的自然宿主,其中猪是受感染最普遍的动物。EMCV感染可引起断奶仔猪的急性心肌炎、突然死亡和母猪的繁殖障碍[1-2]。啮齿类动物和带毒成年动物是该病的主要传染源。鼠作为EMCV的自然宿主和传播者,对于该病毒在猪群中的流行起到重要作用[3]。
EMCV基因组全长约7.8 kb。EMCV的衣壳蛋白由4种结构蛋白VP1、VP2、VP3和VP4组成。除这4种结构蛋白外,还含有非结构蛋白2A、2B、2C、2D、3A、3B以及蛋白酶3C和RNA依赖性聚合酶3D[4]。1958年,猪EMCV作为仔猪致死性心肌炎的病原首次被报道[5]。2005年盖新娜等首次在国内分离和鉴定了该病毒[6],至今我国已分离到4株猪源和1株鼠源EMCV,并对其全基因组序列进行分析[7-9]。本研究目的是对本实验室所分离的EMCV NJ08株进行全基因测序,并与国内外分离病毒株进行同源性比较,从而确定该分离株的来源和遗传变异,同时可以丰富国内分离株的基因序列数据库,为研究EMCV的基因遗传和变异提供实验依据。
1 材料和方法
1.1 病毒株及主要试剂 EMCV NJ08分离株由本实验室分离保存;BHK21细胞购自美国ATCC;AccuPrimeTMPfx DNA Polymerase、dNTP Mixture和M-MLV等均购自Invitrogen公司;pMD18-T vector、DNA A-Tailing Kit、5'-Full RACE Kit和 3'-Full RACE Core Set Ver.2.0均购自TaKaRa公司。
1.2 引物设计 参考EMCV BJC3株(DQ464062)的核苷酸序列设计并合成覆盖ORFs的特异性引物(如读者需要作者可以提供)进行PCR扩增。这7个重叠片段含盖了整个基因组。
1.3 总RNA提取 取EMCV病毒培养液,按照TRIzol试剂说明书,提取总RNA。
1.4 RT-PCR 反转录:利用各片段特异性引物,以总RNA为模板反转录生成相应片段的cDNA链。以cDNA为模板进行PCR扩增,并优化PCR的最佳反应体系(25 μL)和反应条件。PCR反应条件设定为:95℃ 2 min;95℃ 30 s、57℃~60℃ 45 s、68℃1 min,共35个循环;72℃10 min。基因组3'末端和5'末端扩增方法参见3'-RACE试剂盒和5'-RACE试剂盒说明书。扩增产物经1%琼脂糖凝胶电泳鉴定。
1.5 扩增基因片段克隆测序 RT-PCR扩增的目的基因片段纯化回收后与pMD18-T连接、转化感受态细胞、筛选阳性克隆。鉴定的阳性克隆重组质粒由Invitrogen公司测序。利用DNAStar软件包中的MegAlign程序和测序峰图软件Chromas2.22对各片段测序结果进行拼接,获得NJ08株全基因组序列。
1.6 同源性比较及系统进化分析 采用DNAStar软件包中的MegAlign程序,将NJ08株与GenBank中登录的其它EMCV参考株的全基因组序列、各片段的核苷酸序列及其推导的氨基酸序列进行同源性分析,并构建系统进化树。
2 结果与讨论
2.1 EMCV基因组分段扩增、测序及序列拼接 利用RT-PCR和RACE方法扩增出了包含EMCV NJ08分离株全部基因组的7个cDNA片段,经1%琼脂糖凝胶电泳(图1)。各片段产物经纯化、连接、转化及测序,测序由上海英骏生物工程有限公司完成。测序结果表明,3'-UTR、F1~F5、5'-UTR这7个片段的大小分别为316 bp、1526 bp、1580 bp、2146 bp、1002 bp、1316 bp和714 bp。经拼接 EMCV NJ08分离株基因组全序列(NO:HM641897)。
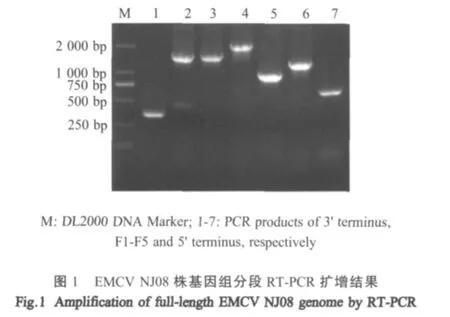
2.2 EMCV NJ08分离株的基因组组成和结构分析NJ08分离株基因组全长7724 bp其中5'-UTR为714 bp,3'-UTR为 131 bp,中间为一个大 ORF(6879 bp)编码由2292个氨基酸组成的多聚蛋白,该多聚蛋白最终切割成11个蛋白终产物:VP1~VP4、2A~2C、3A~3D和一个病毒自身编码的前导蛋白(L蛋白),其基因组组成简图如图2所示。

2.3 EMCV NJ08基因组核苷酸和推导的氨基酸与其他病毒株的比较 将NJ08株的核苷酸序列与国内外EMCV参考株全基因组序列比较分析,同源性在81.2%~99.8%之间,其中与国内分离株GXLC、GX0602、BJC3、HB1以及国外分离株CBNU、K3、K11、BEL-2887A、PV21、EMCV-R、PEC9等同源性很高,达99.6%以上,与国外分离株D variant、EMC-B、EMC-D、Mengo-M、Mengo Rz-pMwt等同源性较低,为81.2%~83.9%。各个蛋白编码区的核苷酸、氨基酸序列的同源性分析结果表明,该病毒非结构蛋白编码区比结构蛋白编码区保守,非结构蛋白中3D最为保守、2A变异最大,结构蛋白中VP2最为保守、VP1变异最大。
2.4 系统进化分析 系统进化分析结果表明,EMCV可分为2个亚群:Ia亚群和Ib亚群,猪源EMCV属于Ia或Ib亚群,鼠源EMCV属于Ia亚群。NJ08与国内分离株GXLC,GX0602,BJC3及HB1同属于Ia亚群,而且与BEL-2887A、CBNU、K3、K11等国外分离株亲缘关系较近,提示EMCV存在一定的地域差异(图3)。
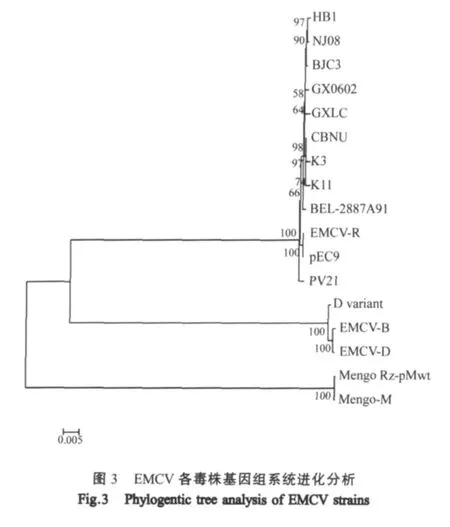
EMCV是近几年来我国新出现的一种病毒,但研究报道还不多。NJ08株分离于南京某发病仔猪,可以引起小鼠发病死亡。本研究应用RT-PCR、3'-RACE和5'-RACE技术克隆获得该病毒全基因组。分析结果表明,NJ08株与 BEL-2887A、CBNU、K3、EMCV-R、PV21等国外分离株同源性达99%以上,与分离于1990年的K3、K11株[10]全基因组序列的同源性达到99.6%,猪源病毒株NJ08与鼠源病毒株GX0602全基因组序列的同源性达到99.6%。表明近几年来源于不同国家的猪源EMCV基因组序列高度同源,分离于不同时间的猪源EMCV基因组序列高度同源,猪源EMCV与鼠源EMCV的亲缘关系较近。综上所述,对NJ08株全基因序列的测定与分析,将为相关疫苗的研究提供实验依据。
[1]Billinis C,Paschaleri-Papadopoulou E,Psychas V,et al.Persistence of encephalomyocarditis virus(EMCV)infection in piglets[J].Vet Microbiol,1999,70(3-4):171-177.
[2]Gelmetti D,Meroni A,Brocchi E,et al.Pathogenesis of encephalomyocarditis experimental infection in young piglets:a potential animal model to study viral myocarditis[J].Vet Res,2006,37:15-23.
[3]Maurice H,Nielen M,Brocchi E,et al.The occurrence of encephalomyocarditis virus(EMCV)in European pigs from 1990 to 2001[J].Epidemiol Infect,2005,133(3):547-557.
[4]Duke G M,Hoffman M A,Palmenberg A C.Sequence and structural elements that contribute to efficient encephalomyocarditis viral RNA translation[J].J Virol,1992,66(3):1602-1609.
[5]Murnane T G,Craighead J E,Mondragon H,et al.Fatal disease of swine due to encephalomyocarditis virus[J].Science,1960,131:498-499.
[6]盖新娜,杨汉春,郭鑫,等.猪脑心肌炎病毒的分离与鉴定[J].畜牧兽医学报,2007,38(1):59-65.
[7]Zhang Guo-qing,Ge Xin-na,Guo Xin,et al.Genomic analysis of two porcine encephalomyocarditis virus strains isolated in China[J].Arch Virol,2007,152:1209-1213.
[8]赵婷,张家龙,盖新娜,等.猪源和鼠源脑心肌炎病毒分离株基因组的比较分析[J].畜牧兽医学报,2009,40(6):873-878.
[9]施开创,屈素洁,陈进喜,等.猪源脑心肌炎病毒GXLC株全基因组序列测定与分析[J].病毒学报,2010,26(2):134-141.
[10]Denis P,Liebig H D,Nowotny N,et al.Genetic variability of encephalomyocarditis virus(EMCV)isolates[J].Vet Microbiol,2006,113:1-12.
