植物磷酸烯醇式丙酮酸羧化酶的功能及其在基因工程中的应用
2011-02-09魏绍巍黎茵
魏绍巍,黎茵
广东省热带亚热带植物资源重点实验室 中山大学生命科学学院,广州 510275
PEPC是一种细胞质酶,它以Mg2+或Mn2+为辅助因子,催化磷酸烯醇式丙酮酸 (PEP) 和HCO3-生成草酰乙酸 (OAA) 和无机磷酸的不可逆反应。1953年,磷酸烯醇式丙酮酸羧化酶 (Phosphoenolpyruvate carboxylase,PEPC) 从菠菜叶中首次被分离出来,此后发现PEPC不仅广泛存在于光合生物,如植物、藻类、蓝细菌和光合细菌中,还存在于很多非光合细菌和原生动物中[1-2]。
植物 PEPC具有多种生理功能,主要体现在以下几个方面:在C4和景天酸代谢 (CAM) 植物中作为C4二羧酸循环和景天酸代谢两个代谢途径的关键酶,起着固定大气中CO2并作为CO2泵而提高光合效率的作用[3-4];在C3植物中,它为Krebs循环补充四碳二羧酸从而调节回补反应[5-6];参与种子和果实的物质合成和代谢,在种子发育和萌发,以及果实成熟方面有促进作用[5,7-8];调节细胞pH,保持离子平衡,调节气孔保卫细胞的渗透压及运动[2];为豆科植物根瘤共生氮固定提供碳骨架[1-2]等等。
1 植物PEPC的种类与结构
1.1 植物PEPC的种类和基因序列特征
PEPC多肽段的大小变化非常大,在高等植物中,PEPC主要以4种同工酶形式存在,即C4光合型、C3光合型、CAM型以及非光合型[9]。在大多数高等植物中存在着3~4个编码PEPC的小ppc基因家族,每个家族包含 1个到多个基因。例如在拟南芥中发现的ppc基因有4个,在水稻和高粱中均发现6个,黄花菊属植物Flaveria trinervia和甘蔗中发现3~4个,而在玉米、欧洲油菜和尖刺莲子草中均发现3个。目前的研究发现大多数植物ppc都含有1个非常保守的由10~11个外显子和9~10个内含子组成的结构,外显子和内含子数目的差别在于一些ppc的5¢非翻译区存在或缺失1个内含子[2]。
从正在发育的蓖麻种子胚乳中发现两类PEPC,其中Class-1 PEPC是一个典型的410 kDa大小的同源四聚体,每个亚基为 107 kDa;而Class-2 PEPC是大小为681 kDa的异源八聚体,包括相同的107 kDa亚基及与之连接但免疫反应和结构都无相关性的64 kDa的肽段[10]。从拟南芥基因组中鉴定出ppc基因家族有4个基因,其中3个具有高度相似性,第4个ppc基因编码的PEPC大小与细菌PEPC相似,并且缺乏N端磷酸化位点,同时在水稻Oryza sativa中也发现了细菌型的PEPC[11]。系统发育分析发现,PEPC的线性序列显示大约有100个残基高度保守 (相同) 而且有另外100个残基也是保守的 (相似),因为C末端的高度保守,来源不同的PEPC长度的差异似乎是在N末端或内部区域中添加或缺失额外序列引起的[1,12]。蔡小宁等通过对GenBank上登录的PEPC的cDNA序列、氨基酸序列进行 Blast搜索,找到了 62个 PEPC蛋白质序列,绝大多数属于C3植物,9个属于C4植物,比较发现62个PEPC蛋白的亚细胞定位基本一致,并且这62个PEPC蛋白质氨基酸序列的同源性为86.70%,而其中19个C3PEPC和9个C4PEPC同源性分别为 89.63%和 89.61%,说明植物PEPC具有高度同源性[6]。
1.2 PEPC的蛋白质结构
目前所有已知的PEPC均是四聚体,4个相同亚基的分子量范围为 95~110 kDa[12]。2002年Matsumura等发表了从玉米叶片中纯化的结合了硫酸根离子的ZmPEPC蛋白质晶体结构,这是目前唯一报道的植物 PEPC的三维蛋白晶体结构[12-13]。X-衍射分析发现ZmPEPC酶蛋白由4个相同的亚基组成“双二聚体”构型,排列成四方形,中间是 1个大空隙。二聚体中 2个单体的接触面积较大,而 2个二聚体的接触面积较小,这正好解释了 ZmPEPC一旦被稀释之后具有解离成二聚体的趋势[1,13]。ZmPEPC单体由1个8股链的β桶形结构 (β barrel)和它周围的42个α螺旋构成,β链由40个氨基酸构成;α螺旋由576个氨基酸构成,α螺旋大部分集中在β桶的C末端,只有少部分在β桶的N末端[13]。高度保守的氨基酸残基在β桶C末端区呈束状排列,在外围区域附近分散[1]。研究还发现ZmPEPC的催化活性位点位于β桶C末端区域,在催化位点中有4个精氨酸残基 (Arg456、Arg647、Arg759和Arg773)与底物PEP结合,2个带负电的氨基酸 (Glu566和Asp603) 与镁离子结合,这些氨基酸在所有的PEPC中严格保守[2,12-13]。
生物学信息预测分析表明其他植物 PEPC的结构与ZmPEPC类似。González等分析了小麦PEPC的蛋白结构,发现小麦 PEPC单体二级结构中预测的活性部位同样是由β桶和较多的α螺旋构成,相应的活性部位有Arg457、Arg648、Arg760和Arg895等保守氨基酸残基[14]。陈明娜等对碱蓬 PEPC蛋白质结构预测结果显示,二级结构以 α螺旋为主,占60.46%,无规则卷曲占35.61%,β折叠占3.93%;三级结构呈紧密的球状结构;跨膜结构域信号肽分析表明该蛋白不含信号肽,属于非分泌性蛋白[15]。
2 植物组织中PEPC的活性调节
绝大多数的 PEPC是变构酶,其变构作用的调节过程往往与代谢产物的反馈调节作用相关[4,8]。双子叶植物 PEPC的活性可被正调控代谢效应物葡萄糖-6-磷酸 (G-6-P) 和丙糖磷酸等激活,并被负调控反馈抑制剂L-苹果酸或Asp等抑制,同时单子叶植物PEPC活性还可被Gly或Ala激活[1,5]。另外,一些包含磷酸基的化合物以及有机酸或 PEP/丙酮酸的类似物还分别是常用的激活剂和抑制剂[16]。除此之外,C4植物 PEPC氨基端的 1个保守丝氨酸残基还受到可逆磷酸化的调节[2]。植物细胞中的PEPC可被一种高水平底物专一性的Ca2+依赖性丝氨酸/苏氨酸激酶——PEPC激酶 (PEPCK) 磷酸化[7,17],但是修饰PEPC试验以及RNAi或反义技术抑制PEPCK活性试验暗示:至少对于C4植物光合PEPC来说,PEPCK对于提高光合效率并不具有重要的调节功能[2]。
引起 ppc基因的差别表达的可能因素还包括特化的细胞核因子、DNA甲基化和不同启动子的存在[14],因此ppc的表达也依赖于器官或细胞的类型、发育时期以及叶片的位置。所有的非 C4植物PEPC在C3植物叶片所有组织中通常表现为低水平表达,在C4植物进化过程中ppc的表达发生了改变,从而大大提高了表达水平[2,18]。对黄花菊属 C4植物Flaveria trinervia光合ppc基因 (ppcA) 启动子的研究支持了ppc表达受到转录调控的事实,这种C4启动子包含2个区域,1个从起始位置到-570的近端片段 (PR) 和 1个从-1 566到-2 141的远端片段(DR),这两个区域是保持ppcA在叶肉细胞中高水平特异性表达的充分必要条件,PR和DR是作为一个加性的协同转录控制系统来起作用的。通过启动子删除和重组研究,发现DR包含1个41 bp的MEM1 (叶肉表达元件 1),主要与亮氨酸拉链 (bZIP) 蛋白家族反式转录因子进行结合[18-19]。PEPC除受到体内调节因子的调节之外,还受到光、温度、pH、氮代谢、盐及干旱等外界环境因子的影响[9,16]。
3 PEPC在基因工程中的应用
3.1 提高作物光合效率
C4PEPC对其底物有较高的亲合力,将C4光合关键酶基因导入 C3植物从而在重要的 C3作物中建立起 C4循环,提高光合生产能力一直是人们关注的问题[20]。Ku等首次将完整的玉米 C4型ppc转入水稻中,体外测定表明PEPC活性增加110倍,植株中的PEPC蛋白含量占叶片总可溶蛋白的12%[21],同时光呼吸有一定程度削弱,但光合速率未发生明显变化。Bandyopadhyay等将完整的玉米ppc整合进籼稻中,观察到在高温下转基因籼稻的光合速率得到了提高,但是产量并没有变化[22]。方立锋等对转ppc水稻的研究结果表明,在旱作条件下,2个转 ppc株系单株产量分别比未转基因的对照增加28%和42%,分蘖增加27%和40%;同时转ppc水稻有较强的抗氧化能力和渗透调节能力[23]。周宝元等对转玉米 ppc水稻株系的研究发现,单纯导入ppc并不能提高水稻的光合速率,而干旱胁迫下由于PEPC参与水稻的抗旱反应而减轻了胁迫对光合作用的抑制[24]。向珣朝等发现不同遗传背景的转 ppc水稻材料在提高其净光合速率方面表现不一致,但ppc单个基因的表达在不同保持系遗传背景下能够大幅度提高千粒重和单株产量[25]。张建福等对转甘蔗 ppc籼稻进行分析,结果表明转基因株系的光合效率显著高于非转基因对照[26]。陈绪清等将玉米ppc完全整合到小麦基因组中,测定发现部分转基因植株叶片中PEPC酶活性提高了3~5倍,光合速率有所提高,并且气孔的开放程度与叶片中的 PEPC活性紧密相关,说明完整的玉米C4ppc在小麦中可以正确表达并起到一定的生理作用[27]。
由于C4循环是在多种酶和转运蛋白的协同作用下完成的,增加个别基因的表达并不能在C3植物中建立完整的 C4循环,还可能扰乱 C3植物的正常代谢,因此目前的研究也尝试将两个或更多C4关键酶基因转入同一种C3植物中[18]。袁定阳等将PEPC、PPDK (磷酸丙酮酸二激酶) 基因导入水稻恢复系R299,PEPC酶活性和光合速率分析结果表明,其PEPC活性比对照提高了1.5~4.9倍,光合速率提高了19%~40%[28]。张边江等发现转PEPC+PPDK 双基因水稻高表达了C4光合酶活性,在高光和高温条件下,喷施外源ATP后,其光合能力增强,光氧化伤害减轻,产量明显提高[29]。傅元婷等研究了转PEPC+PPDK+ME (苹果酸酶) 基因水稻 (PKM)的生理特性,结果表明,PKM水稻高表达了相关的C4光合酶,在高光和高温条件下 PKM 水稻光合速率比原种提高55%以上,水稻耐光氧化能力进一步增强[30]。Taniguchi等通过建立水稻转基因系研究了PEPC、PPDK、NADP-ME (NADP-苹果酸酶) 和NADP-MDH (NADP-苹果酸脱氢酶) 4种C4酶单独或联合在水稻叶肉细胞中超表达对水稻光合特性的影响,结果表明,联合 4种酶的超表达在叶绿体中并不能浓缩CO2。同时发现PEPC和ME超表达导致轻微的发育迟缓,而超表达 MDH可以缓和这种效应[31]。
3.2 提高油料作物种子脂肪酸含量
PEPC是控制植物种子中蛋白质和脂肪酸合成的关键酶之一,抑制其活性可以提高植物体中脂肪酸的含量。PEPC在植物种子中参与种子贮藏蛋白和脂肪酸代谢的调控方面的功能引起了农业界的关注[32-34],当PEPC的表达受到抑制时,PEPC通过改变代谢模式使碳原子的流向从糖类物质的合成转向脂肪酸的合成[35-37]。Kubis等对油菜种子的研究表明,种子质体中的物质代谢流向显示碳原子通过形成多种中间代谢物经质体转运蛋白进入质体参与脂肪酸的合成 (图1)[37]。陈锦清等研究发现反义ppc转化油菜籽粒含油量比对照最高增加了 15%以上,蛋白质含量与油脂含量呈显著负相关,该研究证明了底物竞争调控籽粒蛋白质油脂比率的理论,并在此基础上经多年选育得到超高油油菜转基因品种[33]。其后在大豆、稻米等作物中应用这一技术也得到了籽粒高油品质的转基因植物[38-39]。说明 PEPC在作物种子中的调控能够显著改变种子的物质代谢过程并且可以实际应用到农业生产当中。彭苗苗等利用海岛棉 ppc非保守序列的片段分别构建了带有种子特异性napin启动子和α球蛋白B基因启动子的PEPC-ihpRNA载体,为后期高含油量棉花材料的选育打下基础[40]。范正琪等从能源植物麻疯树中克隆了 ppc基因全长 cDNA,并对其所编码的氨基酸序列进行了生物信息学分析,为鉴定麻疯树 ppc基因功能和进一步培育高油麻疯树新品种奠定了前期基础[41]。而潘昱名等利用 RT-PCR技术克隆花生ppc片段,构建了由35S启动子所控制的反义表达载体[34,42]。本实验室也利用花生 ppc片段分别构建了35S启动子和花生油体蛋白种子特异启动子[43]控制的 RNAi植物表达载体,并在烟草中进行了转基因检测,为获得转基因高油花生种质提供基因和表达载体 (魏绍巍和黎茵,未发表资料)。
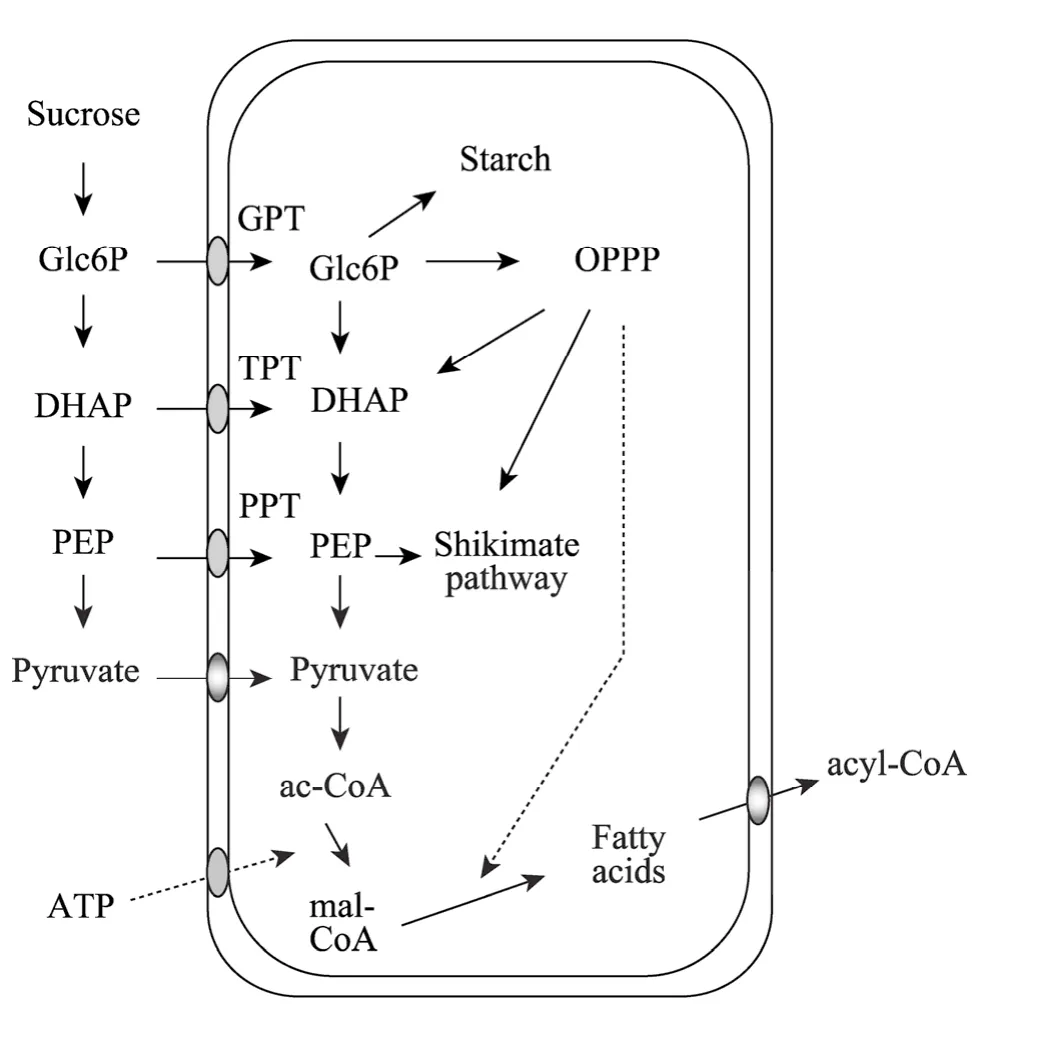
图1 油料作物种子质体中的物质代谢流向[37]Fig. 1 Pathways of metabolic flux in plastids of oil seeds[37]. Glc6P: glucose 6-phosphate; DHAP: dihydroxyacetone phosphate; PEP: phosphoenolpyruvate; ac-CoA: acetyl-CoenzymeA; mal-CoA: malonyl-CoenzymeA; acyl-CoA: fatty acyl-CoA.
3.3 促进种子蛋白质合成
在种子中,与抑制 PEPC表达促进脂肪酸的合成相反,提高 PEPC的表达可激活糖酵解过程中碳原子流向蛋白质方向的分支代谢途径[8,44]。在种子发育的早期和中期,子叶中 PEPC的表达量会明显上升,它的活性通常被磷酸化所激活,并受到植物此时碳源和氮源供给的影响,种子发育后期蛋白质合成基本完成后 PEPC的活性明显下降,因而从一个侧面反映了发育子叶中 PEPC对于蛋白合成当中通过合成有机酸为氨基酸提供碳骨架的重要性[4,44-46]。Rolletschek等将谷氨酸棒杆菌的PEPC转入豆科牧草法国野豌豆并使其在种子中特异表达,发现转基因植物的种子中通过 CO2代谢进入种子蛋白的碳含量增加了3倍之多,虽然总糖含量有所下降,但转基因种子中蛋白质相对于种子干重的含量增加了20%,并且可能由于碳同化作用的提高,种子干重也增加了 20%~30%,在此基础上,Rolletschek等总结得出了过量表达 PEPC的转基因植物种子中糖代谢途径的可能调控途径 (图2)[8]。该研究还发现过量表达 PEPC可促进豆球蛋白的积累,说明在转基因植物中通过调控 PEPC的表达不仅提高豆类种子的蛋白含量,也使其中的一些组分发生变化[8]。张占琴等在油菜中的研究结果表明,PEPC活性直接影响了不同发育时期籽粒的油脂含量/蛋白含量比值,PEPC活性升高期间蛋白积累速度快于油脂,油脂含量/蛋白含量比值低,说明PEPC促进蛋白的合成,该研究对种子蛋白组分的分析结果也表明PEPC的活性与部分种子贮藏蛋白的积累变化有关[47]。因此利用 PEPC相关的生物工程途径改变作物种子的蛋白合成模式并提高种子的品质是完全可行且有效的[8,48]。
3.4 在微生物和藻类基因工程中的应用
PEPC在种类繁多的微生物中也广泛分布,并具有多重生物功能,是糖异生作用和氨基酸合成之间的重要纽带[49],因此也是许多微生物基因工程的调控靶标。Lin等发现在大肠杆菌中同时过表达高粱ppc和乳酸菌pyc (丙酮酸羧化酶基因),能够有效提高具有化工利用价值的琥珀酸产量[50]。近年来微藻作为生物柴油生产原料越来越受到人们的关注。抑制PEPC活性有助于ACCase (乙酰辅酶A羧化酶)催化底物进入脂肪酸途径,侯李君等在大肠杆菌中构建了鱼腥藻 ppc基因 (pepcA) 的正反义表达载体,发现 PEPC的过表达促进了蛋白质的形成,抑制了脂类的合成;而下调 PEPC的表达会促进脂类的合成,减少蛋白的合成[51]。宋东辉等开展了利用反义技术调控微藻 PEPC代谢途径的工作,初步结果表明,反义抑制 PEPC酶活性可显著提高微藻细胞中脂类物质的含量[52]。

图2 PEPC转基因植物种子中糖代谢途径的调控[8]Fig. 2 Proposed changes in the metabolic pathway of carbonhydrates in PEPC transgenic seeds[8]. Metabolites which are decreased are given in red. Metabolites which are increased are shown in green. ADPG: adenosine diphosphate glucose; ATP, adenosine triphosphate; F6P: fructose-6-phosphate; F1,6P2: fructose-1,6-bisphosphate; G1P: glucose-1-phosphate; G3P: glucose-3-phosphate; G6P: glucose-6-phosphate; OAA: oxaloacetate; 3PGA: 3-phospho-glycetate; UDPG: uridine diphosphate glucose.
4 展望
植物 PEPC的结构特点以及在植物细胞中参与的代谢途径已有深入的研究,其生理生化机制在分子水平上也逐步得到阐明。ppc基因的来源、功能、遗传操作等研究取得的成果,为利用 PEPC调控改良作物碳同化途径、提高油料作物和蛋白来源种子作物营养品质提供了理论和实践基础,也为能源植物和微生物基因工程的应用展现了很好的前景。本实验室的部分研究结果也显示,PEPC在植物中尤其是叶片组织细胞中的表达会受到多种因素调节,从而直接影响碳同化的效率,整体降低 PEPC的表达水平会对植物的代谢和生长造成影响,因此应考虑采用组织特异表达的方式来下调 PEPC表达,促进脂肪酸合成。而整体水平上大量提高 PEPC的表达则有可能同时提高植物的干物质总量、蛋白质含量和抗胁迫能力,从而达到高产优质的目的。
REFERENCES
[1] Izui K, Matsumura H, Furumoto T, et al. Phosphoenolpyruvate carboxylase: a new era of structural biology. Annu Rev Plant Biol, 2004, 55: 69−84.
[2] Gowik U, Westhoff P. C4-phosphoenolpyruvate carboxylase//Raghavendra AS, Sage RF. C4Photosynthesis and Related CO2Concentrating Mechanisms. Advances in Photosynthesis and Respiration. Dordrecht: Springer, 2011, 32: 257−275.
[3] Guillet C, Just D, Bénard N, et al. A fruit-specific phosphoenolpyruvate carboxylase is related to rapid growth of tomato fruit. Planta, 2002, 214(5): 717−726.
[4] Jeanneau M, Vidal J, Gousset-Dupont A, et al. Manipulating PEPC levels in plants. J Exp Bot, 2002, 53(376): 1837−1845.
[5] Chollet R, Vidal J, O’Leary MH. Phosphoenolpyruvate carboxylase: a ubiquitous highly regulated enzyme in plants. Annu Rev Plant Physiol Plant Mol Biol, 1996, 47: 273−298.
[6] Cai XN, Chen Q, Yang P, et al. Bioinformatics analysis of phosphoenolpyruvate carboxylase. J Anhui Agri Sci, 2008, 36(3): 914−916.蔡小宁, 陈茜, 杨平, 等. 磷酸烯醇式丙酮酸羧化酶的生物信息学分析. 安徽农业科学, 2008, 36(3): 914−916.
[7] Vidal J, Chollet R. Regulatory phosphorylation of C4PEP carboxylase. Trends Plant Sci, 1997, 2(6): 230−237.
[8] Rolletschek H, Borisjuk L, Radchuk R, et al. Seed-specific expression of a bacterial phosphoenolpyruvate carboxylase in Vicia narbonensis increases protein content and improves carbon economy. Plant Biotech J, 2004, 2(3): 211−219.
[9] Dong LY, Ling J. Structure and expression of phosphoenolpyruvate carboxylase gene in higher plants. Plant Physiol Commun, 1991, 27(6): 407−412.董龙英, 凌俊. 高等植物磷酸烯醇式丙酮酸羧化酶的基因结构及其基因表达的调节. 植物生理学通讯, 1991, 27(6): 407−412.
[10] Gennidakis S, Rao S, Greenham K, et al. Bacterial-and plant-type phosphoenolpyruvate carboxylase polypeptides interact in the hetero-oligomeric class-2 PEPC complex of developing castor oil seeds. Plant J, 2007, 52(5): 839−849.
[11] Sánchez R, Cejudo FJ. Identification and expression analysis of a gene encoding a bacterial-type phosphoenolpyruvate carboxylase from Arabidopsis and rice. Plant Physiol, 2003, 132(2): 949−957.
[12] Zhao Y, Chen LM, Li KZ. Research progresses on the molecular structure of phosphoenolpyruvate carboxylase. Biotech Bull, 2007(5): 9−13.赵艳, 陈丽梅, 李昆志. 磷酸烯醇式丙酮酸羧化酶的分子结构研究进展. 生物技术通报, 2007(5): 9−13.
[13] Matsumura H, Xie Y, Shirakata S, et al. Crystal structures of C4form maize and auaternary complex of E. coli phosphoenolpyruvate carboxylases. Structure, 2002, 10(12): 1721−1730.
[14] González MC, Echevarría C, Vidal J, et al. Isolation and characterisation of a wheat phosphoenolpyruvate carboxylase gene. Modelling of the encoded protein. Plant Sci, 2002, 162(2): 233−238.
[15] Chen MN, Yang QL, Yu SL, et al. Cloning and analysis of phosphoenolpyruvate carboxylase gene from Suaeda glauca. Marine Sci, 2009, 33(6): 67−72.陈明娜, 杨庆利, 禹山林, 等. 碱蓬 PEPCase基因的克隆与分析. 海洋科学, 2009, 33(6): 67−72.
[16] Rajagopalan AV, Devi MT, Raghavendra AS. Molecular biology of C4phosphoenolpyruvate carboxylase: structure, regulation and genetic engineering. Photosynth Res, 1994, 39(2): 115−135.
[17] Hartwell J, Gill A, Nimmo GA, et al. Phosphoenolpyruvate carboxylase kinase is a novel protein kinase regulated at the level of expression. Plant J, 1999, 20(3): 333−342.
[18] Gowik U, Burscheidt J, Akyildiz M, et al. cis-Regulatory elements for mesophyll-specifc gene expressioninthe C4plant Flaveria trinervia, the promoter of the C4phosphoenolpyruvate carboxylase gene. Plant Cell, 2004, 16(5): 1077−1090.
[19] Akyildiz M, Gowik U, Engelmann S, et al. Evolution and function of a cis-regulatory module for mesophyll-specifc gene expression in the C4dicot Flaveria trinervia. Plant Cell, 2007, 19(11): 3391−3402.
[20] Luo ZX, Zhang SZ, Yang BP. Transformation of genes of C4photosynthetic key enzyme into C3plants. Plant Physiol Commun, 2008, 44(2): 187−193.罗遵喜, 张树珍, 杨本鹏. C4光合关键酶基因转化C3植物. 植物生理学通讯, 2008, 44(2): 187−193.
[21] Ku MSB, Agarie S, Nomura M, et al. High-level expression of maize phosphoenolpyruvate carboxylasein transgenic rice plants. Nat Biotech, 1999, 17(1): 76−80.
[22] Bandyopadhyay A, Datta K, Zhang J, et al. Enhanced photosynthesis rate in genetically engineered indica rice expressing pepc gene cloned from maize. Plant Sci, 2007, 172(6): 1204−1209.
[23] Fang LF, Ding ZS, Zhao M. Characteristics of drought tolerance in ppc overexpressed rice seedlings. Acta Agrono Sin, 2008, 34(7): 1220−1226.方立锋, 丁在松, 赵明. 转 ppc基因水稻苗期抗旱特性研究. 作物学报, 2008, 34(7): 1220−1226.
[24] Zhou BY, Ding ZS, Zhao M. Alleviation of drought stress inhibition on photosynthesis by overexpression of PEPC gene in rice. Acta Agrono Sin, 2011, 37(1): 112−118.周宝元, 丁在松, 赵明. PEPC过表达可以减轻干旱胁迫对水稻光合的抑制作用. 作物学报, 2011, 37(1): 112−118.
[25] Xiang XC, He LB, Sun JM, et al. Effect of maize PEPC gene in different genetic backgrounds of CMS maintainers and tolerance to photooxidation in the PEPC transgenic line. Chin J Rice Sci, 2009, 23(3): 257−262.向珣朝, 何立斌, 孙建明, 等. 玉米 PEPC基因在籼型水稻保持系不同遗传背景下的效应及转PEPC基因后代的耐光氧化特性. 中国水稻科学, 2009, 23(3): 257−262.
[26] Zhang JF, Sun YP, Ma HM, et al. Genetic analysis of restorer line N175 with pepc from sugarcane. Mol Plant Breed, 2009, 7(4): 666−670.张建福, 孙亚萍, 马宏敏, 等. 转甘蔗pepc基因籼稻恢复系 N175的遗传学分析. 分子植物育种, 2009, 7(4): 666−670.
[27] Chen XQ, Zhang XD, Liang RQ, et al. Expression of the intact C4type pepc gene cloned from maize in transgenic winter wheat. Chin Sci Bull, 2004, 49(19): 1976−1982.陈绪清, 张晓东, 梁荣奇, 等. 玉米C4型pepc基因的分子克隆及其在小麦的转基因研究. 科学通报, 2004, 49(19): 1976−1982.
[28] Yuan DY, Duan MJ, Tan YN, et al. Generating marker-free transgenic homozygous rice restorer lines with PEPC and PPDK genes by Agrobacterium-mediated transformation using super binary vector. Hybrid Rice, 2007, 22(2): 57−63.袁定阳, 段美娟, 谭炎宁, 等. 共转化法获得无筛选标记的转 PEPC、PPDK基因水稻恢复系纯合体. 杂交水稻, 2007, 22(2): 57−63.
[29] Zhang BJ, Hua C, Zhou F, et al. Photosynthetic characteristics of transgenic rice with PEPC+PPDK gene. Sci Agri Sin, 2008, 41(10): 3008−3014.张边江, 华春, 周峰, 等. 转 PEPC+PPDK双基因水稻的光合特性. 中国农业科学, 2008, 41(10): 3008−3014.
[30] Fu YT, Zhang BJ, Chen QZ. Physiological performance of transgenic rice expressing C4genes. J Henan Agri Sci, 2010(5): 18−22.傅元婷, 张边江, 陈全战. 转 C4基因水稻的生理表现.河南农业科学, 2010(5): 18−22.
[31] Taniguchi Y, Ohkawa H, Masumoto C, et al. Overproduction of C4photosynthetic enzymes in transgenic rice plants: an approach to introduce the C4-like photosynthetic pathway into rice. J Exp Bot, 2008, 59(7): 1799−1809.
[32] Sugimoto T, Tanaka K, Monma M, et al. Phosphoenolpyruvate carboxylase level in soybean seed highly correlates to its contents of protein and lipid. Agri Biol Chem, 1989, 53(3): 885−887.
[33] Chen JQ, Lang CX, Hu ZH, et al. Antisense PEP gene to ratio of protein and lipid content in Brassica napus seeds. J Agri Biotech, 1999, 7(4): 316−320.陈锦清, 郎春秀, 胡张华, 等. 反义 PEP基因调控油菜籽粒蛋白质/油脂含量比率的研究. 农业生物技术学报, 1999, 7(4): 316−320.
[34] Pan YM, Liu FZ, Wan YS. Cloning of peanut gene fragment encoding PEPCase and construction of its plant expression vector. Acta Agri Boreali-Sin, 2009, 24(5): 69−72.潘昱名, 刘风珍, 万勇善. 花生磷酸烯醇式丙酮酸羧化酶基因的克隆及反义表达载体的构建. 华北农学报, 2009, 24(5): 69−72.
[35] Ruuska SA, Girke T, Benning C, et al. Contrapuntal networks of gene expression during Arabidopsis seed filling. Plant Cell, 2002, 14(6): 1191−1206.
[36] Schwender J, Ohlrogge J, Shachar-Hill Y. Understanding flux in plant metabolic networks. Curr Opin Plant Biol, 2004, 7(3): 309−317.
[37] Kubis SE, Pike MJ, Everett CJ, et al. The import of phosphoenolpyruvate by plastids from developing embryos of oilseed rape, Brassica napus (L.), and its potential as a substrate for fatty acid synthesis. J Exp Bot, 2004, 55(402): 1455−1462.
[38] Zhao GL, Chen JQ, Yin AP, et al. Transgenic soybean lines harbouring anti-PEP gene express super-high oil content. Mol Plant Breed, 2005, 3(6): 792−796.赵桂兰, 陈锦清, 尹爱萍, 等. 获得转反义 PEP 基因超高油大豆新材料. 分子植物育种, 2005, 3(6): 792−796.
[39] Wu GT, Lang CX, Hu ZH, et al. Raising fat content in transgenic rice by anti-PEP gene transformation. J Plant Physiol Mol Biol, 2006, 32(3): 339−344.吴关庭, 郎春秀, 胡张华, 等. 应用反义 PEP 基因表达技术提高稻米脂肪含量. 植物生理与分子生物学学报, 2006, 32(3): 339−344.
[40] Peng MM, Yu JW, Zhai HH, et al. Construction and identification of a seed-specific expression ihpRNA vector targeting the PEPC gene of cotton. Genom Appl Biol, 2010, 29(2): 233−238.彭苗苗, 于霁雯, 翟红红, 等. 棉花 PEPC基因种子特异性ihpRNA表达载体的构建及鉴定. 基因组学与应用生物学, 2010, 29(2): 233−238.
[41] Fan ZQ, Li JY, Tian M, et al. Cloning and sequence analysis of full-length cDNA of pepc gene from Jatropha curcas. Forest Res, 2010, 23(3): 349−354.范正琪, 李纪元, 田敏, 等. 麻疯树磷酸烯酮式丙酮酸羧化酶 pepc基因全长cDNA克隆及序列分析. 林业科学研究, 2010, 23(3): 349−354.
[42] Pan YM, Liu FZ, Wan YS, et al. Cloning of peanut gene fragment encoding PEPCase and construction of its plant expression vector. J Shandong Agri Univ: Nat Sci, 2010, 41(1): 1−5.潘昱名, 刘风珍, 万勇善, 等. 花生磷酸烯醇式丙酮酸羧化酶基因的克隆及反义表达载体的构建. 山东农业大学学报: 自然科学版, 2010, 41(1): 1−5.
[43] Li CL, Wu KQ, Fu GH, et al. Regulation of oleosin expression in developing peanut (Arachis hypogaea L.) embryos through nucleosome loss and histone modifications. J Exp Bot, 2009, 60(15): 4371−4382.
[44] Tripodi KE, Turner WL, Gennidakis S, et al. In vivo regulatory phosphorylation of novel phosphoenolpyruvate carboxylase isoforms in endosperm of developing castor oil seeds. Plant Physiol, 2005, 139(2): 969−978.
[45] Golombek S, Heim U, Horstmann C, et al. Phosphoenolpyruvate carboxylase in developing seeds of Vicia faba L.: gene expression and metabolic regulation. Planta, 1999, 208(1): 66−72.
[46] Golombek S, Rolletschek H, Wobus U, et al. Control of storage protein accumulation during legume seed development. J Plant Physiol, 2001, 158(4): 457−464.
[47] Zhang ZQ, Wang JM, Wang XJ, et al. The characteristics of PEPCase activity and accumulation of oil, protein and major protein subunits during seed development of rape (Brassica napus). Chin J Oil Crop Sci, 2009, 31(1): 14−18.张占琴, 王金梅, 王学军, 等. 油菜籽粒发育过程中PEPCase活性与油脂、蛋白及亚基积累的特点. 中国油料作物学报, 2009, 31(1): 14−18.
[48] Weber H, Borisjuk L, Wobus U. Molecular physiology of legume seed development. Annu Rev Plant Biol, 2005, 56: 253−279.
[49] Ettema TJG, Makarova KS, Jellema GL, et al. Identification and functional verification of archaeal-type phosphoenolpyruvate carboxylase, a missing link in archaeal central carbohydrate metabolism. J Bacteriol, 2004, 186(22): 7754−7762.
[50] Lin H, San KY, Bennett GN. Effect of Sorghum vulgare phosphoenolpyruvate carboxylase and Lactococcus lactis pyruvate carboxylase coexpression on succinate production in mutant strains of Escherichia coli. Appl Microbiol Biotechnol, 2005, 67(4): 515−523.
[51] Hou LJ, Shi DJ, Cai ZF, et al. Regulation of lipids synthesis in transgenic Escherichia coli by inserting cyanobacterial sense and antisense pepcA gene. China Biotechnol, 2008, 28(5): 52−58.侯李君, 施定基, 蔡泽富, 等. 蓝藻正反 pepcA基因导入对大肠杆菌中脂类合成的调控. 中国生物工程杂志, 2008, 28(5): 52−58.
[52] Song DH, Hou LJ, Shi DJ. Exploitation and utilization of rich lipids-microalgae, as new lipids feedstock for biodiesel production. Chin J Biotech, 2008, 24(3): 341−348.宋东辉, 侯李君, 施定基. 生物柴油原料资源高油脂微藻的开发利用. 生物工程学报, 2008, 24(3): 341−348.
