圆弧青霉脂肪酶基因序列的生物信息学分析
2010-08-27张慧敏李剑芳邬敏辰
张慧敏, 李剑芳, 邬敏辰
(1.江南大学食品学院,江苏无锡 214122;2.江南大学医药学院,江苏无锡 214122)
圆弧青霉脂肪酶基因序列的生物信息学分析
张慧敏1, 李剑芳1, 邬敏辰*2
(1.江南大学食品学院,江苏无锡 214122;2.江南大学医药学院,江苏无锡 214122)
从圆弧青霉PG37中克隆了碱性脂肪酶(Lip I)基因,并采用生物信息学分析了PG37 Lip I基因携带的遗传信息。序列分析结果表明:Lip I DNA序列全长1 480 bp,不存在重复序列,含有5个内含子,5’端存在TATA box和多个转录因子结合位点;cDNA序列全长1 128 bp,包含了转录起始位点、5’和3’非编码区及858 bp的开放阅读框架;Lip I编码区对TGC、GAC、TTC、CAC、AAG、AAC和TAC这7种密码子使用频率最高;比对Lip I基因与Pichia pastoris基因组中的密码子使用频率,其中有21个密码子使用频率的比值相差较大。
生物信息学;圆弧青霉PG37;碱性脂肪酶;基因序列
脂肪酶(Lipase EC 3.1.1.3)是一类分解三脂酰甘油的水解酶,目前研究最多的是来源于真菌和细菌的脂肪酶,已广泛应用于洗涤剂、食品、造纸、香料、医药、纺织、废物处理、化妆品、有机合成和生物柴油等行业,碱性脂肪酶主要用作洗涤剂添加剂[1-2]。1988年,NoVo Industrial A/B公司最先推出了商品化的重组碱性脂肪酶Liporase,并将其应用于洗涤剂行业。1985年以来,作者所在研究室一直致力于微生物碱性脂肪酶的研究,主要集中在圆弧青霉PG37菌株的诱变育种、发酵工艺条件优化、酶的分离纯化、酶学性质、酶的应用以及酶基因的克隆和表达等方面[3-4]。
鉴于上述的研究结果与基础,有必要进一步了解圆弧青霉PG37碱性脂肪酶(Lip I)基因携带的遗传信息。作者拟采用生物信息学的相关分析软件和数据库,分析、预测PG37 Lip I基因的各种功能单位,例如重复序列、转录起始位点、内含子、转录因子结合位点[5]和开放阅读框架等,从而加深对基因结构、表达和调控机制的认识;同时分析和计算PG37 Lip I编码区密码子使用频率,并与毕赤酵母(Pichia pastoris)基因组密码子使用频率进行比对,为Lip I基因在Pichia pastoris中的高效表达打下基础。有关圆弧青霉PG37 Lip I基因序列的生物信息学分析,国内外尚未见报道。
1 材料与方法
1.1 菌株和质粒
圆弧青霉PG37:为Lip I基因的供体菌,作者所在研究室保藏;大肠杆菌XL1-Blue:用作基因克隆、质粒转化的受体菌;pUCm-T质粒,氨苄青霉素抗性,购自上海生工公司。
1.2 培养基
1.2.1 圆弧青霉培养基 大豆磷脂7.5 g/L,玉米浆30 g/L,豆油4 g/L,豆饼水解液400 g/L,柠檬酸三钠0.5 g/L,K2HPO410 g/L,MgSO41.0 g/L, p H 7.5。
1.2.2 L-Broth培养基 蛋白胨10 g/L,NaCl 10 g/L,酵母粉5 g/L,p H 7.4。对于固体平板LB培养基,在L-Broth的基础上添加15 g/L的琼脂粉。
1.3 试剂、试剂盒和PCR引物
参见文献[3,4]。
1.4 PG37 Lip I基因的克隆
参照Sambrook等[6]的《分子克隆实验指南》一书的方法进行。
1.5 PG37 Lip I基因的生物信息学分析
采用生物信息学相关分析软件及数据库对PG37 Lip I基因序列进行分析、预测,见表1。

表1 生物信息学相关分析软件与数据库网址Tab.1 Website of bioinformatics software and database
1.5.1 PG37 Lip I基因序列分析 采用CENSOR软件分析DNA序列中的重复序列;采用NNPP预测基因转录起始位点;采用GeneMark分析内含子/外显子剪接位点;采用TFSEARCH和PLACE预测转录因子结合位点;采用ORF Finder确定开放阅读框架。
1.5.2 有效密码子数目(ENc)统计 ENc值是指密码子使用频率与同义密码子平均使用频率偏差的量化值,其取值范围为20~61,ENc值越接近20,则密码子偏爱性越强,反之则偏爱性越低。采用CHIPS软件统计PG37 Lip I编码区的ENc值。1.5.3 密码子使用频率(FCU)统计 FCU表示在基因组或基因的所有密码子中某种密码子出现的频率,特指在1 000个密码子中某种密码子被使用的次数。采用CUSP软件统计PG37 Lip I编码区的密码子使用频率FCU。
1.5.4 同义密码子使用的相对概率(RSCU)统计RSCU是指某种密码子在编码对应氨基酸的同义密码子间的相对概率。当RSCU值大于1.0,表示该密码子使用频率相对较高;反之则使用频率相对较低[13]。在同义密码子中若某一密码子RSCU值大于1,而其余密码子RSCU值均小于1,则前者定义为使用频率最高的密码子[14]。采用CodonW软件统计PG37 Lip I编码区的RSCU值。
1.5.5 密码子使用频率比值 当某种密码子FCU比值在0.5~2.0范围内,表示该密码子在不同物种或基因中的FCU较接近;FCU比值超出此范围则FCU相差较大。
2 结果与讨论
2
.1 PG37 Lip I基因克隆
在测定了PG37完整Lip I及其胰蛋白酶酶解肽段N末端部分氨基酸序列的基础上,分别提取圆弧青霉PG37总RNA和基因组DNA作为模板,采用RT-PCR、嵌合式PCR和单侧PCR等技术,分多次扩增和克隆了Lip I基因的相关片段,通过拼接获得了Lip I基因完整的DNA序列,见图1。

图1 圆弧青霉PG37 Lip I基因完整的DNA序列Fig.1 Entire DNA sequence of Lip I gene fromPenicillium cyclopiumPG37
2.2 PG37 Lip I基因重复序列分析
以PG37 Lip I基因完整DNA序列为研究对象,采用CENSOR软件对该序列进行了分析。分析结果表明,该DNA序列中不存在重复序列。
2.3 PG37 Lip I基因转录起始位点预测
采用NNPP软件预测了PG37 Lip I完整DNA序列的转录起始位点。预测结果显示,基因转录起始位点是位于该DNA序列第71位的腺苷酸(AMP),与邬敏辰等的实验结果[15]存在差异,后者是位于DNA序列第92位的鸟苷酸(GMP)。
2.4 PG37 Lip I基因内含子剪接位点分析
GeneMark软件分析结果表明,PG37 Lip I完整DNA序列中含有5个内含子,其5′端剪切位点均为GT,3′端剪切位点均为AG,见表2,符合GTAG法则。GeneMark预测的内含子数目及内含子/外显子剪接位点与邬敏辰的实验结果[3]完全一致。
在Lip I完整DNA序列中剔除转录起始位点AMP的上游序列及5个内含子序列,可获得理论上的Lip I完整cDNA序列,全长1 128 bp,与实验获得的Lip I完整cDNA序列长度(GenBank登录号AF274320)相差21 bp,原因是两者的转录起始位点不同。
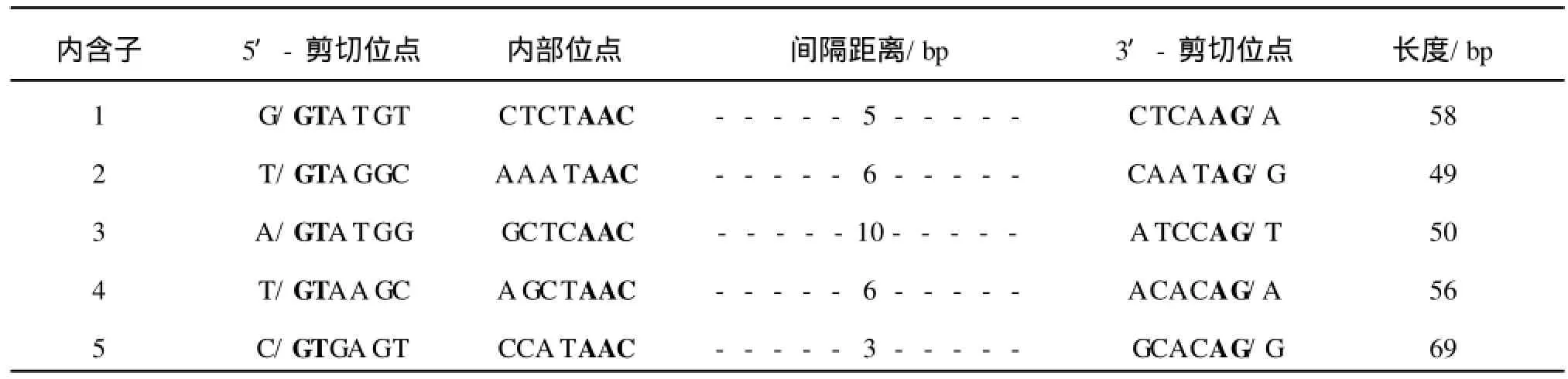
表2 PG37 Lip I基因内含子结构与剪接位点的分析Tab.2 Analysis on the structure and splice sites of introns in Lip I gene from PG37
2.5 PG37 Lip I基因转录因子结合位点预测
采用TFSEARCH软件对PG37 Lip I DNA的5’端序列(从转录起始位点上游的-70 bp位至下游的+95 bp位)进行了转录因子结合位点扫描分析,见图2。扫描结果显示:检测到预测分值在85分以上的有44个转录因子结合位点;分值在90分以上的有23个转录因子结合位点。其中HSF (heat shock factor)9个,其存在表明基因能在较高温度下诱导表达[16];ADR1(alcohol dehydrogenase gene regulator 1)3个,它可调节利用碳源基因的表达,也可与过氧化物酶A等基因的启动子结合[17]; CdxA(caudal type homeobox transcription factor A)3个,它作为启动子的一部分,在基因调控区能增强基因转录的活性[18];NIT2(activator of nitrogen-regulated genes)2个;SRY(sex-determining region Y gene product)2个;cap(cap signal for transcription initiation)1个,它的存在与基因的转录起始有关;GATA-1(GATA-binding factor 1)1个,它通过疏水作用来识别DNA结合位点,可调节氮代谢、光诱导等,还可以提高启动子的强度和活性;SREBP和Sox-5各1个。由于实验手段的限制,生物信息学已成为分析基因转录因子结合位点的重要工具。
此外,在转录起始位点上游的-16~-11 bp处发现存在TATA box,预测分值为85.6,与采用PLACE软件预测的位置相同。
2.6 PG37 Lip I cDNA阅读框架分析
以全长为1 128 bp的PG37 Lip I cDNA序列为研究对象,采用ORF Finder程序对该cDNA进行了分析。分析统计结果表明,Lip I cDNA序列中最长的一个开放阅读框架为858 bp,从95 bp位至952 bp位,起始ATG,终止密码子TGA,编码285个氨基酸,与邬敏辰的实验结果[15]完全一致。
2.7 PG37 Lip I编码区密码子统计分析
采用CHIPS程序计算结果表明,PG37 Lip I

图2 PG37 Lip I DNA序列转录因子结合位点预测Fig.2 Prediction of the transcription factor binding sites in Lip I DNA sequence
编码区有效密码子数目ENc值为48.32,说明Lip I编码区不存在特别明显的密码子偏爱性。采用CUPS、CodonW程序及密码子数据库对Lip I编码区密码子进行了统计分析,见表3。统计结果表明: GCC、GCT、TGC和GAC等28个密码子的RSCU值大于1,且FCU也较大,为Lip I使用频繁的密码子,其中TGC、GAC、TTC、CAC、AAG、AAC和TAC这7种密码子使用频率最高;在Lip I基因与Pichia pastoris基因组中,有21个密码子的使用频率比值相差较大。通过不同物种或基因间密码子使用频率比对,可择优选择表达系统或改变密码子,从而提高外源基因的表达。若某一密码子在Lip I基因中使用频率较高,而在Pichia pastoris基因组中使用频率较低,可以把此密码子突变为Pichia pastoris基因组中使用频率较高的同义密码子,如将密码子CAC突变为CAT,以提高Lip I组氨酸在Pichia pastoris中的表达量。但外源基因的表达受到多种因素的共同作用,如表达的蛋白是否是毒素、需要糖基化或其它修饰等,密码子使用的影响只是其中之一。

表3 Lip I密码子统计分析及与Pichia pastoris密码子比对Tab.3 Statistics on codon of Lip I and comparison with codon ofPichia pastoris

续表3
3 结 语
1)分多次PCR扩增和克隆了PG37 Lip I基因的相关片段,并通过拼接获得了Lip I基因完整的DNA序列。该DNA序列全长1 480 bp,无重复序列,转录起始位点位于序列的第71位,含有5个内含子,5′端存在TA TA box和多个转录因子结合位点。
2)由PG37 Lip I DNA序列推导的cDNA序列全长1 128 bp,包含了转录起始位点、5′和3′非编码区和开放阅读框架,后者编码285个氨基酸。
3)PG37 Lip I编码区的ENc值为48.32,说明Lip I编码区不存在特别明显的密码子偏爱性;Lip I编码区的GCC、GCT、TGC和GAC等28个密码子的RSCU值大于1,FCU也较大,为Lip I使用频繁的密码子,其中TGC、GAC、TTC、CAC、AAG、AAC和TAC这7种密码子使用频率最高。比对Lip I基因与Pichia pastoris基因组中的密码子使用频率,其中有21个密码子使用频率的比值相差较大。
[1]Hasan F,Shah A A,Hameed A.Industrial applications of microbial lipases[J].Enzyme and Microbial Technology, 2006,39:235-251.
[2]王蕾,蔡宇杰,廖祥儒,等.一株碱性低温脂肪酶产生菌发酵条件的优化[J].食品与生物技术学报,2008,27:114-119.
WANG Lei,CAI Yu-jie,LIAO Xiang-ru,et al.Optimization of fermentation conditions for a low-temperature lipase-producing strain[J].Journal of Food Science and Biotechnology,2008,27:114-119.(in Chinese)
[3]邬敏辰.圆弧青霉PG37碱性脂肪酶的发酵生产、分离纯化及基因克隆和表达[D].上海:复旦大学,2000.
[4]邓珊珊,邬敏辰,李江华,等.圆弧青霉BD26碱性脂肪酶基因的克隆及其在大肠杆菌中的表达[J].食品与发酵工业, 2008,34:42-46.
DENG Shan-shan,WU Min-chen,LIJiang-hua,et al.Cloning and expression of alkaline lipase gene fromPenicillium cyclopiummutant inEscherichia coli[J].Food and Fermentation Industries,2008,34:42-46.(in Chinese)
[5]侯琳,钱敏平,朱云平,等.转录因子结合位点生物信息学研究进展[J].遗传,2009,31:365-373.
HOU Lin,QIAN Min-ping,ZHU Yun-ping,et al.Advances on bioinformatic research in transcription factor binding sites[J].Hereditas,2009,31:365-373.(in Chinese)
[6]Sambrook J,Fritsch E F,Maniatis T.Molecular Cloning:A Laboratory Manual[M].New York:Cold Spring Harbor Laboratory Press,1989.
[7]Kohany O,Gentles A J,Hankus L,et al.Annotation,submission and of screening repetitive elements in repbase:repbase submitter and censor[J].BMC Bioinformatics,2006,25:474.
[8]Lomsadze A,Ter-Hovhannisyan V,Chernoff Y,et al.Gene identification in novel eukaryotic genomes by self-training algorithm[J].Nucleic Acids Research,2005,33:6494-6506.
[9]Heinemeyer T,Wingender E,Reuter I,et al.Databases on transcriptional Regulation:TRANSFAC,TRRD,and COMPEL[J].Nucleic Acids Research,1998,26:364-370.
[10]Higo K,Ugawa Y,Iwamoto M,et al.Plant cis-acting regulatory DNA elements(PLACE)database[J].Nucleic Acids Research,1999,27:297-300.
[11]Nakamura Y,Gojobori T,Ikemura T.Condon usuage tabulate form the international DNA sequence databases:status for the year 2000[J].Nucl Acids Research,2000,28:292-295.
[12]Wright F.The effective number of codons used in a gene[J].G ene,1990,87:23-29.
[13]赵耀,刘汉梅,顾勇,等.玉米waxy基因密码子偏好性分析[J].玉米科学,2008,16:16-21.
ZHAO Yao,LIU Han-mei,GU Yong,et al.Analysis of characteristic of codon usage in waxy gene ofZea mays[J]. Journal of Maize Sciences,2008,16:16-21.(in Chinese)
[14]沈庆航,江明锋,钟金城,等.MUC1基因遗传多态性与密码子偏好性的生物信息学分析[J].西南民族大学学报:自然科学版,2009,35:513-518.
SHEN Qing-hang,J IANG Ming-feng,ZHONGJin-cheng,et al.The bioinformatics analysis of genetic polymorphism and code bias of MUC1 gene[J].Journal of Southwest University for Nationalities:Natural Science Edition,2009,35:513-518.(in Chinese)
[15]邬敏辰,朱劼,黄伟达,等.圆弧青霉脂肪酶基因的克隆和表达[J].无锡轻工大学学报,2002,21:218-223.
WU Min-chen,ZHU Jie,HUANG Wei-da,et al.Cloning and expression of the gene encoding alkaline lipase fromPenicillium cyclopium[J].Journal of Wuxi University of Light Industry,2002,21:218-223.(in Chinese)
[16]马志杰,魏雅萍,钟金城,等.藏绵羊GHR基因5’侧翼区序列特征分析[J].遗传,2007,29:963-971.
MA Zhi-jie,WEI Ya-ping,ZHONGJin-cheng,et al.Sequence characterization of the 5’-flanking region of the GHR gene in Tibetan sheep[J].Hereditas,2007,29:963-971.(in Chinese)
[17]Schaufler L E,Klevit R E.Mechanism of DNA binding by the ADR1 zinc finger transcription factor as determined by SPRJ[J].J Mol Biol,2003,329:931-939.
[18]周颖君,杨亦桦,武淑文,等.棉铃虫细胞色素P450 CYP9A12基因5′-上游区的克隆及序列分析[J].昆虫学报,2008, 51:120-125.
ZHOU Ying-jun,YANG Yi-hua,WU Shu-wen,et al.Cloning and sequence analysis of the 5’-upstream region of cytochrome P450 gene CYP9A12 ofHelicoverpa armigera[J].Acta Entomologica Sinica,2008,51:120-125.(in Chinese)
(责任编辑:李春丽)
Bioinformatics Analysis on the Sequence of Lipase Gene fromPenicillium cyclopium
ZHANG Hui-min1, LI Jian-fang1, WU Min-chen*2
(1.School of Food Science and Technology,Jiangnan University,Wuxi 214122,China;2.School of Medicine and Pharmaceutics,Jiangnan University,Wuxi 2141224,China)
The gene encoding alkaline lipase(Lip I)is cloned fromPenicillium cyclopiumPG37 and the genetic information is analyzed by bioinformatic tool.Sequence analysis showed that Lip I DNA is 1480 bp long with no repeat sequence and five introns,and there is a TA TA box and several transcription factor binding sites in the 5’end.Lip I cDNA is 1128 bp long with an open reading frame containing 858 bp.It also has transcription start site,5’and 3’non-coding region. TGC,GAC,TTC,CAC,AAG,AAC and TAC codons are used at highest frequency in Lip I gene.Comparing codon usage frequency of the Lip I withPichia pastorisgenome’showed that the ratios of 21 codon usage frequency are different widely.
bioinformatics,Penicillium cyclopiumPG37,alkaline lipase,gene sequence
Q 811.4
:A
1673-1689(2010)04-0602-07
2009-09-15
国家自然科学基金项目(20776061)。
*通信作者:邬敏辰(1962-),男,江苏无锡人,理学博士,教授,博士生导师,主要从事酶工程与基因工程方面的研究。Email:bioch@163.com
