RAPD和SRAP分子标记在绣球菌菌种鉴定中的应用*
2010-08-08陈瑞鹏贾小宁郭立忠
陈瑞鹏,贾小宁,郭立忠
(青岛农业大学生命科学学院山东省应用真菌重点实验室,山东 青岛 266109)
绣球菌 (Sparassis crispa)属于非褶孔菌目、绣球菌科、绣球菌属[1],称绣球覃。子实体中等至大形,肉质,形似巨大的绣球,直径10 cm~40 cm,白色至污白或污黄色。孢子无色,光滑,卵圆形至球形,4 μm~5 μm[2]。绣球菌是珍稀名贵的食药兼用菌,营养特别丰富,在西欧各国极为畅销,价格昂贵[3]。绣球菌可提高人体免疫功能,治疗心脑血管、糖尿病、高血压等疾病,长期食用可降低胆固醇、血压,能清除面部色斑、色素、雀斑、黄斑及嫩肤美容功效[4]。
随机扩增多态性DNA(RAPD)标记技术和相关序列扩增多态性(SRAP)分子标记技术已用于食用菌的菌种鉴定、遗传育种等研究中。随机扩增多态性DNA技术成功对香菇菌株[5]、灵芝[6]、绿僵菌[7]、蜜环菌[8]等进行了遗传多样性分析、鉴定和分类。相关序列扩增多态性分子标记也在银耳菌[9]、荆半夏[10]、毛木耳[11]等菌株的遗传多样性分析中得到广泛应用。
本实验利用RAPD、SRAP分子标记技术对6个绣球菌菌株进行亲缘关系分析,对绣球菌遗传育种工作有重要的实用价值。
1 实验材料
供试菌株来源及编号情况见表1。

表1 绣球菌供试菌株
2 实验方法
2.1 DNA的提取与检测
选用新鲜菌丝体作为DNA提取的材料,0.1 g菌丝体于1.5 mL离心管中,液氮研磨。按改良后的CTAB法[12]进行DNA的提取,0.8%琼脂糖凝胶电泳检测提取的基因组DNA的质量,紫外分光光度计检测DNA的浓度,于-20℃保存备用。
2.2 RAPD反应引物筛选
64对引物由上海生物工程公司合成,引物名称为S3~S6,S8~S21,S23~S28,S31,S32,S34~S42,S44~S50,S120~S130,S380~S384,S386,S388,S390。
64条引物分别对1号、3号、4号三个绣球菌菌株基因组DNA进行扩增,筛选合适的引物组合用于遗传多样性分析。
PCR反应体积 16 μL,各成分终浓度为:1×PCR Bufer,200 μmol·L-1dNTPs,引物各 0.4 μmol·L-1,0.625U TaqDNA聚合酶,5 ng DNA模板,最后加双蒸水补齐。扩增程序为:94℃预变性 3 min后,94℃ 30 s,35℃ 30 s,72℃ 3 min,32个循环后,72℃延伸10 min。
电泳和检测统计:2%琼脂糖凝胶电泳,恒压70 V,电泳1 h,于凝胶成像系统检测电泳结果。
2.3 SRAP反应引物筛选
应用8条上游引物和8条下游引物合成64对引物,对1号、3号、4号3个绣球菌菌株的DNA进行扩增,筛选出条带多、多态性较好的引物组合用于遗传多样性分析,RAPD引物序列编号及核酸序列情况见表2。
PCR反应体积 16 μL,各成分终浓度为:1×PCR Bufer,200 μmol·L-1dNTPs,引物各 0.4 μmol·L-1,0.625U TaqDNA聚合酶,5 ng DNA模板,最后加双蒸水补齐。循环体系采用复性变温法:94℃预变性5 min,前5个循环(94℃ 1 min,35℃ 1 min,72℃ 1 min),后 35个循环复性温度提高到50℃,最后72℃延伸10 min。
2.4 亲缘关系鉴定
用筛选出的RAPD、SRAP引物对6个绣球菌菌株的基因组DNA进行扩增。
2.5 数据处理分析
用NTSYS-pc(2.02a)软件计算相似性系数,得相似性系数矩阵,用UPGMA法进行聚类分析,生成聚类图。
3 结果与分析
3.1 DNA提取结果
CTAB法提取DNA的检测结果如图1所示。

图1 菌株基因组DNA的琼脂糖凝胶电泳图谱
从图1可以看出,试验得到了清晰的单一总DNA条带,能为RAPD、SRAP分子标记技术的PCR扩增提供可靠模板。
3.2 RAPD多态性分析
用筛选的10条随机引物(S8、S10、S23、S25、S31、S32、S38、S40、S120、S380)对6个菌株的基因组DNA进行扩增,共扩增出257个条带,既有共有带又有特征带,其中多态性条带为245条,多态性比率为91.4%,每个引物平均获得24.5条多态性条带。DNA片段分子量大小在250 bp~3000 bp之间。引物S23、引物S32对供试菌株基因组DNA的扩增图谱见图2。

图2 引物S23、引物S32对供试菌株基因组DNA的扩增图谱
3.3 聚类分析
使用NTSYS(W)202软件对6个菌株的相似性进行聚类分析,结果见图3。
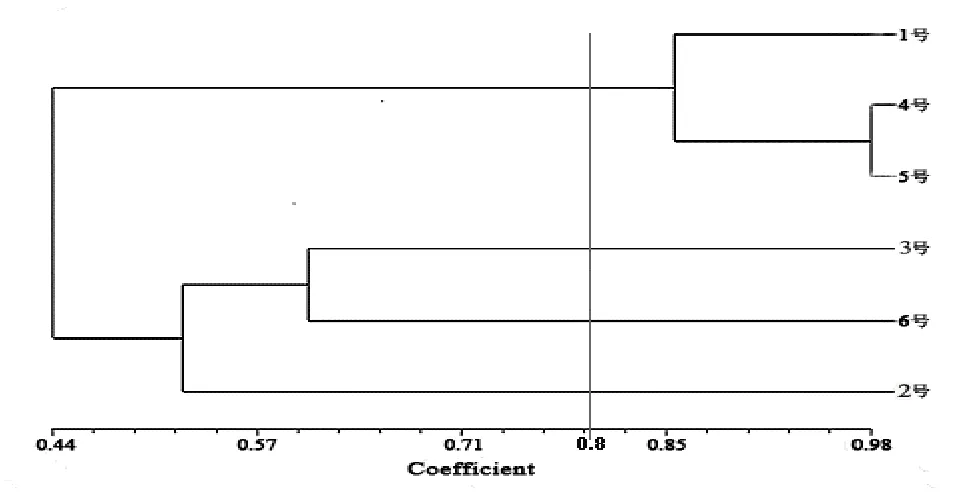
图3 绣球菌菌株的RAPD聚类分析图
供试的6个绣球菌菌株经RAPD分析表明,两两间的相似系数范围在0.439~0.981之间,说明其在遗传上相似程度不同,存在一定的亲缘关系差异。其中菌株4和菌株5的相似性系数最大为0.981,菌株2和菌株4、菌株5的相似性系数为所有菌株中较小的为0.393,菌株2和菌株1相似系数最小为0.383。由图3可知,相似性系数0.800时,6个菌株聚成4组:1号、4号、5号为1组,3号、6号、2号分别为1组。2号与其他4株亲缘关系最远。
3.4 SRAP多态性分析
筛选出的 12对引物(Me6-Em2、Me3-Em3、Me5-Em4、 Me6-Em7、 Me6-Em1、 Me3-Em8、 Me5-Em1、 Me8-Em3、 Me7-Em4、 Me3-Em5、 Me3-Em1、 Me4-Em2),扩增6个供试菌株的基因组DNA,共扩增出312条DNA条带。大部分条带分子量在200 bp~3000 bp之间,多态性条带为246条,占总条带数的78.8%,每对引物平均获得20.5条多态性条带。部分引物的扩增结果见图4。
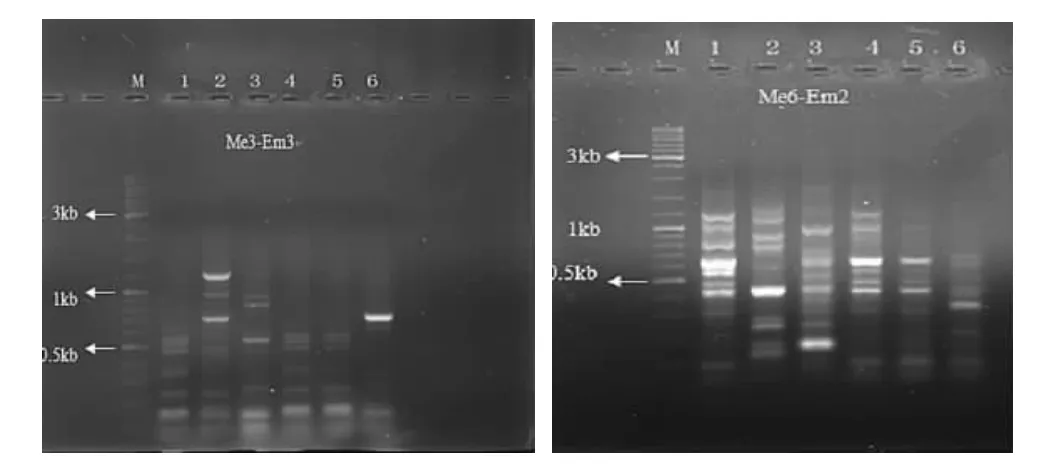
图4 引物Me3-Em3、引物Me6-Em2对绣球菌菌株DNA的扩增图谱
3.5 SRAP聚类分析
使用NTSYS软件对6个菌株的相似性进行聚类分析,分析结果见图5。

图5 绣球菌菌株的SRAP聚类分析图
供试的6个绣球菌菌株SRAP分析表明,两两间的遗传相似系数范围0.411~0.875,表明菌株间丰富的遗传多样性,其中4号和5号的相似系数最大(0.875),亲缘关系最近,2号和4号间的相似系数最小(0.411)。由图5可看出,当相似系数为0.700时,可将6个菌株聚成4组,1号、4号、5号为1组,3号、6号、2号分别为1组。
4 讨论
本研究通过SRAP分子标记技术对绣球菌菌种进行分类鉴定,在相似系数为0.600时,SRAP标记可将6个供试菌株分为3组。在相似系数阈值为0.700时,SRAP标记可将6个供试菌株分为4组。SRAP标记在较低的相似系数水平上,就可将两亲缘关系较近菌株区分开来,所反映的遗传信息丰富,分辨率高且重复性较好。
研究表明[11],RAPD可以快速、灵敏、准确地用于食用菌菌种的检测和鉴定。本研究对64条随机引物进行筛选,获得10个具有多态性的DNA指纹图谱,被测试的6个绣球菌菌种之间均有遗传上的差异,且综合分析,能将所有菌株区分开来。
通过对2种分子标记技术的结果分析,RAPD与SRAP分子标记技术在本实验中分别在以相似系数0.8000和0.7000为阈值时,6个供试菌株的分组是相同的,都分为4组。4号、5号同源性最大,在2种分子标记技术中相似系数分别达到了0.9813和0.8750,表明4号、5号可能是同物异名,而2号与4号相似系数很低,与其他菌株的亲缘关系最远,2种技术结果的一致性,证明2种方法能很好地用于绣球菌菌种间亲缘关系的鉴定。
由于缺乏规范的食用菌品种登记制度,绣球菌菌种同名异物、同物异名的现象普遍存在,这不仅损害了育种工作者和生产者的利益,同时也加速了菌种的退化,严重阻碍了产业的健康发展。本试验采用RAPD、SRAP方法筛选出的部分引物能有效地将6个供试菌株区分开来,从而为绣球菌菌种鉴定提供了依据,但对来源不同的菌种进行分析时,仅依据一种方法的结果具有不确定性,如果再结合子实体形态特征和其他的分子检测手段进行分析,结果将会更准确、更科学。
[1]薛道帆,周立平.绣球菌的研究开发概况[J].杭州农业科技,2008,16(3):27-29.
[2]赖日辉.绣球菌栽培技术初报[J].浙江食用菌,2007,17(2):47-48.
[3]Petrova RD,Wasser SP,Mahajna JA,et al.Potential role of medicinal mushrooms in breast cancer treatment:current knowledge and future perspectives[J].International Journal of Medicinal Mushrooms,2005,7(1):141-155.
[4]朱斗锡,何荣华.珍稀食用菌的经济价值及开发前景[J].资源开发,2009(2):19-20.
[5]龚利娟,李玉,刘淑艳.香菇品种遗传多样性RAPD分子标记的研究[J].菌物研究,2005,23(1):17-21.
[6]许晓燕,余梦瑶,罗霞,等.利用AFLP和SRAP标记分析19株毛木耳的遗传多样性[J].西南农业学报,2008,121(1):121-124.
[7]秦国夫,贺伟,沈瑞祥.中国蜜环菌生物钟的RAPD分析[J].真菌学报,1996,15(1):26-33.
[8]王淑珍,白晨.灵芝与糙皮侧耳原生质体融合子基因组RAPD分析[J].食用菌学报,2003,10(1):1-5.
[9]曲绍轩,高山.SRAP、ISSR和RAPD分子标记技术在银耳菌株鉴别上的应用[J].食用菌学报,2007,14(3):1-5.
[10]邓瑞宁,王沫.荆半夏遗传多样性的SRAP分析[J].农业生物技术学报,2008,16(6):1070-1071.
[11]施庆利,罗信昌.RAPD技术在木耳杂交种及原生质体融合子鉴定分析中的应用[J].中国食用菌,1994,12(6):324.
[12]Porebski S,Bailey LG,Baum BR.Modification of a CTAB DNA extraction protocol for plants containing high polysaccharide and polyphenol components[J].Plant Molecular Biology Reporter,1997,15(1):8-15
