基于术前MRI图像构建影像组学与深度学习的机器学习模型预测胶质瘤IDH1基因表达的研究
2024-05-11胡哲王玉红刘晓龙于昊王皆欢刘德国王唯伟陈月芹
胡哲 王玉红 刘晓龙 于昊 王皆欢 刘德国 王唯伟 陈月芹


【摘要】目的探讨基于术前核磁共振成像(MRI)T2抑脂序列预测胶质瘤异柠檬酸脱氢酶(IDH)1基因表达情况的价值。方法本研究共纳入2016年1月—2023年2月在济宁医学院附属医院经组织病理学证实的124例胶质瘤患者。使用ITKSNAP软件勾画感兴趣区域(ROI),使用Pyradiomics包实现影像组学特征的提取,使用经过预训练的ResNet50深度学习模型提取深度学习特征。使用Pearson相关系数和最小绝对收缩和选择算子(LASSO)回归模型进行特征筛选。最后进行10折交叉验证来评估模型效能。将传统影像组学、深度迁移学习以及融合模型基于支持向量机(SVM)、K近邻(KNN)以及随机森林(RF)三种机器学习模型分别建模。使用受試者工作特征(ROC)曲线评价各模型的预测效能。结果基于影像组学特征的机器学习模型SVM、KNN以及RF的曲线下面积(AUC)分别为0.699、0.628、0.616。基于深度迁移学习特征的机器学习模型SVM、KNN以及RF的AUC分别为0.853、0.753、0.807。基于融合特征的机器学习模型SVM、KNN以及RF的AUC分别为0.868、0.818、0.787。结论基于常规MRI序列中的T2WI抑脂序列的SVM融合模型对预测胶质瘤IDH1基因表达情况具有较高的预测效能。
【关键词】胶质瘤;机器学习;深度学习;磁共振成像;影像组学
【中图分类号】R739.41【文献标志码】A【文章编号】16727770(2024)02018706
Study on predicting IDH1 gene expression in gliomas using machine learning models based on imagomics and deep learning based on preoperative MRI images HU Zhe, WANG Yuhong, LIU Xiaolong, et al. Clinical Medical College, Jining Medical University, Jining 272013, China
Corresponding author: CHEN Yueqin
Abstract: ObjectiveTo investigate value of preoperative magnetic resonance imaging(MRI) T2 fat suppression sequence in predicting isocitrate dehydrogenase(IDH)1 gene expression in gliomas. Methods124 patients with glioma, who were confirmed by histopathology, were collected. Regions of interest(ROI) was delineated using ITKSNAP software. Pyradiomics package was used to extract radiomic features from the imaging data, while a pretrained ResNet50 deep learning model was employed to extract deep learning features. Feature selection was performed using the Pearson correlation coefficient and the Least Absolute Shrinkage and Selection Operator(LASSO) regression model. Model performance was evaluated through 10fold crossvalidation. Traditional radiomics, deep transfer learning, and fusion models were separately constructed based on support vector machine(SVM), knearest neighbors(KNN), and random forest(RF) machine learning algorithms. The predictive performance of each model was assessed using receiver operating characteristic(ROC) curve. ResultsThe area under curve(AUC) for the machine learning models SVM, KNN, and RF based on radiomic features were 0.699, 0.628, and 0.616, respectively. For the machine learning models SVM, KNN, and RF based on deep transfer learning features, the
AUC values were 0.853, 0.753, and 0.807, respectively. The machine learning models SVM, KNN, and RF based on fusion features achieved AUC values of 0.868, 0.818, and 0.787, respectively. ConclusionThe SVM fusion model based on the T2WI fat suppression sequence in routine MRI exhibits higher predictive performance in determining the IDH1 gene expression status in gliomas.
Key words: glioma; machine learning; deep learning; magnetic resonance imaging; radiomics
胶质瘤是成人颅内最常见的恶性原发性脑肿瘤,约占所有中枢神经系统肿瘤的23.3%[1]。胶质瘤恶性程度高,预后偏差。随着第五版《WHO中枢神经系统肿瘤分类指南》发布,使得中枢神经系统肿瘤的分级和诊断更加细致,指南进一步强调了分子基因特征在肿瘤分类和分级中的作用[2]。其中,异柠檬酸脱氢酶(isocitrate dehydrogenase,IDH)1基因作为目前评价分级和预后的关键基因,在临床中具有重要作用[34]。IDH1基因分为突变型和野生型,其中,IDH1突变型胶质瘤预后明显好于IDH野生型胶质瘤[5],并且IDH1突变型胶质瘤对于放、化疗更为敏感[67]。但是目前对于IDH1基因的检测主要依靠免疫组化,但是该方法只能依靠术后的病理组织进行检测。如果在术前可以对胶质瘤患者的IDH1基因分型进行预测,将会对于部分不适宜手术治疗的患者提供个性化的临床治疗方案,同时对于患者预后的判断也具有重要的临床价值。
随着人工智能(artificial intelligence,AI)的发展,基于AI技术的机器学习和深度学习算法模型逐渐被应用于医学影像领域[810]。AI擅长识别高通量的成像数据,并能以自动化的方式进行识别诊断,因此通过影像数据对患者的基因分型的进行无创性预测成为现实[1113]。本研究回顾性分析2016年1月—2023年2月济宁医学院附属医院收治的经组织病理学证实的124例胶质瘤患者,旨在探讨术前MRI图像构建影像组学与深度学习的机器学习模型预测胶质瘤IDH1基因表达的价值。现报告如下。
1资料与方法
1.1一般资料共纳入124例胶质瘤患者,其中男62例,女62例;年龄7~77岁,平均年龄51岁;IDH1阴性81例,阳性43例。纳入标准:(1)符合2021新版WHO中枢神经系统肿瘤诊断标准并经病理诊断证实的胶质瘤患者;(2)术前进行过颅脑磁共振检查的患者;(3)术后病理组织进行过免疫组化检测。排除标准:(1)IDH1分子信息缺失(0例);(2)术前MRI图像不包含T2抑脂序列或图像质量不佳(12例);(3)术前接受过放化疗治疗(0例)。本研究经济宁医学院附属医院伦理委员会批准,免除受试者知情同意(批准文号:202303C030)。
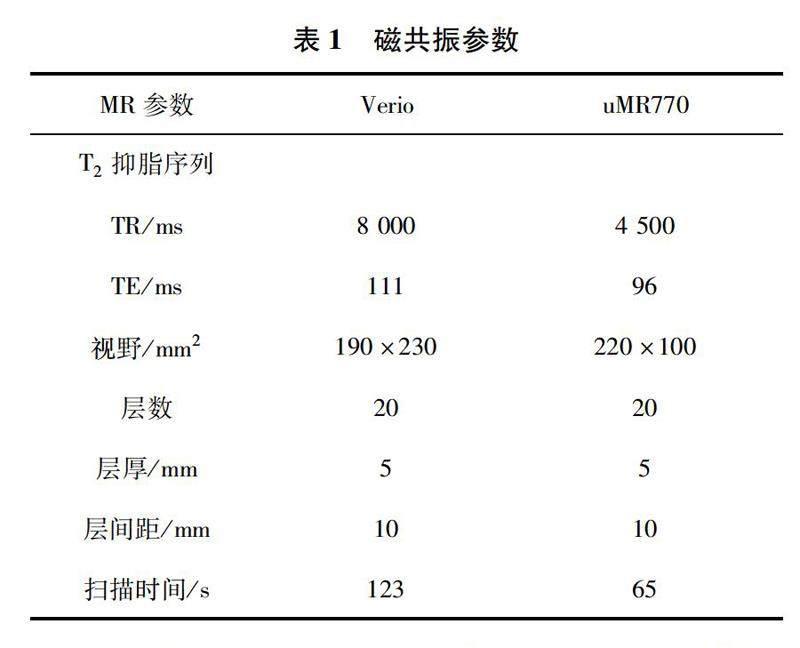
1.2MR扫描方法本研究使用两台磁共振进行扫描,分别是德国西门子MAGNNETOM Verio 3.0 T磁共振及联影uMR770 3.0 T磁共振,参数见表1。
1.3图像预处理本研究首先对磁共振T2抑脂序列进行N4偏置场校正,以消除校正序列的强度不均匀性。然后对图像进行重采样后体素大小为1 mm×1 mm×1 mm,用来减少不同机器采集时的差别。
1.4图像分割本研究采用ITKSNAP软件(http://www.itksnap.org/pmwiki/pmwiki.php)手動分割感兴趣区域(region of interest,ROI )。因为瘤周水肿区可能会存在肿瘤细胞浸润,所以整个ROI的标注包括肿瘤瘤体、周围水肿带、坏死区及囊变区。分别由两位拥有5年和7年影像诊断经验的专家进行双盲标注。由于深度学习模型输入的图像是包含完整ROI病灶的矩形图像,因此选择每个患者的肿瘤病灶的最大切面作为输入图像。磁共振T2抑脂序列用矩形ROI在肿瘤轮廓周围截取。然后将截取后的切片保存为“PNG”格式来进行后续分析(图1)。
1.5特征提取影像组学特征可以分为三类:(1)形态特征;(2)一阶直方图特征;(3)纹理特征。形态特征描述了肿瘤的三维形状特征。一阶直方图特征描述了与ROI内的体素强度分布有关的特征。纹理特征是描述体素空间分布强度等级的特征。本研究使用几种不同的方法提取纹理特征,包括灰度共生矩阵(GLCM)、灰度游程矩阵(GLRLM)、灰度级带矩阵(GLSZM)和邻域灰度差分矩阵(NGTDM)方法。使用Pyradiomics中的内部特征分析程序提取影像组学特征(http://pyradiomics.readthedocs.io)。
深度学习在图像处理和计算机视觉领域中具有广泛的应用。在深度学习中,提取特征是一个非常重要的步骤,因为它可以从原始图像数据中提取高维度的特征表示。深度学习特征通常采用迁移学习算法,预训练的模型参数可以在新的数据集上微调以提高性能。本研究使用的是ImageNet数据集上预训练的ResNet50模型作为基础模型,通过输入的切片图像来提取深度学习特征,这些图像将通过ResNet50模型,使用其已经训练好的特征提取器进行处理。选择avgpool层的深度学习特征作为输出特征,每个患者将获得2 048个深度学习特征,这些特征将用于训练和测试我们的机器学习模型。
1.6特征筛选首先进行影像组学特征在两位影像标注医师组间的一致性检验,一致性检验用组内相关系数(intraclass correlation coefficient,ICC)作为衡量指标。随机选择30例患者,由两名拥有五年影像诊断经验的医师独立勾画ROI并提取特征,评估观察者组间的一致性,仅保留ICC≥0.75的影像组学特征。
其次对第一步保留的影像组学特征进行特征筛选。对于重复性较高的特征,采用Pearson相关系数计算特征之间的相关性,保留任意两个特征之间相关系数大于0.9的特征之一。然后使用最小绝对收缩和选择算子(LASSO)回归模型,通过10折交叉验证的方法,进一步筛选组学特征。
1.7模型选择与模型构建所有数据资料按分层抽样8∶2划分训练集和测试集,其中训练集99例,测试集25例在进行特征筛选和融合后,利用Python SCIKITLearn建立了每个特征组的机器学习分类模型。比较了支持向量机(support vector machine,SVM)、K近邻(Knearest neighbor,KNN)以及随机森林(random forest,RF)三种机器学习模型的性能。利用测试集的受试者工作特征(receiver operating characteristic,ROC)曲线及计算相应曲线下面积(area under curve,AUC)值来评估模型的识别能力(图2)。
1.8统计学分析采用 SPSS 26.0软件对数据进行统计分析,计数资料的差异性分析运用χ2检验; 计量资料以平均数±标准差(x±s)表示,其差异性分析采用独立样本t检验。通过绘制ROC曲线评估各模型的效能,此外计算不同模型特异度、灵敏度、准确度、精确度、召回率和F1分数。以P<0.05为差异具有统计学意义。
2结果
2.1患者临床信息经过统计分析,性别以及年龄不存在统计学差异(P>0.05),见表2。
2.2特征提取和选择的结果影像组学基于ROI的3D图像提取了107个放射学特征。只有ICC≥0.75的影像组学特征被认为是高度稳定的,并保留在后续分析中,因此选择了103个放射学特征进行后续工作。将肿瘤最大ROI图像输入预先训练的卷积神经网络(convolutional neural networks,CNN),从每个MR图像模态中提取2 048个深度学习特征。所提取的深度学习特征从预训练的深度学习模型ResNet50的全连接层输出。通过Pearson相关系数和Lasso回归分析,选取系数不为零的特征构建分类模型。经过特征筛选,最终影像组学保留8个特征(图3A),深度学习保留42个特征(图3B),融合特征保留39个(图3C),见图3。另外通过GradCAM可视化可以发现,深度学习模型对于切片的关注重点在于瘤体中心位置,见图4。
2.3模型比较为了寻找效能最优的机器学习模型来建立模型,我们比较了不同机器学习模型的性能,其中包括SVM、KNN以及RF三种机器学习模型,结果表明,SVM机器学习模型的效能最优。在传统组学中AUC值为0.699(95%CI=0.555~0.843);在深度迁移学习中AUC值为0.853(95%CI=0.754~0.953)。而在融合模型中的AUC值为0.868(95%CI=0.766~0.969)。并且经过计算可得基于融合特征的SVM机器学习模型特异度及精確度均高于单一特征的机器学习模型。由此可得基于融合特征的SVM机器学习模型效能最优,具有诊断IDH1基因状态的价值(图5、表35)。
3讨论
本研究共纳入了124例胶质瘤患者,开发了一个基于MRI影像组学及深度学习特征的融合模型来预测IDH1基因的突变情况,其中该融合模型针对IDH1基因的突变情况具有较高的预测能力(AUC=0.868)。近年来,随着胶质瘤发病率的不断升高以及指南的更新,基因分型也参与到胶质瘤的分级评估以及预后评估。其中IDH1基因的突变情况是影响胶质瘤患者预后的独立危险因素[1415]。IDH突变可通过增加2羟基戊二酸的水平进而抑制组蛋白的去甲基化[16],从而增加IDH突变型患者对化疗的敏感度。并且IDH1作为潜在的治疗靶点[17],未来可以参与临床决策的选择。如术前能够获得IDH1基因的突变情况,将会对胶质瘤患者治疗方案的选择至关重要。
融合模型可以实现IDH1基因状态的无创性预测,主要是由于基因表达的不同,将会导致相关基因位点的关键蛋白表达不同,而不同的蛋白表型将会直接影响肿瘤的形态、生长特点以及肿瘤的异质性。本研究从124例胶质瘤患者的T2抑脂序列图像中提取影像组学和深度学习特征相互叠加,成为融合特征,并通过Pearson相关系数和Lasso回归分析保留39个融合特征(图3),其中包括2个影像组学特征和37个深度学习特征,在这部分特征中显示original_firstorder_Skewness、original_glcm_Imc1和部分深度学习特征与IDH1突变状态相关度最高。这两个影像组学特征可以用来描述胶质瘤图像的亮度、对比度、纹理和肿瘤边缘等信息,其中original_firstorder_Skewness特征(偏度)表示肿瘤内部体素信号不一致,IDH突变型与IDH野生型相比,IDH突变型的胶质瘤边界更清晰,对比度增强更少,即偏度相对越小[18]。这些信息在一定程度上可以反映肿瘤的异质性[19]。同时经过GradCAM可视化分析可以发现,深度学习模型对于肿瘤的关注点在于肿瘤的瘤内部分,也是人工智能模型进行学习的重要位置,同时肿瘤的异质性主要表现在瘤内部分,这与深度学习模型所对于图像的关注点相契合。近年来,人工智能在预测IDH1基因领域已表现出巨大的潜力与价值。Peng等[20]使用CET1、T2、ASL多模态序列使用SVM机器学习模型进行建模预测IDH1基因状态,其AUC值可达0.823。唐薇等[21]使用基于增强MRI图像的影像组学模型来预测脑胶质瘤IDH1基因突变状态,验证集的AUC值为0.889。但以上的研究仅限于单独的机器学习或深度学习算法,并没有将两者进行相互融合。而本研究开发并验证了基于T2抑脂序列的影像组学模型、深度迁移学习模型以及影像组学和深度学习特征融合模型。相比于以往的研究,将影像组学的特征与深度学习的特征相结合。在训练集和测试集中,可以有效区分IDH1基因突变情况。其中基于影像组学和深度学习特征融合模型(SVM模型)表现出较优的效能,SVM机器学习算法是一种性能出色的分类和回归算法,其在处理高维数据时具有很好的泛化能力,可以有效避免过拟合现象。相比其他机器学习算法,SVM的优势在于其能够通过核技巧解决非线性问题,并且适用范围较广。研究结果表明,融合模型具有较好的效能以及在预测IDH基因突变方面具有广阔的应用前景。
本研究也存在一些局限性。首先,在特征提取方面,对于影像组学特征的提取目前大多数是基于Pyradiomics代码包,而对于深度学习特征可能会有更多的深度学习模型来进行选择,如VGG、DenseNet等系列,不同的深度学习模型所包含的卷积层和池化层不同导致所提取出的特征会出现差异,故本研究选择在医学影像领域较为常见的ResNet50模型进行特征提取。在影像数据方面,本研究所采用的样本量较少,可能会限制算法的泛化能力和适用范围。此外,由于仅仅使用医学影像数据可能无法完全反映患者的全部信息,这可能会导致模型过于依赖于影像特征,而忽略了其他重要信息,如患者的基本情况、病史等。这会影响模型的有效性和可靠性。在未来,本研究将希望进一步优化特征提取方式,通过多中心研究以及纳入患者多种临床资料来共同构建模型,进一步增加模型可信度及鉴别效能。
利益沖突:所有作者均声明不存在利益冲突。
[参 考 文 献]
[1]Sung H,Ferlay J,Siegel RL,et al.Global cancer statistics 2020:GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J].CA Cancer J Clin,2021,71(3):209249.
[2]Louis DN,Perry A,Wesseling P,et al.The 2021 WHO classification of tumors of the central nervous system:a summary[J].Neuro Oncol,2021,23(8):12311251.
[3]Hartmann C,Hentschel B,Wick W,et al.Patients with IDH1 wild type anaplastic astrocytomas exhibit worse prognosis than IDH1mutated glioblastomas,and IDH1 mutation status accounts for the unfavorable prognostic effect of higher age:implications for classification of gliomas[J].Acta Neuropathol,2010,120(6):707718.
[4]Turkalp Z,Karamchandani J,Das S.IDH mutation in glioma:new insights and promises for the future[J].JAMA Neurol,2014,71(10):13191325.
[5]Juratli TA,Kirsch M,Geiger K,et al.The prognostic value of IDH mutations and MGMT promoter status in secondary highgrade gliomas[J].J Neurooncol,2012,110(3):325333.
[6]EckelPassow JE,Lachance DH,Molinaro AM,et al.Glioma groups based on 1p/19q,IDH,and TERT promoter mutations in tumors[J].N Engl J Med,2015,372(26):24992508.
[7]Xu SC,Tang L,Dai G,et al.Immunerelated genes with APA in microenvironment indicate risk stratification and clinical prognosis in grade II/III gliomas[J].Mol Ther Nucleic Acids,2021,23:12291242.
[8]Bi WL,Hosny A,Schabath MB,et al.Artificial intelligence in cancer imaging:Clinical challenges and applications[J].CA Cancer J Clin,2019,69(2):127157.
[9]Zhu YB,Man CT,Gong LX,et al.A deep learning radiomics model for preoperative grading in meningioma[J].Eur J Radiol,2019,116:128134.
[10]Avanzo M,Wei LS,Stancanello J,et al.Machine and deep learning methods for radiomics[J].Med Phys,2020,47(5):e185e202.
[11]Avanzo M,Stancanello J,Pirrone G,et al.Radiomics and deep learning in lung cancer[J].Strahlenther Onkol,2020,196(10):879887.
[12]Ma WJ,Zhao YM,Ji Y,et al.Breast cancer molecular subtype prediction by mammographic radiomic features[J].Acad Radiol,2019,26(2):196201.
[13]Saha A,Harowicz MR,Grimm LJ,et al.A machine learning approach to radiogenomics of breast cancer:a study of 922 subjects and 529 DCEMRI features[J].Br J Cancer,2018,119(4):508516.
[14]van den Bent MJ,Brandes AA,Taphoorn MJB,et al.Adjuvant procarbazine,lomustine,and vincristine chemotherapy in newly diagnosed anaplastic oligodendroglioma:longterm followup of EORTC brain tumor group study 26951[J].J Clin Oncol,2013,31(3):344350.
[15]Cairncross G,Wang MH,Shaw E,et al.Phase III trial of chemoradiotherapy for anaplastic oligodendroglioma:longterm results of RTOG 9402[J].J Clin Oncol,2013,31(3):337343.
[16]Lu C,Ward PS,Kapoor GS,et al.IDH mutation impairs histone demethylation and results in a block to cell differentiation[J].Nature,2012,483(7390):474478.
[17]Rohle D,PopoviciMuller J,Palaskas N,et al.An inhibitor of mutant IDH1 delays growth and promotes differentiation of glioma cells[J].Science,2013,340(6132):626630.
[18]van Lent DI,van Baarsen KM,Snijders TJ,et al.Radiological differences between subtypes of WHO 2016 grade IIIII gliomas:a systematic review and metaanalysis[J].Neurooncol Adv,2020,2(1):vdaa044.
[19]Hsieh KLC,Chen CY,Lo CM.Radiomic model for predicting mutations in the isocitrate dehydrogenase gene in glioblastomas[J].Oncotarget,2017,8(28):4588845897.
[20]Peng H,Huo JH,Li B,et al.Predicting isocitrate dehydrogenase(IDH) mutation status in gliomas using multiparameter MRI radiomics features[J].J Magn Reson Imaging,2021,53(5):13991407.
[21]唐薇,段俊艷,余子意,等.增强MRI影像组学预测脑胶质瘤IDH1基因突变的价值分析[J].磁共振成像,2022,13(5):111114.
(收稿20230508修回20230813)
