基于ITS2序列及其二级结构对矩镰荚苜蓿和近缘种的分子鉴定
2024-04-11王霞刘艳贾秀秀方强恩
王霞 刘艳 贾秀秀 方强恩

摘 要 为准确鉴定矩镰荚苜蓿(Medicago archiducis-nicolai)及其近缘种花苜蓿(M.ruthenica )、阔荚苜蓿(M.platycarpos )和毛荚苜蓿(M.edgeworthii ),利用DNA条形码技术对4个物种的ITS2序列进行注释、比对、检验,计算种内种间遗传距离,邻接法(neighbor-joining,NJ)构建系统发育树,通過RNAfold web server预测4个近缘种的ITS2二级结构。结果显示,矩镰荚苜蓿及其近缘种的ITS2序列长度为220~221 bp,稳定性好;种间遗传距离(0.004 55~0.022 73)明显大于种内遗传距离(均为0),存在明显的“Barcoding Gap”区域;NJ系统发育树显示,毛荚苜蓿聚为一支,矩镰荚苜蓿、花苜蓿和阔荚苜蓿聚为另一支,然后矩镰荚苜蓿与阔荚苜蓿又各自形成单系;在二级结构中,矩镰荚苜蓿与阔荚苜蓿、毛荚苜蓿存在明显差异,但与花苜蓿极为相似,难以区分。分析表明:除花苜蓿外,以ITS2序列作为条形码并辅以二级结构,可以达到对矩镰荚苜蓿、阔荚苜蓿和毛荚苜蓿准确、快速鉴定的目的。
关键词 矩镰荚苜蓿;ITS2序列;二级结构;分子鉴定
矩镰荚苜蓿(Medicago archiducis-nicolai)是中国特有植物,和近缘种花苜蓿(M.ruthenica)、阔荚苜蓿(M.platycarpos)、毛荚苜蓿(M.edgeworthii )同属豆科苜蓿属阔荚苜蓿组[1-2]。在中国西北高原地区广泛分布,营养丰富,抗逆性强,极具驯化利用价值[3-4]。另外,矩镰荚苜蓿、花苜蓿与毛荚苜蓿还可为藏药[5-7]。但三者间形态上十分相似,极易混淆[1,5,8],常出现“同名异物”现象,导致误用、混用,给药材使用造成很大隐患[5,8]。为了科学有效利用矩镰荚苜蓿及其近缘种植物资源,有必要对其进行准确分类及鉴定。
传统物种鉴定方法在易混淆近缘种鉴别方面存在很多局限性[9-11]。DNA条形码鉴定技术因不受环境、样品、部位、形态、发育阶段等限制,可以直接从基因水平提供界定依据,从而弥补了传统鉴别方法的不足,成为目前物种鉴定的重要手段[12-13]。内部转录间隔区2(internal transcribed spacer2,ITS2)序列位于5.8S和28S rDNA基因之间,引物通用性强、序列较短、聚合酶链式反应(PCR)扩增成功率和测序成功率高、物种分辨力强,具有易于折叠成二级结构的非编码 RNA片段,在植物物种鉴定上应用广泛,被推荐为核心DNA条形码[14-16]。陈士林等[16]创建的中国中药材DNA条形码鉴定体系选用ITS2序列为物种鉴定的主体序列。在中国中药材鉴定中,ITS2序列已经作为唯一的核心条形码写入第一至四版《中国药典》[16]。郑梦迪等[17]、马丽杰等[18]、刘亚令等[19]研究发现,在植物近缘种鉴定中如果加入ITS2二级结构可获得更多分类信息,能对物种鉴定起到校正、补充和优化的效果,因此,以ITS2序列为主二级结构为辅,可以大大提高物种与近缘种的鉴定准确率。
基于以上综述,本研究在西北主要分布区采集矩镰荚苜蓿与3个近缘种材料进行DNA提取、扩增、测序,结合NCBI获得ITS2序列,通过MEGA 6.0软件对序列进行注释、比对,计算种内种间的遗传距离,构建系统进化树,并通过RNAfold web server 预测ITS2二级结构,以期实现对矩镰荚苜蓿与近缘种的快速、准确鉴定,为用药安全提供科学依据。
1 材料与方法
1.1 材 料
2020年8月至9月,在甘肃、青海、四川、宁夏4个省区选择11个样点取样,共采集到14个自然居群的111个样本,其中矩镰荚苜蓿10个居群88个样本,花苜蓿4个居群23个样本(表1)。各居群间的距离在30 km以上,同一居群内个体间的距离在10 m以上。所选试验材料均为野外采集的新鲜健康叶片,采集后立即用硅胶快速干燥帯回实验室,存放于-20 ℃冰箱中,以备DNA的提取。各居群的凭证标本存放于甘肃农业大学。另外,从GenBank数据库下载阔荚苜蓿的ITS序列3条(MW241773~MW241775),毛荚苜蓿的ITS序列8条(MW241732~MW241739),用于本次分析的序列共122条。
1.2 试验方法
1.2.1 总DNA提取 选取硅胶干燥的叶片约30 mg,加入液氮研磨粉碎,使用生工生物工程(上海)股份有限公司植物基因组DNA 提取试剂盒(离心柱型)提取总DNA。
1.2.2 PCR扩增及测序 ITS2扩增引物由上海生工生物科技有限公司合成。正向引物序列为: 5′-ATGCGATACTTGGTGTGAAT-3′,反向引物序列为:5′-GACGCTTCTCCAGACTACAAT-3′。PCR扩增、测序引物一致。
PCR 扩增体系:总体积为25 μL,其中上游引物和下游引物各1 μL,PCR Master Mix 12.5 μL,DNA 模板 2 μL,并用ddH2O补足。扩增程序:95 ℃预变性 4 min;94 ℃变性 30 s,退火 1 min,72 ℃延伸 1 min,35 个循环;最后 72 ℃延伸 10 min。
PCR扩增产物经电泳获得单一明亮条带后,送交上海生工生物科技有限公司进行双向测序。
1.3 数据处理
1.3.1 ITS2 序列的获得 用SeqMan软件对测序所得序列进行剪切、编辑和比对,并辅以人工校正,删除低质量区,得到ITS2完整序列。从GenBank上下载花苜蓿、阔荚苜蓿和毛荚苜蓿的ITS序列。应用相似性搜索算法(BLAST)检验序列的鉴定成功率;将获得的所有完整序列导入ITS2数据库中(http://its2.bioapps.biozentrum.uni-wuerzburg.de/),通过隐马尔可夫模型(HMMs)[20]注释的方法去除两端5.8S和28S区域,得到标准的ITS2间隔区序列。最后将序列上传至Genbank,得到GenBank登录号。
1.3.2 种内、种间遗传距离计算及Neighbor-Joining(NJ)系统发育树构建 将所有ITS2序列的Fasta格式导入MEGA 6.0软件,利用Muscle比对并进行人工手动校正。利用MEGA 6.0软件对111个样本序列和11条下载ITS2序列按种进行分组,统计分析其碱基含量及序列特征;基于K2-P(Kimura2-parameter)距离模型计算种内种间的遗传距离;以近缘属植物草木樨(Melitoyus-officinalis:Z97687)为外类群,构建Neighbor-Joining (NJ)系统发育树,用bootstrap(1 000次重复)检验各分支置信度,评价分支系统发育关系的可靠性。
1.3.3 矩镰荚苜蓿及其近缘种ITS2序列二级结构预测 利用在线网站RNAfold web server对各样本进行二级结构预测。
2 结果与分析
2.1 PCR 扩增及测序成功率分析
对矩镰荚苜蓿及其近缘种花苜蓿的111个样品的ITS2扩增产物经1.0%琼脂糖凝胶电泳,得到PCR扩增电泳图(图1)。图中条带较亮,没有拖尾现象;ITS2完整序列均在 600 bp左右。所有试验样本的PCR扩增和测序成功率均达到100%,表明ITS2序列引物及其反应条件针对矩镰荚苜蓿及花苜蓿的扩增效果好,具有很好的稳定性。
2.2 种内与种间ITS2 序列差异分析
通过隐马尔可夫模型(HMMs)[20]注释的方法去除ITS2完整序列两端5.8S和28S区域,得到标准的ITS2间隔区序列;采用相似性搜索算法(BLAST)检验,结果显示矩镰荚苜蓿和花苜蓿ITS2 序列与NCBI中已有序列完全一致,序列相似性为100%,表明测序结果质量较好,真实 可信。
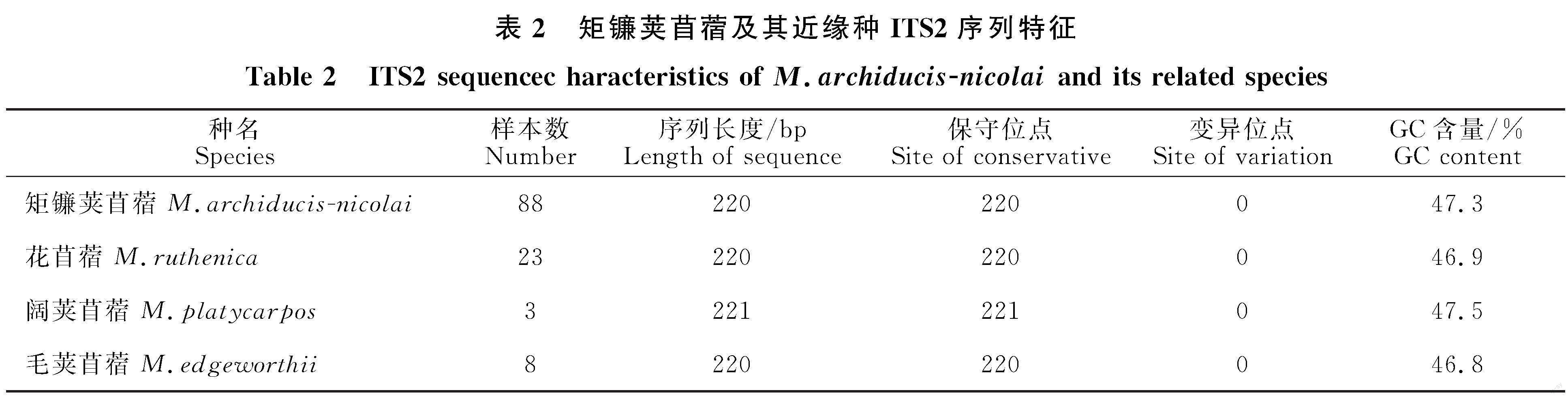
从表2可以看出,矩镰荚苜蓿、花苜蓿及毛荚苜蓿的ITS2序列长度均为220 bp,G+C含量分别为 47.3%、46.9%、46.8%,阔荚苜蓿的ITS2序列长度为221 bp,G+C含量为47.5%。通过MEGA 6.0软件对矩镰荚苜蓿及其近缘种ITS2序列进行比对分析,得到种内差异信息位点(表2)。结果显示,供试的矩镰荚苜蓿88个样本与23个花苜蓿之间无差异位点,下载的阔荚苜蓿、毛荚苜蓿内部也无差异。
种间差异显示(表3),矩镰荚苜蓿与花苜蓿相比,二者只有1个差异位点(141位点的C/T差异);与阔荚苜蓿存在2个差异位点(43和141位点);与毛荚苜蓿有5个差异位点(10、85、141、166、208位点)。花苜蓿與毛荚苜蓿相比有4个差异位点(10、85、166、208位点)。阔荚苜蓿与毛荚苜蓿有5个差异位点(10、43、85、166、208位点)。阔荚苜蓿在165位点处有一个插入/缺失位点。
2.3 种内与种间遗传距离分析及barcoding Gap 检验
基于MEGA 6.0软件中K2-P(Kimura 2-parameter)距离模型,计算矩镰荚苜蓿及其近缘种的种内种间遗传距离,结果显示(表4),矩镰荚苜蓿及其近缘种的种内遗传距离均为0;不同物种间相比,毛荚苜蓿与矩镰荚苜蓿、阔荚苜蓿遗传距离最大(0.022 73),其次是毛荚苜蓿与花苜蓿 (0.018 18),矩镰荚苜蓿与阔荚苜蓿遗传距离较小(0.009 09),花苜蓿与矩镰荚苜蓿、阔荚苜蓿的遗传距离最小(0.004 55)。从种间、种内遗传距离分布图可以看出(图2),矩镰荚苜蓿及其近缘种的种间遗传距离明显大于种内遗传距离,表明种内和种间存在明显的“Barcoding Gap”区域。
2.4 ITS2序列NJ(neighbor-joining)系统进化树鉴别
利用ITS2序列构建矩镰荚苜蓿及其近缘种的邻接法(neighbor-joining,NJ)系统进化树(图3)。结果显示,NJ系统进化树中各分支的支持率都在50%以上。其中毛荚苜蓿聚为一支,与矩镰荚苜蓿、花苜蓿、阔荚苜蓿明显分开。矩镰荚苜蓿、阔荚苜蓿与花苜蓿聚为另一支,矩镰荚苜蓿与阔荚苜蓿又各自聚成单系;但花苜蓿成梳子状拓扑结构,不形成独立的分支,无法与矩镰荚苜蓿、阔荚苜蓿分开。
2.5 ITS2二级结构预测
通过RNAfold web server网站预测得到矩镰荚苜蓿及其近缘种ITS2二级结构(图3),从图中可以看出,这4个近缘种的二级结构较为相似,均符合被子植物一环(主环:Mainloop) 四臂(4个螺旋区:helix) 的特征[17]。每个螺旋上又有大小、数目、形态不同的茎环(loop)结构。比较4个近缘种的ITS2二级结构发现,各物种在4个螺旋区的茎环数目、大小均有明显差异。毛荚苜蓿二级结构最为特殊,臂Ⅰ与臂Ⅱ之间有1个大小不同的突环,臂Ⅰ与臂Ⅳ间多一个突环;矩镰荚苜蓿、花苜蓿和阔荚苜蓿在臂Ⅰ中都有1个发卡环和2个内环,而毛荚苜蓿只有1个发卡环和 1个内环,并且阔荚苜蓿靠近发卡环的内环明显小于矩镰荚苜蓿和花苜蓿的内环;比较臂Ⅲ结构可以看出,阔荚苜蓿在臂Ⅲ有3个内环,而矩镰荚苜蓿、花苜蓿、毛荚苜蓿都有2个。
因此,根据ITS2序列的二级结构可以直观地将矩镰荚苜蓿与近缘种毛荚苜蓿、阔荚苜蓿区分开。但矩镰荚苜蓿与花苜蓿ITS2序列二级结构极为相似,仍然无法将二者区分开。
3 讨 论
序列稳定性指不同产地、不同批次的样品都能稳定地获得DNA条形码序列[21]。稳定性是DNA条形码筛选的重要条件。在国际DNA条形码鉴定联盟组织(CBOL)推荐的7条候选条形码序列(rbcL、trnH-psbA、matK、rpoC1,ycf5、ITS和ITS2)中[22],ITS2序列具有非常好的稳定性[22-23]。这与本研究结果完全一致。本试验中,ITS2序列扩增成功率和测序成功率均达到100%(图1),在不同海拔、产地的样本中并没有产生新的单倍型,ITS2序列十分稳定。
除稳定性之外,ITS2序列因具有引物通用性强、序列较短、扩增成功率和测序成功率高、物种分辨力强等诸多优点[24],一直被植物分子系统学研究所重视。尤其是近十年来,随着DNA条形码技术在中药材易混伪品甄别中广泛应用,中国学者在药用植物ITS2序列分析方面开展了大量工作。Chen等[22]、Yao等[23]通过大样本中药材近缘种鉴定分析,发现ITS2在种水平的分辨率能达到92.7%。据此,Chen等[22]在2011年创建了中药材DNA条形码鉴定体系,推荐ITS2 序列作为物种鉴定的核心条形码,形成了中国特色的药材鉴别技术。自2015年这一技术写入《中国药典》以来,基于ITS2序列的分子鉴定技术在中药材易混伪品、近缘物种鉴定中起到了非常好的应用效果。参考前人研究的方法[21-22],本研究结果显示,矩镰荚苜蓿及其近缘种的ITS2序列长度为220~221 bp,4个物种种内遗传距离都为0,种间遗传距离为0.004 55~0.022 73,种内遗传距离(0)小于种间最小K-2P遗传距离(0.004 55)(表5),即种内和种间存在明显的“BarcodingGap”区域[25](图2)。分析矩镰荚苜蓿及其近缘种NJ树得出,毛荚苜蓿聚为一支,与矩镰荚苜蓿、花苜蓿、阔荚苜蓿明显分开;矩镰荚苜蓿、阔荚苜蓿与花苜蓿聚为另一支,矩镰荚苜蓿与阔荚苜蓿又各自聚成单系,但花苜蓿没有形成独立的分支,无法与矩镰荚苜蓿、阔荚苜蓿分开(图3)。可见,利用ITS2序列可以准确鉴定出矩镰荚苜蓿、阔荚苜蓿和毛荚苜蓿,但不能区分花苜蓿与矩镰荚苜蓿、阔荚苜蓿。
ITS2具有相对保守的二级结构[12],能对一级结构信息进行验证与补充,起到校正优化的作用[12,17-18]。中国学者刘亚令等[19]对两种药用黄芪(Astragalus spp.)、杨美青等[14]对防己(Stephaniatetrandra)及其混伪品的研究得出,加入二级结构可以提供物种分类鉴定的额外信息,成功鉴定出基于一级结构无法界定出的近缘种。为了提高ITS2序列在矩镰荚苜蓿及其近缘种中的鉴定效果,本研究也预测分析了4个近缘种的二级结构。经过比较发现,矩镰荚苜蓿、花苜蓿都与阔荚苜蓿、毛荚苜蓿的二级结构在环的数量和大小方面存在较大的差异(图4)。矩镰荚苜蓿、花苜蓿和阔荚苜蓿在臂Ⅰ中都有1个发卡环和2个内环,而毛荚苜蓿只有1个发卡环和 1个内环;阔荚苜蓿靠近发卡环的内环明显小于矩镰荚苜蓿和花苜蓿的内环;阔荚苜蓿在臂Ⅲ有3个内环,而矩镰荚苜蓿、花苜蓿、毛荚苜蓿都有2个。二级结构为矩镰荚苜蓿及其近缘种的鉴定提供了更加丰富的分类信息。通过二级结构,可以很直观地将矩镰荚苜蓿与阔荚苜蓿、毛荚苜蓿区分开,也可以将花苜蓿與阔荚苜蓿、毛荚苜蓿区分开,但是,矩镰荚苜蓿与花苜蓿具有基本一致的二级结构,仅在臂Ⅲ有C/T差异位点(图4),凭借二级结构难以将二者区分。
前人研究表明,矩镰荚苜蓿与花苜蓿在苜蓿属阔荚苜蓿组中的亲缘关系是最近的[25-27]。吴小培等[7]和Chen等[25] 分别在花苜蓿、矩镰荚苜蓿种间遗传结构和中国苜蓿属系统发育研究中均认为,矩镰荚苜蓿与花苜蓿可能存在不完全的谱系分化或存在杂交渐渗。在本研究中,依据ITS2序列和二级结构仍无法将矩镰荚苜蓿与花苜蓿区分来,可见,二者存在十分复杂的种间关系。为了对矩镰荚苜蓿及其花苜蓿进行准确的分类鉴定,还需结合传统形态学、借助更多基因序列进行试验研究。
参考文献 Reference:
[1] ERNEST S,MARCEL J. A synopsis of the genus Medicago (Leguminosae)[J].Canadian Journal of Botany,1989,67(11):3260-3294.
[2] 中国科学院植物志编辑委员会.中国植物志(第42卷第2分册)[M].北京:科学出版社,1998:312-328.
Editorial Committee of Flora,Chinese Academy of Sciences. Flora of China (Vol. 42,Part 2)[M].Beijing: Science Press,1998:312-328.
[3] 陈默君,贾慎修.中国饲用植物[M].北京:中国农业出版社,2001:9.
CHEN M J,JIA SH X. Forage Plants in China [M].Beijing: China Agriculture Press,2001:9.
[4] 贾笃敬,张映生,张自和,等.高寒地区优良豆科牧草阴山扁蓿豆一些性状的研究[J].甘肃农业大学学报,1984(1):63-69.
JIA D J,ZHANG Y SH,ZHANG Z H,et al. Study on some characters of Medicago archiducis-nicolai in Alpine Region [J].Journal of Gansu Agricultural University,1984(1):63-69.
[5] 江纪武,靳朝东.世界药用植物速查辞典[M].北京:中国医药科技出版社,2015: 14-914.
JIANG J W,JIN CH D. Quick Reference Dictionary of World Medicinal Plants [M].Beijing: China Medical Science and Technology Press,2015:14-914.
[6] 青海省生物研究所,同仁县隆务诊疗所.青藏高原药物图鉴[M].西宁:青海人民出版社,1972: 84.
Qinghai Institute of Biology,Tongren County Longwu Clinic. Drug Atlas of Qinghai-Tibet Plateau [M].Xining: Qinghai Peoples Publishing House,1972:84.
[7] 吴小培,沈迎芳,王海庆. 基于 trnL-trnF 序列的扁蓿豆和青藏扁蓿豆遗传多样性及其群体遗传结构分析. 草业科学,2016,33(6):1136-1146.
WU X P,SHEN Y F,WANG H Q. Genetic diversity and population genetic structure of Medicago ruthenica and Medicago archiducis-nicolai based on trnL-trnF sequence[J]. Pratacultural Science,2016,33(6):1136-1146.
[8] 刘志鹏,任广朋.苜蓿属物种分类研究进展[J].草业学报,2022,31(11):191-203.
LIU ZH P,REN G P. Research progress on species classification of Medicago[J].Acta Prataculturae Sinica,2022,31(11):191-203.
[9] 李晓叶,钱 路,陈茂华.DNA条形码技术在几种苹果园鳞翅目害虫鉴定中的应用[J].西北农业学报,2015,24(5):141-147.
LI X Y,QIAN L,CHEN M H. Application of DNA barcoding technology in identification of lepidoptera pests in apple orchards [J]. Acta Agriculturae Boreali-occidentalis Sinica,2015,24(5):141-147.
[10] 馮鹏龙,韩 睿,王亚艺,等.基于SSR标记辣椒品种DNA指纹图谱的构建[J].西北农业学报,2022,31(3):320-327.
FENG P L,HAN R,WANG Y Y,et al. Construction of DNA fingerprinting of Capsicum annuum varieties based on SSR markers[J]. Acta Agriculturae Boreali-occidentalis Sinica,2022,31(3):320-327.
[11] 刘红梅,张存艳,叶 强,等.基于DNA条形码技术对喉红石斛的植物学分类研究[J].中草药,2021,52(21):6656-6662.
LIU H M,ZHANG C Y,YE Q,et al. Botanical classification of Dendrobium christyanum based on DNA barcoding [J]. Chinese Journal of Traditional Chinese Medicine,2021,52(21):6656-6662.
[12] 杨 烁,薛渊元,李美慧,等.ITS2二级结构系统发育信息在茄属药用植物DNA条形码鉴定中的应用价值[J].中国中药杂志,2017,42(3):456-464.
YANG SH,XUE Y Y,LI M H,et al. Application of phylogenetic information of ITS2 secondary structure in DNA barcoding of Solanum medicinal plant [J]. China Journal of Chinese Materia Medica,2017,42(3):456-464.
[13] SENAPATI A,BASAK S,RANGAN L,et al.A review on application of DNA barcoding technology for rapid molecular diagnostics of adulterants in herbal medicine[J]. Drug Safety :An InternationalJjournal of Medical Toxicology and Drug Experience,2022,45(3):1-21.
[14] 杨美青,杜 燕.基于ITS2序列及其二级结构对防己及其混伪品的鉴定[J].中国实验方剂学杂志,2018,24(10):43-48.
YANG M Q,DU Y.Identification of stephaniae tetrandrae radix and its adulterants based on ITS2 and its secondary structure [J]. Chinese Journal of Experimental Formulae,2018,24(10):43-48.
[15] 陈芙蓉,郭巧生,杨 锋,等.ITS2二级结构系统发育信息在野菊及其近缘种植物DNA条形码鉴定中的应用价值[J].中国中药杂志,2019,44(22):4813-4819.
CHEN F R,GUO Q SH,YANG F,et al. Application of ITS2 secondary structure phylogenetic information in DNA barcode identification of Chrysanthemum indicum and its related plants [J]. Chinese Journal of Traditional Chinese Medicine,2019,44(22):4813-4819.
[16] 陈士林,庞晓慧,姚 辉,等.中药DNA条形码鉴定体系及研究方向[J].世界科学技术(中医药现代化),2011,13(5):747-754.
CHEN SH L,PANG X H,YAO H,et al. Identification system and perspective for DNA barcoding traditional chinese materia medica [J].World Science and Technology (Mmodernization of Traditional Chinese Mmedicine),2011,13(5):747-754.
[17] 郑梦迪,贺紫涵,张 寒,等.基于ITS2一级序列和二级结构对土茯苓及混伪品的鉴别研究[J].中国现代中药,2022,24(6):1026-1032.
ZHENG M D,HE Z H,ZHANG H,et al. Identification of Smilacis glabrae Rhizoma and its adulterants based on ITS2 sequence and secondary structure[J]. Chinese Modern Ttraditional Chinese Medicine,2022,24(6):1026-1032.
[18] 马丽杰,吴 云,谷 巍,等.基于ITS2序列的东北透骨草及其混伪品DNA分子鉴定[J].中草藥,2019,50(23):5830-5837.
MA L J,WU Y,GU W,et al. Molecular identification of Phryma leptostachya and its adulterants based on ITS2 sequence[J].Chinese Herbal Medicines,2019,50(23):5830-5837.
[19] 刘亚令,耿雅萍,王 芳,等.基于ITS2序列及二级结构对药用黄芪及混伪品的鉴别研究[J].药学学报,2020,55(3):522-529.
LIU Y L,GENG Y P,WANG F,et al. Identification of Astragalus and its adulterants based on ITS2 sequence and secondary structure [J].Chinese Journal of Pharmacy,2020,55(3):522-529.
[20] COLEMAN A W. Pan-eukaryote ITS2 homologies revealed by RNA secondary structure.[J]. Nucleic Acids Research,2007,35(10):3322-3329.
[21] CBOL Plant Working Group. A DNA barcode for land plants[J].Proceedings of the National Academy of Sciences,2009,106: 12794.
[22] CHEN SH L,YAO H,HAN J P,et al. Validation of the ITS2 region as a novel DNA barcode for identifying medicinal plant species[J]. Plos One,2010,5(1):e8613.
[23] YAO H,SONG J Y,LIU C,et al. Use of ITS2 region as the universal DNA barcode for plants and animals[J]. PLos One,2010,5(10) : e13102.
[24] 陈士林,姚 辉,韩建萍,等.中药材DNA条形码分子鉴定指导原则[J].中国中药杂志,2013,38(2):141-148.
CHEN SH L,YAO H,HAN J P,et al. Guidelines for molecular identification of DNA barcoding in Chinese medicinal materias[J].Chinese Journal of Materia Medica,2013,38(2):141-148.
[25] CHEN J Y,WU G L,SHRESTHA N,et al. Phylogeny and species delimitation of chinese Medicago (Leguminosae) and its relatives based on molecular and morphological evidence[J]. Frontiers in Plant Science,2021,11:619799.
[26] HU D,LI F,JIE L,et al.Systematic positions of Medicago edgeworthii and M.archiducis-nicolai (Leguminosae) inferred from plastid trnK/matK,nuclear GA30X1 and its sequences[J]. Pakistan Journal of Botany,2014,46(3):775-778.
[27] 田春育,李志勇,劉 倩,等.苜蓿属不同物种叶绿体基因组结构比较及亲缘关系分析[J].中国草地学报,2021, 43(10):1-8.
TIAN CH Y,LI ZH Y,LIU Q,et al. Medicago different species chloroplast genome structure comparison and genetic relationship analysis [J]. Chinese Journal of Grassland,2021,43(10):1-8.
Molecular Identification of Medicago archiducis-nicolai and Its Related
Species Based on ITS2 Sequence and Its Secondary Structure
Abstract In order to accurately identify Medicago archiducis-nicolai and its relative species,M.ruthenica,M.platycarpos and M.edgeworthii,the ITS2 sequences of four species were annotated,compared and examined by DNA barcoding,and the genetic distances of intra-and inter-species were calculated,the neighbor-joining phylogenetic tree was constructed,the ITS2 secondary structures of four related species were predicted by RNA fold web server. The results showed that the length of ITS2 sequence of four species was between 220-221 bp and showed good stability. The genetic distance of inter-species (0.004 55-0.022 73) was significantly larger than that of the intra-species (0),and there was an obvious barcoding gap region. The NJ phylogenetic tree showed that M.edgeworthii clustered into one branch,and Medicago archiducis-nicolai,M.ruthenica and M.platycarpos clustered into another branch,then Medicago archiducis-nicolai and M.platycarpos formed monophyletic,respectively. The ITS2 secondary structure of Medicago archiducis-nicolai was obviously different from that of M.platycarpos and M.edgeworthii,but it was very similar to that of M.ruthenica,and difficult to distinguish. The analysis showed that ITS2 sequence as a barcode and supplemented with secondary structure can be accurately and rapidly used for the identification of M.archiducis-nicolai,M.platycarpos and M.edgeworthii exception for M.ruthenica.
Key words Medicago archiducis-nicolai; ITS2 sequence; Secondary structure; Molecular identification
