珍稀濒危飘带兜兰叶绿体全基因组种内变异研究
2024-03-10高鑫祯唐露汪雨邵士成罗艳
高鑫祯 唐露 汪雨 邵士成 罗艳


摘 要: 飄带兜兰 (Paphiopedilum parishii) 分布范围狭窄,仅在中国、缅甸、泰国以及老挝有少量分布。近年来,因生境破坏和人为滥采而导致飘带兜兰野生种群极度缩减。为开发种内多态性的分子标记用于保护生物学研究,该研究对飘带兜兰4个野生个体经测序、组装、注释获得的叶绿体基因组序列,与已公布的飘带兜兰2个个体的叶绿体全基因组序列进行比对,分析飘带兜兰叶绿体基因组的种内差异。结果表明:(1) 飘带兜兰叶绿体基因组具有典型被子植物叶绿体基因组环状四分体结构,基因组长度为154 403~154 809 bp,共编码129个基因,包括78个蛋白质编码基因、39个tRNA基因、8个rRNA基因,以及4个假基因。(2) 在飘带兜兰6个个体叶绿体基因组中检测到103~107个简单重复序列 (simple sequence repeats,SSRs) 位点,其中21个SSR位点具有多态性。此外,在6个个体叶绿体基因组中还检测到60个长序列重复,包括17~21个正向重复、18~29个反向重复、9~16个回文重复、4~9个互补重复。(3) 通过比较6个个体叶绿体基因组序列的核苷酸多样性,共发现70处变异,包括10个单核苷酸多态性 (single nucleotide polymorphism,SNPs) 、60个插入缺失 (InDels)。其中,有3个SNP位点发生了非同义替换,导致编码功能基因的氨基酸发生改变;19个插入缺失多态性较高,具有开发为分子标记的潜力。(4) 通过计算核苷酸多样性值 (Pi) 共发现8个有变异的区域,Pi值为0~0.006 32,其中变异度较大的是rps3-rpl22、trnL-UAC-rpl32、rpoB-trnC-GCA以及ycf4,这些高变区可开发为分子标记用于评估飘带兜兰遗传多样性。(5) 系统发生分析结果表明,飘带兜兰6个个体叶绿体基因组序列聚在一起,与长瓣兜兰互为姐妹群。综上表明,飘带兜兰叶绿体基因组的SSRs、长序列重复、SNPs、[JP2]InDels以及核苷酸序列呈现了足够的种内多样性,可开发成分子标记用于该种的系统演化及保护生物学研究。
关键词: 叶绿体全基因组, 飘带兜兰, 序列差异, 多态性分子标记, 插入缺失, 微卫星
中图分类号: Q943
文献标识码: A
文章编号: 1000-3142(2024)01-0001-14
Intraspecific genetic variation within chloroplast
genome of a rare and endangered species
Paphiopedilum parishii (Orchidaceae)
GAO Xinzhen1,2, TANG Lu1, WANG Yu1,3, SHAO Shicheng1, LUO Yan1*
( 1. Xishuangbanna Tropical Botanical Garden, Chinese Academy of Sciences, Mengla 666303, Yunnan, China; 2. University of Chinese Academy
of Sciences, Beijing 100049, China; 3. School of Landscape Architecture, Southwest Forestry University, Kunming 650224, China )
Abstract: Paphiopedilum parishii is a rare and endangered species with few localities and fragmented populations found in China, Myanmar, Thailand and Laos. Environmental changes and over-harvesting have led to a decrease in its wild populations. It is important to protect endangered species genetic diversity since it provides the species with the ability to adapt and survive. However, little is known about the genetic information of this species. This study aims to detect intraspecific variation and develop polymorphic genetic markers of P. parishii. The complete chloroplast genome of four individuals of wild P. parishii was obtained by sequencing, assembling and annotating, then compared with the existing genomic data of two individuals available from GenBank to detect the intraspecific variation. Further, simple sequence repeats (SSRs), single nucleotide polymorphisms (SNPs), and insertions and deletions (InDels) were identified. The results were as follows: (1) The four new sequencing chloroplast genomes were quadripartite, with a length between 154 403 bp and 154 809 bp, with 129 genes (78 protein coding genes, 39 tRNAs, 8 rRNAs and four pseudogenes). (2) As a result of comparison of six individuals, 103-107 SSR loci were identified in the chloroplast genome of six individuals of P. parishii, and 21 SSRs were polymorphic. And 60 long repeats were found, including 17-21 forward repeats, 18-29 reverse repeats, 9-16 palindromic repeats, and 4-9 complement repeats. (3) In addition, a total of 10 SNPs and 60 InDels were uncovered across the plastome. Three of the non-synonymous mutations caused amino acid changes in functional domains. 19 InDels might be selected for possible chloroplast DNA markers to determine intraspecific variation. (4) The value of nucleotide diversity (Pi) was calculated ranging from 0-0.006 32 suggesting sequences with low variation. Hyper-polymorphic regions, e.g. intergenic spacers rps3-rpl22, trnL-UAC-rpl32, rpoB-trnC-GCA and ycf4 gene were identified as potential barcoding regions. (5) The phylogenetic analyses based on the complete chloroplast genome supported three lineages in Paphiopedilum, and six individuals of P. parishii form a monophyletic group. SSRs, long repeats, InDels, SNPs and nucleotide sequences showed sufficient intraspecific genetic variation in P. parishii. The molecular markers developed here will contribute to further evolutionary studies and conservation of P. parishii.
Key words: chloroplast genome, Paphiopedilum parishii, sequence divergence, polymorphic DNA markers, InDels, SSRs
遗传多样性代表着物种适应环境和生存发展的能力,保存珍稀濒危物种的遗传多样性是物种保护的重要目标 (Guerrant & Pavlik, 1998; 黄宏文, 2018)。叶绿体是植物细胞内进行光合固碳和胁迫应答的重要半自主遗传细胞器,植物叶绿体基因组为环状DNA分子,单亲遗传,基因组大小为107~218 kb,基因结构和组成稳定 (张韵洁和李德铢, 2011; Ivanova et al., 2017)。叶绿体基因组通常为四分体结构,包括两个反向重复序列(inverted repeat, IR)、一个大单拷贝区 (large single copy region, LSC) 和一个小单拷贝区 (small single copy region, SSC) (Wolfe et al., 1987; 田欣和李德铢, 2002; Jansen et al., 2005; 张韵洁和李德铢, 2011; He et al., 2019)。叶绿体基因组编码110~130个基因,主要包括光合作用相关和叶绿体基因表达相关的基因等 (张韵洁和李德铢, 2011)。叶绿体基因组虽然较小,但包含有大量遗传信息,在核苷酸序列水平和结构重排上呈现出进化与保守性,编码区和非编码区有不同进化速率,在分子水平上有明显差异,可用于区分种间关系、评估种间变异性等,被广泛用于群体遗传多样性、系统演化等研究领域 (Wolfe et al., 1987; 张韵洁和李德铢, 2011; He et al., 2019; 黎若竹等, 2022)。近年来,利用种内不同个体叶绿体基因组的种内变异,为物种进化研究、种质资源利用以及濒危植物保护提供了基础遗传信息(Ishizuka et al., 2017; Muraguri et al., 2020; Zhang RS et al., 2020)。
兜兰属 (Paphiopedilum) 为兰科(Orchi-daceae)杓兰亚科 (subfamily Cypripedioideae)植物,有80余种,主要分布于亚洲至太平洋岛屿的热带亚热带地区的石灰岩山地 (Cribb, 1998; 刘仲健等, 2009)。兜兰的唇瓣因形似拖鞋而被称为“拖鞋兰”,具有极高的观赏价值 (杨颖婕等, 2021)。近年来,兜兰属植物的自然生境局限于生态环境脆弱的喀斯特地区,加上过度采挖等人类活动导致的生态退化和生境严重破坏,使得兜兰属植物野生种群数量急剧减少,部分兜兰属植物面临野外灭绝的风险 (罗毅波等, 2003; 杨颖婕等, 2021)。目前,所有野生兜兰种类均列入《濒危野生动植物种国际贸易公约》 (Convention on International Trade in Endangered Species of Wild Fauna and Flora, CITES),同时我国分布的野生兜兰属植物也全部列入《国家重点保护野生植物名录》(2021年9月颁布实施)。因此,我国野生兜兰属物种的濒危机制与保护工作急需开展。
飘带兜兰 (Paphiopedilum parishii )为附生兰科植物,生于海拔1 000~1 100 m的树干或石头上,为我国一级重点保护植物。飘带兜兰被世界自然保护联盟 (International Union for Conservation of Nature, IUCN) 评估为濒危等级 (Endangered, ER),仅在中国云南南部、泰国、缅甸和老挝分布有少数的野生种群 (Rankou & Averyanov, 2015)。飘带兜兰具有极高的观赏价值,在各植物园均有引种,但野生种群已极为罕见。Chen等 (2012) 在云南南部进行了飘带兜兰繁殖生物学的观察,发现该种具有独特的自动授粉机制。基于叶绿体全基因组发育基因组学的研究,飘带兜兰属于兜兰亚属 (subgenus Paphiopedilum) (Guo et al., 2021)。但是,有关飘带兜兰的遗传信息仍不完善,缺乏评估遗传多样性及种内多样性的分子标记,不利于对其开展综合保护。
本研究选取飘带兜兰的4个野生个体进行浅层基因组测序、组装获得叶绿体全基因组,并与已公开的该种2个个体叶绿体基因组 (Guo et al., 2021; Kao et al., 2021) 进行基因组比较,分析飘带兜兰叶绿体基因组的种内变异,理解其基因进化水平和遗传多样性,以期开发种内多态性的分子标记用于保护生物学研究,为开展飘带兜兰的濒危机制及物种保护研究提供遗传信息基础资料。
1 材料与方法
1.1 研究材料及DNA提取、测序
飘带兜兰4个个体分别采自云南省勐腊县 (P. parishii_1 和P. parishii_2) 及澜沧县 (P. parishii_3 和P. parishii_4) 两个野生居群 (图1),取新鲜叶片放入硅胶中干燥。采用植物基因组DNA提取试剂盒 (TIANGEN, 中国北京) 提取叶片总DNA,使用IlluminaTruSeq文库建立试剂盒 (San Diego, CA, USA) 构建测序文库,利用Illumina Hiseq 2500测序平台 (上海派森诺生物科技有限公司) 进行浅层基因组测序。同时,在GenBank上下载已公开的另外2个飘带兜兰个体的叶绿体基因组序列 (P. parishii_5, MW528213和P. parishii_6, MN587822)进行比较基因组分析。
1.2 叶绿体基因组的组装和注释
采用Fastp软件对下机后的原始数据 (raw data) 进行过滤,获得高质量测序数据 (clean data) 后,用GetOrganelle v1.6.3程序將测序后得到的数据组装成叶绿体基因组 (Jin et al., 2018)。组装完成后的叶绿体基因组数据用Geneious Prime v2021.2.2的MAFFT工具进行比对后,使用在线工具CPGAVAS2 (http://47.90.241.85:16019/analyzer/annotate2) (Shi et al., 2019),GeSeq (https://chlorobox.mpimp-golm.mpg.de/geseq.html) (Tillich et al., 2017) 和tRNA-scan-SE v2.0.3 (Lowe & Chan, 2016) 对叶绿体基因组的蛋白质编码区、核糖体RNA (rRNA) 和转运RNA (tRNA) 进行注释,参考已发表的兜兰属叶绿体基因组对注释结果进行校正。通过OGDRAW (https://chlorobox.mpimp-golm.mpg.de/OGDraw. html) 在线网站绘制飘带兜兰叶绿体基因组图谱 (Lohse et al., 2007)。使用Geneious Prime 2021.2.2软件分析6个飘带兜兰叶绿体基因组的基本结构,统计LSC、SSC、IR的长度以及GC含量、蛋白质编码基因、tRNA基因、rRNA基因的数目。
1.3 叶绿体基因组重复序列和SSR分析
通过MISA在线网站 (https://webblast.ipk-gatersleben.de/misa) (Beier et al., 2017) 测算叶绿体基因组中简单重复序列 (simple sequence repeats, SSR) 的分布,将核苷酸最小重复数分别设置为10 (单核苷酸重复mononucleotide repeats)、5 (二核苷酸重复dinucleotide repeats)、4 (三核苷酸重复trinucleotide repeats)、3 (四核苷酸重复tetranucleotide repeats)、3 (五核苷酸重复pentanucleotide repeats)、3 (六核苷酸重复hexanucleotide repeats)。
通过REPuter (https://bibiserv.cebitec.uni-bielefeld.de/reputer) (Kurtz et al., 2001) 在线网站测算叶绿体基因组中其他重复序列的分布,参数设置为最小重复序列30 bp,汉明距离(Hamming distance) 设置为3 (Liang et al., 2019)。
1.4 种间叶绿体基因组的比较分析
通过mVISTA (https://genome.lbl.gov/vista) 在线网站 (Frazer et al., 2004),以P. parishii_1的叶绿体基因组作为参考,将其余5条飘带兜兰的叶绿体基因组的编码区、基因间隔区、内含子区进行比对分析,评估各基因组序列之间的相似性和差异。使用R v4.0.2软件运行IRscope (Amiryousefi et al., 2018) 脚本,对6个个体的叶绿体基因组序列的相邻边界区域进行绘制,比较分析边界区域的扩张和收缩情况。使用DnaSP v5.10.0.1软件,统计飘带兜兰不同个体间序列的单核苷酸多态性 (SNPs) 和插入缺失 (InDels) 情况,计算各序列间核苷酸多样性 (Pi)。
1.5 系统发育分析
从GenBank上下载15种兜兰属叶绿体全基因组序列,与本研究新测序拼装的4个个体以及已发表的2条飘带兜兰叶绿体基因组序列,以3种杓兰属植物为外类群来构建系统发育树 (物种的基因登录号见图5)。使用PhyloSuite v1.2.2软件,利用ModelFinder计算最佳碱基替换模型 (Zhang et al., 2020a),使用叶绿体基因组全序列,经MAFFT进行多序列比对后,采用IQ-TREE构建最大似然法 (maximum likelihood, ML) 系统发育树 (Trifinopoulos et al., 2016),系统发育树分支的支持率基于bootstrap自展法检测,将bootstrap值设置为5 000、重复次数设置为1 000次。
2 结果与分析
2.1 飘带兜兰叶绿体基因组基本特征
对原始数据进行过滤后,飘带兜兰4个样品分别获得2.4~3.1 G的高质量clean data。组装和注释的叶绿体全基因组序列已登录至GenBank (登录号为OP 604356~OP 604359)。将本研究获得的飘带兜兰4个个体和已公布的2个飘带兜兰叶绿体基因组一起比对分析,发现6条序列的叶绿体基因组均为环状四分体结构,包括LSC区、SSC区和2个IR区 (图2)。叶绿体基因组序列全长为154 403~154 809 bp,其中LSC的长度为86 581~86 983 bp,SSC的长度为2 436~2 446 bp,IR的长度为32 690~32 693 bp。总GC含量为35.9%,LSC的GC含量为33.4%,SSC的GC含量为29.1%~29.2%,IR的GC含量为39.5% (表1)。
飘带兜兰6个个体叶绿体基因组的基因总数均为129个,包括78个蛋白质编码基因、39个tRNA基因、8个rRNA基因,以及4个假基因 (ndhJ、ndhD、2个ycf15) (附表1)。22个基因在IR区重复,具有2个拷贝,包括9个蛋白质编码基因 (psaC、ndhB、rps7、rps15、rps19、rpl2、rpl23、ycf1、ycf2)、9个tRNA基因 (trnA-UGC、trnH-GUG、trnI-CAU、trnI-GAU、trnL-CAA、trnL-UAG、trnN-GUU、trnR-ACG、trnV-GAC) 和4个rRNA基因 (rrn16、rrn23、rrn4.5、rrn5)。18个基因有内含子,其中15个基因含有1个内含子,包括9个蛋白编码基因 (petB、petD、atpF、ndhB、rpoC1、rps16、rpl2、rpl16、accD) 和6個tRNA基因 (trnA-UGC、trnG-UCC、trnI-GAU、trnK-UUU、trnL-UAA、trnV-UAC);2个基因含有2个内含子 (clpP、ycf3);rps12为反剪接基因 (附表1)。
2.2 SSR位点分析
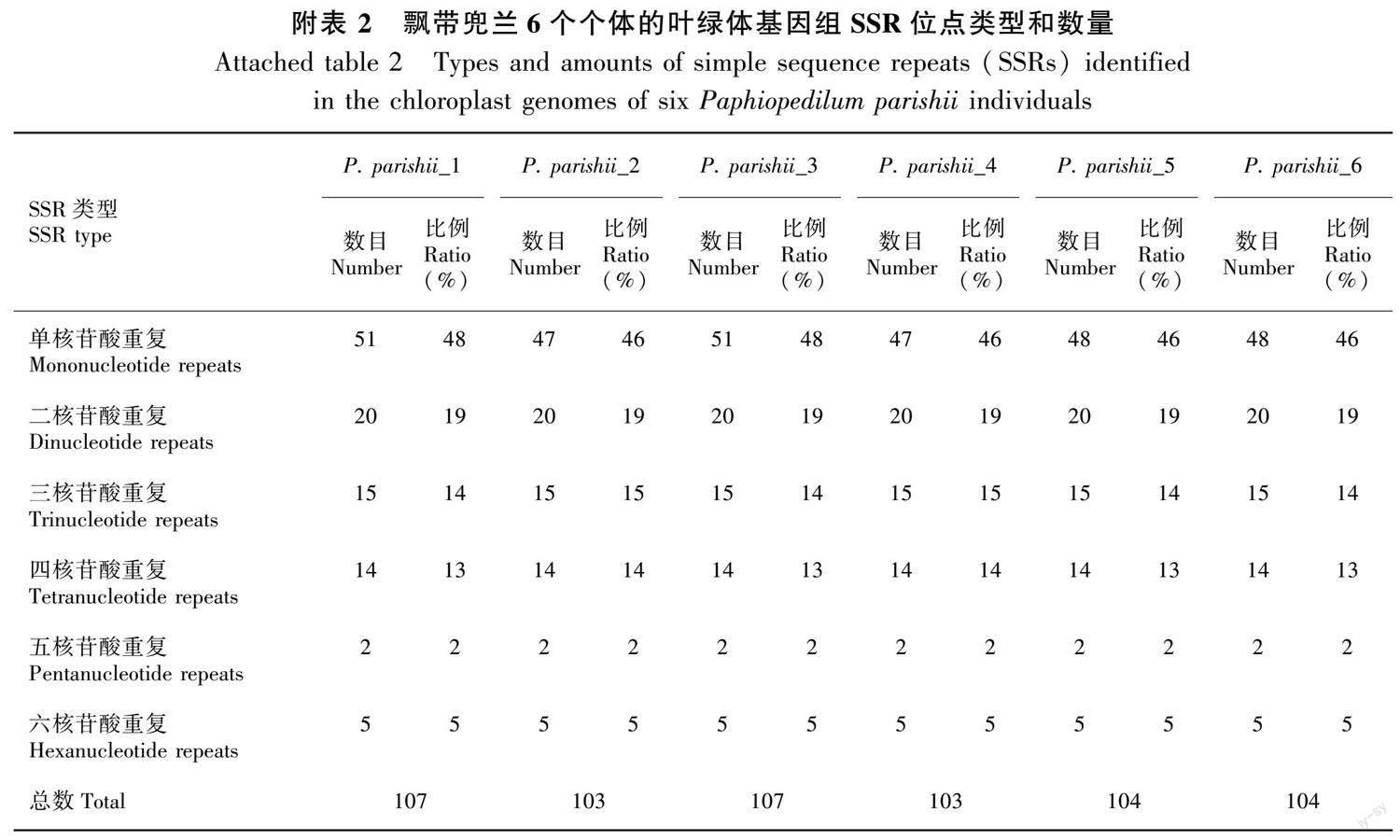
利用MISA软件在飘带兜兰6个个体的叶绿体基因组中分别检测到103~107个SSR位点,包括47~51个单核苷酸重复 (主要以A、T碱基重复为主)、20个二核苷酸重复、15个三核苷酸重复、14个四核苷酸重复、2个五核苷酸重复和5个六核苷酸重复类型 (图3: A;附表2)。SSR位点大部分位于LSC区 (84~88个),其次分布于IR区(18~19个),SSC区无SSR位点 (图3: B)。另外,SSR位点主要分布于叶绿体基因组非编码区的基因间隔区 (intergenic spacer, IGS),为72~76个;位于基因内含子区 (intron) 的有17个;位于编码区的有14个(图3: C, D)。
将飘带兜兰6个个体同源区域的SSR位点进行详细的比对,筛选出具有相同类型且重复数目不同的SSR位点,可作为种内多态性位点,共发现21个SSR位点在6个个体中有差异 (表2)。
2.3 重复序列分析
利用REPuter对6条飘带兜兰叶绿体基因组序列进行重复序列分析。在6个叶绿体基因组中均鉴定到60个重复序列,包括17~21个正向重复(forward repeats)、18~29个反向重复(reverse repeats)、4~9个互补重复 (complement repeats)、9~16个回文重复 (palindromic repeats) (图4: A)。6条叶绿体基因组重复序列的长度范围虽然跨度较大,但都集中在30~40 bp之间 (图4: B)。
2.4 飘带兜兰种间叶绿体基因组序列的多样性
以P. parishii_1为参考序列,利用mVISTA软件对6条飘带兜兰序列进行序列多样性比较 (附图1)。6条飘带兜兰序列相似性较高,LSC区的多样性相对较高,略高于IR区和SSC区。多样性水平较高的片段主要在非编码区,如atpH-atpI、psaA-ycf3、atpB-rbcL、accD-psaI、trnP-UGG-psaJ。編码区变异度较低,ycf1基因的变异度稍高,tRNA和rRNA基因在飘带兜兰中极为保守。
6条序列边界比较图(附图2)显示,rpl22、trnL-UAG、rps19以及psbA分别位于6条序列的LSC/IRb、IR/SSC、IRa/LSC边界或边界附近。rpl22位于LSC/IRb边界上,有58 bp的片段位于IRb区域;重复基因trnL-UAG位于IR区域内,离IR/SSC边界相距176~186 bp;重复基因rps19位于IR区,离IR/LSC边界282 bp;psbA位于LSC区,离IRa/LSC边界96 bp (附图2),6条序列边界基因较为保守,扩张收缩现象不显著,仅P. parishii_4的IR区有微小扩张。
2.5 飘带兜兰种间叶绿体基因组核苷酸多样性
通过对6条飘带兜兰叶绿体基因组核苷酸多样性的分析,发现6条叶绿体基因组序列中共存在10个SNPs和60个InDels,其中基因间隔区有4个SNPs和43个InDels,内含子区有1个SNP和13个InDels,编码区有5个SNPs和4个InDels (表3)。在10个SNPs中有3个发生了非同义替换,导致密码子改变,分别是在rpoC1基因中碱基A替换为碱基C,使编码的氨基酸由异亮氨酸转变为丝氨酸;在rpoB基因中碱基C替换为碱基T,使编码的甘氨酸转变为精氨酸;在ycf4基因中碱基G替换为碱基C,使编码的蛋氨酸转变为异亮氨酸。此外,还有2个SNPs发生同义替换,密码子未改变,其余SNPs均位于非编码区。对大于3 bp的InDels进行多态性分析,发现共有19个位点呈现了种内的多态性 (表4),如基因间隔区pasA-ycf3分别在P. parishii_1、3、4中发生插入,在P. parishii_2、5、6中缺失;而在accD基因的编码区及内含子分别存在的2个InDels在个体间具有多态性。
利用DnaSP分别计算了飘带兜兰6个个体的叶绿体基因组基因和基因间隔区的核苷酸多样性值 (Pi),发现编码区的Pi值为0~0.000 61,仅有5个基因的核苷酸多样性值大于0,其中ycf4基因的Pi值最高;基因间隔区的Pi值为0~0.006 32,仅有3个基因间隔区的核苷酸多样性值大于0,其中rps3-rpl22的Pi值最高 (附图3),表明飘带兜兰种内表现了较低水平的核苷酸多样性,基因间隔区的核苷酸多样性相对较高。此外,还发现变异程度较高的位点主要位于LSC区,而IR区和SSC区相对保守。
2.6 飘带兜兰的系统发育关系
通过PhyloSuite计算出最佳碱基替换模型为TVM+F+R2,采用此模型构建系统发育树(图5)。16种兜兰属植物构成了支持率很高 (支持率均为100%) 的3个分支,其中德氏兜兰 (P. delenatii)、杏黄兜兰 (P. armeniacum)、白花兜兰 (P. emersonii)和硬叶兜兰 (P. micranthum) 构成了基部分支,归属于小萼亚属 (Subgenus Parvisepalum);文山兜兰 (P. wenshanense)、同色兜兰 (P. concolor) 和巨瓣兜兰 (P. bellatulum) 形成一个单系分支,归属于宽瓣亚属 (Subgenus Brachypetalum),本研究中的飘带兜兰6个个体未分开,形成1个分支,与长瓣兜兰形成姐妹群。
3 讨论与结论
本研究利用二代高通量测序技术对飘带兜兰野生居群不同个体进行浅层基因组测序,通过组装和注释得到飘带兜兰叶绿体全基因组。6个个体的叶绿体基因组的基因数量和顺序一致,没有发生基因组的重排或倒位事件。6个叶绿体基因组均为叶绿体基因组的典型四分体式结构,由IR区、LSC区、SSC区组成 (Jansen et al., 2005)。飘带兜兰叶绿体基因组长度为154 403~154 809 bp,符合此前报道的兰科及兜兰属植物的叶绿体基因组大小 (Kim et al., 2014; Guo et al., 2021)。飘带兜兰不同个体间的叶绿体基因组长度变化主要在LSC区,为86 581~86 983 bp;而SSC区和IR区基本保持一致,分别为2 436~2 446 bp和32 690~32 693 bp。IR区较其他被子植物有显著的扩张,SSC区则有较大程度的收缩,仅包含rpl32和ccsA 2个基因,这些特点符合兜兰属叶绿体基因组特征 (Guo et al., 2021)。与其他已报道的兰科属种的叶绿体全基因组的SSC区长度相比,如凌氏石豆兰 (Bulbophyllum lingii, 18 244 bp)、王氏石斛 (Dendrobium wangliangii, 18 373 bp)、扇脉杓兰 (Cypripedium japonicum, 21 911 bp) (Kim et al., 2020; Shao et al., 2020; Tang et al., 2021),兜兰属的SSC区收缩严重,导致整个基因组的长度远低于兰科植物其他物种。大量SSC区的基因转移至IR区,使得SSC区的收缩在兜兰属极为常见,杏黄兜兰中甚至一些典型的SSC区基因,如ycf1、psaC、ndhD转移至IR区 (Kim et al., 2015; Lin et al., 2015; Niu et al., 2017)。有研究认为,由于IR区的基因可以利用同源重组机制修复DNA损伤,较大的IR区域更有利于质体基因组的稳定性, 因此基因的转移可能更利于该基因的表达 (Palmer & Thompson, 1982; Wicke et al., 2011)。这种基因转移使得兜兰属SSC区具有极高多样性,序列长度差异大,长度为524~5 913 bp,基因数目显著不同,其种间多样性和种内的稳定性使其具有开发成用于物种鉴定的分子标记的潜力 (Guo et al., 2021)。
飘带兜兰的叶绿体基因组共编码129個基因,包括78个蛋白质编码基因、39个tRNA基因、8个rRNA基因,以及4个假基因,与Guo等(2021)报道的相同。4个假基因都是因大量基因片段缺失而造成功能丧失,其中ndhJ因片段缺失而导致基因缺少起始密码子,而ndhD和ycf15的碱基缺失在50%以上。飘带兜兰叶绿体基因组中存在大量的ndh基因丢失,只保留了ndhB、ndhD和ndhJ,其中ndhD和ndhJ为假基因,这是SSC区收缩的原因之一。兰科植物普遍存在ndh基因的丢失现象 (Yang et al., 2013; Feng et al., 2016; Niu et al., 2017; Zavala-Páez et al., 2020),但ndh基因的丢失程度不同,如石豆兰属叶绿体基因组中存在12个ndh基因 (Tang et al., 2021),兰属植物叶绿体基因组中存在10个ndh基因 (胡国家, 2020),石斛属的ndh基因丢失现象极为严重 (牛志韬, 2017)。叶绿体ndh基因与核基因组部分基因共同编码NAD (P) H脱氢酶复合体,参与环式电子传递链 (CET) 途径,在植物抗逆胁迫中起重要作用。除了兰科植物外,在裸子植物松杉类和麻黄类中也发现ndh基因的丢失 (Braukmann et al., 2009; Wu et al., 2009)。Lin等(2017)研究认为,ndh基因的缺失可能是植物由自养转为异养的进化过程。
本研究在飘带兜兰6个个体的基因组中共鉴定到103~107个SSR位点,其中数量最多的是单核苷酸重复序列,其次是二核苷酸重复序列,多以A、T碱基为基本重复单元,显示了高度的A/T偏好,与Qin等(2015)、陈模舜和杨仲毅(2022)在被子植物叶绿体基因组中观察到的情况一致。重复序列在植物基因组中扮演重要角色,因多态性较高而在群体遗传和进化研究中经常被用来做分子标记 (Muraguri et al., 2020)。本研究共筛选到21个SSR位点具有多态性,这些位点可开发为分子标记用于评估飘带兜兰种群的遗传多样性。此外,本研究发现非编码区的SSR数量远多于编码区,说明非编码区比编码区具有更高遗传多样性,这可能由于非编码区面临着更大的选择压力 (Shaw et al., 2007)。正向重复、反向重复、互补重复和回文重复在6个个体的叶绿体基因中都表现了多样性。同种植物长序列重复的不同可能因序列的插入缺失而导致重复序列的类型发生改变,可能与基因重组有关 (Somaratne et al., 2019)。
插入缺失和突变引起基因结构的差异是遗传变异的重要来源, 代表生物个体适应环境变化的能力(Han & Xue, 2003)。种间或种内的叶绿体基因结构差异可用于近缘种间和种内的系统发育关系及群体遗传学研究 (McCauley, 1995; Kersten et al., 2016)。本研究在飘带兜兰的6个个体的叶绿体基因组中鉴定到60个InDels和10个SNPs,主要分布在基因间隔区。与其他种内叶绿体基因组比较分析相比,飘带兜兰的InDels和SNPs数量并不多。Muraguri等 (2020) 在蓖麻 (Ricinus communis) 12个个体的叶绿体基因组中共鉴定到162个SNPs和92个InDels;Zhang等 (2020b) 在麻栎 (Quercus acutissima) 3个个体中共检测到77个SNPs和255个InDels。Alexander等 (2014) 在红槲栎 (Quercus rubra) 4个个体中鉴定到8个SNPs和45个InDels。本研究共发现19个位点呈现种内多态性,通过mVISTA基因组比对和核苷酸多样性 (Pi) 值的计算,发现非编码区的核苷酸多样性远高于编码区,其中rps3-rpl22的Pi值最高 (0.006 32)。在飘带兜兰叶绿体基因组中存在的这些基因组的结构变异可广泛用于种间和种内关系以及遗传多样性研究。
遗传多样性是评估物种对栖息地环境适应能力以及抗逆能力的重要指标,研究遗传多样性和遗传结构是制定物种保护措施的前提 (张亚红等, 2019)。通过叶绿体全基因组的比较分析,本研究筛选出了一些多态性叶绿体基因组分子标记,为评估飘带兜兰的遗传多样性,探讨系统发生、进化以及保护遗传学研究提供遗传学基础。
致谢 中国科学院西双版纳热带植物园Sven Landrein、杨国平协助野外调查,云南野兰堂生物有限公司张泽提供照片,谨致谢意。
参考文献:
AL EXANDER LW, WOESTE KE, 2014. Pyrosequencing of the northern red oak (Quercus rubra L.) chloroplast genome reveals high quality polymorphisms for population management [J]. Tree Genet Genomes, 10: 803-812.
AMIRYOUSEFI A, HYVONEN J, POCZAI P, 2018. IRscope: an online program to visualize the junction sites of chloroplast genomes [J]. Bioinformatics, 34(17): 3030-3031.
BEIER S, THIEL T, MUNCH T, et al., 2017. MISA-web: a web server for microsatellite prediction [J]. Bioinformatics, 33(16): 2583-2585.
BRAUKMANN TW, KUZMINA M, STEFANOVIC S, 2009. Loss of all plastid ndh genes in gnetales and conifers: extent and evolutionary significance for the seed plant phylogeny [J]. Curr Genet, 55(3): 323-337.
CHEN LJ, LIU KW, XIAO XJ, et al., 2012. The anther steps onto the stigma for self-fertilization in a slipper Orchid [J]. PLoS ONE, 7(5): e37478.
CHEN MS, YANG ZY, 2022. Genealogical structure and differentiation analysis of Carpinus tientaiensis based on single nucleotide polymorphism of chloroplast genome [J]. Guihaia, 42(10): 1703-1716. [陳模舜, 杨仲毅, 2022. 基于叶绿体基因组SNP的天台鹅耳枥谱系结构与分化分析 [J]. 广西植物, 42(10): 1703-1716.]
CRIBB P, 1998. The Genus Paphiopedilum [M]. Borneo, Malaysia: National History Publications.
FENG YL, WICKE S, LI JW, et al., 2016. Lineage-specific
reductions of plastid genomes in an orchid tribe with partially and fully mycoheterotrophic species [J]. Genome Biol Evol, 8(7): 2164-2175.
FRAZER KA, PACHTER L, POLIAKOV A, et al., 2004. VISTA: computational tools for comparative genomics [J]. Nucl Acid Res, 32: W273-W279.
GUERRANT EO, PAVLIK BM, 1998. Reintroduction of rare plants: Genetics, demography, and the role of ex situ conservation methods [M]// FIEDLER PL, KAREIVA PM, Conservation Biology: For the Coming Decade. Boston, MA: Springer US: 80-108.
GUO YY, YANG JX, BAI MZ, et al., 2021. The chloroplast genome evolution of Venus slipper (Paphiopedilum): IR expansion, SSC contraction, and highly rearranged SSC regions [J]. BMC Plant Biol, 21(1): 248.
HAN B, XUE YB, 2003. Genome-wide intraspecific DNA-sequence variations in rice [J]. Curr Opin Plant Biol, 6(2): 134-138.
HE J, YAO M, LYU RD, et al., 2019. Structural variation of the complete chloroplast genome and plastid phylogenomics of the genus Asteropyrum (Ranunculaceae) [J]. Sci Rep, 9(1): 15285.
HU GJ, 2020. The complete chloroplast genomes of Paphiopedilum and Cymbidium (Orchidaceae) species: comparative genomic and phylogenetic analyses [D]. Xian: Northwest University: 28 -30. [胡国家, 2020. 兰科兜兰属与兰属植物比较叶绿体基因组学与系统发育研究 [D]. 西安: 西北大学: 28-30.]
HUANG HW, 2018. The principle and practice of ex situ plant conservation [M]. Beijing : Science Press: 176-179. [黄宏文, 2018. 植物迁地保育原理与实践 [M]. 北京: 科学出版社: 176-179.]
ISHIZUKA W, TABATA A, ONO K,et al., 2017. Draft chloroplast genome of Larix gmelinii var. japonica: insight into intraspecific divergence [J]. J For Res, 22(6): 393-398.
IVANOVA Z, SABLOK G, DASKALOVA E, et al., 2017. Chloroplast genome analysis of resurrection tertiary relict Haberlea rhodopensis highlights genes important for desiccation stress response [J]. Front Plant Sci, 8: 204.
JANSEN RK, RAUBESON LA, BOORE JL,et al., 2005. Methods for obtaining and analyzing whole chloroplast genome sequences [M]//ZIMMER EA, ROALSON EH, Molecular Evolution: Producing the Biochemical Date: Part B: 348-384.
JIN J, YU WB, YANG JB, et al., 2018. GetOrganelle: a simple and fast pipeline for de novo assembly of a complete circular chloroplast genome using genome skimming data [J]. Genome Biol, 21(1): 241.
KAO H, ZHAO Y, YANG M, et al., 2021. The complete chloroplast genome sequences of an endangered orchid species Paphiopedilum parishii (Orchidaceae) [J]. Mitochondrial DNA B, 6(9): 2521-2522.
KERSTEN B, RAMPANT PF, MADER M, et al., 2016. Genome sequences of Populus tremula chloroplast and mitochondrion: Implications for holistic poplar breeding [J]. PLoS ONE, 11(1): e0147209.
KIM HT, KIM JS, MOORE M, et al., 2015. Seven new complete plastome sequences reveal rampant independent loss of the ndh gene family across orchids and associated instability of the inverted repeat/small single-copy region boundaries [J]. PLoS ONE, 10(11): e0142215.
KIM JS, KIM HT, KIM JH, et al., 2014. The largest plastid genome of monocots: a novel genome type containing AT residue repeats in the slipper orchid Cypripedium japonicum [J]. Plant Mol Biol Rep, 33(5): 1-11.
KIM YK, JO SJ, SE-HWAN C, et al., 2020. Plastome evolution and phylogeny of Orchidaceae, with 24 new sequences [J]. Front Plant Sci, 11: 22.
KURTZ S, CHOUDHURI JV, OHLEBUSCH E, et al., 2001. REPuter: the manifold applications of repeat analysis on a genomic scale [J]. Nucl Acid Res, 29(22): 4633-4642.
LIANG CL, WANG L, LEI J, et al., 2019. A comparative analysis of the chloroplast genomes of four Salvia medicinal plants [J]. Engineering, 5(5): 907-915.
LIN CS, CHEN JJ, HUANG YT, et al., 2015. The location and translocation of ndh genes of chloroplast origin in the Orchidaceae family [J]. Sci Rep, 5(1): 9040.
LIN CS, CHEN JJW, CHIU CC, et al., 2017. Concomitant loss of NDH complex-related genes within chloroplast and nuclear genomes in some orchids [J]. Plant J, 90(5): 994-1006.
LIU ZJ, CHEN XQ, CHEN LJ, 2009. The genus Paphiopedilum in China [M]. Beijing : Science Press: 8-10. [劉仲健, 陈心启, 陈利君, 2009. 中国兜兰属植物 [M]. 北京: 科学出版社: 8-10.]
LI RZ, CAI J, YANG JB, et al., 2022. Plastid phylogenomics resolving phylogenetic placement and genera phylogeny of Sterculioideae (Malvaceae s.l.) [J]. Guihaia, 42(1): 25-38. [黎若竹, 蔡杰, 楊俊波, 等, 2022. 利用叶绿体基因组数据解析锦葵科梧桐亚科的系统位置和属间关系 [J]. 广西植物, 42(1): 25-38.]
LOHSE M, DRECHSEL O, BOCK R, 2007. OrganellarGenome-DRAW (OGDRAW): a tool for the easy generation of high-quality custom graphical maps of plastid and mitochondrial genomes [J]. Curr Genet, 52(5/6): 267-274.
LOWE TM, CHAN PP, 2016. tRNAscan-SE On-line: integrating search and context for analysis of transfer RNA genes [J]. Nucl Acid Res, 44(1): W54-W57.
LUO YB, JIA JS, WANG CL, 2003. A general review of the conservation status of Chinese orchids [J]. Biodivers Sci, 11(1): 70-77. [罗毅波, 贾建生, 王春玲, 2003. 中国兰科植物保育的现状和展望 [J]. 生物多样性, 11(1): 70 -77.]
MCCAULEY DE, 1995. The use of chloroplast DNA polymorphism in studies of gene flow in plants [J].Trend Ecol Evol, 10(5): 198-202.
MURAGURI S, XU W, CHAPMAN M, et al., 2020. Intraspecific variation within Castor bean (Ricinus communis L.) based on chloroplast genomes [J]. Ind Crops Prod, 155: 112779.
NIU ZT, XUE QY, ZHU SY, et al., 2017. The complete plastome sequences of four orchid species: Insights into the evolution of the Orchidaceae and the utility of plastomic mutational hotspots [J]. Front Plant Sci, 8: 715.
NIU ZT, 2017. Comparative plastomic studies of Dendrobium species and comparison of the physiogical effects and transcriptome responses of Dendrobium officinale under different abiotic stresses [D]. Nanjing: Nanjing Normal University: 25-26. [牛志韬, 2017. 石斛属植物叶绿体基因组研究及其代表种逆境下光合作用途径探讨 [D]. 南京: 南京师范大学: 25-26.]
PALMER JD, THOMPSON WF, 1982. Chloroplast DNA rearrangements are more frequent when a large inverted repeat sequence is lost [J]. Cell, 29(2): 537-550.
QIN Z, WANG YP, WANG QM, et al., 2015. Evolution analysis of simple sequence repeats in plant genome [J]. PLoS ONE, 10(12): e0144108.
RANKOU H, AVERYANOV L, 2015. Paphiopedilum parishii. The IUCN red list of threatened species, 2015: e. T193512A2240580.
SHAO SC, TANG L, LUO Y, 2020. The complete chloroplast genome sequence of Dendrobium wangliangii (Orchidaceae) [J]. Mitochondrial DNA B, 5(3): 3513-3515.
SHAW J, LICKEY EB, SCHILLING EE, et al., 2007. Comparison of whole chloroplast genome sequences to choose noncoding regions for phylogenetic studies in angiosperms: The tortoise and the hare III [J]. Am J Bot, 94(3): 275-288.
SHI LC, CHEN HM, JIANG M, et al., 2019. CPGAVAS2, an integrated plastome sequence annotator and analyzer [J]. Nucl Acid Res, 47(1): W65-W73.
SOMARATNE Y, GUAN DL, WANG WQ, et al., 2019. Complete chloroplast genome sequence of Xanthium sibiricum provides useful DNA barcodes for future species identification and phylogeny [J]. Plant Syst Evol, 305(10): 949-960.
TANG HQ, TANG L, SHAO SC, et al., 2021. Chloroplast genomic diversity in Bulbophyllum section Macrocaulia (Orchidaceae, Epidendroideae, Malaxideae): insights into species divergence and adaptive evolution [J]. Plant Diverers, 43(5): 350-361.
TIAN X, LI DZ, 2002. Application of DNA sequences in plant phylogenetic study [J]. Acta Bot Yunnan, 24(2): 170-184. [田欣, 李德铢, 2002. DNA 序列在植物系统学研究中的应用 [J]. 云南植物研究, 24(2): 170-184.]
TILLICH M, LEHWARK P, PELLIZZER T, et al., 2017. GeSeq-versatile and accurate annotation of organelle genomes [J]. Nucl Acid Res, 45(1): W6-W11.
TRIFINOPOULOS J, LAMTUNG N, VON HAESELER A, et al., 2016. W-IQ-TREE: a fast online phylogenetic tool for maximum likelihood analysis [J]. Nucl Acid Res, 44(1): W232-W235.
WICKE S, SCHNEEWEISS G, DEPAMPHILIS C, et al., 2011. The evolution of the plastid chromosome in land plants: gene content, gene order, gene function [J]. Plant Mol Biol, 76(3-5): 273-297.
WOLFE KH, LI WH, SHARP PM, 1987. Rates of nucleotide substitution vary greatly among plant mitochondrial, chloroplast, and nuclear DNAs [J]. Proc Natl Acad Sci USA, 84(24): 9054-9058.
WU CS, LAI YT, LIN CP, et al., 2009. Evolution of reduced and compact chloroplast genomes (cpDNAs) in gnetophytes: Selection toward a lower-cost strategy [J]. Mol Phylogenet Evol, 52(1): 115-124.
YANG JB, TANG M, LI HT, et al., 2013. Complete chloroplast genome of the genus Cymbidium: lights into the species identification, phylogenetic implications and population genetic analyses [J]. BMC Evol Biol, 13(1): 84.
YANG YJ, HUANG JL, HU H, et al., 2021. Progress on conservation and utilization of Paphiopedilum species in China [J]. J W Chin For Sci, 50(5): 108-112. [楊颖婕, 黄家林, 胡虹, 等, 2021. 中国兜兰属植物种质资源保护和利用研究进展 [J]. 西部林业科学, 50(5): 108-112 .]
ZAVALAPAEZ M, VIEIRA LDN, DEBAURA VA,et al., 2020. Comparative plastid genomics of neotropical Bulbophyllum (Orchidaceae; Epidendroideae) [J]. Front Plant Sci, 11: 799.
ZHANG D, GAO FL, JAKOVLIC I, et al., 2020. PhyloSuite: An integrated and scalable desktop platform for streamlined molecular sequence data management and evolutionary phylogenetics studies [J]. Mol Ecol Resour, 20(1): 348-355.
ZHANG RS, YANG J, HU HL, et al., 2020. A high level of chloroplast genome sequence variability in the Sawtooth Oak Quercus acutissima [J]. Int J Biol Macromol, 152: 340-348.
ZHANG YH, JIA HX, WANG ZB, et al., 2019. Genetic diversity and population structure of Populus yunnanensis [J]. Biodivers Sci, 27(4): 355-365.
ZHANG YJ, LI DZ, 2011. Advances in phylogenomics based on complete chloroplast genomes [J]. Plant Divers Resour, 33(4): 365-375. [张韵洁, 李德铢, 2011. 叶绿体系统发育基因组学的研究进展 [J]. 植物分类与资源学报, 33(4): 365-375.]
( 责任编辑 蒋巧媛 )
收稿日期: 2023-01-25
基金项目: 国家自然科学基金 (31870183, 32270225)。
第一作者: 高鑫祯 (1997-),硕士,研究方向为保护生物学,(E-mail)gaoxinzhen@xtbg.ac.cn。
通信作者: 罗艳,博士,研究员,研究方向为兰科植物多样性与保护,(E-mail)luoyan@xtbg.org.cn。
