小麦穗长QTL鉴定及其遗传分析
2023-12-28姚琦馥周界光王健陈黄鑫杨瑶瑶刘倩闫磊王瑛周景忠崔凤娟蒋云马建
姚琦馥,周界光,王健,陈黄鑫,杨瑶瑶,刘倩,闫磊,王瑛,周景忠,崔凤娟,蒋云,马建
小麦穗长QTL鉴定及其遗传分析
姚琦馥1,周界光2,王健2,陈黄鑫2,杨瑶瑶2,刘倩2,闫磊2,王瑛2,周景忠3,崔凤娟3,蒋云4,马建2
1铜仁学院农林工程与规划学院/贵州省梵净山地区生物多样性保护与利用重点实验室,贵州铜仁 554300;2四川农业大学小麦研究所,成都 611130;3通辽市农牧科学研究所,内蒙古通辽 028015;4四川省农业科学院生物技术核技术研究所,成都 610000
【目的】穗长在决定小麦穗的构造和产量潜力方面具有重要作用。挖掘具有育种利用价值的小麦穗长数量性状位点(quantitative trait loci,QTL),并解析其遗传效应,为分子标记辅助育种提供理论依据。【方法】以自然突变体和川农16构建的198份F6代重组自交系(recombinant inbred lines,RIL)群体(MC群体)作为研究材料,于2020—2021和2021—2022年生长季,在四川温江区、崇州市和雅安市(2021WJ、2022WJ、2021CZ、2022CZ和2021YA)进行试验,对5个环境下的穗长进行表型鉴定。利用基于16K SNP芯片构建的高质量遗传连锁图谱对穗长性状位点进行定位。另外,根据穗长主效QTL侧翼标记的基因型分析主效位点对产量相关性状的遗传效应,从而评估其对产量提升的潜力。【结果】共鉴定到14个控制穗长发育的QTL,主要分布在1A(1个)、1B(1个)、2B(1个)、3D(3个)、4A(1个)、4D(2个)、5A(1个)、5B(1个)、7A(1个)、7B(1个)和7D(1个)染色体。其中,在4个环境及最佳线性无偏预测(best linear unbiased prediction,BLUP)值中被检测到,可解释6.46%—20.12%的表型变异率,定位于1A染色体侧翼标记—间,被视为主效QTL。的正效应位点来源于亲本。在多环境QTL分析结果中也检测到,表明其受环境影响较小,为主效且稳定表达的QTL。的效应在2个具有不同遗传背景的验证群体中得到进一步验证。除旗叶长无显著变化以外,携带正效应位点株系的每穗籽粒数(12.68%)、每穗粒重(14.99%)、千粒重(5.79%)、旗叶宽(2.94%)和小穗数(1.48%)显著增加,花期(0.61%)显著提前,而株高(-6.47%)和有效分蘖数(-36.11%)显著减少。【结论】在1A染色体定位到1个主效且稳定的穗长位点。正效应位点显著提高穗粒数、穗粒重、千粒重和小穗数,具有一定的育种价值。
小麦;16K SNP芯片;QTL;穗长;产量
0 引言
【研究意义】小麦作为主要的谷类作物之一,对全球的粮食安全有至关重要的作用[1]。全球可耕地面积的持续减少和人口的增加导致对小麦的需求剧增[2-4]。因此,提高小麦单产成为保障粮食安全、满足人民谷物需求的主要措施。整个麦穗着生小麦的花序,所以穗作为小麦的繁殖器官,是小麦植株的重要组成部分[5-6],穗长是重要的穗部性状,与单位面积穗粒数密切相关,对小麦产量提高有积极作用[7-9]。因此,挖掘穗长相关的基因/QTL,并研究穗长与农艺性状的遗传关系,可为小麦现代分子育种提供参考[10]。【前人研究进展】迄今为止,在小麦上已经鉴定到许多控制穗长的基因/QTL,Fan等[1]检测到26个和穗长相关的QTL,其中,、和是在试验环境中稳定的QTL,其中主效QTL和(478.65—541.29 Mb)分别可解释7.99%— 16.58%和13.09%—30.43%的表型变异。Li等[2]用分别种植在5个不同生态环境中的双亲群体川麦42/科成麦1和川麦42/川农16进行QTL定位,共鉴定到34个穗密度和穗长共定位的QTL,其中,6个主效QTL至少在4个生态环境中被检测到,BLUP值解释7.13%—33.60%的表型变异。这些主效QTL均定位在5A和6A染色体,分别命名为和,这两个QTL对株高、粒长和千粒重有一因多效的作用。Xu等[11]利用2个骨干亲本碧蚂4号/百农矮抗58构建的包含248个株系的RIL群体为材料,分别在染色体2D、3A、3B、3D、4A、4D、5A、5B、5D、7B和7D中检测到18个穗长相关的QTL,可解释2.04%—22.31%的表型变异。其中,2个稳定的主效QTL(13.25—36.89Mb)和506.65—524.73 Mb在4个生态环境中都可以检测到,其增效位点来自于碧蚂4号,其中可解释11.03%—22.31%的表型变异,可解释4.65%—8.38%的表型变异。陈黄鑫等[12]以矮兰麦和野生二粒小麦LM001构建的F8代重组自交系(recombinant inbred lines,RIL)群体为材料,共检测到17个与穗长相关的QTL,分布在2A、2B、3A、4A、4B、5A和6B染色体上,(—)在5个环境中被检测到,可解释10.41%—16.29%的表型变异,为稳定表达的主效位点,其加性效应来源于父本LM001。Ji等[13]以13F10和川麦42杂交构建的F8代RIL群体为材料,检测到1个与穗长相关的主效QTL(516.60—521.27 Mb),可解释7.88%—26.60%的表型变异,其早前研究表明5A染色体上控制穗长的主效位点也是春化作用基因位点。Xiong等[14]利用和LX987构建的包括400个单株的RIL群体为材料,在3A、4A、5B、6A、6B和7D染色体上共检测到9个控制穗长的QTL,可解释3.00%—22.00%的表型变异,其中染色体6A上的是稳定的主效QTL(—)。姚俭昕等[15]以小偃81和西农1376构建的包含120个株系的F9:10RIL群体为材料,共检测到2个控制穗长的QTL(—)和(—),可分别解释18.34%—22.51%和9.57%— 14.94%的表型变异,这两个主效QTL的增效等位基因均来源于小偃81。其中在基因组上的物理距离为20.77—22.45 Mb,此位点和为同一个基因,而Chai等[16]以Y8679/J411构建的包含191个株系的RIL群体为材料,检测到2个位于2D染色体与株高和穗长相关的主效QTL和,分别解释30.94%—40.63%和31.31%—41.95%的表型变异。其增效位点都来自于亲本J411。并证明此位点为同时控制株高和穗长的一致位点。Xu等[17]以人工合成异源六倍体小麦TAA10和重新合成异源六倍体小麦XX329杂交构建的包括198个单株的F7:8RIL群体为材料,共检测到7个与穗长相关的QTL,其中5个是环境稳定的QTL,可解释7.22%—11.59%的表型变异,其中稳定的主效QTL位点、和LOD值为6.19—12.89,可解释21.77%—33.29%的表型变异,其增效位点来自于TAA10。和分别解释14.75%和7.36%的表型变异,其增效位点来自于XX329。Ma等[18]以南大2419和望水白构建的包含136个株系的RIL群体株系间随机交配构成的永久F2群体为材料,检测到1个穗长相关的主效QTL(—),解释29.70%— 36.30%的表型变异。该QTL在永久F2群体中也可解释31.40%的表型变异。望水白在染色体区间的等位基因增加穗长、小穗数和不育小穗数,减少穗密度,通过选择育种将会产生大穗的材料。水志杰等[19]以西农389×人工合成小麦材料KU98衍生的F7:8RIL群体为试验材料,基于小麦55K SNP芯片对该群体进行基因分型,对小麦穗长和穗宽性状进行了QTL定位,在1A、2D、3A、5A和7B染色体上共检测到10个与穗长性状相关的QTL。许多研究在不同染色体上鉴定出穗长稳定的QTL。Yao等[20]报道大约有350个穗长相关的QTL被鉴定,其中一些主效QTL分布在1D、2A、2B、2D、3A、3D、4A、4B、5A、5B、5D、6A、6B、6D、7A、7B和7D。【本研究切入点】小麦21条染色体上都有影响穗长的基因/QTL[1-2, 11-20]。但由于不同定位群体得出的结论有一定差异,且同时鉴定到的主效QTL数量很少,使得这些QTL在实际生产中的应用十分有限。因此,发掘优异的穗长基因并解析其遗传机制,有利于小麦产量的提升。【拟解决的关键问题】本研究以穗长较长的自然变异株为母本、小麦品种川农16(CN16)为父本构建的F6代RIL群体MC为材料,利用基于16K SNP芯片技术构建的高密度遗传图谱对穗长性状进行遗传定位。结合产量相关性状,分析穗长主效QTL对产量的潜在影响,为分子育种提供理论依据。
1 材料与方法
1.1 试验材料
采用自然突变体和小麦品种CN16杂交构建的含有198个单株的F6代RIL群体MC为研究对象。具有多小花、长穗、多小穗等特点,是一个自然突变的优异材料。CN16小麦品种具有合适的株型。此外,2个具有不同遗传背景的F2分离群体/20828(包含218个单株)和/蜀麦969(包含178个单株)作为验证群体用于主效QTL效应的验证。所有材料均由四川农业大学小麦研究所搜集并提供。
1.2 试验设计
将MC群体及亲本分别于2021、2022年种植于崇州(2021CZ和2022CZ)、温江(2021WJ和2022WJ)和雅安(2021YA)共5个环境。采用单籽粒播种法,按照行长0.75 m、株距0.1 m、行间距0.3 m,将每个株系进行单行播种。种植MC群体的5个环境均进行2次重复试验。/20828和/蜀麦969于2021年种植于崇州。这两个验证群体的种植方式与MC群体的相同。所有种植材料的田间管理根据当地气候进行田间常规管理。
1.3 表型鉴定
当小麦生长至成熟期时,在5个环境对MC群体的穗长表型进行调查。单株主穗的基部到顶部(不包含芒)的长度为穗长。对每个株系选取至少4株长势一致的单株进行表型测量,4个单株的平均值被认定为该株系的穗长。MC群体的株高、穗长、有效分蘖数、千粒重、开花期、每穗粒重、旗叶长、旗叶宽的最佳线性无偏预测(BLUP)值已被用于相关性分析[21-22],而本研究对调控穗长的遗传位点进行鉴定和分析。此外,验证群体/20828和/蜀麦969的穗长表型调查方式与MC群体的相同。
1.4 遗传图谱构建和QTL定位
利用Zhou等[22]通过16K SNP芯片构建的遗传连锁图谱对控制穗长发育的位点进行遗传定位。运用IciMapping 4.2软件中完备区间作图法对单个环境的QTL进行检测,参数设置为Step=0.1 cM、PIN=0.001和LOD≥2.5。同时,运用IciMapping 4.2软件对QTL进行多环境分析,参数设置为Step=0.1 cM、PIN=0.001和LOD≥5。按照国际遗传命名规则(https://wheat.pw. usda.gov/ggpages/wgc/98/Intro.htm)对检测到的QTL命名。利用WheatOmics v1.0公布的中国春参考基因组(CS RefSeq v2.1)获得QTL侧翼标记序列的物理位置。
1.5 主效QTL验证
基于QTL的定位结果,将与主效QTL连锁的SNP标记转化为kompetitive allele specific PCR(KASP)标记。具体的转化方法参考Zhou等[22]的描述。KASP标记包含2条特异引物(5′-GAAGGTGACCAAGTT CATGCTGCAACATGTATGTCCGACCTa-3′和5′-GA AGGTCGGAGTCAACGGATTGCAACATGTATGTCCGACCTg-3′)与1条通用引物(5′-GCAAATGGTGC GTTGATGGT-3′),KASP引物的合成由北京擎科生物科技有限公司完成。利用开发的KASP引物对/蜀麦969和/20828 F2分离群体的每个单株进行基因型分型。最后,对携带正效应位点和不携带正效应位点的纯合单株的穗长进行统计分析,而基因型为杂合类型的单株不参与统计分析。
1.6 统计分析
穗长表型数据的多环境方差分析(analysis of variance,ANOVA)通过IciMapping 4.2软件获得。使用SAS 9.4软件对穗长的BLUP值和广义遗传力(2)进行计算。用Excel 2019对穗长表型数据的平均值进行计算。运用Origin 2021软件绘制穗长的频率分布图。利用IBM SPSS Statistics 20进行穗长表型数据的描述性统计分析、独立样本检验和显著性分析。
2 结果
2.1 表型分析
通过对5个种植环境下的MC群体穗长表型值进行鉴定。亲本的穗长均显著高于亲本CN16(表1和图1,<0.05)。MC群体穗长2为0.70,表明其主要受遗传因子影响。RIL中穗长表型值范围为7.05—18.68 cm,在不同环境中都存在超亲分离现象,且其频率分布呈近似正态分布(表1和图2),表明MC群体穗长性状具有典型的数量遗传特点。ANOVA分析表明环境、基因型和环境 × 基因型互作对多环境的穗长有显著影响(表2)。
2.2 相关性分析
根据BLUP值对MC群体在不同环境的穗长表型进行了相关性分析,其相关系数介于0.15—0.81(图2)。除2021CZ和2021YA之间不存在显著相关外,其余环境之间均表现出极显著的正相关(<0.01)。

表1 MC群体亲本及其RIL穗长的表型分布
WJ:温江;CZ:崇州;YA:雅安;BLUP:最佳线性无偏预测;N:无重复值;*和**:在0.05和0.01水平差异显著。下同
WJ: Wenjiang; CZ: Chongzhou; YA: Ya’an; BLUP: Phenotype values based on the best linear unbiased prediction;N: no duplicate values; * and **: Significant difference at level 0.05 and 0.01. the same as below
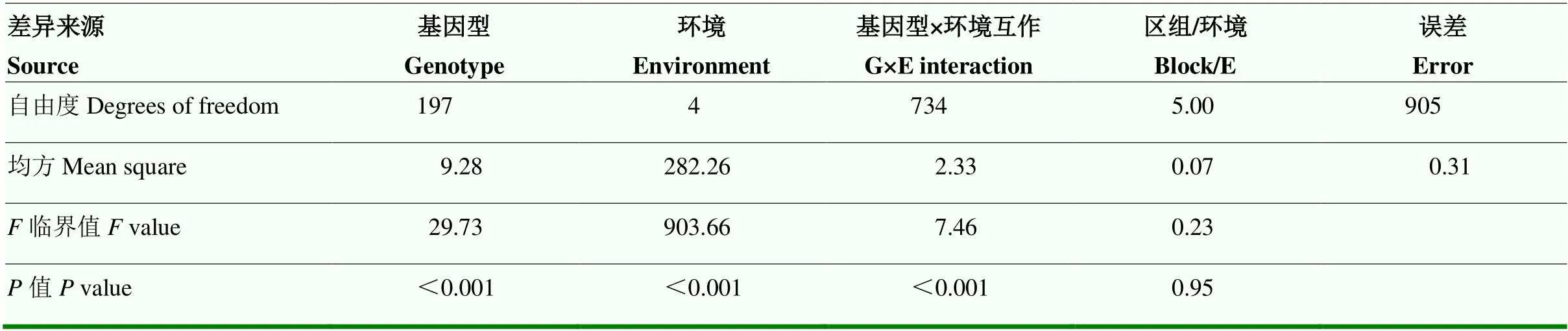
表2 MC群体穗长的方差分析

bar=2 cm
同时评估了MC群体穗长和其他产量相关性状的相关性(表3)。结果显示,穗长与小穗数、每穗籽粒数和每穗粒重之间存在极显著的正相关(<0.01),相关系数分别为0.43、0.64和0.50。穗长和有效分蘖数之间存在极显著的负相关(<0.01),相关系数为-0.45。穗长与株高、千粒重、旗叶长、旗叶宽和开花期之间无显著相关性。
2.3 QTL分析
根据Zhou等[22]构建的遗传连锁图谱和5个环境的穗长表型数据,共鉴定到14个控制穗长发育的QTL,主要分布在1A(1个)、1B(1个)、2B(1个)、3D(3个)、4A(1个)、4D(2个)、5A(1个)、5B(1个)、7A(1个)、7B(1个)和7D(1个)染色体(表4)。其中,在4个环境及BLUP值中被检测到,可解释6.46%—20.12%的表型变异率,定位于1A染色体侧翼标记和之间,被视为主效QTL。的正效应位点来源于突变体亲本。其余13个QTL均在2个环境及以下被检测到,最高可解释8.40%的表型变异率,都被视为微效QTL。多环境QTL分析结果中同样检测到,且可解释表型变异率大于10%,表明其受环境影响较小,为主效且稳定的QTL(表5)。

表3 MC群体穗长与产量相关性状的相关性
2.4 穗长主效QSl.sau.1A遗传分析
根据穗长主效侧翼标记的基因型,将MC群体分为携带增效位点的株系和不携带增效位点的株系,并对这2类株系进行分析(图3)。结果显示,除2021YA之外的所有环境中,携带正效应位点株系的穗长均极显著高于没有携带正效应位点的株系,最高能显著提高11.53%的穗长(图3)。

**:在0.01水平差异显著。下同 **: Significant difference at level 0.01. the same as below
2.5 QSl.sau.1A位点的验证
为了验证的遗传效应,将其连锁的SNP标记转化为KASP标记,序列见材料方法1.5部分。KASP引物在/20828和/蜀麦969群体中的基因型分型结果显示,在/20828和/蜀麦969群体中,携带正效应位点的单株分别有56株和30株,不携带正效应位点的单株分别有55株和52株(图4)。通过对这些单株所对应的穗长进行统计分析,发现在/20828和/蜀麦969群体中携带正效应位点的单株比不携带正效应位点的单株分别极显著增加7.42%和9.99%的穗长(<0.01,图4)。结果表明,的确是一个具有显著增加穗长效应的主效QTL。
2.6 穗长主效QTL QSl.sau.1A对产量相关性状的影响
进一步根据穗长主效QTL侧翼标记的基因型分析其对产量相关性状的影响。除旗叶长无显著变化外,携带正效应位点株系的每穗籽粒数(12.68%)、每穗粒重(14.99%)、千粒重(5.79%)和旗叶宽(2.94%)极显著提高,开花期(0.61%)极显著提前,小穗数(1.48%)显著增加,而株高(-6.47%)和有效分蘖数(-36.11%)极显著减少(图5)。暗示正效应位点对于产量可能有着积极的影响,具有较大的育种价值。

表4 MC群体穗长相关的QTL
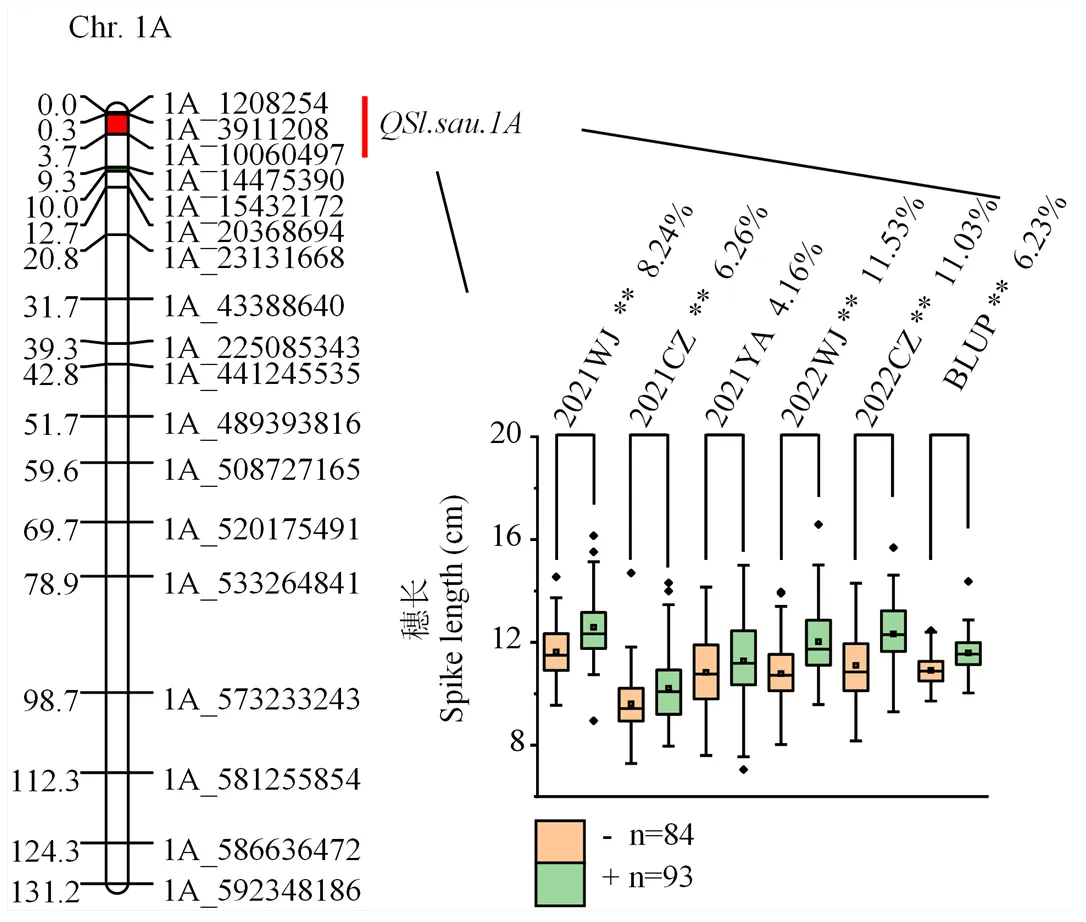
Chr.:染色体;+和−:携带和不携带QSl.sau.1A正效应位点的株系;n:株系数。下同
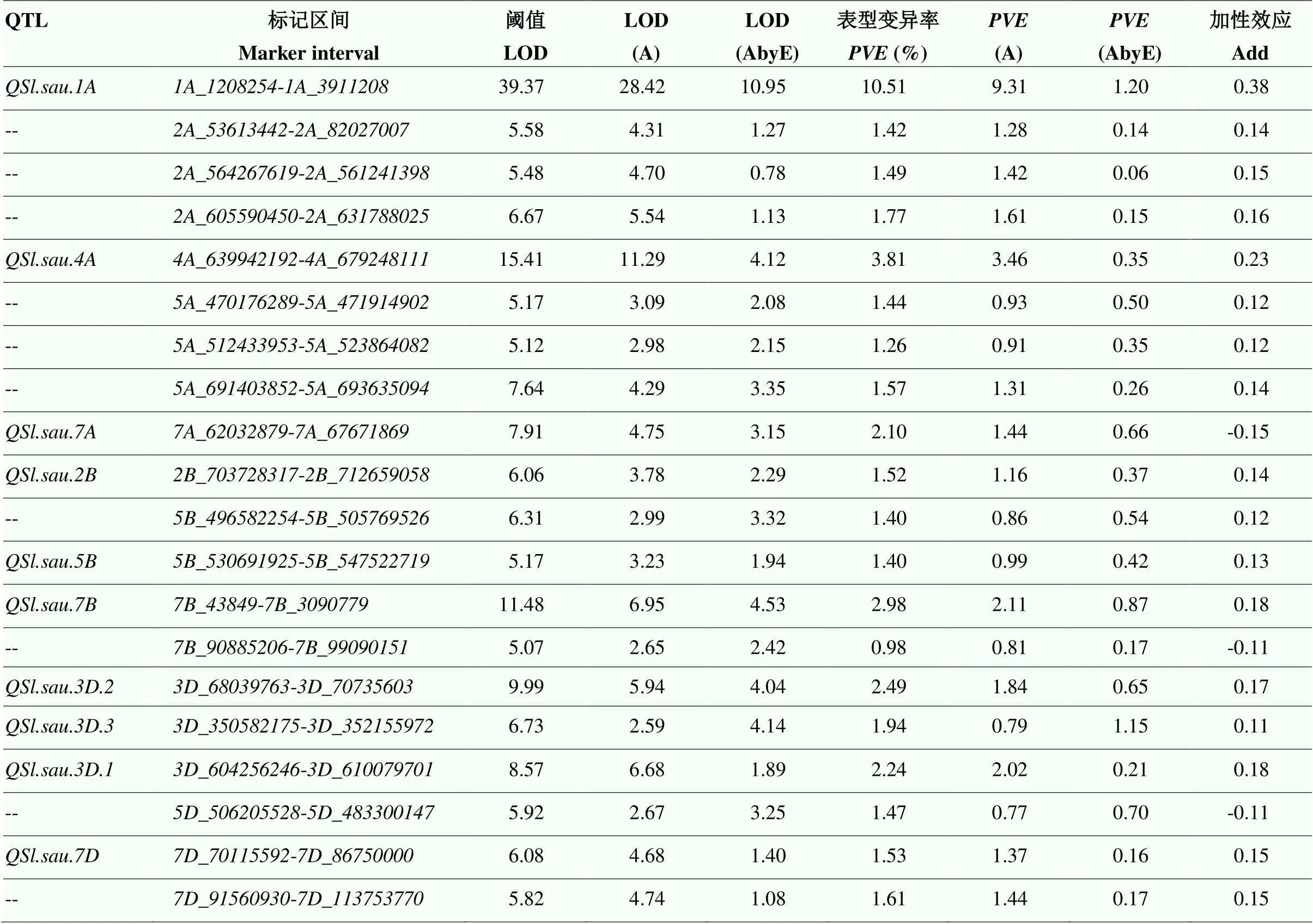
表5 MC群体穗长相关的多环境QTL
LOD(A):加性和显性效应的阈值;LOD(AbyE):环境对加性和显性效应影响的阈值;(A):加性和显性效应的表型变异率;(AbyE):环境对加性和显性效应影响的表型变异率;--:与表4中的穗长QTL未对应上的QTL
LOD(A): Logarithm of the odds of additive and dominant effects; LOD(AbyE): Logarithm of the odds of the influence of environment on additive and dominant effects;(A): Phenotypic variation explained of additive and dominant effects;(AbyE): Phenotypic variation explained of environmental impact on additive and dominant effects; --: The QTL not corresponding to the spike length QTL in Table 4
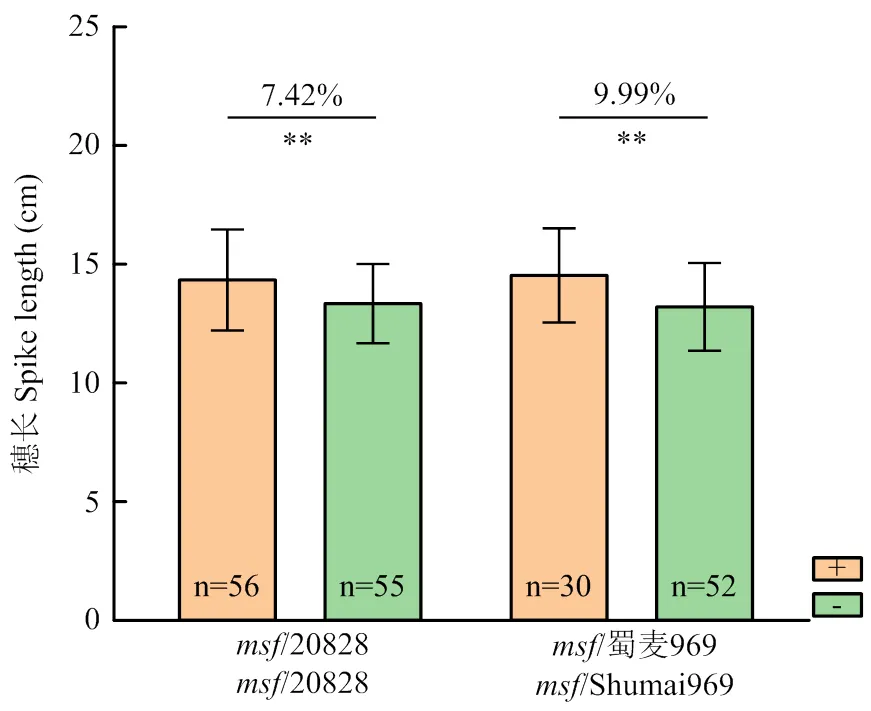
+和−:携带和不携带对应QSl.sau.1A正效应位点的单株。下同
3 讨论
3.1 与前人研究的比较分析
在CS RefSeq v2.1中检索本研究和前人研究中穗长QTL的侧翼标记序列,通过比对其物理位置来确定QTL是否重叠[23-24]。水志杰等[19]检测到1个与穗长性状相关的主效QTL,位于1A染色体标记区间—(51.96— 54.49 Mb)。Kumar等[8]在1A染色体短臂检测到1 个与穗长性状相关的位于标记区间—(23.57—67.92 Mb)。本研究鉴定到1个控制穗长发育的主效定位于1A染色体上的侧翼标记—(1.21—11.43 Mb)。其物理区间无重叠,可能为新的QTL,由于不同材料间遗传背景的差异,所以需对进行候选基因克隆来进一步确定其是否为新的稳定遗传的主效QTL。
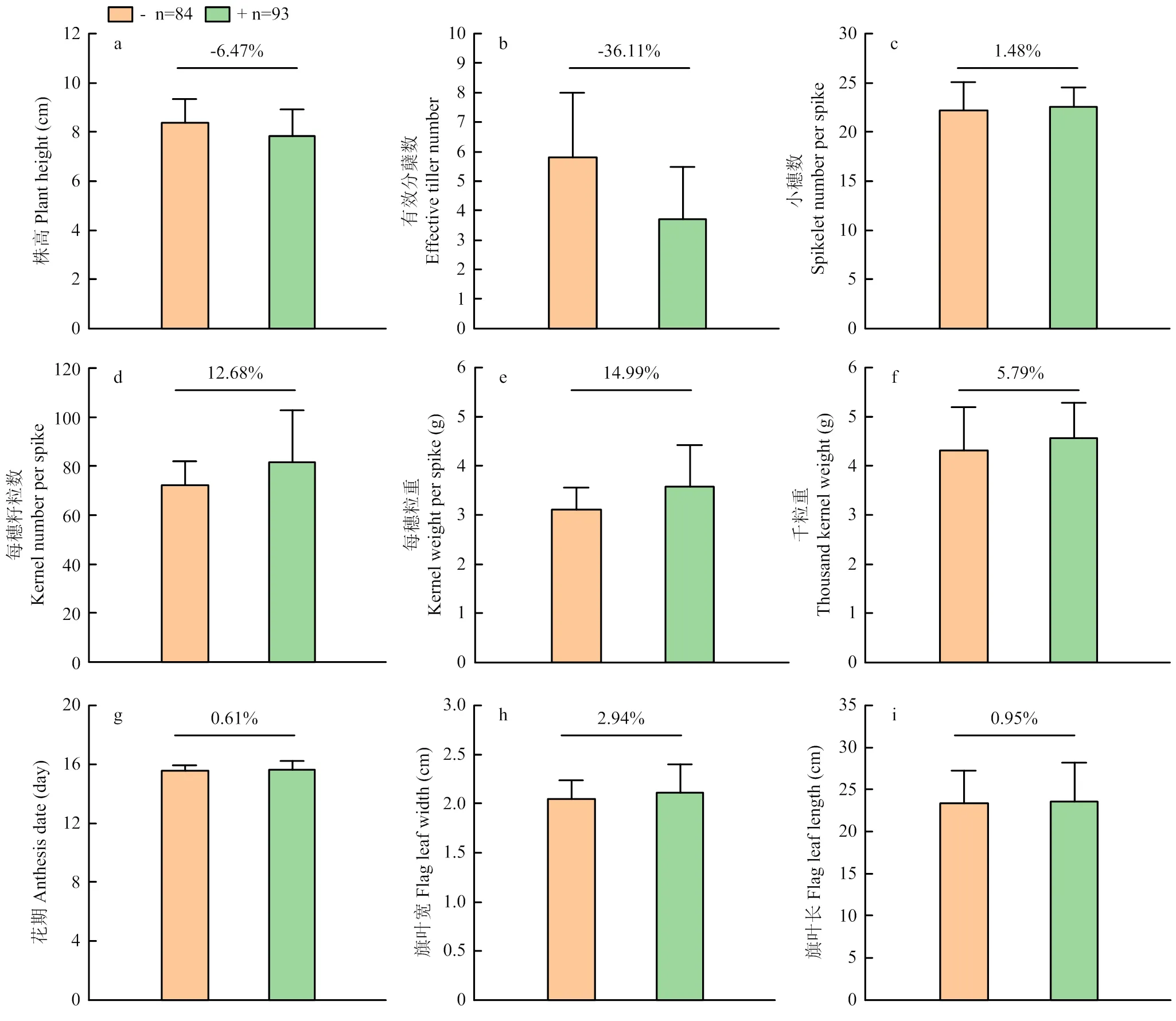
*:在0.05水平差异显著;a—i:QSl.sau.1A对于其他农艺性状的影响
3.2 穗长与产量相关性状的关系
穗长是受多基因控制的小麦重要农艺性状。与产量三要素亩穗数、穗粒数和粒重之间存在一定关系[25],深入研究小麦穗长性状对提高小麦产量有重要意义[26-27]。此外,小麦为异源六倍体生物,基因组大,遗传关系复杂,很难阐释清楚小麦产量相关性状的遗传关系。增加穗长而未影响穗密度需提高可育穗粒数来增加产量。因此,在育种过程中要协调穗长与其他产量性状间的关系,才能达到更大的收获指数[28]。基于此,本研究分别用种植在5个生态环境的MC群体评价穗长和产量相关性状间的相关性。正效应位点可显著增加每穗籽粒数、每穗粒重、千粒重、小穗数和旗叶宽,促进开花期提前,显著减少有效分蘖数和株高。李涛等[29]研究表明,当同时聚合和时,在川麦42×川农16 RIL群体中株高、穗长和千粒重分别增加13.10%、8.00%和7.50%,对穗粒数基本没有影响;而在川麦42×川麦39 RIL群体中,株高和千粒重分别增加7.40%和4.70%,而穗长和穗粒数基本不受影响。CUI等[30]基于2个定位群体的条件QTL分析株高与其组成成分穗长和节间长之间的遗传关系,结果表明,在QTL水平上,穗长对株高的贡献最小。唐华苹等[10]基于穗长的条件QTL结果,利用与穗长主效位点、和紧密连锁的侧翼标记基因型分析它们对其他农艺性状的影响,表明对于株高、穗茎长、每穗小穗数和千粒重没有显著影响,对千粒重有显著影响,对株高和穗茎长有极显著影响。Ji等[13]研究发现穗长与籽粒长呈极显著正相关(<0.01),与穗密度呈极显著负相关(<0.01)。以上研究表明,穗长与产量性状之间的关系在不同背景下表现并不一致,暗示正效应位点对于产量提高可能有着积极的影响,具有潜在的育种价值。为了能真正评价该位点的育种效应,后期应该设置小区试验进一步评价是否对单位面积穗数、千粒重以及单位面积产量有实际效应,以明确其真实效应。
3.3 QSl.sau.1A区间内候选基因预测
基于CS RefSeq v2.1,的侧翼标记1和的区间大小为10.22 Mb,区间内共包含414个基因,其中,高可信基因202个。根据202个高可信基因的功能注释,预测了8个(、、、、、、、)可能影响穗长发育的基因。
4 结论
在1A染色体定位到1个主效且稳定地控制穗长发育的QTL,定位于侧翼标记—(1.21—11.43 Mb)。正效应位点除对旗叶长无显著影响外,可显著增加每穗粒数、每穗粒重、千粒重、小穗数和旗叶宽,促进开花期提前,显著减少有效分蘖数和株高。其中,的侧翼标记和的区间大小为10.22 Mb,区间内预测了8个(、、、、、、、)可能影响穗长发育的基因。
[1] Fan X L, Chi F, Ji J, Zhang W, Zhao X Q, Liu J J, Meng D Y, Tong Y P, Wang T, Li J M. Dissection of pleiotropic QTL regions controlling wheat spike characteristics under different nitrogen treatments using traditional and conditional QTL mapping. Frontiers in Plant Science, 2019, 10: 187.
[2] Li T, Deng G B, Su Y, Yang Z, Tang Y Y, Wang J H, Qiu X B, Pu X, Li J, Liu Z H, Zhang H L, Liang J J, Yang W Y, Yu M Q, Wei Y M, Long H. Identifcation and validation of two major QTLs for spike compactness and length in bread wheat (L.) showing pleiotropic effects on yield-related traits. Theoretical and Applied Genetics, 2021, 134: 3625-3641.
[3] Butterworth M H, Semenov M A, Barnes A, Moran D, West J S, Fitt B D L. North-South divide: contrasting impacts of climate change on crop yields in Scotland and England. Journal of the Royal Society Interface, 2010, 7(42): 123-130.
[4] Su Z Q, Jin S J, Lu Y, Zhang G R, Chao S, Bai G H. Single nucleotide polymorphism tightly linked to a major QTL on chromosome 7A for both kernel length and kernel weight in wheat. Molecular Breeding, 2016, 36(2): 15.
[5] Faris J D, Zhang Z C, Garvin D F, Xu S S. Molecular and comparative mapping of genes governing spike compactness from wild emmer wheat. Molecular Genetics and Genomics, 2014, 289(4): 641-651.
[6] Koppolu R, Schnurbusch T. Developmental pathways for shaping spike inflorescence architecture in barley and wheat. Journal of Integrative Plant Biology, 2019, 61(3): 278-295.
[7] Jantasuriyarat C, Vales M I, Watson C J W, Riera- Lizarazu O. Identification and mapping of genetic loci affecting the free-threshing habit and spike compactness in wheat (L.). Theoretical and Applied Genetics, 2004, 108(2): 261-273.
[8] Kumar N, Kulwal P L, Balyan H S, Gupta P K. QTL mapping for yield and yield contributing traits in two mapping populations of bread wheat. Molecular Breeding, 2007, 19(2): 163-177.
[9] Wu X Y, Cheng R R, Xue S L, Kong Z X, Wan H S, Li G Q, Huang Y L, Jia H Y, Jia J Z, Zhang L X, Ma Z Q. Precise mapping of a quantitative trait locus interval for spike length and grain weight in bread wheat (L.). Molecular Breeding,2014, 33(1): 129-138.
[10] 唐华苹, 陈黄鑫, 李聪, 苟璐璐, 谭翠, 牟杨, 唐力为, 兰秀锦, 魏育明, 马建. 基于55K SNP芯片的普通小麦穗长非条件和条件QTL分析. 中国农业科学, 2022, 55(8): 1492-1502.doi: 10.3864/j. issn.0578-1752.2022.08.002.
Tang H P, Chen H X, Li C, Gou L L, Tan C, Mu Y, Tang L W, Lan X J, Wei Y M, Ma J. Unconditional and Conditional QTL analysis of wheat spike length in common wheat based on 55K SNP array. Scientia Agricultura Sinica, 2022, 55(8): 1492-1502. doi: 10. 3864/j.issn.0578-1752.2022.08.002.(in Chinese)
[11] Xu X, Li X J, Zhang D H , Zhao J S, Jiang X L, Sun H L, Ru Z G. Identification and validation of QTLs for kernel number per spike and spike length in two founder genotypes of wheat. BMC Plant Biology, 2022, 22(1): 146.
[12] 陈黄鑫, 李聪, 吴坤燕, 王岳, 牟杨, 唐华苹, 唐力为, 兰秀锦, 马建. 四倍体小麦株高和穗长性状的QTL定位及其遗传效应分析. 麦类作物学报, 2022, 42(7): 799-807.
Chen H X, Li C, Wu K Y, Wang Y, Mu Y, Tang H P, Tang L W, Lan X J, Ma J. Detection of QTLs for plant height and spike length in tetraploid wheat and analysis of their genetic effect. Journal of Triticeae Crops, 2022, 42(7): 799-807. (in Chinese)
[13] Ji G S, Xu Z B, Fan X L, Zhou Q, Yu Q, Liu X F, Liao S M, Feng B, Wang T. Identification of a major and stable QTL on chromosome 5A confers spike length in wheat (L.). Molecular Breeding, 2021, 41(9): 56.
[14] Xiong H C, Li Y T, Guo H J, Xie Y D, Zhao L S, Gu J Y, Zhao S R, Ding Y P, Liu L X. Genetic mapping by integration of 55K SNP array and KASP markers reveals candidate genes for important agronomic traits in hexaploid wheat. Frontiers in Plant Science, 2021, 12: 628478.
[15] 姚俭昕, 张传量, 宋晓朋, 许小宛, 邢永锋, 吕栋云, 宋鹏博, 杨孟于, 孙道杰. 基于90K芯片的小麦穗长和旗叶长QTL分析. 麦类作物学报, 2020, 40(11): 1283-1289.
Yao J X, Zhang C L, Song X P, Xu X W, Xing Y F, LÜ D Y, Song P B, Yang M Y, Sun D J. QTL analysis of wheat spike length and flag leaf length based on 90k SNP assay. Journal of Triticeae Crops, 2020, 40(11): 1283-1289. (in Chinese)
[16] Chai L L, Chen Z Y, Bian R L, Zhai H J, Cheng X J, Peng H R, Yao Y Y, Hu Z R, Xin M M, Guo W L, Sun Q X, Zhao A J, Ni Z F. Dissection of two quantitative trait loci with pleiotropic effects on plant height and spike length linked in coupling phase on the short arm of chromosome 2D of common wheat (L.). Theoretical and Applied Genetics, 2018, 131(12): 2621-2637.
[17] Xu H W, Zhang R Q, Wang M M, Li L H, Yan L, Wang Z, Zhu J, Chen X Y, Zhao A J, Su Z Q, Xing J W, Sun Q X, Ni Z F. Identification and characterization of QTL for spike morphological traits, plant height and heading date derived from the D genome of natural and resynthetic allohexaploid wheat. Theoretical and Applied Genetics, 2022, 135(2): 389-403.
[18] Ma Z Q, Zhao D M, Zhang C Q, Zhang Z Z, Xue S L, Lin F, Kong Z X, Tian D G, Luo Q Y. Molecular genetic analysis of five spike-related traits in wheat using RIL and immortalized F2populations. Molecular Genetics and Genomics, 2007, 277(1): 31-42.
[19] 水志杰, 安沛沛, 刘天相, 吴洪启, 刘乐, 史雪, 王中华. 利用人工合成小麦RIL群体进行小麦穗长和穗宽性状的QTL分析. 麦类作物学报, 2020, 40(6): 656-664.
Shui Z J, An P P, Liu T X, Wu H Q, Liu L, Shi X, Wang Z H. QTL analysis of spike length and width using RIL population of synthetic wheat. Journal of Triticeae Crops, 2020, 40(6): 656-664. (in Chinese)
[20] Yao H N, Xie Q, Xue S L, Luo J, Lu J K, Kong Z X, Wang Y P, Zhai W L, Lu N, Wei R, Yang Y, Han Y Z, Zhang Y, Jia H Y, Ma Z Q.on chromosome 7D of wheat (L.) regulates both head length and spikelet number. Theoretical and Applied Genetics, 2019, 132(6): 1789-1797.
[21] 姚琦馥, 陈黄鑫, 周界光, 马瑞莹, 邓亮, 谭陈芯雨, 宋靖涵, 吕季娟, 马建. 基于16K SNP芯片的小麦株高QTL鉴定及其遗传分析. 中国农业科学, 2023, 56(12): 2237-2248.doi: 10.3864/j.issn.0578- 1752.2023.12.001.
Yao Q F, Chen H X, Zhou J G, Ma R Y, Deng L, Tan C X Y, Song J H, Lü J J, Ma J. QTL identification and genetic analysis of plant height in wheat based on 16K SNP array. Scientia Agricultura Sinica, 2023, 56(12): 2237-2248. doi: 10. 3864/j.issn.0578-1752.2023. 12.001.(in Chinese)
[22] Zhou J G, Li W, Yang Y Y, Xie X L, Liu J J, Liu Y L, Tang H P, Deng M, Xu Q, Jiang Q F, Chen G Y, Qi P F, Jiang Y F, Chen G D, He Y J, Ren Y P, Tang L W, Gou L L, Zheng Y L, Wei Y M, Ma J. A promising QTLlikely superior tofor the number of spikelets per spike of wheat shows no adverse effects on yield-related traits. Theoretical and Applied Genetics, 2023, 136(9): 181.
[23] INTERNATIONAL WHEAT GENOME SEQUENCING CONSORTIUM(IWGSC). Shifting the limits in wheat research and breeding using a fully annotated reference genome. Science, 2018, 361(6403): eaar7191.
[24] Ma S W, Wang M, Wu J H, Guo W L, Chen Y M, Li G W, Wang Y P, Shi W M, Xia G M, Fu D L, Kang Z S, Ni F. WheatOmics: A platform combining multiple omics data to accelerate functional genomics studies in wheat. Molecular Plant, 2021, 14(12): 1965-1968.
[25] Wang M X, Lu J, Liu R, Li Y F, Ao D H, Wu Y, Zhang L. Identification and validation of a major quantitative trait locus for spike length and compactness in the wheat (L.) line chuanyu12D7. Frontiers in plant science, 2023, 14: 1186183.
[26] Li W L, Nelson J C, Chu C Y, Shi L H, Huang S H, Liu D J. Chromosomal locations and genetic relationships of tiller and spike characters in wheat. Euphytica, 2002, 125(3): 357-366.
[27] 王梦可, 赵德辉, 曾占奎, 陈鹏, 张雷宜, 兰彩霞, 刘瑞芳, 王春平. 小麦穗长性状基因的发掘与标记开发. 西北农林科技大学学报(自然科学版), 2023, 51(2): 11-21.
Wang M K, Zhao D H, Zeng Z K, Chen P, Zhang L Y, Lan C X, Liu R F, Wang C P. Gene detection and marker development of spike length traits in wheat. Journal of Northwest A&F University (Natural Science Edition), 2023, 51(2): 11-21. (in Chinese)
[28] Zhang X Y, Jia H Y, Li T A, Wu J Z, Nagarajan R, Lei L, Powers C, KAN C C, Hua W, Liu Z Y, ChEN C, Carver B F, Yan L L.modifies spike architecture and enhances grain yield in wheat. Science, 2022, 376(6589): 180-183.
[29] 李涛, 陆炳, 李俊, 邓光兵, 张海莉, 梁俊俊, 余懋群, 杨武云, 龙海. 2个小麦株高QTL位点验证及其对产量相关性状的效应分析. 西南农业学报, 2019, 32(3): 476-483.
Li T, Lu B, Li J, Deng G B, Zhang H L, Liang J J, Yu M Q, Yang W Y, Long H. Validation of two plant height QTLs and their effects on yield-related traits in common wheat. Southwest China Journal of Agricultural Sciences, 2019, 32(3): 476-483. (in Chinese)
[30] Cui F, LI J, Ding A M, Zhao C H, Wang L, Wang X Q, Li S S, Bao Y G, Li X F, Feng D S, Kong L R, Wang H G. Conditional QTL mapping for plant height with respect to the length of the spike and internode in two mapping populations of wheat. Theoretical and Applied Genetics, 2011, 122(8): 1517-1536.
Identification and Genetic Analysis of QTL for Spike Length in Wheat

1College of Agroforestry Engineering and Planning, Tongren University/Guizhou Key Laboratory of Biodiversity Conservation and Utilization in the Fanjing Mountain Region, Tongren 554300, Guizhou;2Triticeae Research Institute, Sichuan Agricultural University, Chengdu 611130;3Tongliao Institute of Agriculture and Animal Husbandry Sciences, Tongliao 028015, Inner Mongolia;4Institute of Biotechnology and Nuclear Technology Research, Sichuan Academy of Agricultural Sciences, Chengdu 610000
【Objective】Spike length (SL) plays an important role in determining spike structure and yield potential of wheat. Quantitative trait loci (QTL) for spike length were excavated and their genetic effects were further analyzed to provide theoretical basis for molecular breeding.【Method】This study consisted of a population of 198 F6recombinant inbred lines (RIL) derived from the cross between the natural mutantand the cultivar Chuannong 16 (MC population). The MC population and its parents were planted in five different environments including Wenjiang in 2021 and 2022 (2021WJ and 2022WJ); Chongzhou in 2021 and 2022 (2021CZ and 2022CZ); and Ya’an in 2021 (2021YA) for spike length measurement. The 16K SNP chip-based constructed high-quality and high-density genetic linkage maps were used to map QTL for spike length. Additionally, the genotype of the flanking markers for the major spike length QTL was used to analyze its genetic effect on yield-related traits and thus to evaluate its potentiality for yield improvement.【Result】A total of 14 QTL for spike length were identified and they were mainly distributed on chromosomes 1A (one), 1B (one), 2B (one), 3D (three), 4A (one), 4D (two), 5A (one), 5B (one), 7A (one), 7B (one), and 7D (one). Among them,was detected in four environments and the best linear unbiased prediction (BLUP) value, explained 6.46% to 20.12% of the phenotypic variation, and thus was regarded as a major QTL. The positive allele atcame from the parental line. QTL analysis across multiple environments also detected, indicating it exhibits minimal environmental influence and represents a major and stably expressed QTL. The effect ofwas successfully verified in two populations with different genetic backgrounds. Genetic effects analysis showed that the positive allele ofshowed a significant effect on improving grain number per spike (12.68%), grain weight per spike (14.99%), 1000-grain weight (5.79%), flag leaf width (2.94%), spikelet number (1.48%), and flowering date (0.61%), and a significant effect of reducing plant height (-6.47%) and effective tiller number (-36.11%).【Conclusion】A major and stably expressed spike length QTL,, was detected on chromosome 1A. Its positive allele significantly increased grain number per spike, grain weight per spike, thousand grain weight, and spikelet number per spike, indicating its great breeding value.
wheat; 16K SNP array; QTL; spike length; yield

10.3864/j.issn.0578-1752.2023.24.002
2023-08-11;
2023-10-10
贵州省科技计划基础研究项目(黔科合基础-ZK[2021]一般131)、铜仁市科技计划基础研究项目(铜市科研〔2023〕36号)
姚琦馥,E-mail:yaoqifu@126.com。周界光,E-mail:351062153@qq.com。姚琦馥和周界光为同等贡献作者。通信作者马建,E-mail:jianma@sicau.edu.cn。通信作者蒋云,E-mail:m13438880787@163.com
(责任编辑 李莉)
