大豆和苜蓿基因置换比较分析
2023-11-10高昕彤王佳琪张晓敏李晓楠
高昕彤, 王佳琪, 张晓敏, 张 岩, 李晓楠, 张 岚
(华北理工大学 生命科学学院,河北 唐山 063210)
大豆(Glycinemax)中富含蛋白质,是世界上最重要的豆类,在世界各地广泛种植,是重要的经济作物和油料作物,也是我国重要的粮食作物之一[1].苜蓿是优质的牧草资源,可以提供廉价、营养丰富的饲料,同时还具有食用、药用等多种价值[2].
多倍化事件在被子植物进化过程中普遍发生,在被子植物进化和多样性中发挥重要作用[3-4].在豆科植物中,多倍化可影响其物种多样化[5]及豆科植物与根瘤菌共生固氮的效果[6].与其他真双子叶植物分化后,豆科植物经历了一次豆科共同四倍体事件(legume-common tetraploid,LCT)[7-8],使豆科植物基因组中产生了大量重复基因.多倍化产生的大量重复基因往往会使植物基因组不稳定,从而导致基因置换事件的发生[9-11].
基因置换(gene conversion)指遗传信息从一个基因向其旁系同源基因单向传递,是高度相似的序列在同源染色体间的一种非法重组[12].基因置换有利于保持基因间的相似性,为植物进化提供原材料[13].在禾本科植物基因置换的研究中表明,基因置换能加快物种进化[14],促进分化[15].目前在大豆和野生大豆,以及花生的相关研究中发现,靠近染色体两端的基因更容易发生基因置换[16-17],且有些功能基因偏向于发生基因置换[17].全基因组加倍(whole-genome duplication)事件成为豆科植物基因间发生基因置换的材料来源和重要契机.本研究采用比较基因组学分析方法,推断了大豆、野生大豆(Glycinesoja)、蒺藜苜蓿(Medicagotruncatula)以及新测序的南苜蓿(Medicagopolymorpha)[18]、花苜蓿(Medicagoruthenica)[19]、紫花苜蓿(Medicagosativa)[20]基因组中重复基因间的基因置换事件,这有助于解析豆科植物基因组的进化机制和分化问题.
大豆和苜蓿都属于豆科植物,大多数豆科植物能够与根瘤菌互作形成根瘤,从而进行共生固氮[21].NIN-like protein(NLP)转录因子对植物的根瘤固氮能力至关重要[22],研究基因置换对NLP基因家族的影响,能够为研究植物的根瘤发育、固氮机制提供新的思路.
1 材料和方法
1.1 物种基因组数据
从Phytozome(https://phytozome-next.jgi.doe.gov/)数据库中下载大豆、野生大豆、蒺藜苜蓿的数据;从Figshare数据库中下载紫花苜蓿的数据(https://figshare.com/articles/dataset/genome_fasta_sequence_and_annotation_files/12327602)和花苜蓿的数据(https://figshare.com/articles/dataset/Genome_assembly_and_annotation_files_of_Medicago_ruthenica/12726932);从BIGD(https://ngdc.cncb.ac.cn/gwh/Assembly/17540/show)数据库下载南苜蓿的数据.数据主要包括基因在染色体上的注释文件、核苷酸序列文件及氨基酸序列文件.
1.2 基因共线性分析
将选择的所有物种基因组进行双向BLASTP比对(score>100,E-value<1e-5).编写程序,根据基因在染色体上的位置,绘制基因组内、基因组间的同源结构点阵图.并使用共线性分析工具ColinearScan,筛选物种基因组内、基因组间的共线基因对,其中最大间隙设置为50个间隔基因.然后以蒺藜苜蓿为参考,构建豆科植物间的共线性基因列表,并绘制多基因组联合比对图谱.
1.3 基因置换推断
根据共线性分析结果,分别将6个豆科植物基因组内由LCT事件产生的旁系同源基因,以及这些旁系同源基因在基因组间分别对应的直系同源基因进行比较,获得基因间的对应关系,即同源基因四联子.根据基因置换可能导致的基因树的拓扑结构,使用Bioperl中的模块和多序列比对工具ClustalW,对得到的同源基因四联子的基因序列进行同源性比对,将比对结果中空位占比大于50 %及氨基酸一致性小于40 %的同源基因四联子删除.使用Nei-Gojobori方法计算同义核苷酸替换率(Ks)比较序列间的相似性,再构建同源基因四联子的系统发育树,分析其拓扑结构,使用Bootstrap值为1 000的检验方法得到基因置换的置信度,推断可能的基因置换事件.基因置换会使旁系同源基因间的相似性大于直系同源基因间的相似性,可能会导致部分受体序列被供体序列替代,即部分基因置换,或受体序列全部被供体序列替代,即全基因置换.
1.4 基因表达差异分析
从NCBI网站下载各物种的原始RNA-seq数据,包括大豆的根(SRR17276224)、野生大豆的根和叶(SRR12769149,SRR12549549)、花苜蓿的根和叶(SRR10488430,SRR12407154)、紫花苜蓿的叶(SRR9026566)、蒺藜苜蓿的根和叶(SRR13299546,SRR16004944).首先使用测序数据过滤工具Trimomatic对各物种原始RNA-seq reads进行处理,去除接头序列和低质量的reads.再使用带有默认参数的序列比对软件HISAT2,将处理后的reads比对到各物种基因组.最后使用StringTie计算基因表达丰度值,设置参数为“-e -A”,得到基因丰度信息.删除未表达的基因,计算置换基因对与非置换基因对的表达差异.
1.5 基因本体论分析
使用InterProScan软件分别鉴定了6个豆科植物基因组内基因的GO分类,并使用在线网站WEGO分别对置换基因和非置换基因的GO注释结果进行可视化.
1.6 NLP基因家族相关分析
从Pfam数据库下载NLP家族序列结构域的隐马尔可夫模型(hidden-markov model,HMM)(PF02042),使用HMMER3.0对氨基酸序列文件进行检索(E-value<1e-5).通过SMART,CDD等在线数据库对获得的氨基酸序列进行结构域分析,确定基因家族成员.
根据1.4中得到的基因表达丰度,使用TBtools绘制聚类热图.
2 结果与分析
2.1 多基因组联合比对图谱
以豆科模式植物蒺藜苜蓿为参考,根据基因组间的同源关系,构建了6种豆科植物基因组间的共线基因图谱.图谱展示了由豆科植物共同经历的LCT事件,以及大豆单独经历的四倍体事件(soybean-specifically tetraploid,SST)[7-8]产生的重复基因.根据直系同源基因的保留情况发现,在豆科植物进化过程中,产生了大量重复基因,并且在苜蓿和苜蓿、苜蓿和大豆之间,这些重复基因间保持着良好的直系同源关系.这些重复基因为基因置换的发生提供了材料来源(图1).
2.2 推断基因置换
根据基因间的共线性关系,分别鉴定了6个豆科植物基因组内由LCT事件产生的重复基因,并以任意2个物种互为参考,构建了同源基因四联子.同源基因四联子由一个物种基因组内的2个旁系同源基因M1和M2,以及M1和M2在另一物种基因组中分别对应的直系同源基因S1和S2组成(图2a).基因置换存在部分基因置换及全基因置换2种形式,会使基因树的拓扑结构出现差异(图2b,c,d,e).若在物种分化后基因间未发生基因置换,则直系同源基因间序列的相似性会高于旁系同源基因间的相似性(图3a),若发生了基因置换,则情况相反(图3b,c).
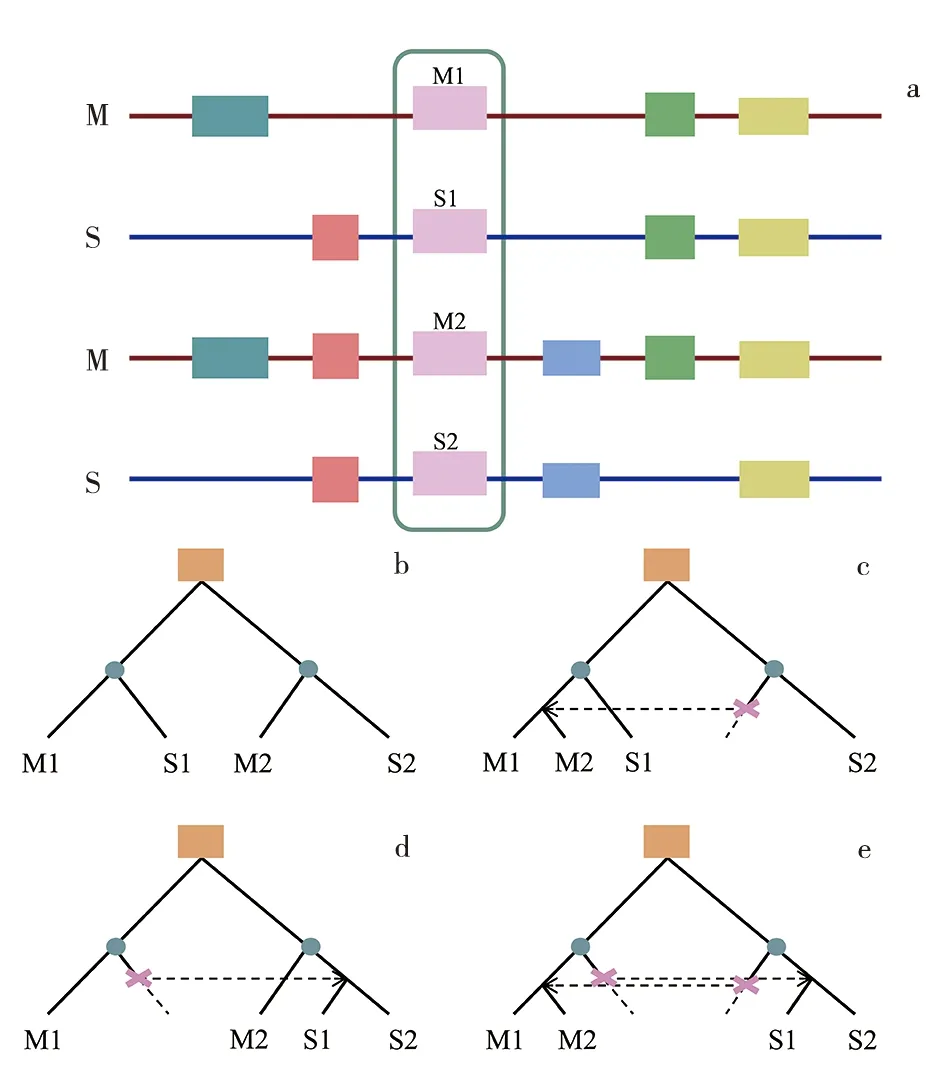
a.中矩形代表基因,颜色相同即同源基因.M,S代表2个近缘物种;M1,M2为物种M的一对旁系同源基因;S1是与M1对应的物种S上的直系同源基因,S2是与M2对应的物种S上的直系同源基因;M1,S1,M2,S2构成一个同源基因四联子; b~e中橙色矩形代表全基因组加倍事件,蓝色圆形代表物种分化; b.无基因置换; c.M2被M1置换; d.S1被S2置换; e.M2被M1置换,S1被S2置换.图2 同源基因四联子及基因树的拓扑结构模型Fig.2 Homologous Gene Quartets and Topological Models of Gene Trees
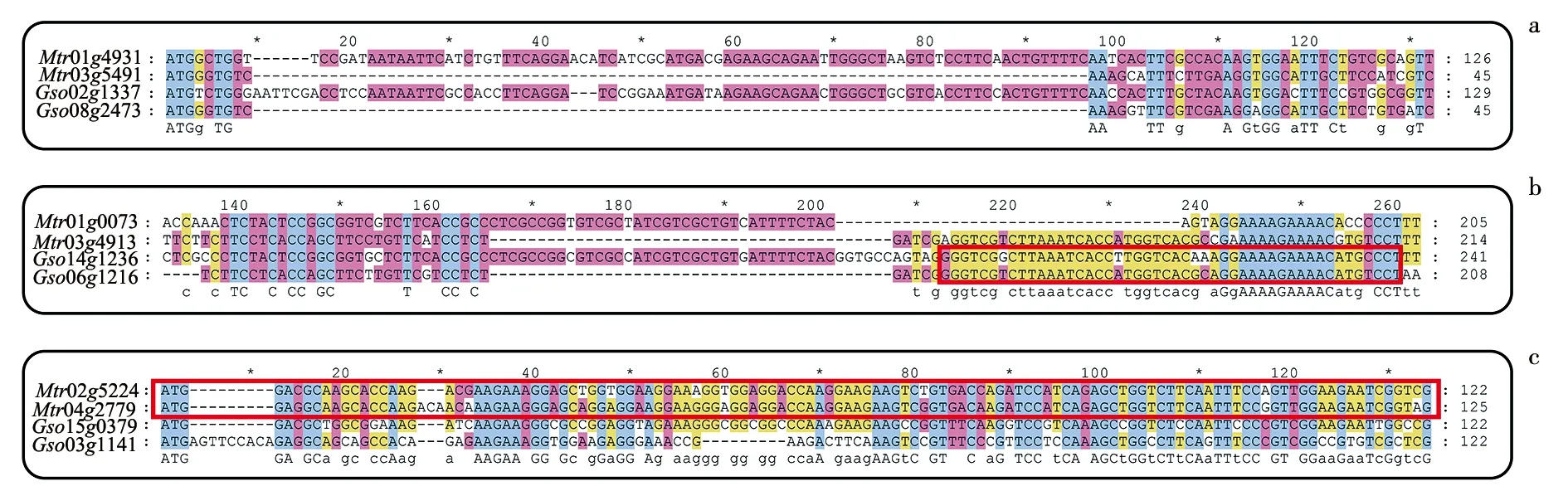
图中为大豆和蒺藜苜蓿基因组间的3组同源基因四联子.a.未发生基因置换的四联子序列; b.Gso14g1236和Gso06g1216发生了部分基因置换; c.Mtr02g5224和Mtr04g2779发生了全基因置换.红色矩形中的序列表示发生了基因置换的部分序列.图3 置换基因和非置换基因序列差异Fig.3 Sequence Difference Between Converted Genes and Non-converted Genes
将6个豆科植物间的同源基因四联子进行序列比对及系统发育分析,获得了四联子中发生全基因置换的基因对及发生部分基因置换的基因对(表1).可以发现,野生大豆和苜蓿基因组间四联子的数量(4 316~18 452)普遍多于大豆和苜蓿基因组间四联子的数量(3 993~14 696).并且在任意2物种组成的四联子中,都有重复基因发生了基因置换,这表明在LCT事件中产生的重复基因间,基因置换普遍存在.此外,通过比较发生全基因置换和部分基因置换的基因对分别占所有发生基因置换的四联子的比例,发现当2种大豆和苜蓿互为参考时,各物种基因组发生全基因置换的比例在4.33 %~18.30 %,而发生部分基因置换的仅有1.85 %~5.13 %.当各苜蓿互为参考时,各物种基因组发生部分基因置换的比例更高(4.45 %~10.59 %),而发生全基因置换的仅有0.10 %~0.67 %.
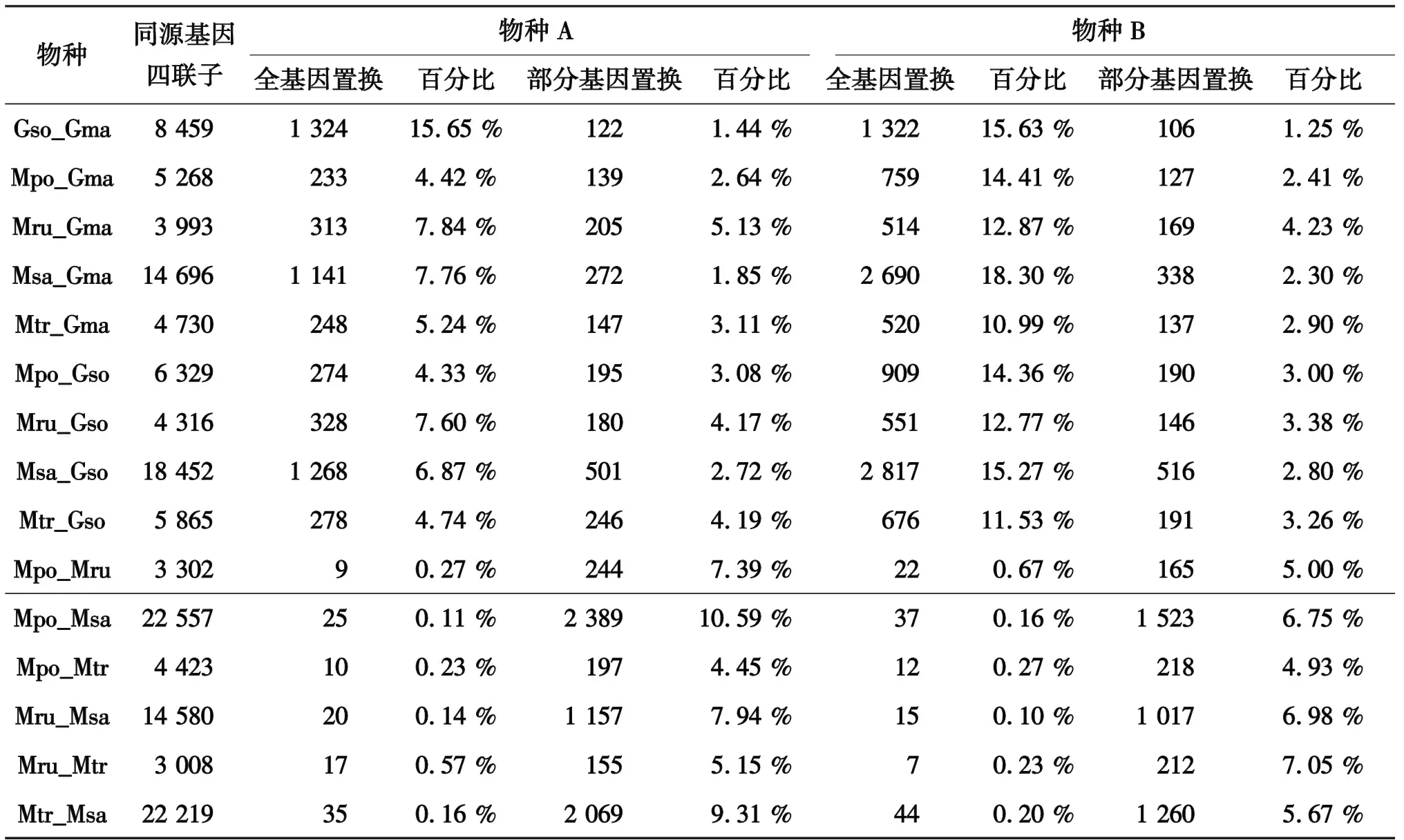
表1 基因置换结果统计Tab.1 Summary of Converted Genes
2.3 基因置换发生位置
分别统计各物种基因组内发生基因置换的所有基因,并计算这些基因与染色体端粒的距离.研究发现,大豆、野生大豆和紫花苜蓿在距离端粒30~40 Mb长度范围内发生基因置换的概率较高(43.38 %,45.34 %,30.62 %),花苜蓿在0~10 Mb、南苜蓿在40~50 Mb、蒺藜苜蓿在20~30 Mb长度范围内发生基因置换的概率较高(43.95 %,39.18 %,44.44 %)(表2).
2.4 基因表达差异
为了探究基因置换是否会影响基因的表达模式,从大豆、野生大豆、花苜蓿、紫花苜蓿和蒺藜苜蓿中分别选取了根和叶的转录组数据用于分析.发现,大豆的根中有66.77 %(1 300/1 947)的置换基因对,基因间的表达差异大于2倍;73.42 %(3 174/4 323)的非置换基因对,基因间的表达差异大于2倍.野生大豆、花苜蓿、紫花苜蓿和蒺藜苜蓿与大豆类似(表3),即当表达差异大于2倍时,置换基因对比非置换基因对数量少,这说明置换基因比非置换基因可能有更相似的表达模式.
2.5 基因置换对功能的影响
为了探究基因置换对重复基因功能的影响,对6个豆科植物的重复基因进行了GO分析(图4).在大豆、野生大豆、南苜蓿、花苜蓿、紫花苜蓿和蒺藜苜蓿中分别鉴定了6 013,6 824,2 803,2 349,6 473,2 880个置换基因,以及12 464,14 407,5 559,5 735,22 066,5 021个非置换基因.结果显示,在大豆和野生大豆的重复基因中,与转录调节活性(transcription regulator activity)相关的置换基因(2.8 %~3 %)多于非置换基因(2.4 %~2.6 %),在苜蓿的重复基因中,与定位(localization)过程相关的置换基因(4 %~6.4 %)多于非置换基因(3.5 %~5.7 %).此外,不同物种中还有特定的功能基因倾向于发生置换.例如在南苜蓿的重复基因中,参与生物调节(biological regulation)的置换基因(6.9 %)多于非置换基因(6 %),在花苜蓿的重复基因中,与转运活性(transporter activity)相关的置换基因(3.1 %)多于非置换基因(2.4 %).
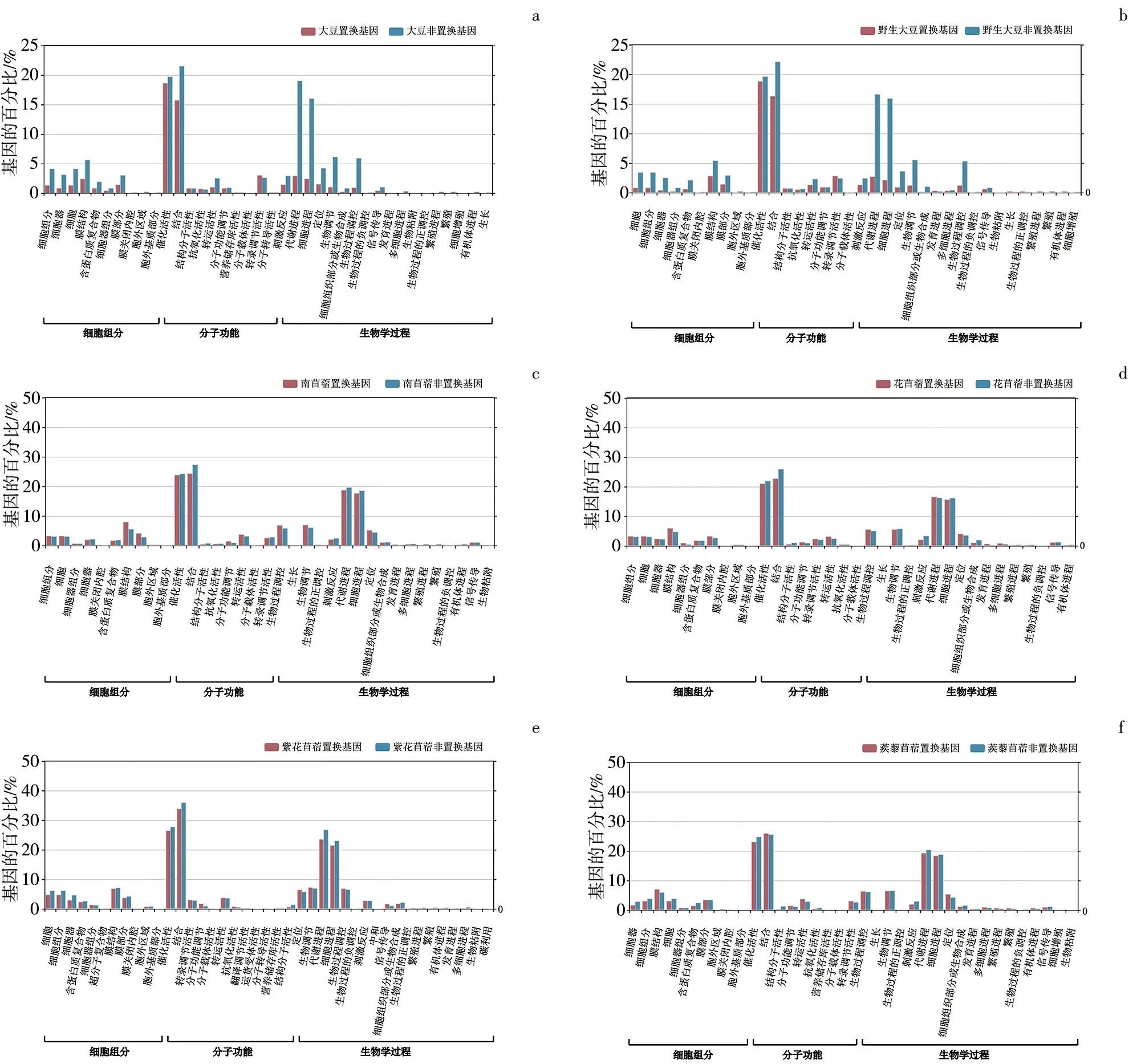
a~f分别为大豆、野生大豆、南苜蓿、花苜蓿、紫花苜蓿和蒺藜苜蓿基因组中重复基因间发生置换的基因和GO注释结果Fig.4 GO Annotation Results for Converted Genes and Non-converted Genes
2.6 基因置换对基因家族影响
为了研究基因置换对NLP基因家族成员固氮能力的影响,分别鉴定了大豆、野生大豆、花苜蓿和蒺藜苜蓿基因组内的NLP基因,并对根中NLP基因的表达模式进行了分析(图5).在大豆、野生大豆、花苜蓿和蒺藜苜蓿中分别有20 %(2/10),41.7 %(5/12),40 %(2/5)和40 %(2/5)的NLP基因发生了置换.并且,大豆中有100 %(2/2)的置换基因表达,62.5 %(5/8)的非置换基因表达;野生大豆中有100 %(5/5)的置换基因表达,85.7 %(6/7)的非置换基因表达;花苜蓿中有100 %(2/2)的置换基因表达,100 %(3/3)的非置换基因表达;蒺藜苜蓿中有100 %(2/2)的置换基因表达,100 %(3/3)的非置换基因表达.
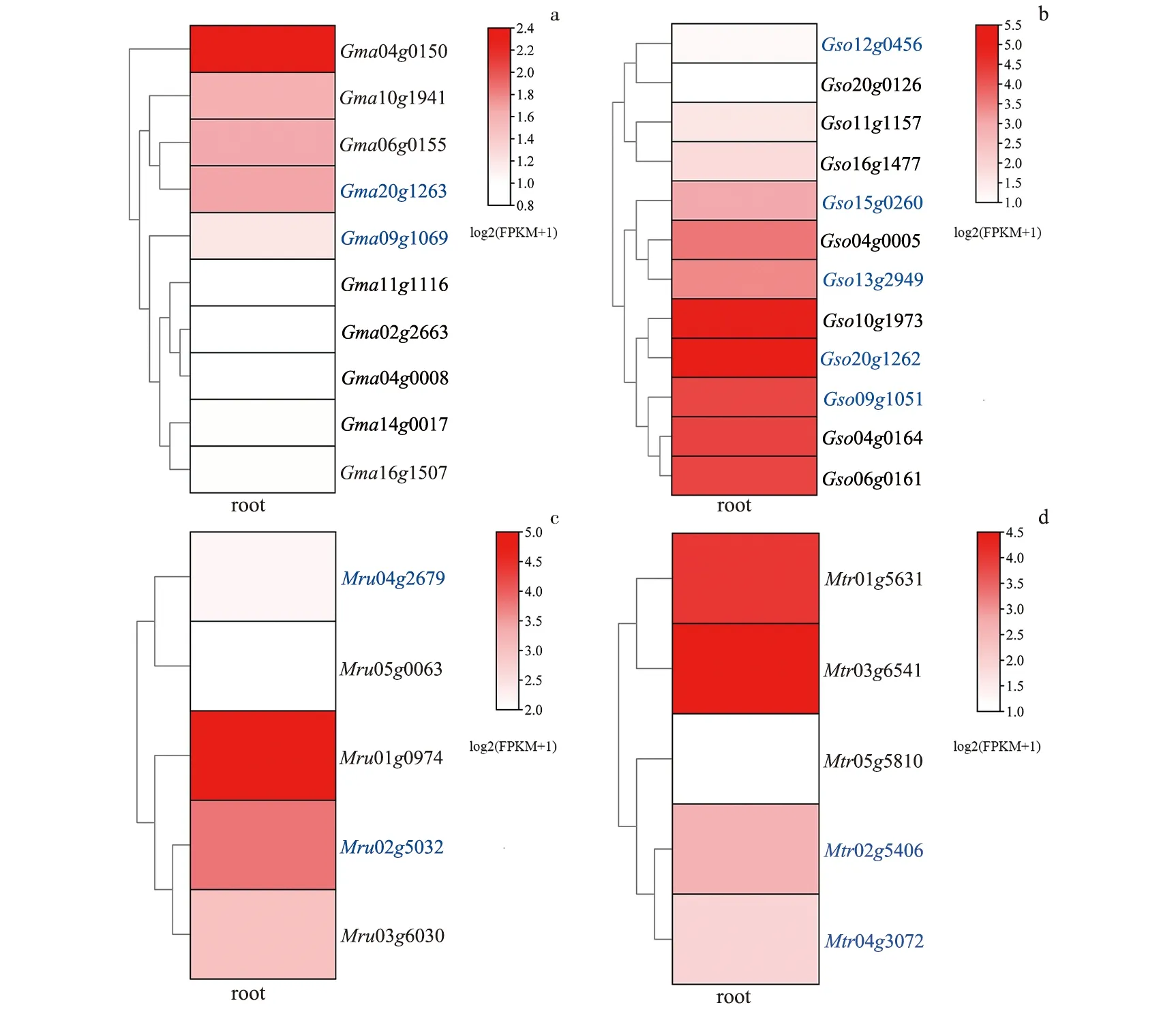
a~d分别为大豆、野生大豆、花苜蓿和蒺藜苜蓿的NLP基因在根中的表达模式,蓝色基因为发生基因置换的NLP基因,黑色基因为未发生基因置换的NLP基因.图5 4种豆科植物NLP基因在根中的表达模式Fig.5 Expression Model of NLP Genes In Roots of Four Legumes
3 讨论与结论
多倍化事件会导致植物基因组内产生大量的重复基因.本研究通过比较基因组学方法,对6个豆科植物重复基因间发生的基因置换事件进行了比较分析,发现这6个豆科植物在LCT事件中产生的重复基因,在物种基因组内发生了大规模的基因置换.在之前研究中发现,大豆和野生大豆在SST事件中产生的重复基因间也发生了基因置换[16].这表明在豆科植物进化过程中,基因置换持续发生.由于在不同豆科植物中发生基因置换的规模不同,所以基因置换可能通过影响物种基因组内基因间的相似性,而影响豆科植物的分化.
以往有研究认为,靠近染色体末端的重复基因相对于其他位置的重复基因更容易受到基因置换的影响[15,17].在本研究中,大豆、野生大豆、南苜蓿、紫花苜蓿和蒺藜苜蓿发生基因置换的位置并不靠近染色体末端,这种现象可能是由于这些豆科植物各自的染色体之间由于染色体融合、基因转座和染色体片段的转移,导致重复基因的位置发生了改变[18,20,23],这需要进一步的研究进行验证.
基因置换可能会影响重复基因的功能.转录组数据分析可用于研究基因对植物功能表达的影响,对置换基因对和非置换基因对的表达差异分析,揭示了基因置换对基因表达模式的影响.对大豆、野生大豆、花苜蓿、紫花苜蓿和蒺藜苜蓿的检测表明,置换基因对之间的表达差异小于非置换基因对,因此,基因置换可能会使重复基因具有更相似的表达模式,这与先前的报道一致[15,24].另外,通过对置换基因和非置换基因的功能注释分析,发现在不同物种中,具有特定功能的基因似乎更倾向于发生基因置换.
基因置换分析为研究基因家族在植物中的表达机制提供了新的角度.通过对小麦中调控淀粉合成的基因进行基因置换分析发现,基因置换可能影响小麦中淀粉的合成[25].此外,对水稻等动植物的研究发现,大基因家族更容易发生基因置换,并且基因置换可能有利于这些大基因家族成员的扩张或发生有利突变[15,24,26].通过对大豆、野生大豆、花苜蓿和蒺藜苜蓿的根组织中NLP基因的表达能力进行分析,发现置换基因在这4个豆科植物中全部表达,这表明基因置换可能会影响豆科植物的根瘤固氮能力.
