腐乳中菌群基因组DNA提取方法的对比研究
2023-11-06张娜娜黎鸿艺
张娜娜,徐 琼,黎鸿艺
(上海市质量监督检验技术研究院,上海 200233)
发酵作为一种传统的食品储存方式,其过程在过去是难以控制的,这是因为微生物系统极其复杂,含有大量的有益微生物和有害微生物[1]。作为我国具有民族特色的传统大豆发酵即食产品,腐乳被称为“东方奶酪”,具有很好的保健功能[2-5]。传统的腐乳在发酵过程中主要采用开放式发酵工艺,需要人工进行手工操作的环节较多,主要工序包括前发酵、腌坯、后发酵、装瓶、储存、运输等,而在这众多工序中,可能会有细菌、酵母、霉菌等微生物对腐乳造成污染,其中微好氧菌、兼性厌氧菌和专性厌氧菌在促进产品成熟、改变风味和口感的同时也会带来一定的污染,这会导致食源性疾病的风险增加[6-9]。因此,微生物多样性研究对腐乳特征质量指标具有重要意义。
腐乳属于高盐发酵产品,其含有的多种微生物属于耐高盐微生物,目前很多微生物无法通过培养的方式获得[10]。另外,腐乳中含有大量的大豆蛋白质、大豆脂肪和大豆基因组脱氧核糖核酸(deoxyribonucleic acid,DNA)等物质,这些物质也增加了腐乳中微生物的基因组DNA提取的难度。分子生物学因具有高度特异性,被广泛应用于食品微生物的研究。分子生物学技术不需要分离培养,而是通过分析微生物基因序列,对食品发酵过程中微生物的多样性和功能性进行更全面和客观的研究,同时能快速分析发酵过程中微生物群落的变化。近年来,基于细菌16S rRNA及真菌ITS区域扩增的测序技术已成为发酵产品微生物学研究的主要方法之一[11-12]。谭强来等[13]借助高通量测序分析了江西特色发酵豆豉中微生物的多样性;赵恒等[14]通过对不同地区花色腐乳真菌多样性进行分析比较得出,产地和工艺对豆腐中的细菌有明显影响;黄郑朝等[15]在对中国不同区域的发酵香肠进行细菌多样性研究时发现,我国不同地区发酵香肠的细菌多样性存在差异,其中四川地区和哈尔滨地区的细菌多样性高于其他地区;刘怡萱等[16]借助高通量测序技术对西藏农、牧区牦牛酸奶菌群多样性进行了分析;武亚婷等[17]对新疆自然发酵辣椒酱中微生物的群落结构和多样性进行了分析;宁亚丽等[18]通过高通量测序技术分别对吉林延边朝鲜族地区的酒曲及其发酵后粗滤和精滤米酒中的细菌和真菌菌群进行了分析。
目前,针对腐乳中存在的大量真菌与细菌,学者们致力于研究开发复杂基质中菌种基因组DNA抽提技术,并试图借助前沿的分子生物学技术找到一种快速、获得率高且不影响扩增效果的基因组DNA提取方法[19-23]。同时,通过对我国市场上目前有贩售的腐乳进行菌群研究,了解其中有益菌和有害菌的种类及分布,为更加有效地分析发酵食品的微生物多样性提供依据[24-25]。目前,借助高通量测序对发酵食品进行菌群分析的方法正在快速发展,但仍然不够成熟。例如该技术在样本的采集、保存,基因组DNA提取、扩增的引物、测序平台的选择上仍然存在不足,这可能会直接导致实验结果无法有效反映微生物的多样性。为更好研究腐乳中微生物菌群的多样性,本实验在样品前处理时通过低速和高速离心相结合的方式降低样品中含有的高盐、高糖成分及豆制品,从而降低了大豆基因组DNA的存在,同时重复离心、提取,并按比例加入有机试剂苯酚、氯仿、异丙醇等,降低了蛋白质、脂类的含量,保证了试验有序进行。在排除其他影响因素的情况下,本研究以非培养方式对腐乳中的微生物进行了基因组DNA提取,对比改良十六烷基三甲基溴化铵(cetyl trimethyl ammonium bromide,CTAB)法与玻璃珠法的提取效率与提取效果,分析了样品全基因提取的不同方法对高通量测序的影响,并结合检测结果,分析腐乳中的菌落结构,最终对两种基因组DNA提取方法进行比较分析,为快速、准确的建立非培养方式提取腐乳中基因组DNA奠定基础。
1 材料与方法
1.1 材料与试剂
红腐乳(LA5)、白腐乳(LA6)样品:市售;玻璃珠试剂盒:天根生物技术有限公司;CTAB:实验室自制;蛋白酶K(>30 U/mg)、溶菌酶(≥20 000 U/mg):美国Promega公司;Tris饱和酚(分析纯):生工(上海)股份有限公司;三氯甲烷、异戊醇、无水乙醇(均为分析纯):国药集团化学试剂有限公司;Premix ExTaqMix:日本宝生物公司。其他试剂均为国产分析纯。
1.2 仪器与设备
5430R振荡水浴槽:德国艾本德有限公司;5810R台式离心机:美国Eppendorf公司;DS-11微量紫外分析仪:美国丹诺尔公司;ESCO LA2-4A1生物安全柜:新加坡艺思高科技有限公司;VERITI梯度聚合酶链式反应(polymerase chain reaction,PCR)仪:美国ABI公司;Sub-Cell电泳仪、Geldoc XR+型凝胶成像仪:美国Bio-Rad公司;MiSeq高通量测序平台:美国Illumina公司。
1.3 方法
1.3.1 腐乳样品前处理
取适量样品于无菌袋中混匀,取40 mL混匀液于50 mL离心管中,2 000×g离心5 min,将上清液全部移至新的50 mL离心管中,9 000×g离心5 min;去上清,沉淀中加入25 mL无菌水,振荡混匀,再次9 000×g离心10 min;去上清,沉淀中加入20 mL无菌水,涡旋振荡混匀,备用。
1.3.2 玻璃珠法提取基因组DNA
取1.8 mL混匀液,按照玻璃珠试剂盒说明书对样品中微生物菌群的基因组DNA进行提取。
1.3.3 改良CTAB法提取基因组DNA
取5 mL混匀液至离心管中,9 000×g离心10 min,去上清,在沉淀物中加入10mL无菌水,混匀,9000×g离心10 min,去上清,得到的沉淀中加入溶菌酶(10 mg/mL)800 μL,37 ℃水浴1 h,加入蛋白酶K 40 μL、CTAB裂解液800 μL,再次水浴(56 ℃,2 h)。参考徐琼等[26]的方法提取基因组DNA。
1.3.4 测定方法
采用DS-11微量紫外分析仪测定提取的基因组DNA浓度、A260nm值/A280nm值以及A230nm值/A280nm值;为验证其比值对PCR扩增的影响,分别以两种方法提取得到的基因组DNA为模板,采用细菌16S rDNA区域引物16SF(5'-AGAGTTTGATCCTGGCTCAG-3')和1495R(5'-CTACGGCTACCTTGTTACGA-3')进行PCR扩增。PCR扩增条件:95 ℃预变性10 min;95 ℃变性45 s,60 ℃退火60 s,72 ℃延伸45 s,共40个循环;72 ℃再延伸10 min。PCR扩增体系:Premix ExTaqMix 10 μL,上、下游引物(5 μmol/L)各1 μL,DNA模板2 μL,去离子水补足至20 μL。PCR扩增产物采用1%琼脂糖凝胶电泳进行检测,对比扩增条带的亮度及纯度。
1.3.5 高通量测序
以提取的总基因组DNA为模板,以细菌16S rRNA基因V1~V3区合成引物27F(5'-AGAGTTTGATCCTGGCTCAG-3')、534R(5'-ATTACCGCGGCTGCTGG-3')和真菌ITS1区域扩增引物ITS 1F(5'-CTTGGTCATTTAGAGGAGTAA-3');ITS 1R(5'-GCTGCGTTCTTCATCGATGC-3')进行PCR扩增。PCR扩增条件及参数参照文献[26-27]。纯化回收扩增产物,采用Illumina MiSeq高通量测序平台进行测序及生物信息学分析。
1.3.6 数据处理
使用软件FastQC(http://www.bioinformatics.babraham.ac.uk/projects/fastqc)对高通量测序得到的双端序列数据进行质量控制,使用软件Cutadapt(http://code.google.com/p/cutadapt/)去除原始数据的接头,使用软件FLASH(https://sourceforge.net/projects/flashpage/files/)连接两端数据,使用软件QIIME2[28]筛选序列和嵌合体,并使用Usearch[29]算法去除嵌合体,参数默认。使用软件QIIME2在97%相似度下进行操作分类单元(operational taxonomic units,OTU)聚类。使用软件QIIME2进行Alpha多样性(Chao1指数、Shannon指数、Simpson指数)和β多样性分析,比较不同样品的微生物群落构成。
2 结果与分析
2.1 两种方法提取基因组DNA质量的比较
以红腐乳和白腐乳作为实验样本,采用玻璃珠法和改良CTAB法提取得到的基因组DNA质量见表1。由表1可知,改良CTAB法提取得到的基因组DNA质量浓度(>200 ng/μL)远高于玻璃珠法提取得到的基因组DNA质量浓度(<5ng/μL),因此,玻璃珠法无法再次进行核酸纯化,且无法一次性进行大量实验。用于PCR扩增的DNA最佳A260nm值/A280nm值在1.8~2.0之间,两种方法提取得到的基因组DNA的A260nm值/A280nm值均在此范围;改良CTAB法提取得到的基因组DNA的A260nm值/A230nm值(0.71~0.79)相较于玻璃珠法提取得到的DNAA260nm值/A230nm值(1.28~1.49)较差,说明改良CTAB法较玻璃珠法提取得到的DNA中蛋白含量较高,纯化蛋白能力较差。
为更加直观地观察改良CTAB法和玻璃珠法提取得到的基因组DNA对PCR扩增结果的影响,现对两种方法提取得到的基因组DNA进行PCR扩增,结果见图1。由图1可知,以两种方法提取得到的基因组DNA为模板均PCR扩增出相应的条带,玻璃珠法PCR扩增产物条带比改良CTAB法PCR扩增产物条带更亮,但拖带情况相对严重,说明玻璃珠法提取得到的基因组DNA相较于CTAB法提取得到的基因组DNA杂质更多。通过基因组DNA提取浓度结果和PCR扩增结果来看,改良CTAB法提取得到的DNA浓度比玻璃珠法提取得到的DNA浓度更高,质量更好,更适合进行PCR扩增。
2.2 两种DNA提取方法下不同腐乳样品微生物菌群多样性分析
2.2.1 Alpha多样性分析
不同腐乳样本的Alpha多样性分析结果见表2。由表2可知,各样本的覆盖率均>0.9,表明样本库中的序列大多被检测到,结果可以反映实际样本情况。对于红腐乳和白腐乳而言,玻璃珠法和改良CTAB法两种方法得到的细菌和真菌菌群的Alpha多样性相差较小,说明在不同腐乳样本中两种基因组DNA提取方法均实用。
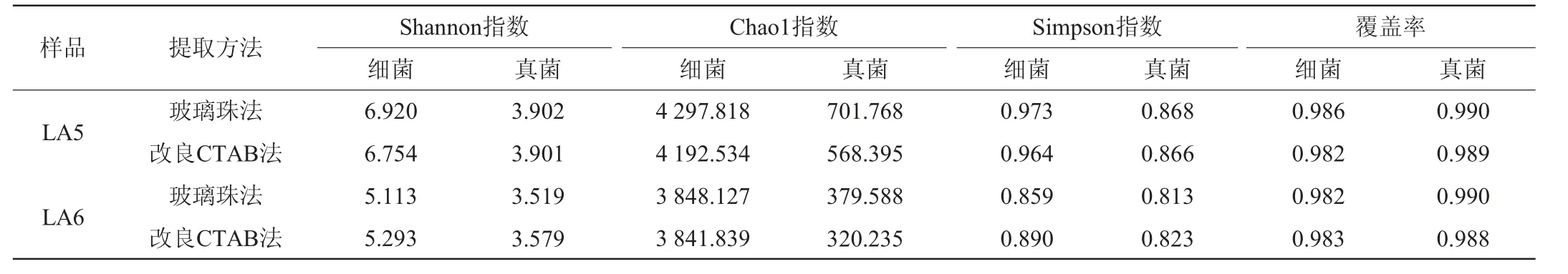
表2 不同腐乳样品Alpha多样性分析结果Table 2 Alpha diversity analysis results of different sufu samples
2.2.2 微生物菌群结构分析
(1)细菌菌群结构分析
基于门水平和属水平对不同腐乳样品细菌菌群结构进行分析,结果见图2。由图2a可知,在门分类水平上,样品LA5经两种提取方法得到的基因组DNA均鉴定到4个优势细菌门(相对丰度>1%),样品LA6经两种提取方法得到的基因组DNA均鉴定到3个优势细菌门。其中厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)和变形菌门(Proteobacteria)在样品LA5和样品LA6中的相对丰度之和均>95%。从相对丰度来看,玻璃珠法得到的厚壁菌门(Firmicutes)(LA5:57.81%;LA6:97.10%)较改良CTAB法得到的厚壁菌门(Firmicutes)(LA5:45.32%;LA6:95.76%)高,但玻璃珠法得到的拟杆菌门(Bacteroidetes)(LA5:9.12%)、变形菌门(Proteobacteria)(LA5:28.47%;LA6:1.53%)较改良CTAB法得到的拟杆菌门(Bacteroidetes)(LA5:20.20%)、变形菌门(Proteobacteria)(LA5:31.04%;LA6:2.13%)低。
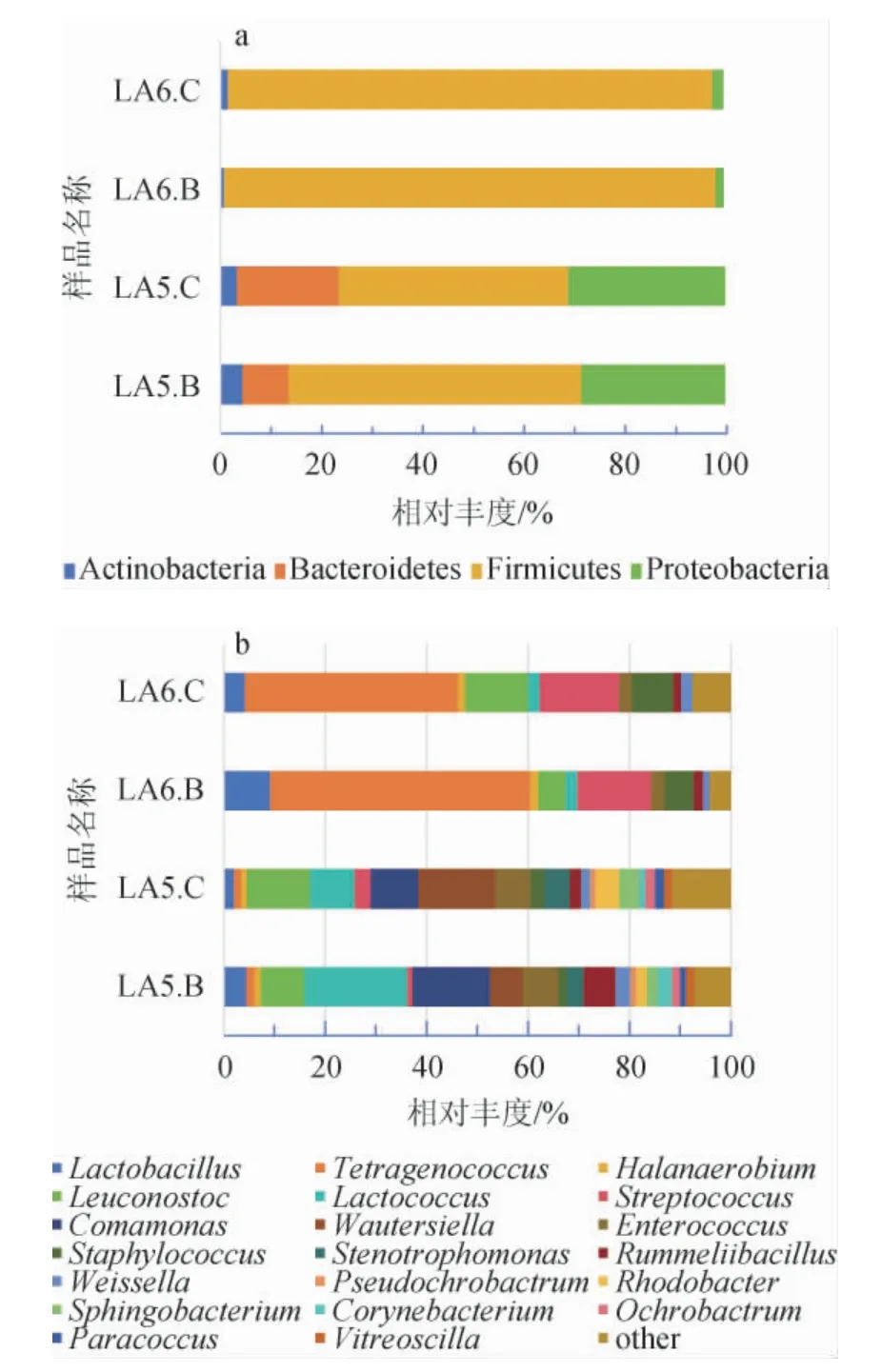
图2 基于门(a)和属(b)水平腐乳样品细菌菌群结构分析结果Fig.2 Analysis results of bacterial community structure based on phylum (a) and genus (b) levels
由图2b可知,在属分类水平上,样品LA5经两种提取方法得到的基因组DNA均鉴定到20个优势细菌属,样品LA6经两种提取方法得到的基因组DNA均鉴定到10个优势细菌属。在相对丰度>5%的优势细菌属中,样品LA5中玻璃珠法的乳球菌属(Lactococcus)(20.24%)、丛毛单胞菌属(Comamonas)(15.29%)、水原拉梅尔芽胞杆菌(Rummeliibacillus)(6.31%)的相对丰度高于改良CTAB法(8.67%、9.44%、2.25%),改良CTAB法的明串珠菌属(Leuconostoc)(12.52%)、法氏沃氏菌(Wautersiella)(15.18%)的相对丰度高于玻璃珠法(8.66%、6.48%);样品LA6中玻璃珠法的四链球菌属(Tetragenococcus)(51.24%)、乳酸菌属(Lactobacillus)(9.22%)的相对丰度高于改良CTAB法(41.95%、4.16%),改良CTAB法的明串珠菌属(Leuconostoc)(12.37%)、链球菌属(Streptococcus)(15.70%)、葡萄球菌属(Staphylococcus)(8.13%)的相对丰度高于玻璃珠法(5.28%、14.48%、5.79%)。
综上,两种基因组DNA提取方法的高通量测序结果分析得到的细菌门和属种类相差不大。但根据革兰氏分类可以看出,玻璃珠法更适用于革兰氏阳性菌的基因组DNA提取,而改良CTAB法则适用革兰氏阴性菌的基因组DNA提取。
(2)真菌菌群结构分析
腐乳中除大量细菌外还有一定量的真菌存在,基于真菌门和属水平对不同腐乳样品真菌菌群结构进行分析,结果见图3。由图3a可知,在门分类水平上,样品LA5和样品LA6经两种提取方法得到的基因组DNA均鉴定到2个优势真菌门,且玻璃珠法和改良CTAB法得到的真菌门相对丰度相差不大。
由图3b可知,在属分类水平上,样品LA5经两种提取方法得到的基因组DNA均鉴定到5个优势真菌属,样品LA6经两种提取方法得到的基因组DNA均鉴定到7个优势真菌属。在相对丰度>5%的优势真菌属中,样品LA5中玻璃珠法的隐球菌属(Cryptococcus)(19.71%)、瑟氏哈萨克斯坦酵母(Kazachstania)(6.18%)的相对丰度高于改良CTAB法(18.14%、5.65%);样品LA6中玻璃珠法的红曲霉属(Monascus)(30.48%)、曲霉属(Aspergillus)(7.18%)的相对丰度高于改良CTAB法(26.94%、5.89%),而改良CTAB法的隐球菌属(Cryptococcus)(42.43%)的相对丰度高于玻璃珠法(39.00%)。
综上,两种基因组DNA提取方法的高通量测序结果分析得到的真菌门和属种类相差不大,说明两种提取方法均可用于腐乳中真菌菌落的基因组DNA提取,且对真菌的提取效率影响甚微。
3 结论
在基因组DNA提取方面,改良CTAB法得到的基因组DNA浓度高于玻璃珠法。借助PCR进行扩增,结果显示,以玻璃珠法提取得到的基因组DNA为模板PCR扩增得到的条带拖尾较改良CTAB法严重,说明其含有一定量的杂质,玻璃珠法提取基因组DNA得到的基因组DNA浓度低,无法再次进行纯化,且受试剂盒中试剂的影响,无法一次性进行大批量的基因组DNA提取。在微生物菌群多样性和结构方面,通过高通量测序发现,两种基因组DNA提取方法均能有效提取到样品中的微生物基因,微生物菌群Alpha多样性相差不大,不同样品的两种基因组DNA提取方法得到的各微生物菌群的相对丰度存在一定差异,玻璃珠法更适用于革兰氏阳性菌的基因组DNA提取,改良CTAB法则适用于革兰氏阴性菌的基因组DNA提取,但微生物菌群的种类差异很小,证明两种基因组DNA提取方式对微生物基因组DNA的提取对高通量测序结果影响甚微。综上,玻璃珠法和改良CTAB法提取的基因组DNA均可用于腐乳中非培养方式的基因组DNA提取,但改良CTAB法具有更大的优势。
