高产聚苹果酸黑色素短梗霉CGMCC18996全基因组组装注释及关键蛋白分析
2023-09-13王舸楠李佳谦李雨桐陈世伟王淑贤赵廷彬贾士儒乔长晟
王舸楠,李佳谦,李雨桐,陈世伟,王淑贤,赵廷彬,贾士儒,乔长晟,,
(1.天津科技大学生物工程学院,天津 300457;2.工业发酵微生物教育部重点实验室暨天津市工业微生物重点实验室,天津 300457;3.天津市微生物代谢与发酵过程控制技术工程中心,天津 300457;4 .天津慧智百川生物工程有限公司,天津 300457)
聚苹果酸(polymalic acid,PMLA)是以苹果酸为唯一单体的均聚高分子聚合物,属于聚酯类聚合物,具有高生物相容性、高水溶性、生物可吸收性、化学可衍生性、可降解性和无免疫原性等多种优良性能[1]。PMLA分子的侧链带有可修饰性较强的羧基,一些分子能够通过官能团反应引入其聚合链上,同时PMLA水解产生的苹果酸是理想的调酸剂,这使PMLA在生物医药、食品和生物材料领域具有潜在的应用前景[2-6]。
研究表明产黑色素短梗霉(Aureobasidium melanogenum)是一种具有较强产PMLA能力的类酵母真菌[4],但目前对其PMLA生物合成研究并不透彻,对通路中关键酶的研究并不清晰。产量还有很大的提升空间[7]。同时,该菌基因组信息依旧较少,限制了该菌株的开发利用。因此,对该菌种进行基因组测序及组装可为改造菌种,提高产量提供理论依据。
目前,主流的基因测序分为两种,单分子测序(如Pacbio三代测序平台)和高通量测序(如二代Illumina平台),其中单分子测序具有读长长组装结果好的特点,但其测序成本与错误率较高;高通量测序采用双端测序策略,结果兼备质量高、价格低的特点,但其测序长度较短,组装会产生较多的片段(contigs)。因此采用三代测序结果组装再通过二代测序结果进行纠错(基因组polish),能够很大程度降低contigs数目同时保持基因组准确率,在完成基因组组装后,可通过转录组测序结果对基因组进行结构注释,以同时获得高质量基因组注释结果[8]。
本研究通过PacBio Sequel II及Illumina NovaSeq 6000测序平台对高产PMLA产黑色素短梗霉基因组进行测序,通过不同组装软件对测序的下机文件进行组装及优化;结合转录组数据对组装结果进行基因结构注释。之后对基因组注释结果进行不同数据库的功能注释,同时分析PMLA合成关键蛋白。通过这种方法,期望得到适合于基因组分析以及后续分子生物学实验的高质量基因组,为产黑色素短梗霉的开发利用提供一定生物信息学参考,并同时为其他类似物种的基因组组装提供思路。
1 材料与方法
1.1 材料与试剂
菌株黑色素短梗霉,已保存至中国普通微生物菌种保藏管理中心(保藏号:CGMCC18996);基因组提取试剂盒 湖南艾科瑞生物工程有限公司;乙腈(色谱纯)北京鼎国昌盛生物技术有限公司;磷酸二氢钾、磷酸氢二钾、磷酸(均为色谱纯)天津市大茂化学试剂厂。
1.2 仪器与设备
UC7超薄切片机 德国Leica公司;LC-10高效液相色谱仪 日本岛津公司;ZWYR-D2403恒温培养振荡器上海智城分析仪器制造有限公司;LRH-250A生化培养箱 泰宏医疗器械有限公司;NanoDrop One超微量分光光度计 美国赛默飞世尔科技公司。
1.3 方法
1.3.1 基因组提取与测序
本实验的基因组提取、建库以及测序委托诺禾致源科技股份有限公司(北京)完成,实验流程均按照诺禾致源公司测序方法进行(https://www.novogene.com/novo/sd_cpjs_6.html,https://www.novogene.com/novo/ed_cpjs_2.html)。
1.3.2 数据处理及生物信息学分析
使用曙光云计算服务器(曙光信息产业股份有限公司)进行生物信息学分析。最终得到相关的测序下机文件以及组装的基因组文件已上传至国家生物信息中心(项目号:PRJCA011444)。
1.3.2.1 原始下机文件获取及质控
通过Illumina NovaSeq 6000平台获取高通量测序产生的fastq基因组测序文件,通过PacBio Sequel II测序平台获取单分子测序产生的bam基因组测序文件,同时验证高通量测序和单分子测序文件的md5值与平台提供是否一致。
1.3.2.2 基因组组装及评估
使用SPAdes-3.15.5[9]组装软件对二代测序clean reads进行组装,为了得到最优N50及L50值,将k-mer大小设定为77、87、97、107、117及127进行组装。并通过QUAST[10]软件对组装结果进行评估。三代测序通过Flye[11]、Canu[12]、NextDenovo(https://github.com/Nextomics/NextDenovo)以及MECAT2[13]组装软件进行组装,通过quickmerge[14]将最优结构与其他组装结果进行融合,之后对得到的融合后结果进行去重复处理进一步降低contigs数目。
1.3.2.3 基因组注释
使用BRAKER2[15]软件进行基因组注释,首先通过HISAT2将转录组序列比对到基因组上,生成bam文件,再将生成的bam文件与基因组文件作为输入参数使用BRAKER2进行注释。将获得的氨基酸序列通过InterProScan[16]与eggNOG-mapper[17]进行注释得到UniProtKB AC/ID(基因名),将这些基因名进行京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)(https://www.kegg.jp/kegg/)、基因本体论(Gene Ontology,GO)[18]及直系同源集(Clusters of Orthologous Groups,COG)[19]数据库注释,并通过antiSMASH[20]对次级代谢产物合成基因簇进行预测。
1.3.3 菌体形态
透射电镜拍摄方法如下,将电子显微镜固定剂(2.5%戊二醛溶于磷酸缓冲液)添加到经离心分离的菌株中,重悬菌体,使用 0.1 mol/L磷酸漂洗液漂洗3 次,1%锇酸固定液固定1 h;之后使用0.1 mol/L磷酸漂洗液漂洗3 次,丙酮脱水3 次。使用纯EMBed 812树脂(90529-77-4)包埋,移入65 ℃烘箱中聚合48 h;使用超薄切片机切至60~80 nm薄片,用2%乙酸铀饱和醇溶液和柠檬酸铅染色15 min。最后,使用JEM1200电镜进行观察拍摄,得到放大4000 倍的产黑色素短梗霉CGMCC18996图像。
1.3.4 蛋白质结构预测
蛋白质结构预测使用Alphafold2-2.2.0[21],将基因组注释的氨基酸序列作为输入参数,运行模式参数选择monomer,其余均选用默认选项进行蛋白质结构预测,最终在生成文件中选择最优结果(即rank0结果)。之后将蛋白预测结果与蛋白质数据库(Protein Data Bank,PDB)[22]中已上传的晶体结构进行比对(磷酸烯醇式丙酮酸羧化酶激酶(phosphoenolpyruvate carboxykinase,PCKA):1YLH[23];苹果酸合成酶(malate synthase,MASY):3CUZ[24])。
2 结果与分析
2.1 通过二代测序数据进行基因组预组装
基于Illumina NovaSeq 6000测序平台对产黑色素短梗霉基因组进行测序,共得到35.85 Gb×2的双端测序结果。且该测序平台得到的raw reads具有较高的质量评分(Q>35)。之后,选取质控后clean reads进行后续的组装实验。
不同k-mer长度对产黑色素短梗霉clean reads进行组装结果见表1,组装结果显示,产黑色素短梗霉基因组大小约为44 Mb。其中,在k-mer长度为127时得到N50值为908694,L50值为14,基因组覆盖度为121.2×。基于该结果可以判断该菌基因组长度理论值在44 Mb左右;且随着k-mer长度的增加,N50值增大,L50值减小;若继续增大k-mer值会进一步优化组装参数。但由于SPAdes软件进行组装的k-mer值最大为127,这可能是考虑到继续加大k-mer值对测序深度要求较高,从而提高测序成本。
2.2 三代测序组装结果及基因组结构注释
基于PacBio平台的单分子测序共产生46 Gb大小的bam基因组测序文件,转换为fasta文件后,根据二代组装结果设置基因组大小为44 Mb进行组装。各组装软件组装结果如表2所示,其中,选用Canu组装的最优结果通过quickmerge软件与其他组装结果进行融合,并结合二代测序文件进行基因组纠正(polish),在删除重复contigs后的最终组装结果见表3。

表2 三代组装结果Table 2 Results of third-generation assembly
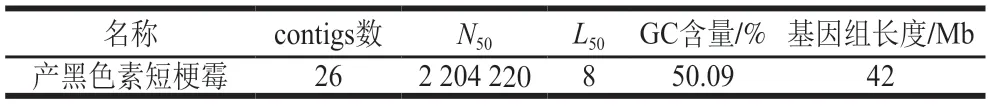
表3 产黑色素短梗霉基因组装结果Table 3 Results of genome assembly of A.melanogenum
结合转录组测序文件进行基因组结构注释共注释出15684 个基因,并找到基因编码区与氨基酸预测区,因结合转录组测序文件进行结构注释,这些基因可能包含有可变剪切和重复注释的结构,会增加注释出的基因数目。因此,进行功能注释和基因名称注释后需删除重复的基因名;最终,共获得6202 个基因注释结果。
2.3 GO、KEGG、COG以及antiSMASH次级代谢注释结果
对注释出的6202 个基因进行GO、KEGG与COG数据库注释,结果如图1所示。其中,COG注释结果显示大部分基因与碳水化合物转运及代谢、氨基酸转运代谢、转录后修饰、RNA加工及修饰有关。KEGG注释结果显示大部分基因所处代谢通路与核糖体、过氧化物体、RNA转运有关。GO注释结果显示大部分基因与RNA、过氧化物体以及线粒体有关。最终,antiSMASH次级代谢物预测结果共发现4 个非核糖体肽合成酶(nonribosomal peptide synthetase,NRPS)基因簇、5 个一类聚酮合酶(polyketide synthetase,pks)基因簇、3 个β-内酯类合成基因簇以及7 个萜类合成基因簇,其中1 个一类pks基因簇和黑色素合成有关(相关性100%),1 个一类pks基因簇与黑麦酮酸类化合物合成有关(相关性18%)。

图1 COG(a)、KEGG(b)、GO(c)数据库注释结果Fig.1 Results of annotation in the COG (a),KEGG (b),and GO (c) database
2.4 产黑色素短梗霉菌体透射电镜结果
如图2所示,在低产组中菌株具有较大细胞核(N)以及周围存在的具有双层膜结构线粒体(M),在高产组中出现了类似乙醛酸循环体的圆形结构,提示黑色素短梗霉中可能存在有乙醛酸循环途径。

图2 产黑色素短梗霉PMLA低产组(a、c)及PMLA高产组(b、d)透射电镜结果Fig.2 TEM results of low-and high-PMLA producing A.melanogenum
2.5 PMLA合成相关基因结构预测
在对基因结构与基因名注释后,找到PCKA、MASY的蛋白质序列,其晶体结构预测结果如图3、4所示,其中,PCKA、MASY与PDB上传晶体结构(PCKA:1YLH[23],MASY:3CUZ[24])进行比对,结果显示预测的蛋白结构与PDB数据库中序列比对结果基本一致,氨基酸比对结果中序列与其晶体结构中小分子配体的结合位点也具有较高的一致性。同时蛋白的保守作用位点如PCKA中86R、140V、146G、287G、288D、289D,MASY中440C、275C、276G、277R、278W在比对结果中一致。

图3 Alphafold对PCKA预测结果(a)及与PDB中已上传晶体结构的比对(b)和序列对比结果(c)Fig.3 Results of PCKA prediction by Alphafold software (a),crystal structure of PCKA versus that in PDB database (b),and amino acid alignment results (c)
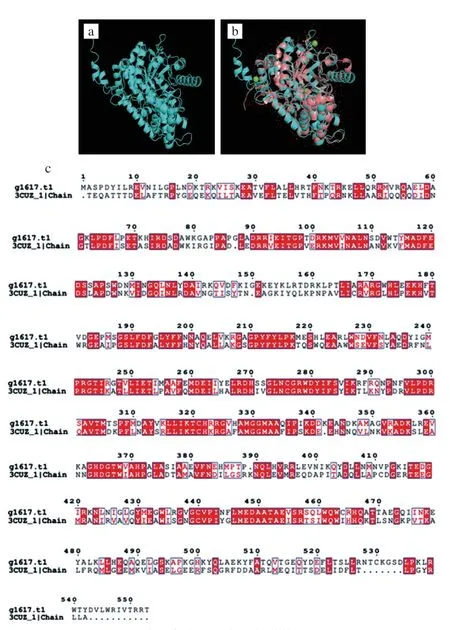
图4 Alphafold对MASY预测结果(a)及与PDB中已上传晶体结构的比对(b)和序列对比结果(c)Fig.4 Results of MASY prediction by Alphafold software (a),crystal structure of MASY versus that in PDB database (b),and amino acid alignment results (c)
3 讨论
结合基因组及转录组测序结果可以组装并注释出质量较高的基因组。本研究通过三代测序(Pacbio sequel II平台)、二代测序(Illumina NovaSeq 6000)平台对产黑色素短梗霉基因组进行测序,因三代组装需要预估基因组大小,因此,首先通过二代数据进行基因组预组装共得到44 Mbp基因组大小。之后考察了不同组装软件对产黑色素短梗霉基因组的组装效果,在一般的默认选项下,Canu软件得到了较好的组装效果。对该组装结果通过二代数据修正并去重后得到包含26 个contigs、N50为2204220、GC值为50.09%、大小为42 Mb的较高质量基因组组装结果。通过转录组数据对该组装结果进行结构注释,共找到6202 个基因。产黑色短梗霉属于出芽短梗霉的亚种,其在不同环境中也会呈现出酵母状与菌丝体状的不同形态[25]。将基因组组装与注释结果与酵母和丝状真菌的模式生物基因组进行比较;其中酿酒酵母(Saccharomyces cerevisiae)基因组大小为12.15 Mb,编码6016 个蛋白,GC含量为38.15%[26];构巢曲霉基因组大小为30.30 Mb,编码10008 个蛋白,GC含量为50.10%[27]。因此,产黑色素短梗霉基因组组成更偏向构巢曲霉,且本实验室先前通过无参转录组注释发现很多与构巢曲霉同源的基因[28]。因构巢曲霉是丝状真菌的模式生物,该结果提示产黑色素短梗霉可能同样适用构巢曲霉的分子转化方法。
本研究通过Illumina NovaSeq 6000平台对产黑色素短梗霉进行了大约35 GB下机文件大小的高通量测序,该测序量的测序深度较大,理论测序深度为1000×。通过二代组装软件进行组装后,最终组装出N50值为908694的基因组,该结果仅达到三代测序组装结果N50的41.4%,因此,考虑到组装结果以及建库与测序成本[29],三代测序进行组装辅以二代测序进行基因组修正的测序方法更具有性价比。
通过COG、KEGG与GO数据库注释结果表明,产黑色素短梗霉中基因表达主要位于核糖体、线粒体以及过氧化物体等细胞器中,其中,有研究表明线粒体中的三羧酸循环、乙醛酸循环体中的乙醛酸循环以及细胞质中的还原性三羧酸途径与PMLA的生物合成有关[7,30-31]。功能注释结果中出现了大量与过氧化物体有关的基因,而能够产生苹果酸的乙醛酸循环体也属于一类过氧化物体。因此,该结果说明在产黑色素短梗霉中可能存在乙醛酸循环体。实验室先前研究发现乙醛酸体中的MASY可通过乙醛酸途径生成苹果酸,相比于线粒体中存在的苹果酸/天冬氨酸穿梭体系,乙醛酸循环体中苹果酸可能可以直接通过单层乙醛酸体膜进入细胞质中而被非核糖体肽合成酶(non-ribosomal peptide synthase,NRPS)聚合成PMLA[28]。透射电镜结果也显示,在加入CaCO3的高产PMLA组中,菌体内部出现了较多的圆形细胞器,提示菌体中存在乙醛酸循环体的可能。antiSMASH注释结果表明在产黑色素短梗霉中存在4 个NRPS基因簇,该结果也同样反映出NRPS蛋白聚合苹果酸形成PMLA的可能性。同时,注释出的与黑色素合成的pks基因簇可解释该菌种在生长过程中逐渐变黑的现象。
实验室先前研究表明[28],PCKA、MASY和NRPS基因在PMLA高产组中发生了较大幅度的上调。因此,通过对基因组注释得到的PCKA和MASY的编码区进行了蛋白结构预测,并将预测结果与PDB上已有的晶体结构进行比对。比对结果表明,蛋白预测结果与已有晶体结构基本吻合,且保守结构域相似,提示预测蛋白与其他菌株中已存在蛋白可能具有相似的功能。PCKA是糖异生途径的限速酶,一般情况下催化草酰乙酸转化为磷酸烯醇式丙酮酸,但有研究表明PCKA在真菌细胞质中可反向催化磷酸烯醇式丙酮酸生成草酰乙酸[23]。而细胞质中的草酰乙酸会进一步转化为PMLA的前体物质苹果酸。MASY可催化乙醛酸循环中的第二部反应,将乙醛酸转化为苹果酸[24],而乙醛酸体的单层膜结构可能使苹果酸被动运输进入细胞质,进而被细胞质中存在的NRPS蛋白聚合成为PMLA[28]。本实验没有对NRPS蛋白预测的原因是因为其氨基酸序列较长(5000 aa左右),软件无法预测,其次是在PDB上还没有找到近似的蛋白晶体结构,比对较为困难。
本研究对产黑色素短梗霉进行二代、三代基因组测序并组装得到最优组装结果,后通过转录组数据对基因组进行结构注释,将得到的结构注释结果进行不同数据库的功能注释。同时对产黑色素短梗霉进行了透射电镜拍摄,电镜结果提示菌株中存在乙醛酸循环体结构,该结构可能与PMLA合成有关,最终,实验通过对PMLA合成相关的PCKA和MASY蛋白进行结构预测并与PDB上已有电镜结果进行比对。发现产黑色素短梗霉中这两种蛋白与PDB上已有晶体结构基本一致,且这两种蛋白功能最终都与苹果酸代谢有关。本实验结果可为产黑色短梗霉菌株的PMLA代谢提供一定的参考,相关的测序下机文件以及组装的基因组文件已上传至国家生物信息中心(PRJCA011444),为后续的菌株开发利用提供基础。
