DNA甲基化谱:流淌的生命脉律
2023-08-14廖端芳,张佳,卢光明,李凯
廖端芳,张佳,卢光明,李凯


〔摘要〕 70年前,随着DNA双螺旋结构的发现,人类进入基因组时代;21世纪初,人类基因组测序完成及对转录組、蛋白质组和表观遗传的研究,人类继而进入后基因组时代;近年对DNA甲基化的深入研究,预示更为神秘宽广的表观遗传组学时代已经到来。人体血液中大量存在的甲基化DNA构成甲基化谱,作为一个整合性指标,有如一曲流淌的生命脉律,反映不同细胞的生理或病理状态。精准分析甲基化谱,具有诊断疾病特别是肿瘤早筛早诊的价值;同时可作为人类健康、乃至天赋的量化评估指标;也可用于中医体质辨识与证型分类及中药材溯源、真伪识别和品种改良。本文介绍表观遗传组学时代甲基化谱的重要意义,以纪念DNA双螺旋结构发现70周年。
〔关键词〕 DNA甲基化;甲基化谱;游离核酸;表观遗传;肿瘤早筛
〔中图分类号〕R2 〔文献标志码〕A 〔文章编号〕doi:10.3969/j.issn.1674-070X.2023.07.026
DNA methylation profile, the flowing melody of life:
Marking the 70th anniversary of discovering DNA double helix
LIAO Duanfang1,2, ZHANG Jia1, LU Guangming3, LI Kai1,2*
1. National Engineering Research Center of Personalized Diagnostic and Therapeutic Technology, Hunan University of
Chinese Medicine, Changsha, Hunan 410208, China; 2. Key Laboratory for Quality Evaluation of Bulk Herbs of Hunan
Province, Hunan University of Chinese Medicine, Changsha, Hunan 410208, China; 3. General Hospital of Eastern
Theater Command, Jinling Hospital of Nanjing University, Nanjing, Jiangsu 210016, China
〔Abstract〕 Seven decades ago, the discovery of double helix structure announced the genetic era of human beings; at the beginning of the century, human genomic sequencing, study of transcriptome, proteome, and epigenetics marked the post-genomic era; in recent years, the study of DNA methylation signifies the era of mysterious epigenetics. DNA methylation profile is composed of numerous DNA methylation markers in human blood. As the integrated parameter, it resembles the flowing melody of life, and reflects the physiological or pathological statues of the cells. The methylation profile can diagnose diseases such as cancer at its early stage, evaluate healthy status and constitution, classify the patterns of Chinese medicine, and standardize the diagnosis of Chinese medicine. In addition, it can help trace, identify and improve the Chinese medicines. To mark the 70th anniversary of discovering double helix structure, this paper introduces the biomedical significance of methylation profile.
〔Keywords〕 DNA methylation; methylation profile; circulating cell-free DNA; epigenetic; early detection of cancer
生命,是一首歌,通过优美的DNA双螺旋结构奏响着由A、T、C、G和甲基化C 5种音符组成的动人旋律[1-2];生命,是一条河,流淌着蕴含亿万细胞交织而成的甲基化谱脉律。每个个体的生命,从诞生到离去,那动人的5种音符,从深奥无底的基因组,透过甲基化胞嘧啶(methylcytosine, mC)组成的甲基化谱,无时无刻不在我们血液中流淌,是我们生命的脉律,影响着我们的人生。
在跨入21世纪之际,随着人类基因组测序计划完成,人类获取了由ATCG和甲基化C 5种核酸构成的长达30亿个碱基音符的生命天书,定义了我们所处的时代为后基因组时代[3-5]。解码生命天书,成为当代生物科学技术的主旋律。
甲基化谱,是生物体DNA不同CpG位点甲基化数量在某一时刻的总和[6-8]。基因组中的每个单一CpG位点,通過不断更新或凋亡细胞释放入血的游离核酸,携带其组织的特异性信息,这些信息的集合反映机体不同系统、器官、组织的生理或病理状态。同一个体在不同时间、不同个体在同一时间,甲基化状态均有不同;血液游离核酸所呈现的甲基化谱亦相应有所区别[9-10]。因此,游离核酸甲基化谱是生命的信息子集,是一个高度动态、由不同细胞类型凋亡或死亡细胞碎片化核酸组成的瞬时集合,与之相对应的细胞甲基化图谱,则属于特定细胞类型各自具有的、相对恒定的甲基化组学序列[10-12]。
核酸甲基化是表观遗传的主要方式[12-15],通过对编码人类大约5万种蛋白质的基因在何时何处以及何种强度进行甲基化调控,使生命乐章得以完美演奏。甲基化调控基因的表达水平,抑癌基因的甲基化可显著增加癌症发生的风险[16-18]。文献报道,超过2 500多种基因参与肿瘤的发生和发展过程,早期肿瘤为单克隆,而晚期肿瘤常常表现出高度异质性,即多克隆或实际为多种疾病的集合[19-20]。DNA甲基化也与循环肿瘤细胞的干细胞特性、解聚及转移能力相关[21]。以上提示,DNA甲基化对指导肿瘤早期诊断、早期治疗及转移监控具有非常重要的价值。
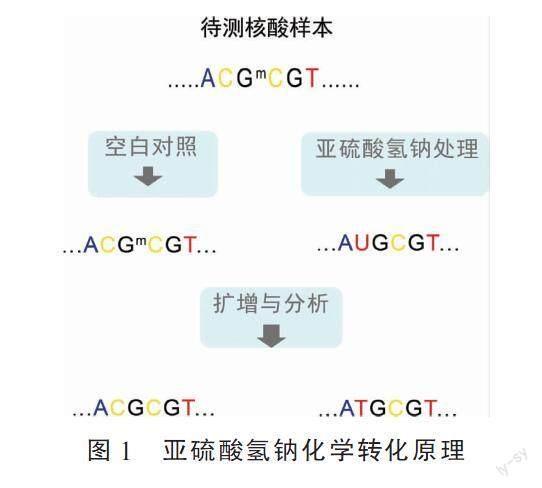
参与特定肿瘤发生的基因种类多样,早期诊断需要对尽可能多的基因甲基化进行高敏感检测[11]。在现有甲基化分析技术中,聚合酶链式反应(polymerase chain reaction, PCR)检测一个或少数几个甲基化位点,而二代测序(next gene sequencing, NGS)可进行组学层面的甲基化测序[22-23]。甲基化分析技术虽已用于肺癌、结直肠癌等筛查,但这些技术大都采用亚硫酸氢钠对未甲基化的胞嘧啶(C)向尿嘧啶(U)进行转化,U在PCR或测序时的后续扩增中表现为T,而甲基化的C则维持不变,比较化学转化前后两个标本,即可得到待测标本的CpG是否甲基化。因此,保留了mC在基因分析时仍呈现为C的基本结构特征(图1)。由于亚硫酸氢钠在上述化学转化过程中大量破坏核酸,相应技术对低拷贝DNA甲基化表观遗传学信息进行检测的敏感性不高,用于肿瘤的早期诊断存在很大局限性[24-25]。
为克服亚硫酸氢钠相关技术的致命缺陷,多种新技术已用于DNA甲基化研究[26-28]。其中,以甲基化依赖酶为核心的单序列特异性引物甲基化检测技术和med-NGS技术,人工创造了一系列抑制接头自连系统,结合广泛用于基因编辑的金门克隆技术,构建动态抑制甲基化接头的自连,并促使可逆反应向接头与待测核酸形成产物的方向进行[28-31](图2)。这些创新性技术,可对数个到成千上万个CpG选定位点设计序列特异性引物(图3),以达到在不破坏待测核酸前提下对甲基化核酸的富集和解码,均可望成为甲基化谱的标配技术。
从基因组时代到后基因组时代,从遗传学时代到表观遗传学时代,从单个甲基化位点检测到甲基化组学测序,生物科技和生命科学领域经历了漫长而艰辛的探索。在21世纪20年代之初,当全球生物医学因为新冠疫情不得不重复开发和广泛应用相对原始的核酸检测RT-PCR技术的特殊时期,甲基化谱技术的出现,拓展了其在重大传染病防治中的应用[32-33]。再次使我们可以通过表观遗传学,去聆听生命的歌,去感受生命的脉律,去呵护生命的美好。
甲基化谱是机体细胞新陈代谢在基因层面的一个瞬时性总体集合,重复检测甲基化谱,可以动态反映组成生命各系统、各器官的细胞更新速率和各种细胞基因表达调控水平的变化程度。人类基因组含有大约2 800万个CpG位点,这些位点的C是否甲基化直接关系到相关基因表达与否以及表达水平的高低。换言之,基因的甲基化与去甲基化,高甲基化与低甲基化,均参与基因表达的调控[34-35]。甲基化谱直接反映生命活动的健康、亚健康,亦或某种疾病状态。
甲基化谱分析技术的出现,突破了长期困扰肿瘤早筛早诊的技术瓶颈[18,20-21]。然而,器官特异性肿瘤甲基化谱和泛癌种甲基化谱要逐步成为基因诊断标准或成熟为临床指南,尚有待积累大量临床检测病例,并通过甲基化谱与早筛早诊所必须的突变数量、突变丰度、突变比例和突变速率等临床数据的不断细化、更新和临床再验证。甲基化谱的临床应用,短期可用于耐药监测和围手术期监测(minimal residual disease, MRD)[35],减少晚期肿瘤患者比例,提高患者生存期和生存质量,使常态化荷瘤生存成为可能,其远期效果有望根治大部分肿瘤患者。
在临床推进上,甲基化谱用于肿瘤的早期诊断,首先需要通过对单一癌种晚期患者手术前后的甲基化谱进行布尔代数运算,以及术前术后甲基化谱的差异性(即病集)运算,从而得到相应肿瘤的疾病特异性甲基化谱[36-37]。多种肿瘤甲基化谱的进一步并集,即得到泛癌种的甲基化谱。甲基化谱在用于肿瘤早期诊断之前,一个合适的应用场景是对影像学发现的微小增生病灶进行良恶性鉴别,然后初步扩大临床应用。正是因为血液游离核酸甲基化的高敏感检测技术,人类得以第一次认识到甲基化谱——这一动态与全息的生命脉律。在肿瘤早期诊断的医学旅程中,可以预期,甲基化谱分析技术的出现对甲基化谱成为肿瘤早期肿瘤诊断标准之一将具有重要推动作用。
甲基化谱分析新技术[25-30]研发的初心,是为防止晚期肿瘤。这些技术避免使用亚硫酸氢钠对待测标本的破环,不仅提高了检测敏感性,更解决了亚硫酸氢钠技术导致的“薛定谔的猫”相类似难题。以亚硫酸氢钠为基础的经典DNA甲基化分析技术,需对化学处理前后标本进行比对,很明显,在对体外受精的单细胞甲基化分析中,临床实际难以提供两个细胞进行比对。新的甲基化谱技术,因其只需对一个细胞进行检测,即可在生命诞生的初始阶段对其进行监控。作为一个机体细胞的整合性指标,甲基化谱除了可以广泛用于多种疾病的早期诊断、疗效评估以及药物毒副作用监测外,还可以用于人种[38-39]、同卵及异卵双胞胎[9]鉴别,甚至还可以用于衰老速率评估及特定工作状态分析[40]。
游離核酸甲基化谱同样可以用于传统医学的全流程分析。传统医学和现代医学都是根据可观察到的症状或证候指标描述病理变化,而甲基化谱揭示了在遗传和表观遗传水平上发生的变化。中医学的整体观念和辨证论治两大特点,很可能与甲基化谱及其变化相吻合。一方面,甲基化谱可以作为一个通用的参数,对中医的基本概念进行分类,如阴阳五行、脏腑及其表现、气血津液、经络等。特别是在辨识中医的不同证型或亚证型方面,甲基化谱可能是中医证素(包括病性证素和病位证素)的重要展现方式,可为中医体质辨识和疗效评估提供一个敏感的定量技术途径,从表观遗传的角度更加精准地指导中医治病防病。另一方面,也可以利用植物DNA甲基化(CpG、CpHG和CpHH)谱辨别中药材的真伪,采用甲基化谱技术结合基因编辑技术对农作物和中药材进行品种改良和优化[15,41-42]。此外,DNA甲基化谱还可用于动物模型的评价[43]。利用甲基化谱对传统医学进行量化评估,可能成为中药通过多途径、多靶标调节人体功能的一种最好诠释方式,使人类历史上的经验性知识升华为现代科学理论。
游离核酸甲基化谱既是生命中属于机体的物理性动态指标,可授益通常意义上的疾病诊断;也可在一定程度上作为人类健康、亚健康的衡量指标,甚至成为心理和智商的判断指标。譬如,机体处于亚健康状态、应激状态或某个特殊工作状态(如夜班工作者)时,可能不存在病理、生化或物理等指标的改变,但以甲基化谱为代表的表观遗传学改变已经悄悄展现[40];又如,当神经细胞中参与思维的基因表达异常时,外周血cfDNA的甲基化谱将可能早于常规的行为学和生化检测指标[44]。
因为基因组主要定性组成生命的元器件,甲基化谱则定量各元器件在生命活动中的实时参与程度[13]。一方面,从更广的视野上,人类有望利用甲基化谱对多种能力如运动、音乐、绘画、管理等进行量化评估;另一方面,甲基化谱技术对植物育种,同样具有实用价值。一个甲基化谱技术,可以连通整个真核生物世界[45],为构建包括人类健康、动植物健康以及环境健康在内的大健康生态打通航道、扬起风帆。
从千万年蛮荒时代走来,得益于一代又一代科学家的努力,人类终于在20世纪50年代发现了DNA双螺旋结构,尽管最早发现DNA双螺旋结构——X射线衍射照片的富兰克林女士因为晚期癌症去世,未能与沃森和克里克分享诺贝尔奖的殊荣,但DNA双螺旋结构的发现,让我们进入了基因组时代,进入了后基因组时代,进入了表观遗传学时代。新年伊始,Nature和Nature Genetics又分别以“正常人细胞甲基化谱”和“DNA甲基化提供了复杂性状背后的分子联系”开篇[46-47]。正常细胞甲基化谱的完成,为肿瘤甲基化谱的确定起到了加速作用,我们有理由期待,我们有理由相信,因为用于血液游离核酸甲基化谱分析技术的出现,因为游离核酸甲基化谱的广泛应用,迎接我们的,将是一个更加健康、更加美好的灿烂明天。
参考文献
[1] WATSON J D, CRICK F H. Molecular structure of nucleic acids: A structure for deoxyribose nucleic acid[J]. Nature, 1953, 171(4356): 737-738.
[2] WATSON J D, CRICK F H C. Genetical implications of the structure of deoxyribonucleic acid[J]. Nature, 1953, 171(4361): 964-967.
[3] LANDER E S, LINTON L M, BIRREN B, et al. Initial sequencing and analysis of the human genome[J]. Nature, 2001, 409(6822): 860-921.
[4] OLIVIER M, AGGARWAL A, ALLEN J, et al. A high-resolution radiation hybrid map of the human genome draft sequence[J]. Science, 2001, 291(5507): 1298-1302.
[5] 周翠兰, 殷宇芳, 张 佳, 等. DNA甲基化的生物学意义及其检测方法[J]. 南华大学学报(医学版), 2005, 33(2): 148-153.
[6] ZILLER M J, GU H C, MüLLER F, et al. Charting a dynamic DNA methylation landscape of the human genome[J]. Nature, 2013, 500(7463): 477-481.
[7] MULQUEEN R M, POKHOLOK D, NORBERG S J, et al. Highly scalable generation of DNA methylation profiles in single cells[J]. Nature Biotechnology, 2018, 36(5): 428-431.
[8] ZHU T Y, LIU J, BECK S, et al. A pan-tissue DNA methylation atlas enables in silico decomposition of human tissue methylomes at cell-type resolution[J]. Nature Methods, 2022, 19(3): 296-306.
[9] KAMINSKY Z A, TANG T, WANG S C, et al. DNA methylation profiles in monozygotic and dizygotic twins[J]. Nature Genetics, 2009, 41(2): 240-245.
[10] GUO H S, ZHU P, YAN L Y, et al. The DNA methylation landscape of human early embryos[J]. Nature, 2014, 511(7511): 606-610.
[11] SHEN S Y, SINGHANIA R, FEHRINGER G, et al. Sensitive tumour detection and classification using plasma cell-free DNA methylomes[J]. Nature, 2018, 563(7732): 579-583.
[12] LORTHONGPANICH C, CHEOW L F, BALU S, et al. Single-cell DNA-methylation analysis reveals epigenetic chimerism in preimplantation embryos[J]. Science, 2013, 341(6150): 1110-1112.
[13] SCH?譈BELER D. Function and information content of DNA methylation[J]. Nature, 2015, 517(7534): 321-326.
[14] RAZIN A, RIGGS A D. DNA methylation and gene function[J]. Science, 1980, 210(4470): 604-610.
[15] MARTIENSSEN R A, COLOT V. DNA methylation and epigenetic inheritance in plants and filamentous fungi[J]. Science, 2001, 293(5532): 1070-1074.
[16] BIRD A P. DNA methylation: How important in gene control?[J]. Nature, 1984, 307(5951): 503-504.
[17] CEDAR H. DNA methylation and gene activity[J]. Cell, 1988, 53(1): 3-4.
[18] COUNTS J L, GOODMAN J I. Alterations in DNA methylation may play a variety of roles in carcinogenesis[J]. Cell, 1995, 83(1): 13-15.
[19] 張 佳, 陈琳玲, 周翠兰, 等. 甲基化遁逸与保守氨基酸的进化[J]. 南华大学学报(医学版), 2005, 33(2): 157-159.
[20] ARAN D, HELLMAN A. DNA methylation of transcriptional enhancers and cancer predisposition[J]. Cell, 2013, 154(1): 11-13.
[21] GKOUNTELA S, CASTRO-GINER F, SZCZERBA B M, et al. Circulating tumor cell clustering shapes DNA methylation to enable metastasis seeding[J]. Cell, 2019, 176(1/2): 98-112.
[22] KRUEGER F, KRECK B, FRANKE A, et al. DNA methylome analysis using short bisulfite sequencing data[J]. Nature Methods, 2012, 9(2): 145-151.
[23] ZILLER M J, HANSEN K D, MEISSNER A, et al. Coverage recommendations for methylation analysis by whole-genome bisulfite sequencing[J]. Nature Methods, 2015, 12(3): 230-232.
[24] WANG Q, GU L, ADEY A, et al. Tagmentation-based whole-genome bisulfite sequencing[J]. Nature Protocols, 2013, 8(10): 2022-2032.
[25] LIU Y B, SIEJKA-ZIELI■SKA P, VELIKOVA G, et al. Bisulfite-free direct detection of 5-methylcytosine and 5-hydroxymethylcytosine at base resolution[J]. Nature Biotechnology, 2019, 37(4): 424-429.
[26] LIU Y B, HU Z Y, CHENG J F, et al. Subtraction-free and bisulfite-free specific sequencing of 5-methylcytosine and its oxidized derivatives at base resolution[J]. Nature Communications, 2021, 12: 618.
[27] YUE X, XIE Z Y, LI M R, et al. Simultaneous profiling of histone modifications and DNA methylation via nanopore sequencing[J]. Nature Communications, 2022, 13: 7939.
[28] BOERS R, BOERS J, DE HOON B, et al. Genome-wide DNA methylation profiling using the methylation-dependent restriction enzyme LpnPI[J]. Genome Research, 2018, 28(1): 88-99.
[29] 廖端芳, 齊 捧, 曾娅玲,等.甲基化位点的检测方法及检测试剂盒: CN115011677A[P]. 2022-09-06.
[30] 肖 莉, 袁梦兮, 罗晶晶, 等. DNA甲基化修饰的检测方法: CN114381497A[P]. 2022-04-22.
[31] CHIASSON D, GIMéNEZ-OYA V, BIRCHENEDER M, et al. A unified multi-Kingdom Golden Gate cloning platform[J]. Scientific Reports, 2019, 9: 10131.
[32] BALNIS J, MADRID A, HOGAN K J, et al. Blood DNA methylation and COVID-19 outcomes[J]. Clinical Epigenetics, 2021, 13(1): 118.
[33] SHIRVALILOO M. Epigenomics in COVID-19; the link between DNA methylation, histone modifications and SARS-CoV-2 infection[J]. Epigenomics, 2021, 13(10): 745-750.
[34] STEVENS M, CHENG J B, LI D F, et al. Estimating absolute methylation levels at single-CpG resolution from methylation enrichment and restriction enzyme sequencing methods[J]. Genome Research, 2013, 23(9): 1541-1553.
[35] PARIKH A R, VAN SEVENTER E E, SIRAVEGNA G, et al. Minimal residual disease detection using a plasma-only circulating tumor DNA assay in patients with colorectal cancer[J]. Clinical Cancer Research, 2021, 27(20): 5586-5594.
[36] HOW-KIT A, DEJEUX E, DOUSSET B, et al. DNA methylation profiles distinguish different subtypes of gastroenteropancreatic neuroendocrine tumors[J]. Epigenomics, 2015, 7(8): 1245-1258.
[37] CAPPER D, JONES D T W, SILL M, et al. DNA methylation-based classification of central nervous system tumours[J]. Nature, 2018, 555(7697): 469-474.
[38] GOKHMAN D, LAVI E, PRüFER K, et al. Reconstructing the DNA methylation maps of the Neandertal and the Denisovan[J]. Science, 2014, 344(6183): 523-527.
[39] ZHANG J, LI K, LIAO D F. Application of Nuc-mtDNA and DNA methylation analysis in life science and medical studies[J]. Digital Chinese Medicine, 2022, 5(3): 233-235.
[40] WACKERS P, DOLL?魪 M E T, VAN OOSTROM C T M, et al. Exploration of genome-wide DNA methylation profiles in night shift workers[J]. Epigenetics, 2022, 1: 1-12.
[41] ZHANG X, YAZAKI J, SUNDARESAN A, et al. Genome-wide high-resolution mapping and functional analysis of DNA methylation in Arabidopsis[J]. Cell, 2006, 126(6): 1189-1201.
[42] LIANG X L, HOU X, LI J Y, et al. High-resolution DNA methylome reveals that demethylation enhances adaptability to continuous cropping comprehensive stress in soybean[J]. BMC Plant Biology, 2019, 19(1): 79.
[43] LIU H Q, ZHOU J T, TIAN W, et al. DNA methylation atlas of the mouse brain at single-cell resolution[J]. Nature, 2021, 598(7879): 120-128.
[44] MENG J, WANG F L, JI L, et al. Comprehensive methylation profile of CSF cfDNA revealed pathogenesis and diagnostic markers for early-onset Parkinson's disease[J]. Epigenomics, 2021, 13(20): 1637-1651.
[45] ZEMACH A, MCDANIEL I E, SILVA P, et al. Genome-wide evolutionary analysis of eukaryotic DNA methylation[J]. Science, 2010, 328(5980): 916-919.
[46] LOYFER N, MAGENHEIM J, PERETZ A, et al. A DNA methylation atlas of normal human cell types[J]. Nature, 2023, 613(7943): 355-364.
[47] DNA methylation provides molecular links underlying complex traits[J]. Nature Genetics, 2023, 55(1): 12-13.
(本文編辑 匡静之)
