生物信息学方法分析CLDN9基因在子宫内膜癌中表达对生存率的影响
2023-06-05诸充康乐瞿晓燕杨尚闫蔷许浩宇和斌
诸充,康乐,瞿晓燕,杨尚,闫蔷,许浩宇,和斌,
(同济大学1.附属杨浦医院临床研究与转化医学中心,2.附属第一妇婴保健院生殖医学中心,上海 200120)
近年来,子宫内膜癌(uterine corpus endometrial carcinoma,UCEC)的发病率逐年升高,UCEC是发达国家最常见的妇科癌症,在发展中国家发病率仅次于宫颈癌[1]。目前对UCEC的治疗提倡早期诊断、早期治疗,晚期UCEC患者的治疗效果和预后情况并不乐观[2]。因此,探索UCEC新的预后指标及治疗靶点具有重要的临床意义。肿瘤微环境(tumor microenvironment,TME)是近年来的研究热点,指由细胞外可溶性化合物、基质细胞、免疫细胞、肿瘤细胞组成的复杂生态系统[3-5],与肿瘤有着密切关系,是肿瘤细胞的“庇护所”,其中的各种成分与肿瘤细胞的增殖、侵袭等有着密切关系[6-8],从而影响癌症的发生发展。因此,通过靶向调节癌基因和相关信号通路来重塑TME成为了治疗肿瘤的新策略。在本研究中,我们通过R软件对TCGA数据库中UCEC样本的基因表达数据进行挖掘分析,鉴定出与UCEC的免疫微环境相关的关键基因,分析其与UCEC的临床病理特征、生存预后、TME中的免疫细胞浸润的关系,探寻潜在的分子机制以及UCEC肿瘤免疫治疗的新靶点,提高其诊治率。
1 资料与方法
1.1 资料
从UCEC XENA(https:∥xena.ucsc.edu/)数据库下载TCGA数据库中UCEC组织和正常子宫内膜组织的基因表达、临床特征和生存数据。
1.2 方法
主要通过R软件(4.2.1版本)进行统计分析、可视化分析。
1.2.1 分析鉴定差异基因(differentially expressed genes,DEGs)、GO/KEGG富集分析 通过R软件中Estimate包(1.0.13版本)对肿瘤样本免疫浸润、肿瘤纯度进行评分,以样本的免疫细胞与基质细胞综合评分(ESTIMATE Score)的中位值将样本分为ESTIMATE Score高、低两组。以DESeq2包版本(1.36.0版本)、tidyvers包(1.3.2版本)计算组间DEGs,两组DEGs取并集。通过org.Hs.eg.db包(3.15.0版本)、clusterProfiler包(4.4.4版本)对DEGs进行GO/KEGG富集分析。
1.2.2 加权基因共表达网络分析(WGCNA)富集分析、鉴定关键基因 通过R软件中WGCNA包(1.71版本)对DEGs进行聚类分析,分析聚类的基因模块中基因与UCEC的病理分级、患者生存时长之间的关系。通过DESeq2包(1.36.0版本)、tidyverse包(1.3.2版本)分析关键基因在UCEC组织和正常内膜组织中的表达水平,结合基因表达水平和在UCEC、正常内膜组织中差异表达程度确定目的基因。
1.2.3 分析目的基因与UCEC的病理特征、患者生存的关系 以目的基因的表达水平(FPKM值)的中位值为界值,将UCEC样本分为目的基因表达高、低两组。分析不同分期(Ⅰ、Ⅱ、Ⅲ、Ⅳ期)、分级(1、2、3、高级别)的UCEC中目的基因的表达水平差异。LinkedOmics(http:∥linkedomics.org/login.php)数据库下载不同组织学分型的UCEC中目的基因的表达数据,并进行分析,以上分析结果通过ggplot2包(3.3.6版本)可视化。
1.2.4 生存曲线 以目的基因在UCEC中表达水平(FPKM值)的1/4界值将样本分为两组,survival包(3.3-1版本)对两组的生存资料进行分析并可视化。
1.2.5 GSEA富集分析、构建目的基因蛋白互作网络 GSEA官网(https:∥www.gsea-msigdb.org/gsea/index.jsp)下载msigdb_v7.5.1_GMTs文件,应用org.Hs.eg.db包(3.15.0版本)、clusterProfiler包(4.4.4版本)进行GSEA富集分析,结果通过enrichplot包(1.16.1版本)进行可视化。String数据库(https:∥cn.string-db.org/)构建目的基因蛋白互作网络。
1.2.6 免疫浸润分析 依据目的基因表达水平(FPKM值)的中位值将UCEC分为高、低表达组,分析两组的免疫评分(Immune Score)、基质评分(Stromal Score)、肿瘤细胞丰度(Tumor Purity)的差异。TISIDB官网(http:∥cis.hku.hk/TISIDB/index.php)下载TILs.txt文件整理为cellMarker文件,通过GSVA包(1.44.2版本)对两组进行定量免疫浸润分析,结果通过ggplot2(3.3.6版本)可视化。通过Corrplot包(0.92版本)分析目的基因的表达与免疫细胞浸润水平的相关性并进行可视化。
2 结 果
2.1 DEGS的鉴定及GO/KEGG富集分析
样本由533个UCEC样本、35个正常内膜组织样本的基因表达数据组成。ESTIMATE包计算得到UCEC样本的ESTIMATE Score区间,为-3 205.840 811~3 976.557 046。以ESTIMATE Score的中位值(-614.951 2)分组,计算组间DEGs(log2FoldChange绝对值>1,P<0.05)取并集,得到1 068个DEGs。对DEGs进行GO功能注释和KEGG途径富集分析。GO功能分析结果显示,DEGs在生物过程(BP)方面主要富集在白细胞介导的免疫、细胞活化正向调节、白细胞-细胞黏附、单核细胞分化、淋巴细胞介导的免疫反应、T细胞活化调节、免疫效应过程的调节、白细胞增生、细胞杀伤等通路上;在细胞成分(CC)方面主要富集在质膜外侧、细胞外基质、内吞小泡等通路及MHC蛋白复合体、内质网膜腔侧等;分子功能(MF)方面主要富集在免疫受体活性、碳水化合物结合、细胞因子结合、细胞因子受体活性、趋化因子受体结合、趋化因子活性、MHC蛋白复合物结合、G蛋白-偶联化学吸引受体活性、趋化因子受体活性,见图1。KEGG分析结果显示,DEGs主要富集在细胞因子-细胞因子受体相互作用、趋化因子信号通路、细胞黏附分子、病毒蛋白与细胞因子和细胞因子受体的相互作用、细胞吞噬体等信号通路上,见图2。
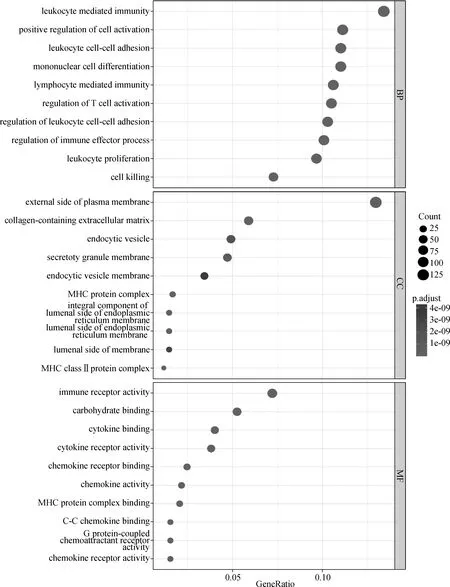
图1 GO富集分析
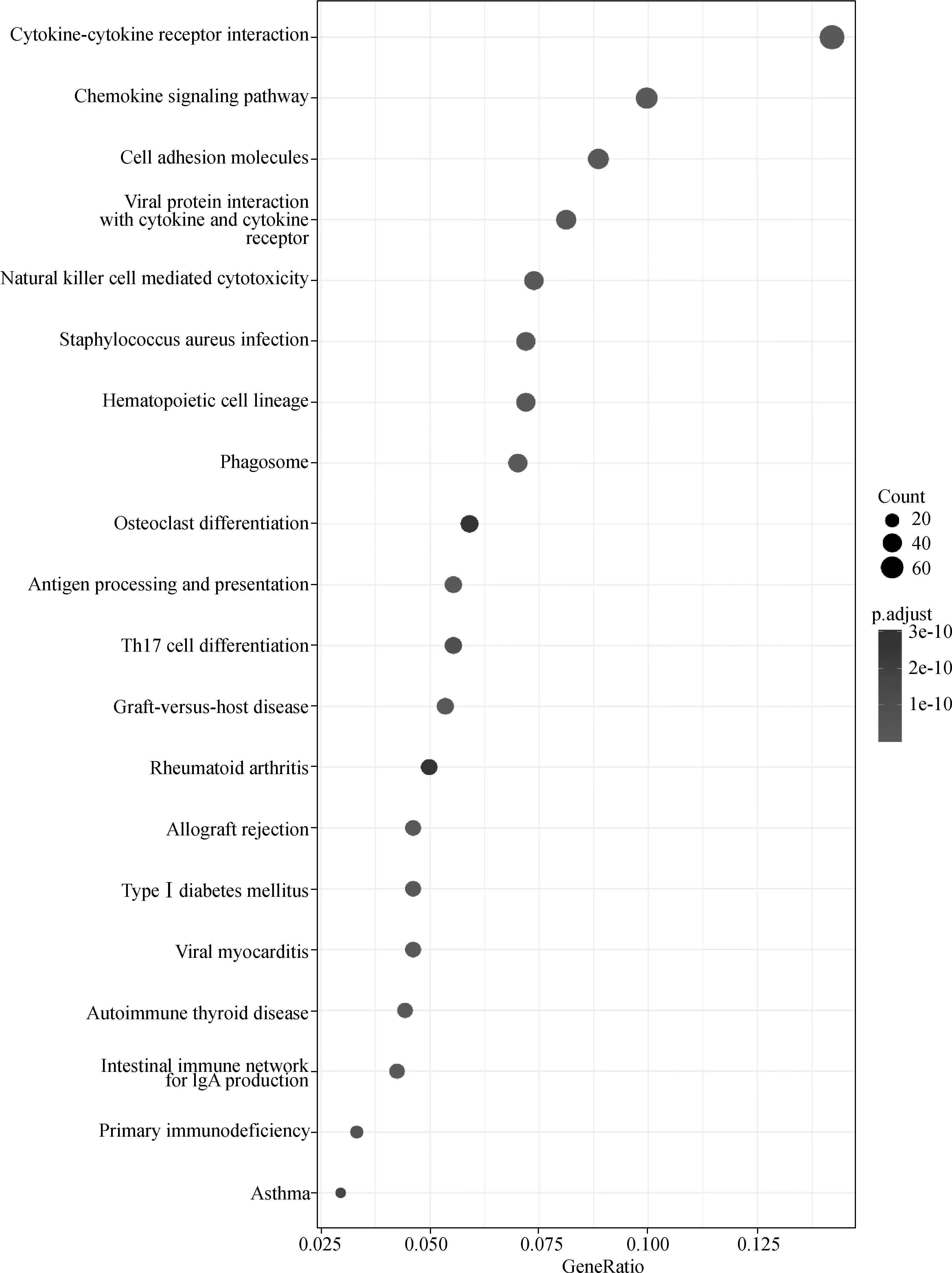
图2 KEGG富集分析
2.2 WGCNA聚类分析结合UCEC和正常内膜组织基因表达水平确定目的基因CLDN9
为了找出与UCEC的病理特征及患者生存相关的DEGs,对1 068个DEGS进行WGCNA,得到10个基因模块。其中包含77个DEGs的棕色(brown)模块与UCEC的分期、分级、生存时间显著相关(图3)。进一步分析这77个DEGs在UCEC、正常内膜组织中的表达水平,DESeq2包对这77个DEGs差异分析的结果中baseMean值前10位基因为CLDN6、HMGA2、SST、FBN3、EGFL6、CLDN9、NKAIN4、PNMA3、CRTAC1、CTCFL(表1)。综合考虑目的基因表达水平和在UCEC、正常内膜组织中差异表达程度,我们将CLDN9作为研究对象,对CLDN9在537个UCEC样本和35个正常内膜组织样本表达水平进行分析后发现CLDN9在UCEC组织中的表达水平显著升高(图4)。
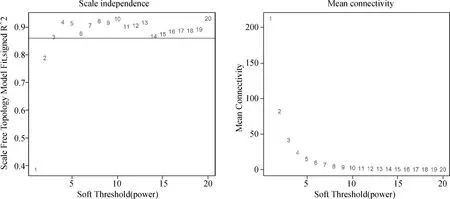
A.最佳软阈值(Soft Threshold)的确定
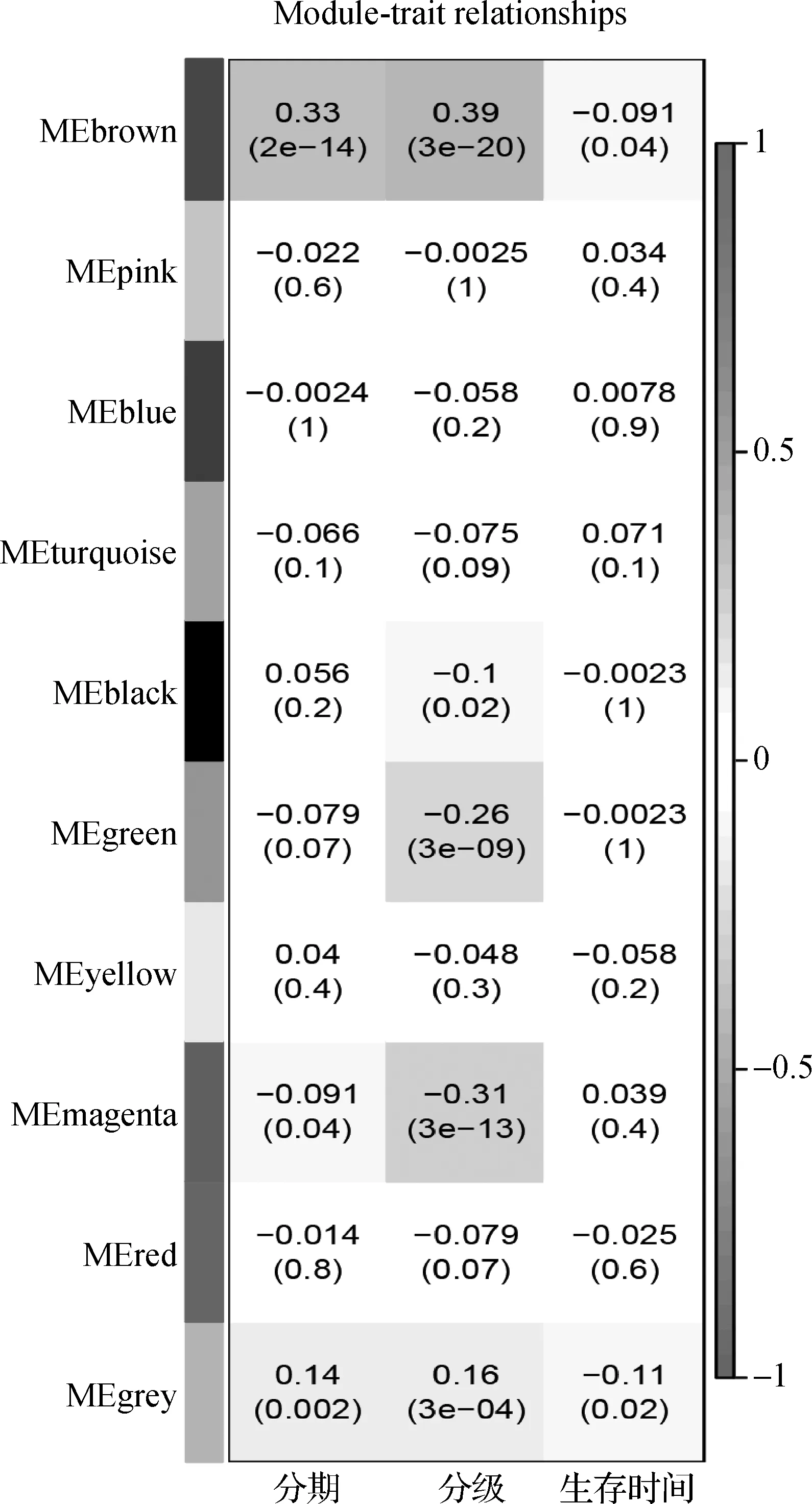
D.不同基因模块与肿瘤分期、分级、生存的相关性图3 WGCNA富集分析
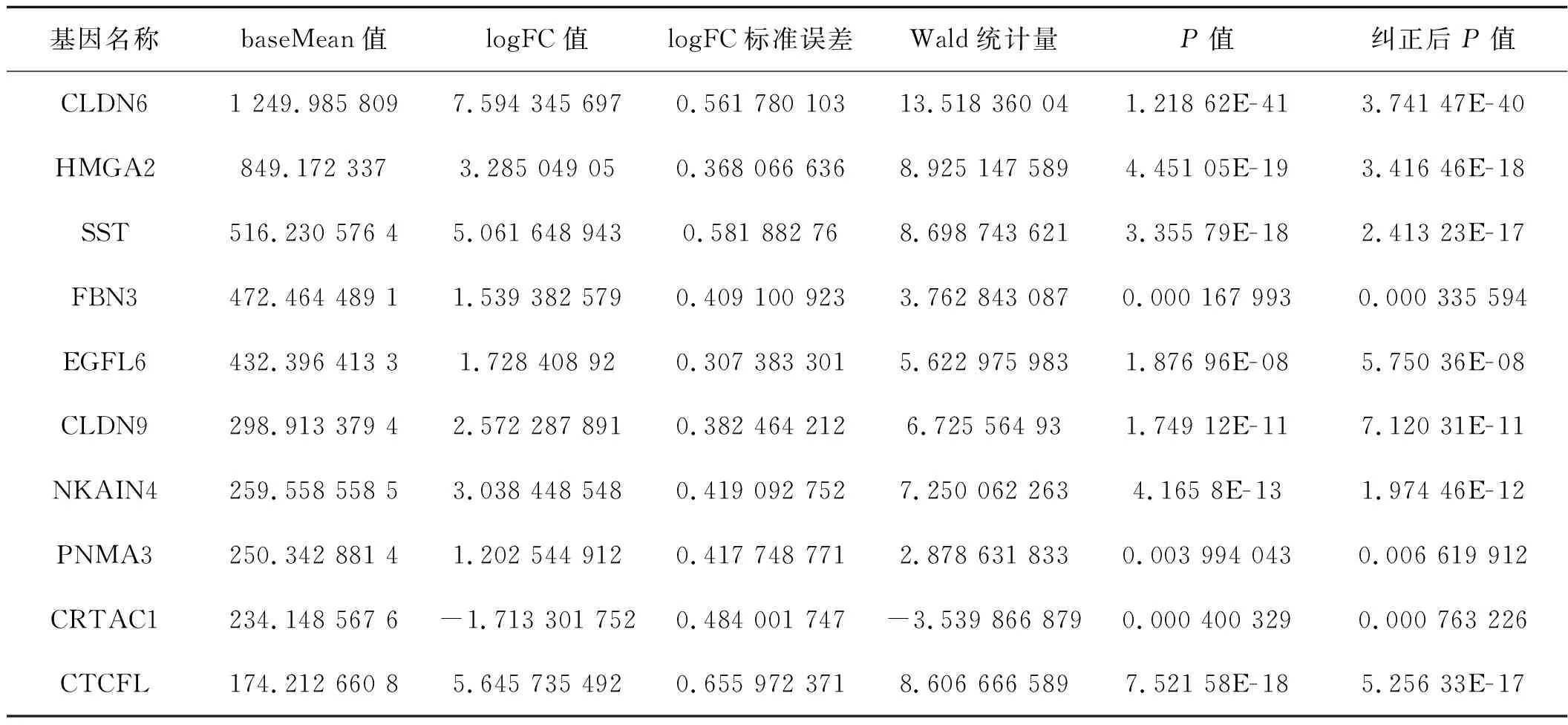
表1 UCEC和正常内膜组织中baseMean值前10 位基因
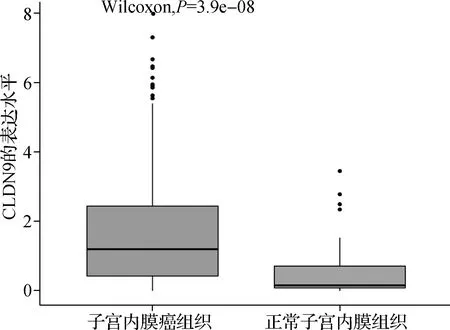
图4 CLDN9在UCEC和正常内膜组织中的表达情况
2.3 CLDN9与UCEC病理特征、患者生存时间的相关性
为了进一步分析CLDN9与UCEC病理特征的相关性,我们将UCEC样本按照分期(Ⅰ、Ⅱ、Ⅲ、Ⅳ期)、分级(1、2、3、高级别)进行分组。并对各组中CLDN9的表达水平进行了分析统计可视化(图5),结果显示不同分期、分级的UCEC组织间CLDN9的表达差异显著,与病理分级呈正相关(P<0.05)。我们还通过LinkedOmics数据库下载了不同组织学分型的UCEC中CLDN9的表达数据,并通过ggplot2包进行可视化,结果显示CLDN9在子宫内膜样子宫内膜腺癌、浆液型和子宫内膜型混合、浆液性子宫内膜腺癌中的表达差异具有统计学意义,其中浆液性子宫内膜腺癌中CLDN9的表达水平最高(图6)。接着我们分析统计UCEC患者的生存数据,并以患者CLDN9的表达水平的中位值(1.203 173)为界值将患者分为CLDN9高、低表达组,构建两组患者的生存曲线(图7),结果显示CLDN9高表达组的生存比率显著低于低表达组(P<0.05)。
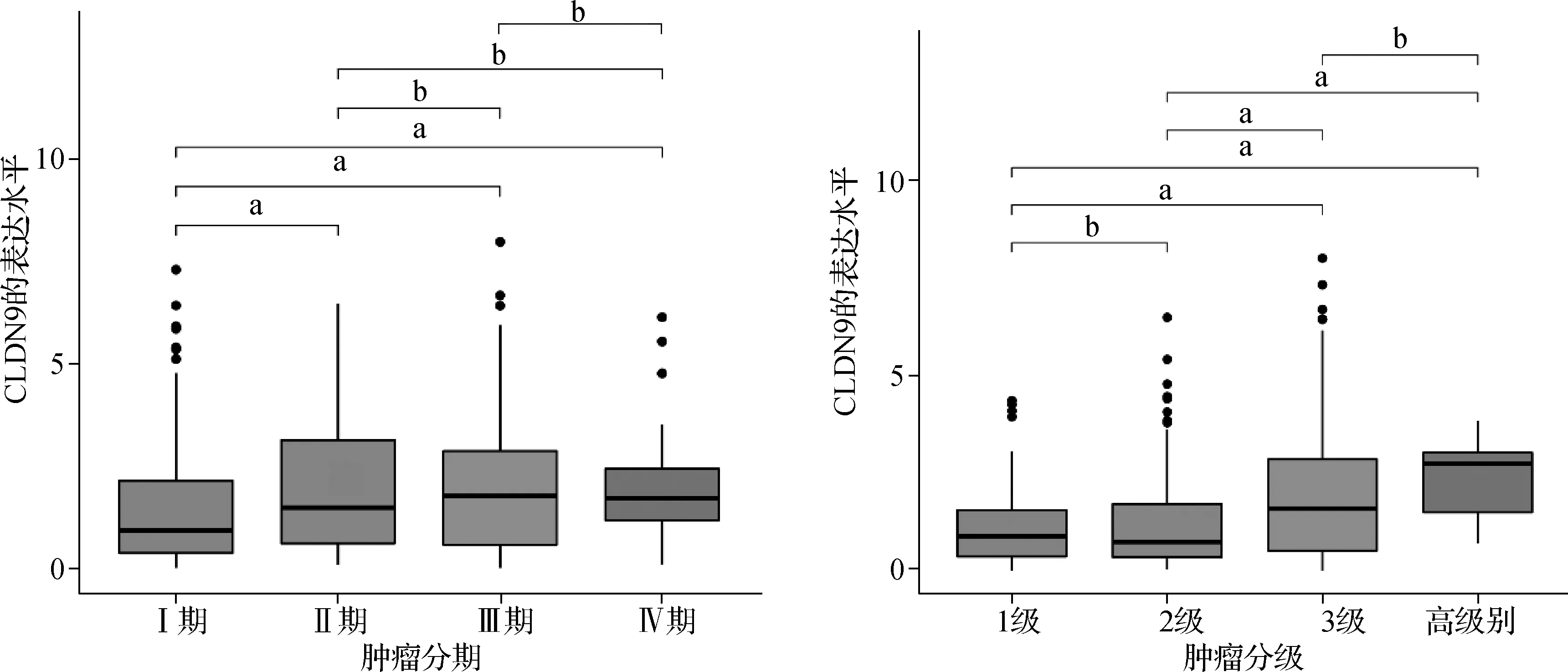
a P<0.05;b P>0.05图5 不同分期、分级UCEC中CLDN9的表达水平
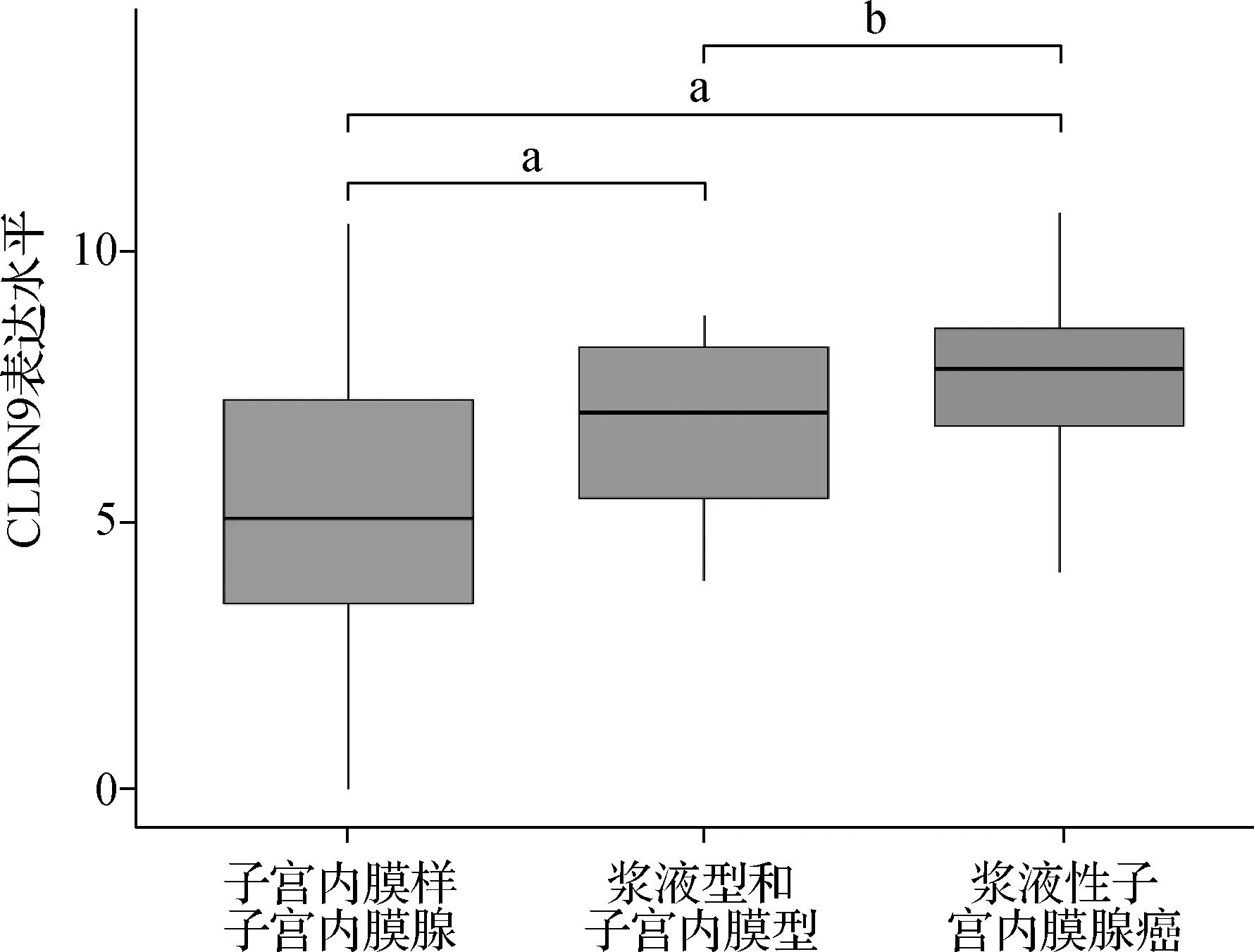
a P<0.05;b P>0.05图6 不同组织学类型的UCEC中CLDN9的表达水平
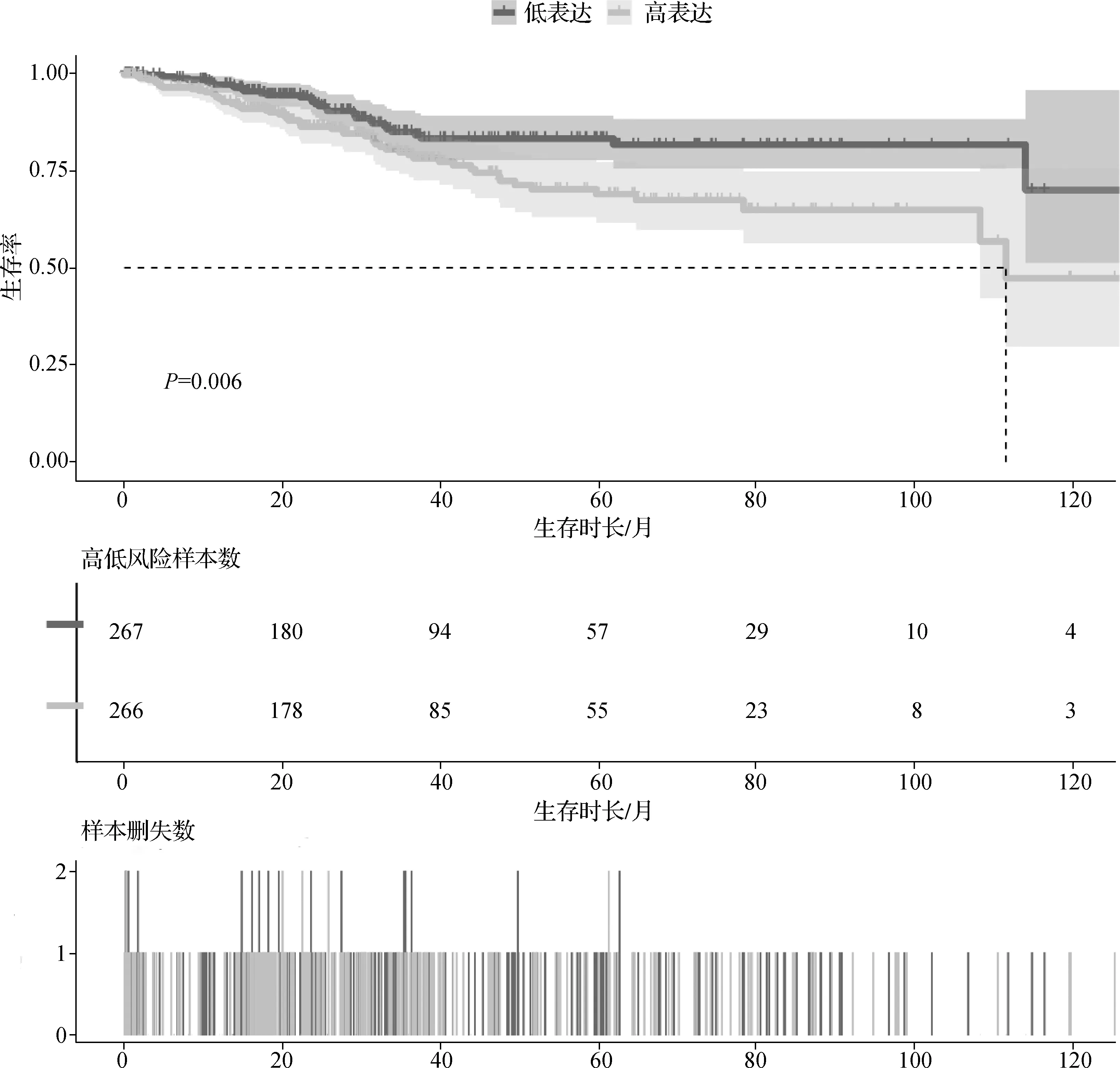
图7 CLDN9的表达水平与UCEC患者生存期的关系
2.4 CLDN9相关的蛋白互作网络
为了了解CLDN9在子宫内膜癌TME及免疫浸润中可能存在的调控机制及相关蛋白,我们基于String数据库对CLDN9为核心的蛋白网络进行了预测(图8),与CLDN9存在互作可能的相关蛋白包括OCLN、TJP3、CLDN23、CLDN10、CLDN22、CLDN12、TJP1、CLDN16、CLDN11、CLDN1。
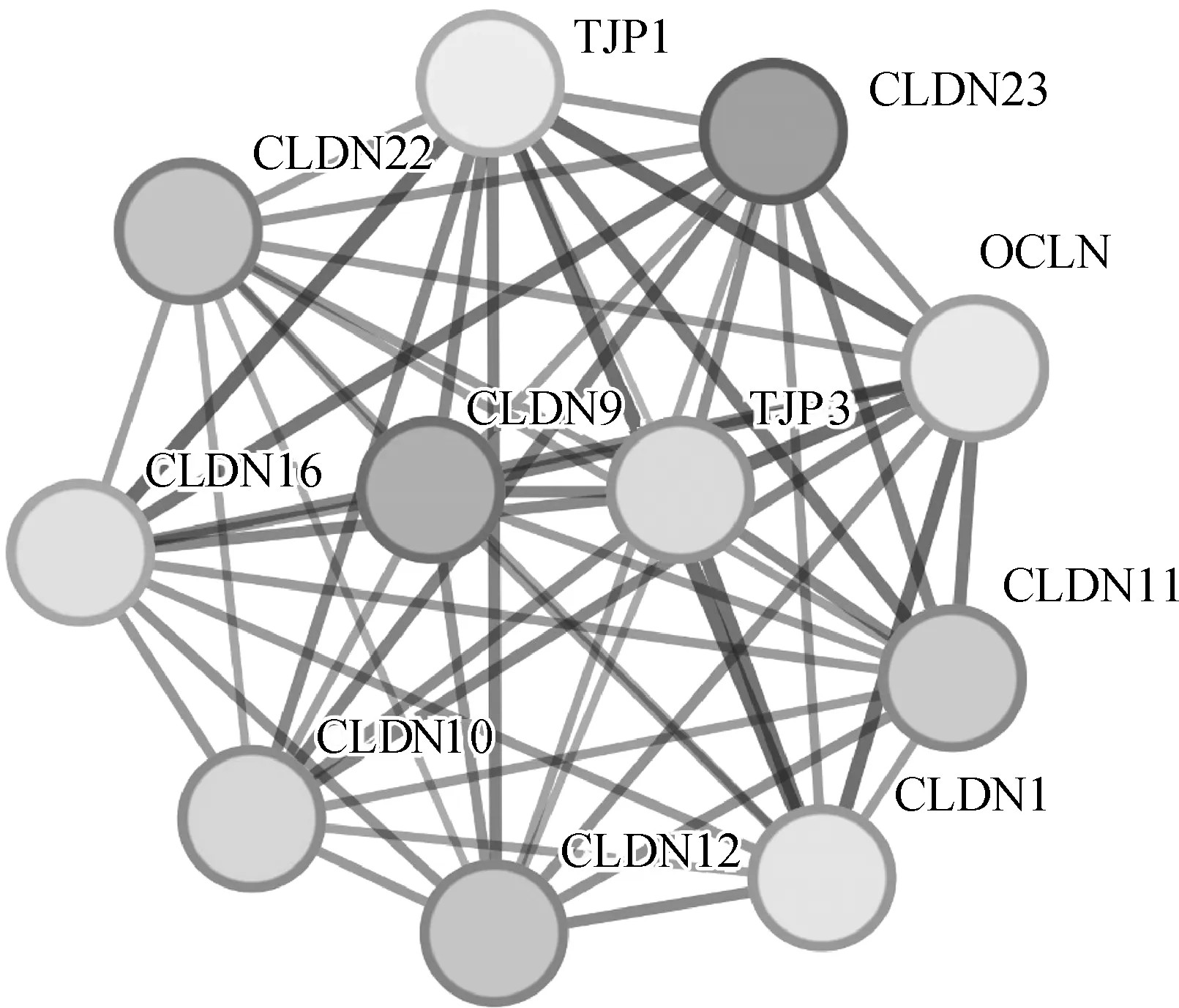
图8 CLDN9蛋白互作网络
2.5 GSEA富集分析
以肿瘤样本中CLDN9的表达水平(FPKM值)的中位值(1.203 173)作为界值,将样本分为CLDN9高、低表达组,分析鉴定组间DEGs(log2FoldChange绝对值>0,P<0.05),对得到的DEGs进行GSEA基因集富集分析。结果显示CLDN9高表达组DEGs主要富集在KRAS基因(KRAS基因是一种在肿瘤细胞生长以及血管生成等过程的信号传导通路中起着重要调控作用的基因)激活下调的信号通路、骨骼肌发育、雄性配子(精子)发生相关信号通路以及免疫相关的外周血中抑制CD8+T细胞免疫反应的生物学通路,CLDN9低表达组DEGs主要富集在异生代谢、脂肪酸代谢、雄激素反应相关信号通路中(图9)。
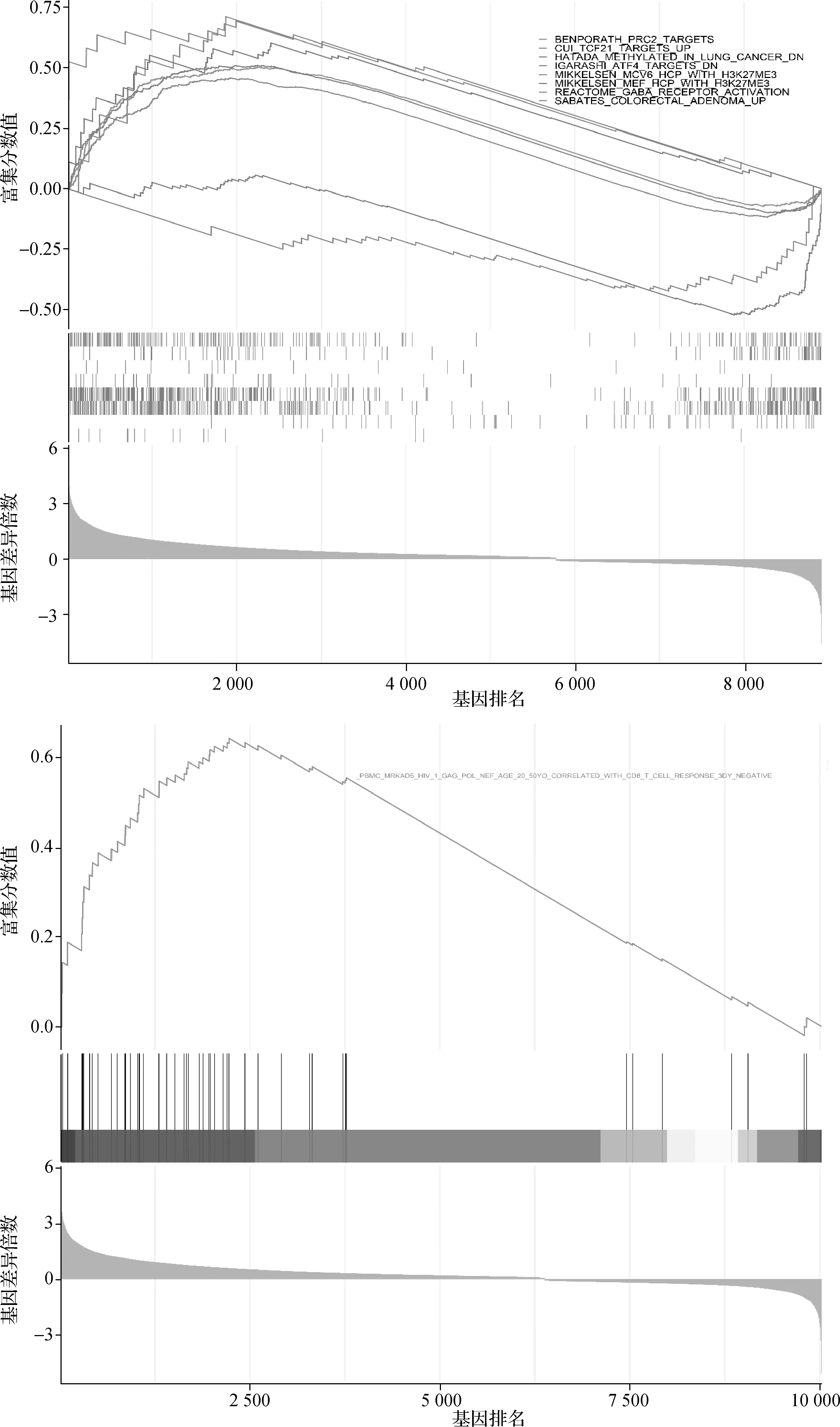
图9 CLDN9的GSEA富集分析结果
2.6 UCEC中CLDN9与免疫细胞浸润的关系
为了进一步了解CLDN9表达与UCEC免疫微环境之间的关系,通过CLDN9表达水平的中位值将样本分为CLDN9高、低表达组,分析两组的免疫评分(Immune Score)、基质评分(Stromal Score)、肿瘤细胞丰度(Tumor Purity)的差异性(图10)。在R中通过GSVA包对UCEC中28种免疫细胞的浸润情况进行了计算分析,以CLDN9表达水平的中位值为界值分组,分析两组间免疫细胞浸润情况的,结果显示CLDN9的低表达组有着更高的免疫细胞浸润水平,差异具有统计学意义(P<0.05),包括被激活的CD4+细胞、CD8+T细胞、CD56自然杀伤细胞、未成熟树突状细胞、巨噬细胞、肥大细胞以及2、17型辅助T细胞。尤其是CD4+、CD8+T细胞浸润水平增高在肿瘤免疫治疗中具有重要意义(图11)。

图10 CLDN9的表达与免疫、基质评分以及肿瘤细胞丰度的关系
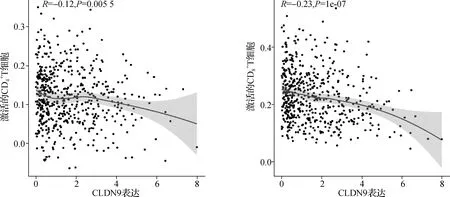
图11 CLDN9表达与UCEC肿瘤微环境中CD4+、CD8+ T细胞浸润水平的关系
3 讨 论
UCEC起源于子宫内膜,是全球常见的妇科恶性肿瘤[9],虽然早期UCEC的治疗效果比较好,但因复发率高、死亡率高,UCEC的整体预后仍然较差[10]。因此,研究UCEC预后相关分子及免疫治疗靶点具有深刻的意义。
TME在肿瘤发生的开始和发展中发挥不可忽视的作用。肿瘤的免疫疗法通过调节抗原释放、抗原呈递、抗原识别和免疫细胞运输来增强抗肿瘤免疫反应改善癌症患者的预后。然而,TME中的多种成分可能通过缺氧、代谢功能障碍、免疫细胞表型转移和肿瘤来源的外泌体等途径形成免疫抑制微环境[11-14],导致肿瘤细胞的免疫逃逸,从而促进肿瘤进展。
CLDN9是属于Claudin蛋白家族的一种细胞黏附因子,Claudins蛋白是膜蛋白和细胞间紧密连接链的组成部分。紧密连接链可作为物理屏障,防止溶质和水自由通过上皮或内皮细胞片之间的细胞旁空间,并且在维持细胞极性和信号转导方面也起着关键作用。CLDN9蛋白是丙型肝炎病毒侵入肝细胞的辅助因子之一[15-16]。Zhu等[17]研究发现CLDN9在宫颈癌组织及癌旁组织的表达水平发生改变,与宫颈癌的淋巴转移相关。Hong等[18]发现CLDN9的过表达与垂体瘤癌细胞的侵袭性有关。Zavala-Zendejas等[19]研究发现过表达CLDN9可以增强AGS细胞(胃腺癌细胞系)的侵袭性、迁移性和增殖率。我们的富集分析结果显示,CLDN9高表达组的DEGs被富集在KRAS基因激活的相关通路、骨骼肌发育等相关通路上,低表达组DEGs富集在异生代谢、脂肪酸代谢、雄激素反应代谢通路中,尤其是在免疫学相关通路中,CLDN9高表达组的DEGs富集在外周血中抑制CD8+T细胞免疫反应的生物学通路上。
在本研究中,我们通过生物信息学分析的方法发现CLDN9与UCEC病理特征、生存预后、免疫浸润密切相关。我们的结果显示,随着UCEC的进展,CLDN9的表达水平呈增高趋势,在3种组织学分型的UCEC中,浆液性子宫内膜腺癌中CLDN9表达水平相对更高。CLDN9的高表达水平对UCEC患者的生存及预后是一种不利因素。CLDN9在UCEC中的表达与免疫细胞CD4+、CD8+T细胞的浸润呈负相关,CD4+、CD8+T细胞在TME中的抗肿瘤作用是众所周知的,因此我们推测CLDN9在UCEC的TME中会减弱免疫细胞对肿瘤杀伤作用。
我们对UCSC Xena数据库中整合的TCGA UCEC样本进行分析鉴定,得到与UCEC的分期、分级、患者生存相关联的基因CLDN9。进一步分析发现,CLDN9的表达与UCEC的TME相关联。我们的分析结果显示,CLDN9的表达与UCEC的发生、发展呈正相关,与肿瘤中免疫细胞的浸润呈负相关。因此,CLDN9可能成为一种新的UCEC生物学标志物及肿瘤免疫治疗新靶点。
